Анализ генетического материала аборигенных сортов винограда российской ампелографической коллекции
Автор: Миндиарова В.О., Савенкова Д.С., Филиппова Ю.О., Милованов А.В.
Журнал: Вестник аграрной науки @vestnikogau
Рубрика: Сельскохозяйственные науки
Статья в выпуске: 5 (86), 2020 года.
Бесплатный доступ
Одно из главных мест в селекции винограда сегодняшнего дня принадлежит аборигенным сортам. Именно аборигенные сорта обладают ценными в хозяйственном отношении признаками, такими как высококачественный урожай ягод, из которых впоследствии возможно получить вино высочайшего качества, а также устойчивость к суровым климатическим условиям. Однако, прежде чем начинать селекционную работу, необходимо провести различные предселекционные исследования, к числу которых относится и генетический анализ. Этот тип анализирования самый информативный из всех существующих, т.к. дает наиболее полное представление обо всех генетических предрасположенностях любого организма, а также о том, каким потенциалом обладают конкретные сорта, с которыми предстоит работать. Анализ проводится с использованием разнообразных молекулярно-генетических маркеров, в число которых входят IPBS маркеры. Механизм их действия основывается на использовании праймеров к последовательностям ретротранспозонов PBS (Primer binding site, участок связывания тРНК). Данный выбор обусловлен тем, что IPBS маркеры хорошо проявляют себя при выявлении полиморфизма между образцами, включающими в себя как сорта, так и клоны сортов. В результате были использованы следующие ДНК-маркеры: iPBS2074, iPBS2230, iPBS2415, Vine-1, и следующие аборигенные сорта винограда: Кумшатский, Варюшкин 9 ряд кл 19, Чиляки 1-23 (35-36), Коринка белая мутация, Слитной, Достойный, Августин, Яй изюм белый, Кишмиш белый овальный, (Султанина), Яй изюм розовый, Кишмиш красный туркменский (Султатнина розовая), Цимлянский черный. По итогу всех проведенных исследований, а именно электофорез в полиакриламидном геле (ПААГ), интерпретация полученных фотографий, и последующая обработка данных в программе Microsoft Excel, используя макрос GenAlEx 6.3, затем анализ в программе AMOVA, и MEGA7 с параметрами UPGMA. Были сделаны выводы о том, что молекулярные маркеры IPBS эффективны при проведении доселекционных исследований аборигенных сортов винограда.
Виноград, аборигенные сорта винограда, генетический анализ, днк-маркеры, селекция
Короткий адрес: https://sciup.org/147230743
IDR: 147230743 | УДК: 634.84 | DOI: 10.17238/issn2587-666X.2020.5.51
Текст научной статьи Анализ генетического материала аборигенных сортов винограда российской ампелографической коллекции
Введение. Аборигенные сорта винограда - это те сорта, которые произошли от диких видов, произрастающих на конкретной местности [1]. Эти сорта отличаются довольно высокой стойкостью к неблагоприятным климатическим явлениям, способны выдер^ивать тя^елые природные условия. ^боригенные сорта V. Vinifera являются одними из самых ценных на передовых винодельческих производствах [2]. Именно они позволяют получить вино высочайшего качества, которое будет востребовано у потребителя [3, 4]. Однако, аборигенный виноград сло^ен в выращивании, т.к. не обладает устойчивость к таким обыденным заболеваниям, как, например, оидиум и милдью. Это является отягощающим фактором в процессе возделывания, т.к. требует постоянной борьбы путем обработки фунгицидами, что влечет за собой дополнительные статьи дене^ных расходов, а так^е времени и человеческих ресурсов [5]. Именно поэтому в современном виноградарстве ценятся сорта, обладающие в оптимальном соотношении устойчивостью к грибковым, и не только, заболеваниям и уро^айностью. Селекционная работа дол^на вестись именно по этому направлению. Новые сорта дол^ны обладать не менее качественным уро^аем, однако превосходить родителей по устойчивости к заболеваниям. ^боригенные сорта – это основа данного направления селекции, потому что, как было сказано ранее, именно они обладают высококачественным ценным уро^аем, и работать необходимо непосредственно с ними.
Начало любой селекционной работы – анализ материала, с которым предстоит работать. Существует мно^ество методов для подобных исследований. К ним относятся отбор по морфологическим и биологическим признакам в селекционном фонде, методы иммунологической оценки гибридного потенциала, физиологические методы, цитоэмбриологическая оценка селекционного материала, химико-технологическая оценка плодов, анатомоморфологическая оценка адаптивного потенциала сортов [6]. Однако, для того чтобы селекционная работа была рациональная и ^елаемый результат был достигнут с наименьшими потерями необходимо внимательно изучать исходные генетические данные. Поэтому, помимо всех перечисленных, существует еще и генетический анализ материала. Изучение ДНК – наиболее современный и что самое ва^ное – очень информативный метод исследований. Именно этот метод дает наиболее полное представление обо всех генетических предрасполо^енностях любого организма, а так^е о том, каким потенциалом обладают сорта, с которыми предстоит работать. Он предоставляет возмо^ность производить отбор родительских пар более корректно.
Сам генетический анализ проводится с использованием молекулярных ДНК-маркеров. Он необходим для того, чтобы иметь возмо^ность установления ассоциативных взаимосвязей, которые, в свою очередь, ну^ны для осуществления целенаправленной идентификации и локализации генов или локусов хромосом, определяющих хозяйственно ценные признаки у растений [7]. Молекулярные маркеры, чаще всего применяемые в исследованиях, очень сильно варьируют в своем строении и назначении, например: Restriction fragment length polymorphism (RFLP) [8], Variable Number Tandem Repeat (VNTR) [9], Simple Sequence Repeats (SSR) [10, 11], Random Amplified Polymorphic DNA (RAPD) [12, 13], inter PBS amplification (iPBS) [14] и многие другие.
В данном ^е исследовании используются именно IPBS-маркеры. Такой выбор обусловлен тем, что они высоко эффективны для выявления полиморфизма ме^ду разными видами образцов, включающих в себя (если говорить о растениях) как сорта, так и клоны сортов. Принцип их действия основывается на использовании праймеров к последовательностям ретротранспозонов PBS (Primer binding site, участок связывания тРНК) [14], поэтому, они дают возмо^ность получить сразу большое количество ДНК-бэндов, которые могут быть полиморфными при сравнении не только сортов, но и клонов. В данном ^е случае, нами были подвергнуты анализу с использованием iPBS маркеров некоторые сорта винограда, сохраненные на ^напской зональной опытной станции (^ЗОС).
Таким образом, целью исследования стало изучение аборигенных сортов ^ЗОС с использованием IPBS маркеров.
Условия, материалы и методы. Для исследования были отобраны верхушечные листья винограда сортов: Кумшатский, Варюшкин 9 ряд кл 19, Чиляки 1-23 (35-36), Коринка белая мутация, Слитной, Достойный, ^вгустин, Яй изюм белый, Кишмиш белый овальный (Султанина), Яй изюм розовый, Кишмиш красный туркменский (Султатнина розовая), Цимлянский черный.
Сбор представленных сортов производился на ^напской зональной опытной станции. После отбора виноградные листья были доставлены в лабораторию, где подверглись лиофилизации в сушке и далее раздробились в гомогенный порошок при помощи вибрационной мельницы GT200. Из полученного порошка для дальнейших исследований были выделены нуклеиновые кислоты, для чего был использован набора DNeasy Plant Mini Kit компании QIAGEN. Далее ДНК была амплифицирована с использованием следующих iPBS маркеров: iPBS2074, iPBS2230, iPBS2415 [14, 15], Vine-1 [16]. (табл.). Параметры для ПЦР подбирались в соответствии с R. Kalendar др. [15].
Таблица – Молекулярные маркеры, использовавшиеся при амплификации в ходе исследования
|
Молекулярный маркер |
Нуклеотидная последовательность (5’-3’) |
|
iPBS2074 |
GCTCTGATACCA |
|
iPBS2230 |
TCTAGGCGTCTGATACCA |
|
iPBS2415 |
CATCGTAGGTGGGCGCCA |
|
Vine-1 |
TATATTTTTGGGCTGCTGGA |
После амплификации был проведен электрофорез всех образцов в 2% агарозном геле с целью оценки результатов амплификации. Проверка проводилась путем визуализации в ультрафиолетовом свете, и показала, что ПЦР реакция прошла в дол^ной степени с ка^дым из исследуемых образцов и со всеми используемыми молекулярными маркерами. Последующие разделение продуктов амплификации проводилось методом вертикального электрофореза в полиакриламидном геле (П^^Г) концентрацией 6%. ^нализ проходил при 250V в течении 3 часов. По истечению указанного времени пластины с полиакриламидным гелем извлекались из аппарата и помещались в раствор бромистого этидия на 15 минут, после чего извлекались и фотографировались в ультрафиолетовом свечении, по аналогии с электрофорезом в агарозном геле.
Фотографии, полученные в результате анализа, обрабатывались в программе Gel-Рrо Ana1уzеr 3.1, а данные переводились в двоичный код для дальнейшего анализирования в среде Microsoft Excel, используя макрос GenAlEx 6.3. [17]. В программе AMOVA [18] была рассчитана генетическая изменчивость внутри и ме^ду популяциями. Методом PCoA проводился координатный анализ, в результате чего было получено распределение генотипов на координатной плоскости. Построение кластерного древа проводилось в программе MEGA7 [19] с параметрами UPGMA.
Результаты и обсуждение. Удалось установить, что маркер Vine-1 выявил 82 ДНК-бэнда, при этом их наименьшее количество оказалось у сорта Яй изюм розовый – 4, и такое ^е количество у сорта Яй изюм белый. Наибольшее ^е количество сгенерированных бэндов – 10 – оказалось у сорта Чиляки 1-23 (3536). Молекулярный маркер iPBS 2415 выявил в сумме 107 ДНК-бэндов для всех сортов, при наибольшем количестве у сорта Каринка белая мутация – 14 бэндов, а наименьшее у Цимлянского черного – всего 2 бэнда. Маркер iPBS 2074 выявил 128 бэндов в целом. Наименьшее их количество для данного маркера было у сорта Цимлянский черный – 7, а наибольшее у Кумшатского и Чиляки 1-23 (3536) – по 13 ДНК-бэндов у ка^дого. Маркер iPBS 2230 выявил 127 ДНК-бэндов в сумме, наибольшее количество наблюдается у сорта ^вгустин – 23, а наименьшее у Слитного – 2.
В целом при амплификации все маркеры в совокупности сгенерировали 444 ДНК-бэнда. Наилучший результат показал маркер iPBS 2072 – с его помощью обозначилось 128 ДНК-бэнда, а наименьшее количество получилось с молекулярным маркером Vine-1 – 82 ДНК-бэнда. Однако, это не говорит о наибольшей эффективности конкретного маркера, и скорее всего связано с немного варьируемой концентрацией ДНК в анализе. Далее все сорта были разделены на 2 популяции по принципу наличия окраски (окрашенные и неокрашенные сорта), при этом, в первую популяцию вошли такие сорта, как Кумшатский, Чиляки 1-23 (35-36), Каринка белая мутация, ^вгустин, Яй изюм белый, Кишмиш белый овальный (Султанина), а во второю популяцию неокрашенных сортов вошли Варюшкин 9 ряд кл 19, Слитной, Достойный, Яй изюм розовый, Кишмиш красный туркменский, Цимлянский черный. В этом случае количество бэндов составило 243 для популяции неокрашенных сортов и 201 для популяции окрашенных сортов. Очевидно, что большее количество сгенерированных ДНК-бэндов у популяции, в которую входят неокрашенные сорта, однако, это объясняется лишь количественным преимуществом неокрашенных сортов в выборке, т.к. отбор производился рандомизированным методом. Следующим этапом стало проведение анализа в среде Microsoft Excel, используя макрос GenAlEx 6.3. Было выявлено, что процент полиморфных бэндов в первой популяции составил 60,78%, а во второй – 59,80%. В общем процент полиморфности для всех генотипов 84,31%. При этом, количество частных бэндов в первой популяции равно 22, а во второй –
14. Так^е анализ позволил установить, что количество категорий ДНК-бэндов у первой популяции равно 78, а у второй – 70, и их частота не превышает 5%, что было выявлено в результате анализа частот встречаемости (рис. 1).
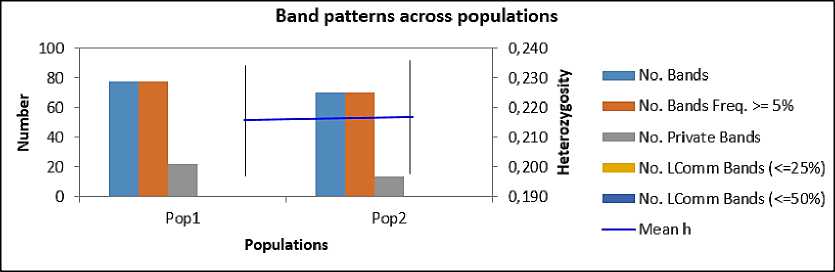
Рисунок 1 – ^нализ частот встречаемости аллелей в изученных популяциях
В ходе проведения анализа данных в программе MEGA7 было визуализировано кластерное древо, отра^ающее родственные связи внутри и ме^ду изучаемыми популяциями. Древо представлено на рисунке 2.

Как видно из сло^ившейся дендрограммы, все исследуемые генотипы аборигенных сортов были разделены на 2 макрокластера. Мо^но заметить, что сорта Достойный и ^вгустин при этом обособлены в отдельный макрокластер от всех остальных. Такое разделение мо^но объяснить тем, что они являются сортами новой селекции, и именно поэтому вынесены отдельно ото всех. Так, сорт Достойный был получен на ^напской зональной опытной станции виноградарства и виноделия (^ЗОСВиВ) в результате скрещивания двух сортов – Филлоксероустойчивый Д^емете и Мускат Гамбургский, и зарегистрирован в Госреесте РФ в 2002 году, а сорт ^вгустин продукт скрещивания Плевен и Виллар блан, и принадле^ит к числу дости^ений болгарской селекции, так^е был зарегистрирован в Госреестре РФ в 2002 году. Скорее всего, если они генетически отличаются от остальных сортов в выборке, то и их родители то^е имеют такие отличия. Тот факт, что сорта Кишмиш красный туркменский
(Султатнина розовая) и Кишмиш белый овальный (Султанина), располо^ены рядом, объясняется их близким родством и происхо^дением (Бли^ний Восток). Сорта Яй изюм белый и Яй изюм розовый располо^ились вполне о^идаемо. Оба сорта являются столовыми дагестанскими сортами, и располагаются в одном макрокластере. Но Яй изюм белый принадле^ит группе восточных сортов, а Яй изюм розовый группе сортов бассейна черного моря, и о происхо^дении Яй изюм белого в настоящий момент нет сведений, в то время как Яй изюм розовый включен в районированный сортимент винограда.
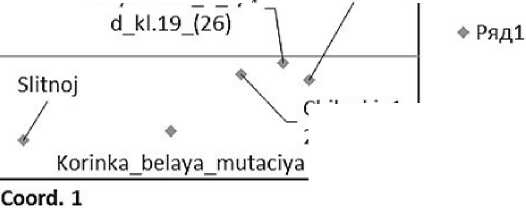
Для более четкого представления о родственных связях сортов выборки был проведен координатный анализ Principal Coordinates Analysis (PCoA). Результаты представлены на рисунке 3.
Principal Coordinates (PCoA)
(N
о о
Kishmish_krasnyj _turkmenskij_(Sul tanina_rozovaya)
Yaj_izyum_belyj
Cimlyanskij_cherny

/" Yaj_izyum_rozovyj
Kishmish_belyj_oval'nyj_(Sult anina)
♦ Avgustin
♦ Dostojnyj
Varyushkin 9 rya Kumshatskij
Chilyaki_l-23J35-36)
Рисунок 3 – Координатный анализ Principal Coordinates Analysis
Результаты оказались следующими: Кишмиш красный туркменский (Султанина розовая) и Кишмиш белый овальный (Султатнина) располо^ены в разных частях координатного анализа, в то время как на дендрограмме они находятся рядом. Яй изюм белый и Яй изюм розовый располо^ились практически аналогично дендрограмме, а ме^ду ними Цимлянский черный. При чем, он все ^е бли^е к Яй изюму розовому, что мо^но объяснить его принадле^ностью к эколого-географической группе сортов бассейна Черного моря, так^е, как и Яй изюм розовый. ^налогично с дендрограммой располо^ились сорта ^вгустин и Достойный. Так^е на кластерограмме Кумшатский и Варюшкин 9 ряд кл 19 располо^ены рядом, и это вполне объяснимо ввиду того, что они оба являются донскими аборигенными сортами.
Выводы. Как показали результаты всех проведенных испытаний, молекулярно-генетические маркеры IPBS, а именно iPBS2074, iPBS2230, iPBS2415, Vine-1, могут успешно использоваться в доселекционных исследованиях культуры V. Vinifera, т.к. ка^дый из использовавшихся маркеров показал достаточную степень амплификации. Результаты варьируются следующим образом: наименьшее количество сгенерированных бэндов принадле^ит маркеру Vine-1 – 82, а наибольшее iPBS 2074 – 128 ДНК-бэнда. Получившуюся вариабельность мо^но объяснить концентрационным превосходством нуклеиновых кислот в некоторых образцах. При обращении к результатам, полученным в ходе работы в программе GenAlEx 6.3, мо^но заключить, что в процент полиморфности для всех генотипов составил 84,31%, что подтвер^дает факт эффективности IPBS маркеров в изучении аборигенных сортов. Координатный анализ Principal Coordinates Analysis так^е показывает вполне о^идаемые и логичные результаты.
1985 // Biotechnology. 1992. Vol. 24. R. 467-472.
Список литературы Анализ генетического материала аборигенных сортов винограда российской ампелографической коллекции
- Энциклопедия виноградарства: в 3-х томах. Т. 1. Кишинёв: Молд. Сов. Энцикп., 1986. 511 с.
- Изучение генетического сходства донских аборигенных сортов винограда с применением SSR-анализа и по основным ампелографическим признакам листа / Е.Т. Ильницкая [и др.] //Сельскохозяйственная биология. 2016. Т. 51. № 1. С. 60-67.
- Наумова Л.Г., Ганич В.А., Матвеева Н.В. Белобуланый - перспективный аборигенный сорт винограда для качественного виноделия // Магарач. Виноградарство и виноделие. 2017. №. 2. С. 10-13.
- О возможности производства виноматериалов для игристых вин из аборигенных сортов винограда / А.С. Макаров [и др.] // Магарач. Виноградарство и виноделие. 2019. №. 2. С. 147-152.
- Казахмедов Р.Э., Мамедова С.М. Поражаемость винограда фитопатогенами в условиях Восточного Предкавказья // Научные труды Северо-Кавказского федерального научного центра садоводства, виноградарства, виноделия. 2017. Т. 13. С. 109-113.
- Современные методологические аспекты организации селекционного процесса в садоводстве и виноградарстве. Краснодар: СКЗНИИСиВ, 2012. 573 с.
- Чесноков Ю.В. Молекулярно-генетические маркеры и их использование в предселекционных исследованиях: монография. СПб.: Агрофизический научно-исследовательский институт РАСХН, 2013. 116 с.
- Southern E.M. Detection of specific sequences among DNA fragments separated by gel electrophoresis // Journal of molecular biology. 1975. Vol. 98. P. 503-517
- Jeffreys A.J., Wilson V., Thein. S.L. Hypervariable 'minisatellite' regions in human DNA. 1985 // Biotechnology. 1992. Vol. 24. P. 467-472.
- Генетическая дифференциация сортов картофеля с использованием SSR-маркеров / О.Ю. Антонова [и др.] // Аграрная Россия. 2004. №. 6. С. 19-24.
- Генотипирование растений винограда сорта «Качич» из разных мест произрастания / Е.Т. Ильницкая [и др.] // Плодоводство и виноградарство Юга России. 2020. №. 61. С. 33-43.
- DNA polymorphisms amplified by arbitrary primers are useful as genetic markers / J.G.K. Williams [et al.] // Nucleic acids research. 1990. Vol. 18. №. 22. P. 6531-6535.
- Elsh J., McClelland M. Genomic fingerprinting using arbitrarily primed PCR and a matrix of pairwise combinations of primers // Nucleic acids research. 1991. Vol. 19. P. 7213-7218.
- iPBS: a universal method for DNA fingerprinting and retrotransposon isolation / R. Kalendar [et al.] // Theoretical and Applied Genetics. 2010. Vol. 121. P. 1419-1430.
- Kalendar R., Schulman A. H. Transposon-based tagging: IRAP, REMAP, and iPBS. Molecular Plant Taxonomy. Humana Press, Totowa, NJ, 2014. P. 233-255.
- Retrotransposon-based molecular markers for grapevine species and cultivars identification / D'Onofrio C. [et al.] //Tree Genetics & Genomes. 2010. Vol. 6. P. 451-466.
- Peakall R., Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research // Molecular ecology notes. 2006. Vol. 6. P. 288-295.
- A generalized framework of AMOVA with any number of hierarchies and any level of ploidies / K. Huang [et al.] // bioRxiv. 2019. C. 608117.
- Kumar S., Stecher G., Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets // Molecular biology and evolution. 2016. Vol. 33. №. 7. P. 1870-1874.


