Анализ коэкспрессии генов плацентарного транскриптома как основа для поиска ключевых сигнальных путей и биомаркеров больших акушерских синдромов
Автор: Трифонова Е.А., Марков А.В., Зарубин А.А., Бабовская А.А., Куценко И.Г., Габидулина Т.В., Ижойкина Е.В., Сереброва В.Н., Степанов В.А.
Журнал: Сибирский журнал клинической и экспериментальной медицины @cardiotomsk
Рубрика: Экспериментальные исследования
Статья в выпуске: 4 т.36, 2021 года.
Бесплатный доступ
Цель: изучение молекулярных механизмов развития заболеваний группы больших акушерских синдромов (БАС) на уровне транскриптома материнской части плаценты человека.Материал и методы. С помощью базы данных Gene Expression Omnibus (GEO) был осуществлен поиск результатов полногеномного профилирования плацентарной ткани человека для следующих фенотипов: физиологическая беременность, преэклампсия (ПЭ), невынашивание беременности и задержка роста плода (ЗРП). Было отобрано 11 наборов данных, которые были дополнены результатами собственного исследования, суммарно в интегративный анализ был включен 481 образец плацентарной ткани человека. Биоинформатическая обработка и статистический анализ данных были выполнены в программной среде R 3.6.1 с использованием пакетов Bioconductor. Итоговые наборы данных, объединенные по трем патологиям беременности, были использованы для поиска общих молекулярных мишеней БАС посредством анализа взвешенных сетей коэкспрессии генов (WGCNA). Функциональную аннотацию генов и полученных кластеров осуществляли в базе данных DAVID, сеть белок-белковых взаимодействий продуктов генов была построена с использованием программного обеспечения STRING, центральные гены для сети были идентифицированы с помощью анализа MCC плагина сytoHubba программного обеспечения Cytoscape 3.7.2.Результаты. Получена таблица уровней экспрессии 15167 генов в 246 образцах. Иерархическая кластеризация этой сети позволила обнаружить 55 модулей коэкспрессирующихся генов в группе с ПЭ, 109 - в группе с преждевременными родами (ПР), 75 - для больных ЗРП и 56 - для контрольной группы. Результаты анализа общности набора модулей коэкспрессии для изучаемых фенотипов свидетельствуют о наличии общего кластера, состоящего из 8 генов, специфичного только для больных ПЭ и ЗРП, а также модуля коэкспрессии из 23 генов, характерного только для больных ПР и ЗРП. Для данных генов была построена сеть белок-белковых взаимодействий, в которой центральное место заняли гены SOD1, TXNRD1 и UBB. Оценив топологию сети в cytoHubba, были идентифицированы 6 наиболее функционально активных генов (rank ˂ 5): SOD1, TKT, TXNRD1, GCLM, GOT1, ACO1.Заключение. Полученные результаты позволили идентифицировать перспективные генетические маркеры преэклампсии, задержки роста плода и невынашивания беременности, а также обозначить наиболее важные общие молекулярные механизмы данных заболеваний, протекающие в плацентарной ткани.
Большие акушерские синдромы, коэкспрессия генов, плацента, интегративный анализ
Короткий адрес: https://sciup.org/149139363
IDR: 149139363 | УДК: 577.214/.215:618.36 | DOI: 10.29001/2073-8552-2021-36-4-144-155
Текст научной статьи Анализ коэкспрессии генов плацентарного транскриптома как основа для поиска ключевых сигнальных путей и биомаркеров больших акушерских синдромов
Появившаяся более 10 лет назад благодаря работам G. Di Renzo и R. Romero [1, 2] концепция об общности патогенетических процессов, ассоциированных с возникновением тяжелых гестационных осложнений, задала новый вектор в изучении патологии беременности и заставила по иному взглянуть на роль провизорных органов, в частности, плаценты, в генезе так называемых больших акушерских синдромов (БАС), к которым относят преэклампсию (ПЭ), задержку роста плода (ЗРП), преждевременный разрыв плодных оболочек, преждевременные роды (ПР) и невынашивание беременности (НБ). К ключевым признакам, характеризующим заболевания из группы БАС, авторы концепции относят полиэтиологичность, длительную преклиническую стадию, частое вовлечение плода, присутствие клинических проявлений, часто являющихся адаптивными по своей природе, наличие предрасположенности к определенному синдрому, зависящей от взаимодействия генов и окружающей среды и/или комплексного ген-ген-ного взаимодействия материнского и/или фетального генотипов [2].
Несмотря на то, что перечисленные акушерские заболевания обусловливают существенную долю перинатальных потерь, а также материнской заболеваемости и смертности в мире и России, анализ современной научной литературы свидетельствует о том, что мы все еще очень далеки от ясного понимания этиологических факторов и патогенеза данных патологических состояний беременности. Однако появившиеся и ставшие доступными в последние годы не только для теоретических изысканий, но и для прикладных научных медицинских исследований современные высокоинформативные молекулярно-генетические методы дают надежду на появление новых знаний, на основе которых будут разработаны патогенетические модели БАС, предложены методы их прогноза и профилактики.
В данном контексте особую актуальность приобретают исследования патофизиологических механизмов осложнений беременности с позиции единого генеза, детерминированного морфофункциональными нарушениями в фетоплацентарной системе, с акцентом на изучение молекулярных процессов, происходящих в плацентарной ткани.
Необходимо отметить, что в последнее десятилетие было проведено более 50 исследований транскриптома с целью поиска генетических факторов, связанных с различными акушерскими заболеваниями, в которых было выявлено достоверное повышение/снижение экспрессии нескольких сотен генов в плацентарной ткани и крови пациенток с репродуктивной патологией по сравнению с женщинами с физиологической беременностью [3–12]. Эти данные полезны для более глубокого понимания патогенеза гестационных осложнений, однако взаимосвязи между экспрессией генов и регуляцией генных сетей остаются малоизученными.
Цель нашей работы: характеристика молекулярных механизмов и поиск новых генов, определяющих развитие заболеваний группы БАС, в рамках анализа генных сетей и сетей коэкспрессии генов, построенных на основе транскриптомных данных, полученных при полногеномном экспрессионном профилировании плацентарной ткани.
Материал и методы
В представленной работе с целью поиска транс-криптомных данных, полученных на выборках пациенток из группы БАС и женщин с физиологической беременностью (объект исследования – материнская (дистальная) часть плаценты), были использованы следующие варианты запроса в поисковой строке баз данных GEO (Gene Expression Omnibus; и ArrayExpress arrayexpress): «preeclampsia, placenta»; «preeclampsia, decidua»; «preeclampsia, basal plate»; «premature birth, premature delivery, prematurity, preterm delivery, obstetric labor, premature, preterm labor, placenta»; «premature birth, premature delivery, prematurity, preterm delivery, obstetric labor, premature, preterm labor, decidua»; «premature birth, premature delivery, prematurity, preterm delivery, obstetric labor, premature, preterm labor, basal plate»; «fetal growth retardation, intrauterine growth restriction, small gestational age, placenta»; «fetal growth retardation, Intrauterine Growth Restriction, small gestational age, decidua»; «fetal growth retardation, Intrauterine Growth Restriction, small gestational age, basal plate»; «abortion, spontaneous abortion, miscarriage, placenta»; «abortion, spontaneous abortion, miscarriage, decidua»; «abortion, spontaneous abortion, miscarriage, basal plate». Для анализа нами были выбраны следующие четыре наиболее распространенных патологии из группы больших акушерских синдромов: ПЭ, невынашивание беременности (ПР и самопроизвольный выкидыш), ЗРП. Всего было найдено 30 исследований с выгруженными наборами данных, характеризующих транскриптом плацентарной ткани человека при выбранных фенотипах (E-MTAB-5353, GSE10588, GSE12216, GSE14722, GSE22490, GSE24129, GSE25861, GSE25906, GSE25906, GSE30186, GSE35574, GSE35574, GSE44711, GSE4707, GSE47187, GSE54618, GSE60438, GSE6573, GSE73374, GSE73377, GSE73685, GSE73712, GSE74341, GSE75010, GSE91077, GSE91189, GSE93839, GSE94643, GSE94644, GSE96985), которые были дополнены результатами собственного исследования [12].
Из всего спектра опубликованных в GEO и ArrayExpress наборов данных были использованы только те, которые удовлетворяли следующим критериям: 1) оценки количества транскриптов в образцах представлены в виде сигналов свечения соответствующих ДНК-зондов (детектируемых технологией одноцветного мечения), сравнимого диапазона интенсивности; 2) не менее 30000 ДНК-зондов на микрочип для более обширного покрытия спектра возможных транскриптов; 3) ткани, в которых проводилось транскриптомное профилирование, включали материнскую (дистальную) часть плаценты; 4) забор материала на исследование проводился в третьем триместре беременности; 5) дизайн эксперимента включал группу больных и группу с нормальной (физиологической) беременностью, количество индивидов в группе сравнения было не менее 2. Согласно данным критериям, было отобрано 11 наборов данных, полученных при использовании коммерческих микрочиповых платформ Illumina и Affymetrix (табл. 1). Суммарное количество анализируемых в интегративном анализе образцов плацентарной ткани человека составило 481. Необходимо отметить, что было найдено всего 4 исследования, в которых было проведено транскриптомное профилирование плацентарной ткани при ПР и ЗРП. Основной массив данных был получен для такого фенотипа, как ПЭ (см. табл. 1).
Таблица 1. Данные, включенные в интегративный анализ (данные из БД GEO и результаты собственных исследований), описывающие результаты профилирования транскриптома плацентарной ткани человека (дистальная часть) при преэклампсии, невынашивании беременности и задержке роста плода
Биоинформатическая обработка и статистический анализ данных были выполнены в программной среде R 3.6.1 с использованием специализированных пакетов проекта Bioconductor (https://www. . Доступ к ресурсам GEO осуществлен с помощью программного пакета GEOquery [24]. Наборы данных были преобразованы с помощью log2-трансфор-мации и нормализации: методом квантильной нормализации для микрочипов Illumina и согласно алгоритму
RMA (Robust Multi-array Average) – в случае микрочипов Affymetrix. С помощью инструмента biomaRt [25] были получены идентификаторы генов из геномного браузера Ensembl для соответствующих ДНК-проб микрочипов. Затем для каждой патологии беременности в отдельности были найдены списки ДНК-проб, общих для разных наборов данных и разных микрочиповых платформ, согласно идентификаторам генов. Если на микрочиповой платформе на один ген приходилось несколько ДНК-проб, опреде-
Table 1. Data included in the integrative analysis (datasets from the GEO database and the results of own studies) describing the results of profiling the transcriptome of human placental tissue (distal part) in preeclampsia, pregnancy loss, and intrauterine growth restriction
Результаты и обсуждение
Благодаря быстро расширяющимся знаниям об инте-рактоме человека как сложной системе белок-белковых взаимодействий, на сегодняшний день подходы, основанные на построении генных сетей, являются наиболее эффективными и информативными при изучении патогенетики заболеваний. В представленной работе с целью детального изучения молекулярных механизмов
развития БАС на уровне транскриптома плацентарной ткани и идентификации ключевых взаимодействий генов был проведен анализ коэкспрессии генов, позволяющий выявлять модули генов, межмодульные хабы и узлы, основываясь на схожести профиля экспрессии. Сеть коэкс-прессии была построена с помощью метода WGCNA [26], для реализации которого проводили предварительную коррекцию известного эффекта группировки по отдельным исследованиям с помощью эмпирического Байесовского алгоритма ComBat в программном пакете sva [28] и выбирали гены, детектированные во всех исследуемых группах. В результате описанных процедур была получена таблица уровней экспрессии 15167 генов в 246 образцах. Иерархическая кластеризация этой сети при выбранных параметрах позволила обнаружить 55 модулей коэкспрессирующихся генов в группе с ПЭ, 109 – в группе с ПР, 75 модулей было выявлено для больных ЗРП, 56 – для контрольной группы (рис. 1).
Результаты анализа соответствия (общности) набора специфических для изучаемых фенотипов модулей ко-экспресии, полученные в парных сравнениях взаимного обогащения модулей транскриптами при помощи точного теста Фишера, свидетельствуют о наличии общего модуля коэкспресии, специфичного только для больных ПЭ и ЗРП, включающего 8 генов, а также модуля коэкспресии, состоящего из 23 генов, характерного исключительно для пациенток с ПР и ЗРП (рис. 2, табл. 2). Необходимо отметить, что в представленной работе не было выявлено общего модуля коэкспрессирующихся генов, специфичного только для групп с ПР и ПЭ, а также для всех трех исследуемых фенотипов.

II Hill III
А. Преэклампсия
A. Preeclampsia
Б. Преждевременные роды B. Premature birth
-
В. Задержка роста плода
Г. Контрольная группа D. Control group
-
C. Intrauterine growth restriction
Рис. 1. Результаты кластеризации профилей экспрессии генов методом WGCNA
Примечание: сверху представлено дерево, каждая ветвь которого соответствует группе генов со сходными профилями экспрессии. Вертикальная ось описывает расстояния между кластерами. Внизу представлен результат автоматического выделения модулей коэкспрессии на основе полученного дерева кластеризации. Области серого цвета соответствуют группам генов, не вошедшим в состав ни одного из модулей коэкспрессии.
-
Fig. 1. The results of gene expression clustering using WGCNA
Note: Cluster dendrogram is presented at the top; each branch corresponds to a group of genes with similar expression profiles. The vertical axis describes the distances between clusters. Bottom colored bands is the result of automatic assignment of genes to co-expressed modules based on clustering algorithm and topological similarity of co-expressed genes. Areas of gray color correspond to gene groups not assigned to any module.
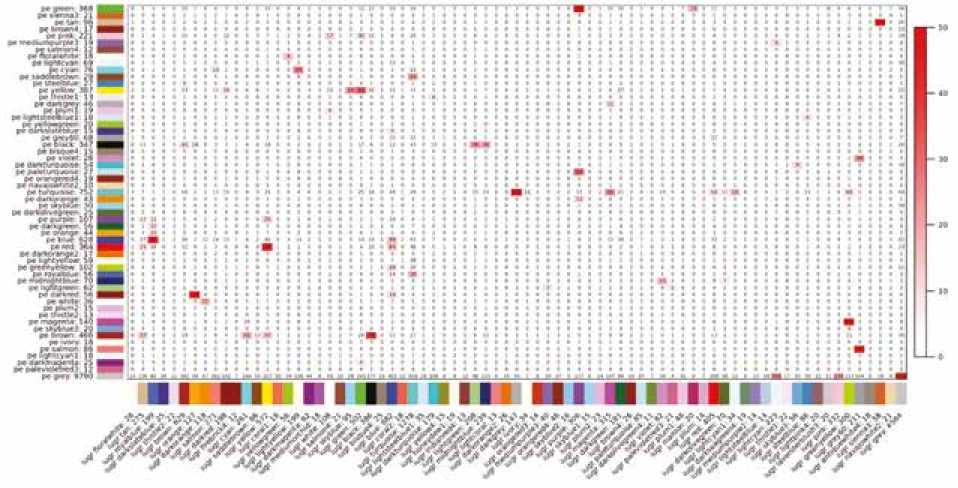
А. Результаты попарного сравнения преэклампсии и задержки роста плода
-
A. Results of pairwise comparison of preeclampsia and intrauterine growth restriction
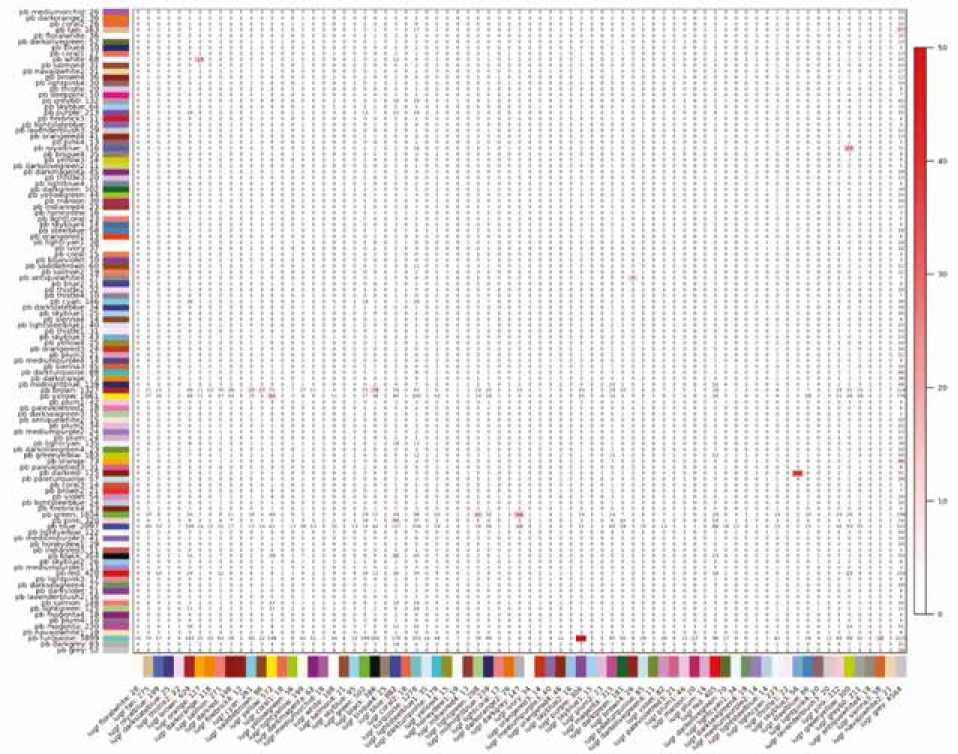
Б. Результаты попарного сравнения преэклампсии и задержки роста плода
-
B. Results the pairwise comparison of preeclampsia and intrauterine growth restriction
Рис. 2. Результаты анализа соответствия набора специфических для изучаемых фенотипов модулей коэкспресии
-
Fig. 2. Results of analysis of co-expression gene modules specific for the study phenotypes
Таблица 2. Гены, входящие в состав общих модулей коэкспрессии, специфичных для больших акушерских синдромов
Table 2. Genes in the co-expression modules common for the great obstetrical syndromes
|
Ген Gene |
Продукт гена Gene product |
Изученные фенотипы/модули Studied phenotypes/modules |
||
|
ПР Premature birth |
ПЭ Preeclampsia |
ЗРП IUGR |
||
|
DSG3 |
Десмоглеин 3 Desmoglein-3 |
Darkred |
grey |
skyblue3 |
|
KRT14 |
Кератин 14 Keratin 14 |
Darkred |
magenta |
skyblue3 |
|
IL22RA1 |
Альфа-1 субъединица рецептора интерлейкина 22 Interleukin 22 receptor subunit alpha 1 |
Darkred |
grey |
skyblue3 |
|
KRT17 |
Кератин 17 Keratin 17 |
Darkred |
magenta |
skyblue3 |
|
KRT6B |
Кератин 6B Keratin 6B |
Darkred |
grey |
skyblue3 |
|
FAM83A |
Белок FAM83A Family with sequence similarity 83 member A |
Darkred |
grey |
skyblue3 |
|
WDR62 |
Белок 62, содержащий повторы WD WD repeat-containing protein 62 |
Darkred |
grey |
skyblue3 |
|
C1orf116 |
Специфически андроген-регулируемый белок Specifically androgen-regulated gene protein |
Darkred |
darkturquoise |
skyblue3 |
|
LAMB3 |
Бета-3 субъединица ламинина Laminin subunit beta 3 |
Darkred |
darkturquoise |
skyblue3 |
|
HAND1 |
Белок 1, экспрессируемый производными сердца и нервного гребня Heart- and neural crest derivatives-expressed protein 1 |
Darkred |
grey |
skyblue3 |
|
ZBED2 |
Цинковый палец, содержащий домен BED 2 Zinc finger BED-type containing 2 |
Darkred |
darkturquoise |
skyblue3 |
|
GABRP |
Субъединица pi рецептора гамма-аминомасляной кислоты Gamma-aminobutyric acid type A receptor pi subunit |
Darkred |
darkturquoise |
skyblue3 |
|
MSLN |
Мезотелин Mesothelin |
Darkred |
darkturquoise |
skyblue3 |
|
AGR3 |
Белок переднего градиента-3 Anterior gradient 3, protein disulphide isomerase family member |
Darkred |
darkturquoise |
skyblue3 |
|
EDN2 |
Эндотелин 2 Endothelin 2 |
Darkred |
grey |
skyblue3 |
|
KRT16 |
Кератин 16 Keratin 16 |
darkred |
grey |
skyblue3 |
|
A2ML1 |
Альфа-2-макроглобулиноподобный белок 1 alpha-2-macroglobulin like 1 |
Darkred |
grey |
skyblue3 |
|
DSG1 |
Десмоглеин 1 Desmoglein 1 |
Darkred |
grey |
skyblue3 |
|
KRT5 |
Кератин 5 Keratin 5 |
Darkred |
grey |
skyblue3 |
|
SFN |
Стратифин Stratifin |
Darkred |
darkturquoise |
skyblue3 |
|
HRASLS2 |
Фосфолипаза А и ацилтрансфераза 2 HRAS-like suppressor 2 |
Darkred |
grey |
skyblue3 |
|
NGEF |
Эфексин-1 Neuronal guanine nucleotide exchange factor |
Darkred |
grey |
skyblue3 |
|
SPRR3 |
Малый белок 3, богатый пролином Small proline-rich protein 3 |
Darkred |
grey |
skyblue3 |
|
PRPF40A |
Фактор 40 пре-мРНК-процессинга, гомолог A Pre-mRNA-processing factor 40 homolog A |
Salmon |
floralwhite |
yellowgreen |
|
ZC3H11B |
Цинковый палец CCCH- тип, содержащий белок 11B Zinc finger CCCH-type containing 11B |
Yellow |
floralwhite |
yellowgreen |
|
ZNF148 |
Белок цинковых пальцев 148 Zinc finger protein 148 |
Lightgreen |
floralwhite |
yellowgreen |
|
AGO2 |
Белок-аргонавт 2 Argonaute RISC catalytic component 2 |
Blue |
floralwhite |
yellowgreen |
|
TNPO1 |
Транспортин-1 Transportin 1 |
Salmon |
floralwhite |
yellowgreen |
Окончание табл. 2
End of table 2
|
Ген Gene |
Продукт гена Gene product |
Изученные фенотипы/модули Studied phenotypes/modules |
||
|
ПР Premature birth |
ПЭ Preeclampsia |
ЗРП IUGR |
||
|
ZRANB1 |
Убиквитин тиоэстераза ZRANB1 Zinc finger RANBP2-type containing 1 |
Thistle |
floralwhite |
yellowgreen |
|
TAOK1 |
Серин/треонин-протеинкиназа TAO1 Serine/threonine-protein kinase TAO1 |
Steelblue |
floralwhite |
yellowgreen |
|
NUFIP2 |
Ядерный белок 2, взаимодействующий с FMR1 Nuclear FMR1 interacting protein 2 |
brown |
floralwhite |
yellowgreen |
Результаты функциональной аннотации генов, входящих в состав общих модулей коэкспрессии, специфичных для БАС, с помощью ресурса Molecular Signatures Database (MSigDB), вычислительного ме- тода «Gene Set Enrichment Analysis» свидетельствуют о статистически значимой принадлежности 19 генов из данного массива к девяти категориям, описанным в таблице 3.
Таблица 3. Наиболее значимые категории, полученные с помощью ресурса MSigDB, обогащенные генами, входящими в состав общих модулей коэкспрессии, специфичных для больших акушерских синдромов
Table 3. The most significant categories obtained using the MSigDB resource and enriched by the genes from the overlapping co-expressed gene modules specific for great obstetrical syndromes
|
Категория Category |
Краткая характеристика категории Brief description of the category |
о ™ 2 ф О ™ if |
FDR FDR |
|
JAEGER_METASTASIS_DN |
Гены, экспрессия которых подавляется в метастазах злокачественной меланомы по сравнению с первичными опухолями Genes down-regulated in metastases from malignant melanoma compared to the primary tumors |
11 |
1.25E-13 |
|
GOBP_KERATINOCYTE_ DIFFERENTIATION |
Процесс дифференцировки неспециализированных клеток в кератиноциты The process in which a relatively unspecialized cell acquires specialized features of a keratinocyte |
10 |
1.43E-10 |
|
SMID_BREAST_CANCER_BASAL_UP |
Гены, экспрессия которых активируется в образцах базальноподобного рака молочной железы Genes up-regulated in samples of basal-like breast cancer |
12 |
3.41E-11 |
|
RICKMAN_TUMOR_DIFFERENTIATED_ WELL_VS_POORLY_DN |
Гены, дифференциально экспрессирующиеся между группами HNSCC (плоскоклеточная карцинома головы и шеи) Genes differentially expressed between HNSCC groups (head and neck squamous cell carcinoma) |
10 |
2.15E-10 |
|
MODULE_153 |
Гены, входящие в опухолевый модуль 153 Genes in the cancer module 153 |
6 |
2.34E-10 |
|
KOINUMA_TARGETS_OF_SMAD2_OR_ SMAD3 |
Гены, промоторы которых связаны с факторами SMAD2 или SMAD3 в клетках HaCaT (кератиноциты), согласно ChIP-chip анализу Genes with promoters occupied by SMAD2 or SMAD3 [GeneID = 4087, 4088] in HaCaT cells (keratinocyte) according to a ChIP-chip analysis |
12 |
2.34E-10 |
|
ONDER_CDH1_TARGETS_2_DN |
Гены, гипоэкспрессирующиеся в клетках HMLE (иммортализованный/ трансформированный эпителий молочной железы) после нокдауна с помощью RNAi гена CDH1 , кодирующего E-кадгерин Genes down-regulated in HMLE cells (immortalized nontransformed mammary epithelium) after E-cadhedrin (CDH1) [GeneID = 999] knockdown by RNAi |
10 |
7.44E-10 |
|
TURASHVILI_BREAST_DUCTAL_ CARCINOMA_VS_DUCTAL_NORMAL_DN |
Гены, гипоэкспрессирующиеся в протоковой карциноме по сравнению с нормальными протоковыми клетками молочной железы Genes down-regulated in ductal carcinoma vs normal ductal breast cells |
8 |
1.41E-9 |
|
SMID_BREAST_CANCER_LUMINAL_B_DN |
Гены, гипоэкспрессирующиеся при люминальном В подтипе протоковой карциномы молочной железы Genes down-regulated in the luminal B subtype of breast cancer |
10 |
3.42E-9 |
Примечательно, что шесть из девяти обнаруженных категорий связаны с различными онкологическими заболеваниями. Необходимо отметить, что проблема взаимоотношений беременности и роста злокачественных новообразований занимает особое место в современной медицине. В первую очередь, интерес к данной проблеме обусловлен сходством обоих процессов по многим признакам на молекулярном, клеточно-тканевом и организменном уровнях, включая изменения нейроэндокринного, иммунного и метаболического статуса организма, сходные фенотипические свойства эмбриональных и злокачественных клеток, такие как способность к инвазивному росту, аутокринной секреции ростовых факторов, индукции микроциркуляции, «ускользанию» из-под иммунного надзора организма и другие [29].
В рамках данной проблемы особое внимание исследователей в последние годы уделяется поиску общих сигнальных путей инвазии клеток трофобласта в децидуальную ткань плаценты и процессов опухолевой инвазии. Показано, что клетки вневорсинчатого трофобласта и раковые клетки демонстрируют очень похожий молекулярный фенотип как на уровне транскрипционной активности генов, так и на уровне метилирования промоторов [30–32]. Следует отметить, что клетки трофобласта с их способностью к пролиферации и дифференцировке, апоптозу и выживанию, миграции, ангиогенезу и иммунной модуляции, использующие в процессе инвазии сход- ные с раковыми процессами молекулярные пути, делают их привлекательной моделью для изучения рака. Таким образом, представляются чрезвычайно перспективными дальнейшие исследования, характеризующие функциональную роль идентифицированных генов в ген-генных сетях БАС и онкологических заболеваний, что может обеспечить не только новые стратегии профилактики и терапии акушерской патологии, но и лучшее понимание молекулярных механизмов развития злокачественных новообразований.
Представленная на рисунке 3 сеть белок-белковых взаимодействий продуктов генов, полученная с помощью базы данных STRING, подтверждает взаимосвязи между генами, выявленные при анализе взвешенных сетей коэкспрессии генов. Центральное место в данной сети с наибольшим числом взаимодействий занимают гены SOD1, TXNRD1 и UBB , кодирующие соответственно супероксиддисмутазу I типа (антиоксидантный фермент, катализирующий превращение супероксид-ани-онов в молекулярный кислород и пероксид водород), цитозольную форму тиоредоксинредуктазы (селеноцистеинсодержащий флавофермент, восстанавливающий тиоредоксины и играющий ключевую роль в окислительно-восстановительном гомеостазе) и убиквитин, принимающий участие в активации или ингибировании активности белков, их транспорте, процессинге, а также деградации.
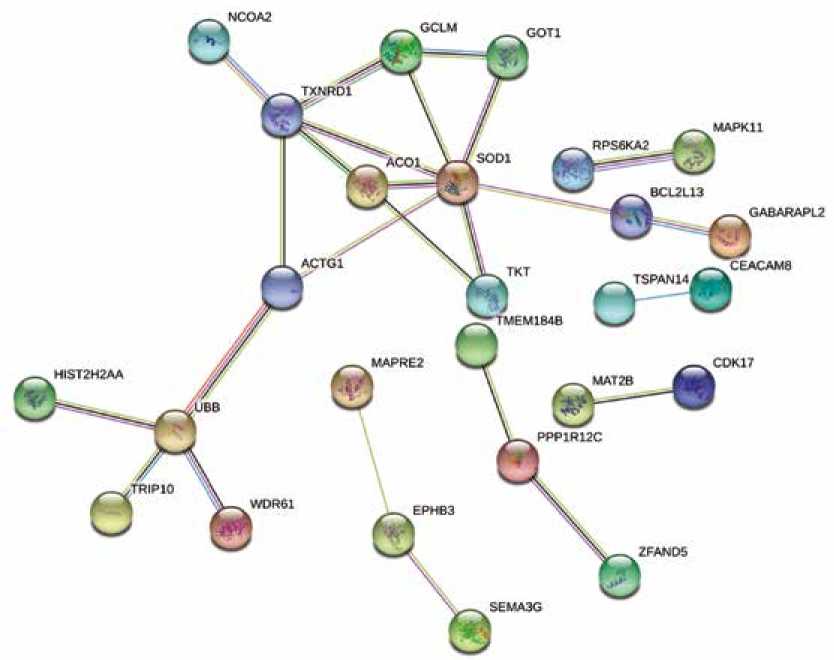
Рис. 3. Белок-белковые взаимодействия продуктов генов, ассоциированных с большими акушерскими синдромами, по данным анализа транс-криптома
Fig. 3. Protein-protein interactions of great obstetrical syndrome-associated genes according to data of transcriptome analysis
При анализе данной сети, включающей 26 протеинов, кодируемых изучаемыми генами, были выявлены только 7 генов, для которых доверительный уровень взаимодействий (combined score) составляет более 0,9: CEACAM8, NCOA2, TRIP10, TSPAN14, TXNRD1, UBB, WDR61 . Продукты некоторых из этих генов, согласно известным на сегодняшний день данным об их функциональных особенностях, вполне могут быть вовлечены в молекулярные механизмы исследуемой патологии.
В представленной работе топология сети была проанализирована также с использованием плагина cytoHubba программного обеспечения Cytoscape. Для каждого узла (белка) сети была посчитана степень, учитывающая количество взаимодействий с соседними узлами, длину связи, а также тенденцию белков к образованию кластеров. В результате были идентифицированы 6 генов, характеризующиеся наиболее высокими значениями данного показателя (rank ˂ 5): SOD1, TKT, TXNRD1, GCLM, GOT1, ACO1. Основными центральными генами рассматриваемой сети выступают локусы SOD1 и TKT (rank = 1), вовлеченные в сигнальные пути, связанные с окислительным стрессом (ген SOD1 ), пентозофосфат-ным циклом и циклом Кальвина (ген TKT ).
Известно, что окислительный стресс обусловливает в плацентарной ткани важные физиологические функции, участвуя в запуске механизмов дифференцировки клеток, повреждении трофобласта и прогрессирующей дегенерации ворсинок при формировании плодных оболочек, стимуляции выработки различных трофобластических белков, таких как хорионический гонадотропин и эстрогены. Тем не менее, при невозможности регуляции свободных радикалов антиоксидантной системой в плацентарной ткани наблюдается резко повышенный уровень маркеров окислительного стресса, что может приводить к повреждению ворсинчатого дерева и патологическому течению беременности, в частности, ПЭ, плацентарной недостаточности, неразвивающейся беременности. Так, в работе F.C. Donabela и соавт. (2015) более высокая экспрессия гена SOD1 обнаружена в кумулюсных клетках женщин, страдающих бесплодием в сочетании с эндометриозом, которые прошли контролируемую стимуляцию яичников для интрацитоплазматической инъекции сперматозоида (ИКСИ) [33].
Гипоэкспрессия гена SOD1 в плацентарной ткани выявлена при развитии преэклампсии по сравнению с физиологической беременностью [34], аналогичные данные были получены и для ПР [35]. Наряду с этим продемонстрировано, что ген SOD1 гиперэкспрессируется при раке молочной железы, а сверхэкспрессия супероксиддисму-тазы (белка, кодируемого этим геном) наблюдается при опухолях легких и молочной железы [36, 37].
Центральным геном рассматриваемой сети является также локус TKT , кодирующий транскетолазу. В литературе отсутствуют данные о вариабельности экспрессии данного гена при физиологической и патологической беременности человека, однако полногеномный анализ ассоциаций, проведенный при исследовании финской когорты новорожденных, выявил ассоциацию маркера rs4687715, являющегося eQTL гена TKT , с таким фенотипом, как гестационный возраст более 42 нед. [38]. Известно, что ген TKT кодирует белок, участвующий в основных метаболических путях углеводов, связывая пентозофосфатный путь с гликолизом. Этот процесс приводит к снижению синтеза никотинамидадениндинукле-отидфосфата (НАДФН), который вовлечен в регуляцию генерации активных форм кислорода, отвечающих за формирование состояния окислительного стресса. Недостаточная экспрессия гена TKT у мышей способствует развитию беременности, характеризующейся меньшим количеством потомства, задержке постнатального роста и снижению уровней жировой ткани у потомства [39]. Показано также, что дисрегуляция активности транскетолазы играет важную роль в нарушении скорости роста клеток, прогрессии клеточного цикла и созревании ооцитов [38].
В контексте рассматриваемой общности молекулярных механизмов онкологических и репродуктивных заболеваний интересными представляются данные, демонстрирующие значимость TKT и пентозофосфатно-го пути в развитии окислительного стресса, этиологии и фармакогенетике рака. Так, выявлено, что ТКТ усиливает пролиферацию и миграцию клеток гепатоцеллюлярной карциномы, а также их жизнеспособность в условиях окислительного стресса in vitro и темп роста и метастазирования in vivo . Более того, нарушение окислительно-восстановительного гомеостаза раковых клеток вследствие генетического нокдауна гена TKT или фармакологического ингибирования транскетолазы повышает чувствительность раковых клеток к существующей антиоксидантной таргетной терапии [40, 41].
Таким образом, проведенный интегративный анализ транскриптомных данных, анализ сетей белок-белковых взаимодействий продуктов генов, экспрессирующихся в плацентарной ткани при патологической (БАС) и физиологической беременности и анализ коэкспрессионных сетей позволили идентифицировать перспективные генетические маркеры ПЭ, ЗРП и НБ, а также обозначить наиболее важные общие молекулярные патогенетические механизмы данных заболеваний, связанные с нарушением окислительно-восстановительного гомеостаза и патологическими процессами при инвазии трофобласта, протекающими в плацентарной ткани.
Список литературы Анализ коэкспрессии генов плацентарного транскриптома как основа для поиска ключевых сигнальных путей и биомаркеров больших акушерских синдромов
- Romero R. Prenatal medicine: The child is the father of the man. 1996. J. Matern. Fetal Neonatal Med. 2009;22(8):636-639. DOI: 10.1080/14767050902784171.
- Di Renzo G.C. The great obstetrical syndromes. J. Matern. FetalNeona-ta. Med. 2009;22(8):633-635. DOI: 10.1080/14767050902866804.
- Eidem H.R., Ackerman W.E., McGary K.L., Abbot P., Rokas A. Gestational tissue transcriptomics in term and preterm human pregnancies: A systematic review and meta-analysis. BMC Med. Genomics. 2015;8:27. DOI: 10.1186/s12920-015-0099-8.
- Rajakumar A., Chu T., Handley D.E., Bunce K.D., Burke B., Hubel C.A. et al. Maternal gene expression profiling during pregnancy and preeclampsia in human peripheral blood mononuclear cells. Placenta. 2011;32(1):70-78. DOI: 10.1016/j.placenta.2010.10.004.
- Krieg S.A., Fan X., Hong Y., Sang Q.X., Giaccia A., Westphal L.M. et al. Global alteration in gene expression profiles of deciduas from women with idiopathic recurrent pregnancy loss. Mol. Hum. Reprod. 2012;18(9):442-450. DOI: 10.1093/molehr/gas017.
- Mayor-Lynn K., Toloubeydokhti T., Cruz A.C., Chegini N. Expression profile of microRNAs and mRNAs in human placentas from pregnancies complicated by preeclampsia and preterm labor. Reprod. Sci. 2011;18(1):46-56. DOI: 10.1177/1933719110374115.
- Toft J.H., Lian I.A., Tarca A.L., Erez O., Espinoza J., Eide I.P. et al. Whole-genome microarray and targeted analysis of angiogenesis-reg-ulating gene expression (ENG, FLT1, VEGF, PlGF) in placentas from pre-eclamptic and small-for-gestational-age pregnancies. J. Matern. Fetal Neonatal Med. 2008;21(4):267-273. DOI: 10.1080/14767050801924118.
- Buimer M., Keijser R., Jebbink J.M., Wehkamp D., van Kampen A.H., Boer K. et al. Seven placental transcripts characterize HELLP-syndrome. Placenta. 2008;29(5):444-453. DOI: 10.1016/j.placenta.2008.02.007.
- Van Dijk M., Oudejans C.B.M. STOX1: Key player in trophoblast dysfunction underlying early onset preeclampsia with growth retardation. J. Pregnancy. 2010;2011:521826. DOI: 10.1155/2011/521826.
- Junus K., Centlow M., Wikström A.K., Larsson I., Hansson S.R., Olovs-son M. Gene expression profiling of placentae from women with early-and late-onset pre-eclampsia: down-regulation of the angiogenesis-re-lated genes ACVRL1 and EGFL7 in early-onset disease. Mol. Hum. Reprod. 2012;18(3):146-155. DOI: 10.1093/molehr/gar067.
- Lyu S.W., Song H., Yoon J.A., Chin M.U., Sung S.R., Kim Y.S. et al. Transcriptional profiling with a pathway-oriented analysis in the placen-tal villi of unexplained miscarriage. Placenta. 2013;34(2):133-140. DOI: 10.1016/j.placenta.2012.12.003.
- Trifonova E.A., Gabidulina T.V., Ershov N.I., Serebrova V.N., Vorozhish-cheva A.Y., Stepanov V.A. Analysis of the placental tissue transcriptome of normal and preeclampsia complicated pregnancies. Acta Naturae. 2014;6(2):71-83.
- Tsai S., Hardison N.E., James A.H., Motsinger-Reif A.A., Bischoff S.R., Thames B.H. et al. Transcriptional profiling of human placentas from pregnancies complicated by preeclampsia reveals disregulation of si-alic acid acetylesterase and immune signalling pathways. Placenta. 2011;32(2):175-182. DOI: 10.1016/j.placenta.2010.11.014.
- Meng T., Chen H., Sun M., Wang H., Zhao G., Wang X. Identification of differential gene expression profiles in placentas from preeclamptic pregnancies versus normal pregnancies by DNA microarrays. OMICS. 2012;16(6):301-311. DOI: 10.1089/omi.2011.0066.
- Guo L., Tsai S.Q., Hardison N.E., James A.H., Motsinger-Reif A.A., Thames B. et al. Differentially expressed microRNAs and affected biological pathways revealed by modulated modularity clustering (MMC) analysis of human preeclamptic and IUGR placentas. Placenta. 2013;34(7):599-605. DOI: 10.1016/j.placenta.2013.04.007.
- Blair J.D., Yuen R.K., Lim B.K., McFadden D.E., von Dadelszen P., Robinson W.P. Widespread DNA hypomethylation at gene enhancer regions in placentas associated with early-onset pre-eclampsia. Mol. Hum. Reprod. 2013;19(10):697-708. DOI: 10.1093/molehr/gat044.
- Yong H.E., Melton P.E., Johnson M.P., Freed K.A., Kalionis B., Murthi P. et al. Genome-wide transcriptome directed pathway analysis of maternal pre-eclampsia susceptibility genes. PLoS One. 2015;10(5):e0128230. DOI: 10.1371/journal.pone.0128230.
- Herse F., Dechend R., Harsem N.K., Wallukat G., Janke J., Qadri F. et al. Dysregulation of the circulating and tissue-based renin-angioten-sin system in preeclampsia. Hypertension. 2007;49(3):604-611. DOI: 10.1161/01.HYP.0000257797.49289.71.
- Martin E., Ray P.D., Smeester L., Grace M.R., Boggess K., Fry R.C. Epigenetics and preeclampsia: Defining functional epimutations in the preeclamptic placenta related to the TGF-ß pathway. PLoS One. 2015;10(10):e0141294. DOI: 10.1371/journal.pone.0141294.
- Garrido-Gomez T., Dominguez F., Quiñonero A., Diaz-Gimeno P., Ka-pidzic M., Gormley M. et al. Defective decidualization during and after severe preeclampsia reveals a possible maternal contribution to the etiology. Proc. Natl. Acad. Sci. USA. 2017;114(40):E8468-E8477. DOI: 10.1073/pnas.1706546114.
- Nishizawa H., Ota S., Suzuki M., Kato T., Sekiya T., Kurahashi H. et al. Comparative gene expression profiling of placentas from patients with severe pre-eclampsia and unexplained fetal growth restriction. Reprod. Biol. Endocrinol. 2011;9:107. DOI: 10.1186/1477-7827-9-107.
- Bukowski R., Sadovsky Y., Goodarzi H., Zhang H., Biggio J.R., Varner M. et al. Onset of human preterm and term birth is related to unique inflammatory transcriptome profiles at the maternal fetal interface. Peer J. 2017;5:e3685. DOI: 10.7717/peerj.3685.
- Rull K., Tomberg K., Koks S., Männik J., Möls M., Sirotkina M. et al. Increased placental expression and maternal serum levels of apoptosis-in-ducing TRAIL in recurrent miscarriage. Placenta. 2013;34(2):141-148. DOI: 10.1016/j.placenta.2012.11.032.
- Davis S., Meltzer P.S. GEOquery: A bridge between the Gene Expression
- Omnibus (GEO) and BioConductor. Bioinformatics. 2007;23(14):1846-1847. DOI: 10.1093/bioinformatics/btm254.
- Durinck S., Spellman P.T., Birney E., Huber W. Mapping identifiers for the integration of genomic datasets with the R/Bioconductor package biomaRt. Nat. Protoc. 2009;4(8):1184-1191. DOI: 10.1038/ nprot.2009.97.
- Zhang B., Horvath S. A general framework for weighted gene co-expression network analysis. Stat. Appl. Genet. Mol. Biol. 2005;4:17. DOI: 10.2202/1544-6115.1128.
- Szklarczyk D., Gable A.L., Nastou K.C., Lyon D., Kirsch R., Pyysalo S. et al. The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets. Nucleic Acids Res. 2021; 49(D1):D605-D612. DOI: 10.1093/ nar/gkaa1074.
- Leek J.T., Johnson W.E., Parker H.S., Jaffe A.E., Storey J.D. The sva package for removing batch effects and other unwanted variation in high-throughput experiments. Bioinformatics. 2012;28(6):882-883. DOI: 10.1093/bioinformatics/bts034.
- Louwen F., Muschol-Steinmetz C., Reinhard J., Reitter A., Yuan J. A lesson for cancer research: Placental microarray gene analysis in preeclampsia. Oncotarget. 2012;3(8):759-773. DOI:10.18632/oncotarget.595.
- Smith Z.D., Shi J., Gu H., Donaghey J., Clement K., Cacchiarelli D. et al. Epigenetic restriction of extraembryonic lineages mirrors the somatic transition to cancer. Nature. 2017;549(7673):543-547. DOI: 10.1038/ nature23891.
- Macaulay E.C., Chatterjee A., Cheng X., Baguley B.C., Eccles M.R., Morison I.M. The genes of life and death: A potential role for placen-tal-specific genes in cancer: Active retrotransposons in the placenta encode unique functional genes that may also be used by cancer cells to promote malignancy. Bioessays. 2017;39(11). DOI: 10.1002/ bies.201700091.
- Kshitiz, Afzal J., Maziarz J.D., Hamidzadeh A., Liang C., Erkenbrack E.M. et al. Evolution of placental invasion and cancer metastasis are causally linked. Nat. Ecol. Evol. 2019;3(12):1743-1753. DOI: 10.1038/ s41559-019-1046-4.
- Donabela F.C., Meola J., Padovan C.C., de Paz C.C., Navarro P.A. Higher SOD1 gene expression in cumulus cells from infertile women with moderate and severe endometriosis. Reprod. Sci. 2015;22(11):1452-1460. DOI: 10.1177/1933719115585146.
- Roland L., Beauchemin D., Acteau G., Fradette C., St-Pierre I., Bilodeau J.F. Effects of labor on placental expression of superoxide dismutases in preeclampsia. Placenta. 2010;31(5):392-400. DOI: 10.1016/j.placen-ta.2010.02.007.
- Lin P., Lai X., Wu L., Liu W., Lin S., Ye J. Network analysis reveals important genes in human placenta. J. Obstet. Gynaecol. Res. 2021;47(8):2607-2615. DOI: 10.1111/jog.14820.
- Gomez M.L., Shah N., Kenny T.C., Jenkins E.C. Jr., Germain D. SOD1 is essential for oncogene-driven mammary tumor formation but dispensable for normal development and proliferation. Oncogene. 2019;38(29):5751-5765. DOI: 10.1038/s41388-019-0839-x.
- Wang X., Zhang H., Sapio R., Yang J., Wong J., Zhang X. et al. SOD1 regulates ribosome biogenesis in KRAS mutant non-small cell lung cancer. Nat. Commun. 2021;12(1):2259. DOI: 10.1038/s41467-021-22480-x.
- Schierding W., Antony J., Karhunen V., Vääräsmäki M., Franks S., Elliott P. et al. GWAS on prolonged gestation (post-term birth): Analysis of successive Finnish birth cohorts. J. Med. Genet. 2018;55(1):55-63. DOI: 10.1136/jmedgenet-2017-104880.
- Xu Z.P., Wawrousek E.F., Piatigorsky J. Transketolase haploinsufficien-cy reduces adipose tissue and female fertility in mice. Mol. Cell Biol. 2002;22(17):6142-6147. DOI: 10.1128/MCB.22.17.6142-6147.2002.
- Xu I.M., Lai R.K., Lin S.H., Tse A.P., Chiu D.K., Koh H.Y. et al. Tran-sketolase counteracts oxidative stress to drive cancer development. Proc. Natl. Acad. Sci. USA. 2016;113(6):E725-734. DOI: 10.1073/ pnas.1508779113.
- Qin Z., Xiang C., Zhong F., Liu Y., Dong Q., Li K. et al. Transketolase (TKT) activity and nuclear localization promote hepatocellular carcinoma in a metabolic and a non-metabolic manner. J. Exp. Clin. Cancer Res. 2019;38(1):154. DOI: 10.1186/s13046-019-1131-1.


