Анализ микробных сообществ термальных источников района озера Фумарольное Кальдеры вулкана Узон, Камчатка
Автор: Дворянчикова Е.Н., Кизилова А.К., Кравченко И.К., Гальченко В.Ф.
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Водные ресурсы
Статья в выпуске: 1-6 т.13, 2011 года.
Бесплатный доступ
Впервые проведено комплексное молекулярно-биологическое исследование микробных сообществ 5 термальных источников, расположенных в районе озера Фумарольное кальдеры вулкана Узон, Камчатка. Были использованы методы, основанные на применении ПЦР технологий (ПЦР-детекция, ПЦР-ДГГЭ, клонирование) и гибридизации in situ для анализа как рибосомальных, так и функциональных генов, отвечающих за синтез ключевых ферментов метанотрофии, метаногенеза, автотрофной фиксации СО2, нитрификации. Установлено, что общая численность микроорганизмов в образцах ила составляла от 0,5 до 36,8×106 клеток/мл и метаболически активные эубактерии составляли 22-35%. Методом ПЦР представители Bacteria и также фототрофные прокариоты были обнаружены во всех исследованных источниках, Archаea в четырех, а метанотрофы в трех. Ни в одном из источников не были обнаружены метаногенные и нитрифицирующие археи. Впервые в гидротермах изучено разнообразие метанотрофных сообществ и обнаружено низкое разнообразие, представленное только метанотрофами I типа, наиболее близкими к Methylotermus и Methylobacter. Получено 5 накопительных высокообогащенных монокультур экстремально термофильных метанотрофов, в состав которых входят организмы, значительно отличающиеся от двух известных видов рода Methylotermus.
Микробные сообщества, метанотрофы, накопительные культуры, гибридизация
Короткий адрес: https://sciup.org/148199878
IDR: 148199878 | УДК: 579.64.631.46
Текст научной статьи Анализ микробных сообществ термальных источников района озера Фумарольное Кальдеры вулкана Узон, Камчатка
типами анаэробного дыхания (серо-, нитрат- и железоредукторам, использующим СО), тогда как другие группы микроорганизмов, например, использующие в своем метаболизме метан и/или другие С 1 соединения, исследованы крайне недостаточно. Основные микробиологические исследования выполнены на объектах, расположенных на Восточном термальном поле, а изучение микробных сообществ в термальных источниках района озера Фумарольное практически не проводились. В то же время образование горячих озер на месте кратероподобных воронок является уникальной особенностью Узонских термопроявлений. Крупнейшим среди них является озеро Фумарольное, которое заполняет обширную котловину размером 300 х 600 м с хорошо выраженным береговым обрывом.
Цель исследования: изучение микробного разнообразия в термальных источниках района озера Фумарольное кальдеры вулкана Узон (Камчатка) с помощью методов молекулярной экологии. Особое внимание было уделено мета-нокисляющим организмам, сведения о которых практически отсутствуют.
Материалы и методы.
Отбор образцов и определение физикохимических параметров среды. Образцы цианобактериальных матов и ила были отобраны в ходе экспедиционных исследований в июле 2010 г. Определение физико-химических параметров гидротерм (температуры, pH, Eh) производили с помощью наборов Aquamerk («Merck», Германия). Результаты представлены в табл. 1. Непосредственно на месте отбора 2 см3 образца вносили во флаконы объемом 120 мл с 20 мл среды «П» [3], разбавленной в 5 раз, и инкубировали в атмосфере метан: воздух (1:1) в течение 7 суток в источнике Заварзина (56,7оС). Полученные обогащенные образцы (первичная накопительная культура) были использованы в лаборатории для проведения исследований методами ПЦР и FISH, а также для выделения накопительных культур метано-трофов.
Выделение ДНК и ПЦР-детекция прокариот различных таксономических и функциональных групп. Препарат тотальной ДНК выделяли с помощью коммерческого набора реактивов (Wizard Genomic DNA Purification Kit, «Prome-ga», США) в соответствии с протоколом производителя с минимальными модификациями. Для увеличения выхода микробной ДНК мы проводили обработку лизирующим буфером при повышенной температуре (80оС) и встряхивании. Для амплификации фрагментов рибосомального гена 16S рРНК бактерий использовали систему праймеров 341 F-907 R [4], для архей – 344F-915R [5]. Для детекции прокариот различных функциональных групп (метанотрофы, фотосинтетики, метаногены, нитрификаторы) использовали ПЦР-амплификацию фрагментов генов ключевых ферментов различных процессов метаболизма. Для метанотрофов применяли систему вырожденных праймеров А189F и A682R [6], амплифицирующих фрагмент гена pmoA , кодирующего синтез β-субъединицы метанмонооксигеназы (ММО), ключевого фермента метанокис-ления, а для обнаружения прокариот, обладающих генами автотрофной фиксации углекислоты в цикле Кальвина ( cbbL ) применяли праймерную систему cbbl1F- cbbLR [7].
Присутствие метаногенных архей детектировали с помощью праймерной системы mcrAF /mcrAR [8], позволяющей амплифицировать фрагмент гена mcrA, кодирующего α-субъеди-ницу фермента метил-коэнзим М, а нитрифицирующих архей – праймерной системой Crena-moA23f и CrenamoA616 [9] для детекции фрагмента amoA архей. Оценку разнообразия метано-трофов проводили методом денатурирующего градиентного гель-электрофореза (ДГГЭ) с использованием праймера A189F(GC) с GC-клампом для обеспечения разделения смеси ампликонов pmoA в полиакриламидном геле [10]. Состав реакционной смеси для ПЦР и температурно-временной профиль реакции соответствовали таковым в упомянутых выше работах. Амплификацию проводили на приборе MyCycler («BioRad», США). Анализ продуктов ПЦР проводили в 1,2% агарозном геле, окрашенном бромистым этидием. Выделение и очистку ПЦР фрагментов осуществляли с помощью набора Wizard PCR Preps («Promega», США), согласно рекомендациям производителя.
Оценка бактериального разнообразия методом клонирования фрагмента гена 16S рРНК. Очищенные ПЦР-фрагменты клонировали с помощь pGEM-T easy vector system I (Promega, США) в компетентных клетках E.coli DH10B. Для создания каждой библиотеки клонов случайным образом было отобрано по 50 колоний, проявивших положительную реакцию (белые). С материалом колоний была поставлена ПЦР реакция с универсальными плазмидными праймерами M13F и M13R. Наличие вставки оценивали методом агарозного электрофореза. Генетический материал клонов, содержащих вставки гена 16S рРНК, был секвенирован в сервисной лаборатории с помощью набора реактивов Big Dye Terminator v.3.1 на автоматическом секвенаторе ABI 3730 (Applied Biosystems Inc., США).
Денатурирующий градиентный гель-электрофорез. Разделение смеси ампликонов pmoA проводили с помощью прибора DCode Universal Mutation Detection System («BioRad»,США) при постоянной температуре 60оС, напряжении 200 V и градиенте денатурантов (формамид, мочевина) 35-60% в течение 6 ч. Полученные гели окрашивали раствором этиди-ум бромида и документировали с помощью имидж-системы Gel Doc System («BioRad», США). Характерные видимые полосы (bands), содержащие ДНК, были вырезаны, полученные после элюирования растворы ДНК были очищены и использованы для определения нуклеотидных последовательностей методом секвенирования.
Гибридизация in situ с флуоресцентномечеными олигонуклеотидными зондами (FISH). Фиксацию образцов раствором параформальдегида и гибридизацию препаратов с флуоресцентно-мечеными олигонуклеотидными зондами проводили при температуре 46оС в соответствии с методикой [11]. Для специфической детекции метанотрофов II типа использовали зонд М-450 (30% формамида в гибридизационном буфере), а метанотрофов I типа – смесь зондов М-84 и М705 (20% формамида) [12].Для детекции представителей домена Bacteria использовали смесь универсальных зондов EUB 338mix [13]. Синтез зондов, меченных флуоресцентным красителем Су3, выполнен компанией «Синтол» (Москва, Россия). Общую численность бактерий определяли в препаратах, окрашенных раствором ДНК-специфичного красителя ДАФИ. Количество гибридизованных с зондами клеток подсчитывали с помощью микроскопа AxioImager D1 («Karl Zeiss», Германия) в 50 полях зрения с использованием светофильтров Zeiss 20 для Cy3-меченных зондов и Zeiss 49 для подсчета клеток, окрашенных ДАФИ, с последующим расчетом на 1 мл обогащенного образца.
Выделение и анализ накопительных культур. Накопительные культуры из обогащенных образцов получали путем регулярных пересевов один-два раза в месяц. Инкубацию вели в статических условиях при температуре 60оС. Процесс роста метанотрофных бактерий при последовательных пересевах контролировали с помощью световой микроскопии, FISH и ПЦР детекции метанотрофов. Представленные результаты анализа были получены для накопительных культур, полученных через 3 месяца инкубации (3-4 пассажа).
Анализ нуклеотидных последовательностей. Анализ полученных нуклеотидных последовательностей фрагментов генов проводили с помощью программного пакета BLAST [].
Результаты и обсуждение. Характеристики гидротерм. В июле 2010 г. был проведен отбор образцов ила и цианобактериальных матов из жерла термальных источников Культурный, Терра, Строма, Квадрат и Глаз дракона, расположенных в районе озера Фумарольное кальдеры вулкана Узон. Физико-химические характеристики вод изученных гидротерм представлены в табл. 1. Все исследованные источники были высокотемпературными со слабокислой или близкой к нейтральной реакцией среды.
Таблица 1. Характеристики термальных источников
|
Название источника |
Координаты |
Температура, оС |
pН |
Eh, мВ |
|
|
N |
E |
||||
|
Культурный |
54030. 115` |
1590 59. 279` |
63,0 |
5,2 |
80 |
|
Терра |
54029. 887` |
1590 59. 410` |
65,0 |
6,2 |
73 |
|
Строма |
54029. 854` |
1590 59. 477` |
56,1 |
5,3 |
5 |
|
Квадрат |
54029. 862` |
1590 59. 485` |
64,5 |
6,3 |
- 22 |
|
Глаз Дракона |
54029. 885` |
1590 59. 468` |
59,8 |
6,2 |
н.о. |
Примечание: н.о. – не определяли.
ПЦР-детекция прокариот. На первом этапе исследования была проведена амплификация фрагментов таксономических и функциональных генов прокариот. Примеры детекции целевых организмов продемонстрированы на рис. 1.
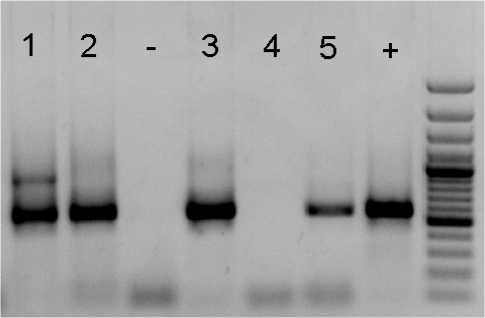
а)

б)
Рис. 1. Визуализация результатов ПЦР с помощью агарозного электрофореза: а – археи, система праймеров 344F-915R; б – метанотро-фы, система праймеров А189F и A682R; 1 – Культурный; 2 – Квадрат; 3 – Терра; 4 –Глаз Дракона; 5 –Строма; «–» отрицательный контроль; «+» положительный контроль, организмы Methanosarcina lacustris (а ), Methylosinus trichosporium (б).
Результаты анализа показали присутствие Bacteria во всех исследованных источниках и Arch-аea в четырех (рис. 2а). Анализ функциональных генов также продемонстрировал различия в составе микробных сообществ. Метанотрофные бактерии были детектированы только в 3 источниках (рис. 1б), а фототрофные – во всех 5 источниках.
Из термальных источников Камчатки с кислой реакцией среды (pH 3,5) были выделены ацидо-термофильные метанокисляющие представители филума Verrucomicrobia [14, 15], в то время как сведения о метанотрофах в высокотемпературных нейтральных источниках Камчатки в литературе отсутствуют. Таким образом, в нашем исследовании впервые было зафиксировано их присутствие в горячих источниках Камчатки.
Распространение фототрофных цианобактерий и анаэробных фотосинтезирующих бактерий в термальных источниках изучено для достаточно большого количества объектов, расположенных в России, Исландии, США и Китае [16], поэтому неудивительным был факт обнаружения организмов, обладающих генами первичной ассимиляции СО 2 во всех исследованных источниках. Для осадков источника Культурный фрагмент гена cbbL был секвенирован, и полученная нуклеотидная последовательность продемонстрировала сходство с депонированной в GenBank последовательностью фототрофной бактерии Chloroflexus . Ни в одном из исследованных источников нам не удалось детектировать присутствие аммонийокисляющих архей, хотя в работе (Zhang et al., 2008) было обнаружено присутствие нитрифицирующих кренархей в высокотемпературных источниках Камчатки [17]. Ни в одном из источников не были обнаружены метаногенные археи. Результаты ПЦР-детекции прокариот в источниках суммированы в табл. 2.
Анализ бактерий в обогащенных образцах методом FISH. Общее количество микроорганизмов, выявленных в обогащенных образцах (окраска ДАФИ), составляло от 5 до 36 х 106 клеток на 1 мл осадка, а количество метаболически активных бактерий (EUB338 mix) варьировало от 2,3 до 21 х 106 клеток на 1 мл осадка (табл. 3). Доля активных бактерий была максимальной (35,6%) в образцах источника Квадрат, а в остальных варьировала от 22,3% до 31,5%.
Таблица 2. Результаты амплификации рибосомальных и функциональных генов бактерий и архей в образцах осадков термальных источников
|
Образование целевого продукта |
|||||||
|
Группа прокариот |
Целевой ген |
Праймерные системы |
Термальные источники |
||||
|
1 |
2 |
3 |
4 |
5 |
|||
|
все Bacteria |
16S рРНК |
341 F -907 R |
+ |
+ |
+ |
+ |
+ |
|
метанотрофные бактерии |
pmoA |
А189F- A682R |
+ |
- |
+ |
+ |
- |
|
фототрофные бактерии |
cbbL |
cbbl1F- cbbLR |
+ |
+ |
+ |
+ |
+ |
|
все Archea |
16S рРНК |
344F-915R |
+ |
+ |
+ |
- |
+ |
|
метаногенные археи |
mcrA |
mcrAF /mcrAR |
- |
- |
- |
- |
- |
|
нитрифицирующие кренархеи |
amoA |
CrenamoA23f -CrenamoA616 |
- |
- |
- |
- |
- |
Примечание: источники: 1 – Культурный, 2 – Терра, 3 – Строма, 4 – Квадрат, 5 – Глаз Дракона; «+» -означает детекцию целевого организма, «-» - не обнаружено.
Анализ обогащенных образцов методом FISH выявил высокие значения численности ме-танотрофов I типа (γ- Proteobacteria ) в источниках Культурный, Строма и Квадрат. Количество клеток здесь достигало 1,5-2,0 х 106 клеток на 1 мл осадка. В источниках Терра и Глаз Дракона количество метанотрофов было значительно ниже
– 105 и 104 клеток на 1 г осадка, соответственно. Доля метанотрофов I типа от метаболически активных эубактерий составляла: Культурный – 4,3%; Терра – 3,2%; Строма – 4,2%; Квадрат – 3,4%; Глаз Дракона – 0,2%. Метанотрофы II группы (зонд М-450) ни в одном из исследованных источников обнаружены не были.
Таблица 3. Численность клеток в обогащенных образцах, определенная методом гибридизации in situ с флуоресцентными олигонуклеотидными зондами и окраской ДАФИ.
|
Источник |
Культурный |
Терра |
Строма |
Квадрат |
Глаз Дракона |
|
окраска / гибридизация |
количество клеток, х106 /мл осадка |
||||
|
ДАФИ |
24±2,2 (68.2%) |
11±3,9 (20,1%) |
33±5,6 (73,5%) |
36±4,5 (61%) |
5±0,68 (68,4%) |
|
EUB338 mix |
9,7±0,39 (27,6%) |
4,2±1,4 (26,8%) |
10±1,6 (22,3%) |
21±3,3(35,6%) |
2,3±0,81 (31,5%) |
|
М-84 + М-705 |
1,5±0,46 (4,3%) |
0,5±0,19 (3,2%) |
1,9±0,61 (4,2%) |
2±0,49 (3,4%) |
0,013±0,002(0,2%) |
Оценка разнообразия бактерий в образце ила источника Культурный методом клонирования 16S рРНК. Клонирование ПЦР-фрагментов позволило оценить разнообразие бактерий в источнике Культурный. Из анализа результатов клонирования (табл. 4) следует, что доминирующим организмом (29 клонов) в источнике является метанотроф, наиболее близкий к штамму НВ [16]. Минорные компоненты (1-5 клонов) представлены термофильными организмами, проявившими высокую степень сходства с бактериями, выделенными из термальных источников Камчатки, Йеллоустонского национального парка, Тибета и геотермальных почв Новой Зеландии.
ПЦР-ДГГЭ анализ фрагмента гена pmoA. Для 4 источников, в которых был амплифициро-ван фрагмент гена pmoA, был проведен ПЦР-ДГГЭ анализ, позволяющий оценить разнообразие метанокисляющих бактерий. В табл. 5 представлены данные по анализу нуклеотидных последовательностей характерных полос. Проведенное исследование показало, что во всех источниках присутствуют метанотрофы, наиболее близкие к представителям рода Methylothermus, но отличающиеся от двух описанных видов (79-91% сходства нуклеотидных последовательностей). В образцах из источников Терра и Глаз Дракона были обнаружены последовательности, наиболее близкие к некультивируемым представителям рода Methylomonas (79-90% сходства). Только в образце из источника Терра обнаружены 2 мета-нотрофных организма, в то время как во всех остальных образцах – по одному. В источниках Культурный, Терра, Квадрат идентифицированы метанотрофы, проявившие высокую степень сходства с термофильным метанотрофом НВ [18]. Для метанотрофа из образца Строма ближайшим родственником был Methylothermus sub-terraneus из горячих подземных водоносных слоев золотодобывающих рудников Японии [19].
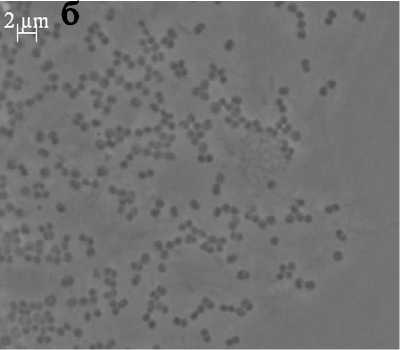
Характеристика накопительных культур термофильных метанотрофов. Из образцов ила источника Культурный, а также источников Глаз дракона и Строма путем последовательных пересевов на селективных средах были выделены 5 накопительных культур метанотрофов, присутствие которых в обогащенных образцах нам не удалось детектировать. Накопительные культуры представляли собой ассоциации метанокисляю-щих бактерий с гетеротрофными спутниками, однако доля метанокисляющих бактерий в них была высокой. Морфология метанотрофных клеток, дающих сигнал с зондом М-84+М-705 (I группа метанотрофов), была сходной для всех накопительных культур. Это были короткие, крупные (около 1 мкм) округлые палочки, которые хорошо идентифицировались методом фазового контрастирования препаратов живых клеток. Электронно-микроскопический анализ интактных культур метанотрофов культуры К12 выявил присутствие полярного жгутика, а анализ срезов обнаружил мембранные структуры, характерные для метанотрофов I типа. Анализ последовательностей pmoA показал, что во всех накопительных культурах присутствуют метанотрофы, наиболее близкие к Methylotermus.
Таблица 4. Разнообразие бактерий в образце ила источника Культурный методом клонирования 16S рРНК
|
Номер образца |
Ближайший представитель |
Процент сходства покрытие/сходство |
|
1 (Культурный, 29 клонов) |
Thermophilic methanotroph HB (U89302) Uncultured bacterium clone SL_5.95 (AY550713) |
98%(99%) 98%(94%) |
|
2 (Камчатка 14, 15 клонов) |
Uncultured Chlorobi bacterium clone VSM5Q1u76 16S ribosomal RNA gene, partial sequence (EU631213) Uncultured green sulfur bacterium clone SM1H02 16S ribosomal RNA gene, partial sequence (AF445702)---- |
95% 94% |
|
3 (Культурный, 5 клонов) |
Thermoactinomyces sacchari (AM161154) Uncultured bacterium clone LaC15L24(EF667582) Propionibacterium acnes (AM161153) |
98% (83%) 73%(89%) 9%(83%) |
|
4 (Культурный, 1 клон) |
Uncultured bacterium clone ZB_P10_C06 (GQ328567) из источника Заварзина в Узоне Uncultured Acidobacteriaceae bacterium (AM935486) |
83% (100%) 83% (95%) |
|
5 (Культурный, 1 клон) |
Pseudomonas putida (AM161157) |
98% (95%) |
|
6 (Культурный, 2 клона) |
Uncultured bacterium clone ZB_P14_B04 16S (GQ328675 из источника Заварзина в Узоне Candidate division OP10 clone OPB80 (AF027089) из горячего источника Йеллоустонского парка |
73%(97%) 73%(96%) |
|
7 (Культурный, 1 клон) |
Uncultured bacterium clone ZB_P13_F12 (GQ328655) из источника Заварзина в Узоне |
73%(85%) |
|
8 (Культурный, 1 клон) |
Uncultured bacterium clone TP146 (EF205576) hot springs in central Tibet Uncultured Acidobacteria (AM749745) from geothermal soils in New Zealand |
78%(97%) 78%(94%) |
Таблица 5. Результаты BLAST анализа нуклеотидных последовательностей характерных ДГГЕ полос, полученных при разделении ПЦР фрагментов гена.
|
Источник |
Ближайший родственник |
Покрытие/сходство |
|
Культурный |
Uncultured bacterium clone SL_5.95 (AY550713) Thermophilic methanotroph HB (U89302) |
99/80 99/79 |
|
Терра (смесь) |
Uncultured bacterium clone SL_5.32 (AY550731) Thermophilic methanotroph HB (U89302) |
100/95 100/95 |
|
Uncultured bacterium (AM910134) Uncultured Methylomonas sp . (AB500822) |
100/80 100/79 |
|
|
Строма |
Methylothermus subterraneus (AB536748) |
100/91 |
|
Квадрат |
Uncultured bacterium clone SL_5.95 (AY550713) Thermophilic methanotroph HB (U89302) |
93/88 93/88 |
|
Глаз Дракона |
Uncultured bacterium (AM910134) Uncultured Methylomonas sp . (AB500822) |
95/90 95/87 |
Выводы: в результате проведенных исследований впервые получены характеристики микробных сообществ гидротерм Камчатки с помощью комплексного молекулярно-биологического подхода, основанного на использовании ПЦР технологий (ПЦР-детекция, ПЦР-ДГГЭ, клонирование) и гибридизации in situ для анализа рибосомальных генов, а также функциональных генов, отвечающих за синтез ключевых ферментов метанотрофии, метаногенеза, автотрофной фиксации СО2, нитрификации). Впервые изучено распространение и разнообразие метанотрофных сообществ в осадках нейтральных высокотемпературных источников. Установлено, что разнообразие термофильных метанотрофов в исследованных источниках ограничивается бактериями, наиболее близкими к Methylotermus и Methylobacter. Получено 5 накопительных высокообогащенных (20-50%) монокультур метанотрофов, в состав которых входят организмы, значительно отличающиеся от двух известных видов рода Methy-lotermus. Культуры являются экстремальными термофилами и их рост осуществляется при значениях температур не ниже 50оС. Дальнейшие исследования предполагают более детальные исследования обнаруженных организмов, выделение чистых культур и описание новых таксонов термофильных метанотрофов.
Работа выполнена при поддержке грантов РФФИ № 08-04-00164-а, 09-04-10113-к и 10-04-10127-к.
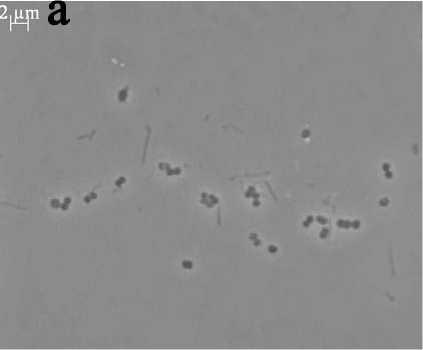
Рис. 2. Морфология клеток накопительных культур метанотрофов: а – К14, источник Культурный; б – К12, источник Строма (фазовый контраст)
Список литературы Анализ микробных сообществ термальных источников района озера Фумарольное Кальдеры вулкана Узон, Камчатка
- Brock, T.D. Microbiological studies of thermal habitats of the central volcanic region, North Island, New Zealand./T.D. Brock, M.L. Brock//N. Z. J. Marine and Freshwater Res. 1971. V.5. P. 233-258.
- Труды института микробиологии имени С.Н. Виноградского: Вып.16: Термофильные микроорганизмы//Ин-т микробиологии им. С.Н. Виноградского РАН. -М.: МАКС Пресс, 2011. 364 с.
- Гальченко, В.Ф. Метанотрофные бактерии. -М.: ГЕОС, 2001. 500 с.
- Muyzer, G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA/G. Muyzer, E.C. de Waal, A.G. Uitterlinde//Appl. Environ. Microbiol. 1993. V. 59. P. 695-700.
- Casamayor, E.O. Identification of and spatiotemporal differences between microbial assemblages from two-neighboring sulfurous lakes: comparison by microscopy and denaturing gradient gel electrophoresis/E.O. Casamayor, H. Schafer, L. Baneras//Appl. Environ. Microbiol. 2000. 66. С. 499-508.
- Holmes, A.J. Evidence that particulate methane monooxigenase and ammonia monooxigenase may be evolutionarily related/A.J. Holmes, A. Costello, M.E. Lidstrom, G.C. Murrell/FEMS Microbiol. Lett. 1995. V. 132. P. 203-208.
- Спиридонова, Е.М. Система олигонуклеотидных праймеров для амплификации генов рибулозо-1,5-бисфосфаткарбоксилазы/оксигеназы у бактерий различных таксономических групп/Е.М. Спиридонова, И.А. Берг, Т.В. Колганова и др.// Микробиология. 2004. № 2. С. 377-380.
- Luton, P.E. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill/P.E. Luton, J.M. Wayne, R.J. Sharp, P.W. Riley//Microbiology. 2002. V. 148. P. 3521-3530.
- Tourna, M. Growth, Activity and Temperature Responses of Ammonia-Oxidising Archaea and Bacteria in Soil Microcosms/M. Tourna, T.E. Freitag, G.W. Nicol, J.I. Prosser//Environ. Microbiol. 2008. Vol. 10. P. 1357-1364.
- McDonald, I.R. Molecular ecology techniques for the study of aerobic methanotrophs/I.R. McDonald, L. Bodrossy, Y. Chen, C.J. Murrell//Appl. Environ. Microbiol. 2008. V. 74. P. 1305-1315.
- Stahl, D.A. Nucleic Acid Techniques in Bacterial Systematics/D.A. Stahl, R. Amann//Development and Application of Nucleic Acid Probes. Eds. E. Stakebrandt and M. Goodfellow Chichester: Wiley,1991. P. 205-248.
- Eller, G. Group-specific 16S rRNA targeted probes for the detection of type I and type II methanotrophs by fluorescence in situ hybridization/G. Eller, S. Stubner, P. Frenzel//FEMS Microbiol. Lett. 2001. V. 198. P. 31-37.
- Diams, H. The domain-specific probe EUB338 is insufficient for the detection of all Bacteria: Development of a more comprehensive probe set/H. Diams, A. Bruhl, R. Amann et al./Syst. Appl. Microbiol. 1999. V. 22. P. 434-444.
- Pol, A. Op den Camp. Methanotrophy below pH 1 by a new Verrucomicrobia species/A. Pol, K. Heijmans, H.R. Harhangi//Nature. 2007. V. 450. P. 874-878.
- Bodrossy, L. A novel methane-oxidising -Proteobacterium/L. Bodrossy, K. Kovács, I.R. McDonald, K. Murrell//FEMS Microbiol. Lett. 1999. V. 170. P. 335-341.
- Madigan, M.T. Anoxygenic phototrophic bacteria from extreme environments//In: Discoveries in Photosynthesis. Advances in Photosynthesis and Respiration, 2005. Vol. 20, XII., 969-983, DOI: DOI: 10.1007/1-4020-3324
- Chuanlun, L. Zhang. Global Occurrence of Archaeal amoA Genes in Terrestrial Hot Springs/Chuanlun L. Zhang, Qi Ye, Zhiyong Huang et al.//Appl. Environ. Microbiol. 2008. V.74. P. 6417-6426.
- Bodrossy, L. A novel methane-oxidising -Proteobacterium/L. Bodrossy, K. Kovács, I.R. McDonald, K. Murrell//FEMS Microbiol. Lett. 1999. V. 170. P. 335-341.
- Hirayama, H. Methylothermus subterraneus sp.nov., a moderately thermophilic methanotrophic bacterium from a terrestrial subsurface hot aquifer in Japan/H. Hirayama, Y. Suzuki, M. Abe et al.//Int. J. Syst. Evol. Microbiol. 2010. DOI 10.1099/ijs.0.028092-0.


