Молекулярный анализ микологического поражения древесины
Автор: Андронов Е.Е., Чижевская Е.П., Пинаев А.Г., Павлов С.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Методика
Статья в выпуске: 5 т.48, 2013 года.
Бесплатный доступ
Оценка микологического поражения образцов древесины традиционно проводится с помощью культивирования, продолжительность которого может составлять до нескольких месяцев. Нами продемонстрирована возможность применения методов молекулярной биологии для таксономического и количественного анализа при грибном поражении древесины. С использованием секвенирования библиотеки диагностического фрагмента межгенного участка ITS рибосомального оперона и ПЦР с детекцией в реальном времени обнаружено, что 75 % популяции, населяющей проанализированный образец древесины (фрагмент эксплуатируемой деревянной конструкции), относится к виду Aureobasidium pullulans. Показано, что примененный подход имеет большие преимущества перед традиционными методами микологической экспертизы, так как он универсален, доступен для большинства современных отечественных лабораторий и не зависит от культивирования. Метод характеризуется высокой скоростью выполнения анализов (3-4 сут) и довольно низкими затратами (1-2 тыс. руб. на образец). Основное достижение разработанного протокола заключается в возможности выявлять разнообразие в микробных сообществах, которое до последнего времени, по всей видимости, недооценивалось. Без существенных модификаций предложенный метод может быть перенесен в рутинную сельскохозяйственную практику для детекции грибных патогенов.
Грибное поражение, древесина, пцр с детекцией в реальном времени
Короткий адрес: https://sciup.org/142133438
IDR: 142133438 | УДК: 632.4:577.29:632.08
Текст научной статьи Молекулярный анализ микологического поражения древесины
Проблема микологического поражения образцов древесины традиционно решается с использованием методов культивирования, продолжительность которого может составлять до нескольких месяцев. То же характерно для микологического тестирования других объектов. Однако по следнее десятилетие ознаменовано широким внедрением в практику микологического анализа методов молекулярной экологии микроорганизмов, основывающихся на исследовании общей ДНК, выделенной из различных объектов (1, 2).
Существующие молекулярные подходы по большей части основаны на детекции и анализе таксономически значимых участков генома грибов. Прежде всего это фрагменты рибосомального оперона: гены субъединиц рибосом и межгенные участки ITS и IGS (1, 3, 4).
Рис. 1. Образец пораженной древесины, представленный для анализа.
Нашей целью была разработка протокола для таксономического и количественного анализа микологического поражения образца на основе двух молекулярных подходов — анализа библиотеки клонов суммарного амплификата ITS-участка рибосомального оперона и ПЦР с детекцией в реальном времени.
Описание методики. Для исследования был представлен образец деревянной конструкции (сельский
16.552.11.7047).
дом), на котором в течение первого месяца эксплуатации было выявлено интенсивное окрашенное микологическое поражение (рис. 1).
Для выделения ДНК 100-250 мг образца (в виде опилок) разрушали перетиранием в фарфоровой ступке с добавлением абразивов. Первый этап экстракции проводили при температуре 65 ° C в СТАВ-содержащем буфере (СТАВ — 2 %; NaCl — 1,4 M; EDTA — 20 мМ), с добавлением стеклянных шариков (0,3 мм) и периодическом интенсивном встряхивании на вортексе (3 раза по 5 мин). Смесь дважды экстрагировали хлороформом, после чего суммарную ДНК осаждали изопропанолом. Окончательную очистку ДНК проводили посредством селективной сорбции на диоксиде кремния в соответствии с типовой методикой (5). Качество ДНК проверяли визуально после электрофоретического разделения по степени деградации основного массива (20-30 т.п.н.).
ПЦР выполняли со стандартными компонентами. Для амплификации участка рибосомального оперона, содержащего районы ITS1, ITS2 и ген 5,8-рРНК, применяли праймеры ITS1 и ITS4 (3). В качестве термостабильной ДНК-полимеразы использовали Taq-полимеразу («Хеликон», г. Москва). Температура отжига — 52 ° C. Матрицей служила ДНК, выделенная из исследуемого образца, в количестве 50 нг на реакцию в общем объеме 30-50 мкл. Для реамплификации клонированной в плазмиде вставки использовали стандартные праймеры M13F (-46, 22-mer, «Fermentas», Литва) и M13R (-46, 24-mer, «Fermentas», Литва), температура отжига — 55 ° C. Вместо матричной ДНК в реакционную смесь добавляли небольшое количество интактных клеток Escherichia coli , содержащих плазмиду с клонированной вставкой. При ПЦР с детекцией в реальном времени анализ выполняли на амплификаторе ICycler («Bio-Rad», США). Для определения общего количества грибной ДНК в пробе использовали праймеры 5,8S и ITS1f, условия ПЦР соответствовали опубликованным рекомендациям (6). При амплификации применяли стандартный буфер для ПЦР с добавлением SYBR green («Amresco LLC», США) до конечной концентрации 0,3* и изотиоцианата флюоресцеина («Sigma-Aldrich», США) до конечной концентрации 10 нмоль. Калибровочную кривую строили на основе серии разведений ДНК, выделенной из штамма дрожжей Saccharomyces cerevisiae 1B-D1606 (7).
Для клонирования амплифицированных фрагментов ДНК после электрофоретического разделения продуктов ПЦР полосу, содержащую необходимый фрагмент ДНК, вырезали из агарозного геля. ДНК выделяли в соответствии с обычной методикой (5) с той лишь разницей, что в первом растворе гель выдерживали при 65 ° С. Очищенный амплифициро-ванный фрагмент клонировали в Т-векторе pAL («Евроген», г. Москва). Лигирование и трансформацию проводили по стандартным методикам. Для рестрикционного анализа клонированные фрагменты реамплифи-цировали, как описано выше. К аликвоте ПЦР-буфера, содержащей 300500 нг неочищенного продукта, добавляли эквивалентный объем 1* буфера Tango («Fermentas», Литва) и 2-3 ед. эндонуклеазы MspI («Fermentas», Литва). Продукты рестрикции разделяли электрофоретически в 3 % агарозном геле. Рестрикционный анализ проводился для 40 клонов каждой из исследованных проб.
После анализа выявили группу клонов с одинаковыми рестрикционными профилями (рис. 2).
На заключительном этапе выполняли секвенирование и определение таксономического положения клонированных последовательностей ITS-участка. Три клона, представляющие основной рестрикционный тип,
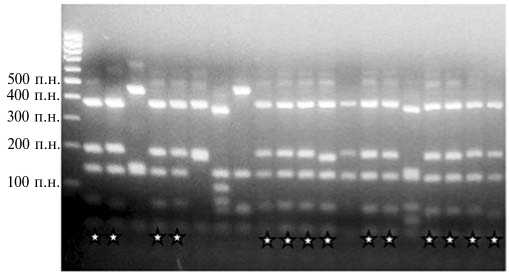
Рис. 2. Рестрикционный анализ библиотеки клонов ITS-участка грибной ДНК, выделенной из образца эксплуатируемой деревянной конструкции, представленного для исследования. На первой дорожке слева — маркер молекулярной массы («Си-бэнзим», Россия); звездочками отмечены доминантные рестрикционные типы, характеризующиеся одинаковым набором фрагментов ДНК.
подвергли циклическому секвенированию по Сэнгеру с помощью капиллярного автоматического секвенатора Beckman Coulter CEQ8000 и набора DTCS Quick Start kit («Beckman Coulter, Inc.», США) в соответствии с рекомендациями изготовителя. Секвенирование проводили с использованием реампли-фицированных фрагментов, выделенных из геля, и стандартных праймеров для секвенирования M13. Таксономическую принадлежность клонов определяли с применением программы BLASTn (8), доступной на сервере NCBI (National Center for Biotechnology Information, США).
При количественной оценке содержания общей грибной ДНК методом ПЦР с детекцией в реальном времени и пересчете на число дрожжевых клеток на 1 г субстрата в образцах было выявлено более 108 грибных клеток на 1 г субстрата. Анализ видового состава грибов в одном из образцов пораженной древесины с использованием амплификации общей последовательности ITS, клонирования и секвенирования показал, что более 75 % всей популяции составляет вид Aureobasidium pullulans. Это очень распространенный, поражающий поверхности деревянных строений аскомицет, вызывающий такой же тип поражений, как на обследованном объекте . Отдельного внимания заслуживает тот факт, что, согласно опубликованным на указанном интернет-ресурсе данным, этот гриб способен расти даже на виниловом сайдинге, используя питательные вещества, накапливающиеся на поверхностях. Таким образом, в работе была полностью решена поставленная задача — выявлен основной агент микологического поражения и оценено его количество.
Следует отметить, что описанный метод, доступный фактически любой молекулярно-биологической лаборатории, не требует много времени для идентификации грибных агентов. Так, суммарные затраты составили около 1,5 тыс. руб. и 2 человеко-дня (всего анализ продолжался 4 рабочих дня). По сравнению с методиками, основанными на культивировании, описанный прием, несомненно, представляет собой исключительно быстрый, точный, а главное, универсальный протокол для микробиологического анализа образцов различного происхождения, причем это касается не только грибов. Во Всероссийском НИИ сельскохозяйственной микробиологии с использованием аналогичных подходов проводят анализы бактериальных и архейных сообществ, населяющих различные объекты окружающей среды. Мы полагаем, что эти методы с успехом могут быть использованы и в практике сельского хозяйства.
Итак, в настоящей публикации на примере исследования образца древесины предложен быстрый и универсальный протокол, доступный большинству современных отечественных лабораторий, который обеспечивает количественный и детальный таксономический анализ популяции гри- бов, заселяющих тестируемый образец. Метод характеризуется высокой скоростью определения (3-4 сут) и довольно низкими затратами (1-2 тыс. руб. на образец). Его основное достижение заключается в возможности выявлять разнообразие микробных сообществ, которое до последнего времени, по всей видимости, недооценивалось.


