Автоматическая сегментация изображений процесса интрацитоплазматической инъекции сперматозоида
Автор: Ковалев Валентин Юрьевич, Шишкин Алексей Геннадьевич
Журнал: Компьютерная оптика @computer-optics
Рубрика: Обработка изображений, распознавание образов
Статья в выпуске: 4 т.46, 2022 года.
Бесплатный доступ
Данная работа посвящена решению методами глубокого обучения задачи мультиклассовой семантической сегментации изображений процесса интрацитоплазматической инъекции сперматозоида. В качестве входных данных использованы видеопоследовательности, на которых изображена вышеназванная процедура интрацитоплазматической инъекции сперматозоида. Для обучения нейросети выполнена ручная разметка 656 кадров, в результате которой каждый пиксель изображения был отнесен к одному из 4 классов: микроинъектор, микропипетка, яйцеклетка, фон. Проведен анализ современных методов решения, и экспериментальным путем выбраны наилучшие архитектура, кодировщики и гиперпараметры нейронной сети: сверточная нейронная сеть FPN (feature pyramid network) с кодировщиком resnext101, имеющим глубину 101 слой с 32 параллельными разделяемыми свертками. Построенная нейросетевая модель позволила получить эффективность сегментации IOU = 0,96 при скорости работы алгоритма 15 кадров в секунду.
Интрацитоплазматическая инъекция сперматозоида, семантическая сегментация, сверточные нейронные сети
Короткий адрес: https://sciup.org/140295021
IDR: 140295021 | DOI: 10.18287/2412-6179-CO-1060
Текст научной статьи Автоматическая сегментация изображений процесса интрацитоплазматической инъекции сперматозоида
Интрацитоплазматическая инъекция сперматозоидов (ИКСИ, ICSI – Intra Cytoplasmic Sperm Injection) – это один из методов вспомогательной репродуктивной технологии, в котором искусственно проводится процесс введения сперматозоида непосредственно в женскую яйцеклетку (рис. 1). Этот метод разработан как альтернатива естественному оплодотворению для пар с тяжелым мужским фактором бесплодия или пар, которые не смогли добиться успеха в предыдущей попытке экстракорпорального оплодотворения (ЭКО), в котором для оплодотворения требуется большое количество спермы, помещаемой в одну пробирку с яйцеклеткой. Процедура ИКСИ позволяет парам, имеющим невысокие шансы на успешную беременность, получить оплодотворенные эмбрионы. Существует большое число факторов, оказывающих влияние на результат операции, как физиологического, так и технического характера [1 –4]. Несмотря на широкое распространение метода ИКСИ, некоторые технические аспекты этой процедуры и их влияние на окончательный результат являются недостаточно изученными [5]. Хотя существуют подробные рекомендации выполнения ИКСИ [5], успешность процедуры, в первую очередь, помимо степени подвижности сперматозоида, зависит от конкретного оператора, выполняющего ее [6–9]. Например, в работе [9] продемонстрировано, что модификация методики внедрения сперматозоида приводит к увеличению числа успешных беременностей у женщин, для кото- рых предыдущие операции ИКСИ были неудачными. В другом исследовании [7], в котором был произведен ручной анализ 535 видеозаписей процедур ИКСИ, было показано, что при определенном способе введения микроинъектора в яйцеклетку вероятность успешного оплодотворения может быть значительно ниже, чем в среднем. Благодаря тому, что во время выполнения операции ИКСИ производится ее видеозапись, представляется весьма перспективным, используя методы компьютерного зрения, проанализировать весь процесс выполнения и выработать рекомендации оператору для успешного проведения процедуры, а также предсказать с высокой вероятностью результат операции. Первым этапом такого автоматизированного анализа является семантическая сегментация видеоизображений.
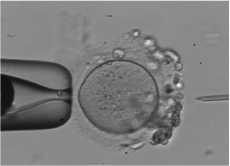
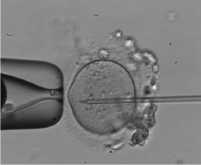
а)
Риc. 1. Схема метода ИКСИ: (а) фиксация микропипеткой яйцеклетки; (б) введение сперматозоида в яйцеклетку с помощью микроинъектора
К сожалению, использование исходных необработанных видеоизображений процесса ИКСИ для автоматизации контроля, улучшения качества и предсказания результатов операции затруднено, так как на изображениях присутствуют шумы, различные арте- факты, а также посторонние объекты – яйцеклетка находится в питательной живой среде. В связи с этим возникает проблема предварительной обработки исходных видеопоследовательностей и устранения присутствующего шума.
Существует только несколько работ, посвященных сегментации изображений ИКСИ. Так, в [10] на основе применения морфологических операций и фильтрации с помощью вейвлет-преобразований выполняется сегментация сперматозоидов на стадии их отбора. В работе [11] с помощью градиент-взвешенного метода Хафа производится выделение контуров ооцитов. В качестве тестового множества рассматривались наборы данных с разными видами аугментаций (разный цвет фона, разное положение ооцита на кадре, разный масштаб изображений) – 4 набора данных по 80 кадров. В последние годы наилучшие результаты для задач семантической сегментации изображений, в том числе в области медицинских снимков, показывают сверточные нейронные сети. Поэтому неслучайно их применение и для задачи сегментации изображений ИКСИ. В [12, 13] используются различные архитектуры глубоких сверточных сетей для сегментации яйцеклетки, а в [14] с помощью модификации сети U-Net выполняется семантическая сегментация микропипетки, яйцеклетки и наконечника иглы микроинъектора. Основным недостатком работы [14] является неполное выделение иглы микроинъек-тора, хотя её положение и методика введения во многом определяют результат операции [6–9].
Целью данной работы является построение и программная реализация нейросетевой модели для семантической сегментации на изображениях процесса оплодотворения с помощью метода ИКСИ важных для исследования объектов: микропипетки, микро-инъектора и яйцеклетки. Данная модель обладает рядом достоинств по сравнению с единственным существующим на данный момент аналогичным решением на основе нейросетей [14]: в отличие от архитектуры U-NET, предложенной в [14], используется обладающая большей обобщающей способностью модель feature pyramid network, которая состоит из множества слоев, по которым делается итоговый прогноз. Помимо этого, на изображениях игла микроинъекто-ра выделяется целиком, а не только ее наконечник, как в [14], что не позволяет, например, определить, под каким углом микроинъектор вводится в яйцеклетку. Следует отметить, что иглу по сравнению с другими объектами выделить сложнее всего. Это связано с тем, что ее изображение является часто размытым, так как микроскоп сфокусирован, в первую очередь, на яйцеклетке; помимо этого, во многих случаях игла резко перемещается оператором, из-за чего её контуры на изображении наиболее сильно размыты. Следует дополнительно отметить, что в силу ограниченных возможностей по ручной разметке изображений по сравнению с [14] для обучения использовалось намного меньше данных – 656 кадров в данной работе против 7983 кадров в работе [14]. Однако, как можно видеть из раздела, посвященного экспериментальному анализу разработанной модели, результаты по метрике пересечения над объединением (intersection over union – IOU) практически идентичны –0,961 и 0,962 в данной работе и в работе [14] соответственно.
Данные
В качестве входного набора рассматривались видеокадры операции ИКСИ, предоставленные клиникой Центр репродукции «Линия Жизни». На момент проведения исследования имелось 230 видеопоследовательностей, они обладают высоким разрешением 1024×768, но на большинстве кадров присутствуют артефакты и шумы (рис. 1), не относящиеся к исследованию .
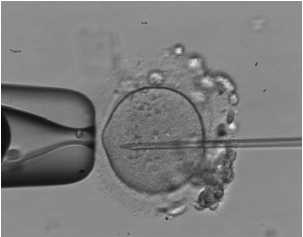
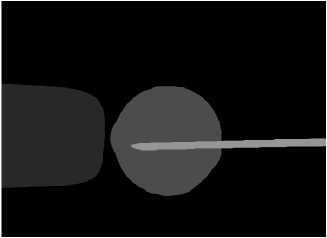
Риc. 2. Исходное изображение (а) и полученная вручную маска в соответствии с выбором цветов для каждого класса (б)
Для исследования случайным образом были отобраны 40 видеопоследовательностей, из которых выбирались 20 кадров, распределенных на протяжении всей видеопоследовательности, обладающих полезной информацией и имеющих относительно хорошее качество. Так, не рассматривались размытые, без необходимых объектов исследования изображения, а также кадры, где визуально сложно определить, и тем более выделить границы объекта. Итого было получено 656 изображений. В качестве предварительной обработки все они были переведены из цветной палитры RGB в палитру оттенков серого цвета (grayscale). С использованием редактора изображений производилась ручная разметка – каждому пикселю был сопоставлен определенный цвет в зависимости от его класса (табл. 1, рис. 2).
Табл. 1. Соответствие классов в разметке и их кодировке соответствующих цветов в палитре (R, G, B)
|
Фон |
Черный цвет (0, 0, 0) |
|
Яйцеклетка |
Красный цвет (255, 0, 0) |
|
Микропипетка |
Синий цвет (0, 0, 255) |
|
Микроинъектор |
Зеленый цвет (0, 255, 0) |
Для обучения использовались 80% данных, для валидации и тестирования – по 10% оставшихся данных.
Отметим, что для нейронных сетей с архитектурой типа U-Net и Feature Pyramid Network, которые использовались в экспериментах данной работы, крайне важно, чтобы размер входных данных был кратен 32, так как при использовании операций свертки и обратной свертки необходимо сохранить заданные про- порции и не потерять часть важных данных. С целью увеличения скорости обучения и повышения обобщающей способности нейронной сети было решено выбирать часть исходного изображения с размерами, кратными 32 [15]. В данном случае был выбран размер 512×768 пикселей.
При исследовании были применены различные типы аугментации с целью генерализации выборки и повышения устойчивости обучающейся нейронной сети к шуму. В данной работе использовались следующие преобразования в указанном порядке:
-
1. Эффекты размытия (каждый раз при обучении выбирается один из эффектов):
-
а. Использование размытия (blur).
-
б. Использование размытия при движении (motion blur).
-
в. Использование медианного размытия (median blur).
-
г. Использование Гауссова размытия (Gaussian blur).
-
2. Вырезание части изображения заданного размера (512×768 пикселей).
-
3. Эффекты шума (каждый раз при обучении выбирается один из эффектов):
-
а. Гауссов шум.
-
б. Цифровой шум изображения (ISO Noise).
-
в. Добавление эффекта резкости.
-
4. Эффекты, связанные с пространственными изменениями (каждый раз выбирается один из эффектов):
-
а. Поворот.
-
б. Оптическое искажение.
-
в. Растровое искажение (grid distortion).
Использование эффектов размытия связано с тем, что на многих видеопоследовательностях объекты могут быть размытыми, но для разметки по большей части использовались кадры, на которых объекты были хорошо видны. Использование эффектов, связанных с шумом, объясняется тем, что, как правило, изображения являются зашумленными, что ухудшает способности нейронной сети для выделения объектов. Применение преобразований, изменяющих форму изображения, обусловлено тем, что часто форма яйцеклетки далека от круглой, а игла может попадать в кадр с разных сторон. Добавление онлайн-аугментаций позволяет увеличить размер набора данных примерно в десять раз.
Результаты экспериментов
Вначале была проведена серия экспериментов с целью выбора наилучшей комбинации гиперпараметров нейросетей. Для сравнения качества моделей и их оценки было предложено зафиксировать размер и данные в обучающей и тестовой выборках, а также выбрать метрику для оценки качества модели: пересечение над объединением (intersection over union, или IOU). Данная метрика является наиболее популярной при решении задачи сегментации изображений.
Самым важным этапом является этап выбора архитектуры нейронной сети и кодировщика. В качестве архитектуры нейронной сети для решения задачи многоклассовой семантической сегментации рассматривались архитектуры U-Net [15] и Feature Pyramid Network [16]. Для описанных выше моделей U-Net и FPN было важно также выбрать оптимальную архитектуру кодировщика. В данный момент существует большое число предварительно обученных кодировщиков: ResNet [17], Inception [18], resNext [19], SeResNet [20], EfficientNet [21], Xception [22]. Помимо этого, архитектуры в качестве начального приближения использовали веса, предварительно обученные (transfer learning [23]) на корпусе данных ImageNet [24], с применением методики noisy student [25].
Кроме того, необходимо правильно выбрать функцию потерь, а также оптимизатор, который позволит получить наилучшие результаты. В данной работе рассматривались следующие функции потерь: функция кросс-энтропии, фокальная функция потерь, вычисление коэффициента Сёренсена. Помимо этого, были рассмотрены различные методы оптимизаторов. Наилучшие результаты были получены с использованием функции потерь перекрестной энтропии с равными весами для каждого класса и оптимизатора AdamW [26] со значением шага обучения, равным 3^10 – 4.
В табл. 2 представлены эксперименты по выбору наилучшей архитектуры для фиксированных параметров аугментаций, функции потерь, оптимизатора и кодировщиков.
Табл. 2. Результаты экспериментов по выбору архитектуры. Все значения даны в единицах IOU
|
Архитектура сети |
Фон |
Яйцеклетка |
Инъектор |
Пипетка |
Усредненное значение метрики |
|
U-Net resnet50 |
0,988324 |
0,92981 |
0,77941 |
0,93231 |
0,9074635 |
|
U-Net resnet101 |
0,989242 |
0,93481 |
0,79421 |
0,94532 |
0,9158955 |
|
U-Net se_resnext50_32×4d |
0,989899 |
0,93798 |
0,790541 |
0,94721 |
0,916407 |
|
U-Net se_resnext101_32×4d |
0,990582 |
0,945986 |
0,787482 |
0,947283 |
0,917833 |
|
FPN resnet50 |
0,995315 |
0,960872 |
0,85393 |
0,977218 |
0,946834 |
|
FPN resnet101 |
0,995924 |
0,98507 |
0,841078 |
0,973926 |
0,948999 |
|
FPN se_resnext50_32×4d |
0,995365 |
0,974233 |
0,847012 |
0,971088 |
0,946924 |
|
FPN se_resnext101_32×4d |
0,995873 |
0,976564 |
0,86294 |
0,976605 |
0,952995 |
Из табл. 2 видно, что наилучшие результаты среди архитектур U-Net и FPN для различных кодировщиков при остальных фиксированных параметрах показывает архитектура FPN.
В табл. 3 представлены результаты экспериментов по выбору наилучшего кодировщика для фиксированных остальных параметров.
Можно заметить, что среди различных кодировщиков наилучшее качество показывает resnext_101_32×8d – кодировщик ResNext, описанный выше, имеющий глубину 101 слой с 32 параллельными разделяемыми свертками. Несмотря на то, что эта сеть является достаточной большой, для изображений исходного размера нейронная сеть работает с оригиналь- ной частотой кадров видеопоследовательностей, а именно, 15 кадров в секунду.
Как следует из результатов экспериментов, разработанная нейросеть имеет наименьшую точность сегментации для микроинъектора. Это может быть связано с тем, что в большинстве случаев микроинъек-тор на изображении является размытым (рис. 3, 4). Обычно оператор фокусирует линзу микроскопа на яйцеклетке, поэтому все остальные объекты на изображении имеют большую или меньшую степень размытия. Помимо этого, достаточно часто встречаются изображения, на которых игла резко перемещается (рис. 4, правый нижний угол). В этом случае сложно выделить её контуры и произвести ручную разметку, что приводит к ошибкам нейронной сети.
Табл. 3. Результаты экспериментов по выбору архитектуры. Все значения даны в единицах IOU
|
Кодировщик |
Фон |
Яйцеклетка |
Инъектор |
Пипетка |
Усредненное значение метрики |
|
Fpn resnet50 |
0,995315 |
0,960872 |
0,85393 |
0,977218 |
0,946834 |
|
Fpn resnet101 |
0,995924 |
0,98507 |
0,841078 |
0,973926 |
0,948999 |
|
Fpn resnet152 |
0,996282 |
0,984465 |
0,86103 |
0,990109 |
0,957972 |
|
Fpn seresnet50 |
0,995635 |
0,983885 |
0,851969 |
0,981264 |
0,953188 |
|
Fpn seresnet101 |
0,995692 |
0,976324 |
0,847174 |
0,981681 |
0,950218 |
|
Fpn seresnet152 |
0,995744 |
0,977127 |
0,830368 |
0,974314 |
0,944388 |
|
Fpn resnext50_32×4d |
0,99557 |
0,984956 |
0,838627 |
0,983907 |
0,950765 |
|
Fpn resnext101_32×4d |
0,99557 |
0,984956 |
0,838627 |
0,983907 |
0,950765 |
|
Fpn resnext101_32×4d |
0,996178 |
0,976765 |
0,866408 |
0,976078 |
0,953857 |
|
Fpn resnext101_32×8d |
0,996511 |
0,992908 |
0,868213 |
0,984737 |
0,960592 |
|
Fpn se_resnext50_32×4d |
0,995365 |
0,974233 |
0,847012 |
0,971088 |
0,946924 |
|
Fpn se_resnext101_32×4d |
0,995873 |
0,976564 |
0,86294 |
0,976605 |
0,952995 |
|
Fpn effnet-b0 |
0,995698 |
0,982065 |
0,849835 |
0,970896 |
0,949623 |
|
Fpn effnet-b1 |
0,995428 |
0,971273 |
0,847722 |
0,984819 |
0,949811 |
|
Fpn effnet-b2 |
0,994488 |
0,973422 |
0,845179 |
0,974954 |
0,947011 |
|
Fpn effnet-b3 |
0,994437 |
0,977884 |
0,850502 |
0,970106 |
0,948232 |
|
Fpn inceptionv4 |
0,995902 |
0,983716 |
0,850977 |
0,976076 |
0,951668 |
|
Fpn xception |
0,996139 |
0,982156 |
0,870264 |
0,991512 |
0,960018 |

Рис. 3. Исходное изображение и полученная для него маска с помощью нейронной сети resnext с наилучшим качеством
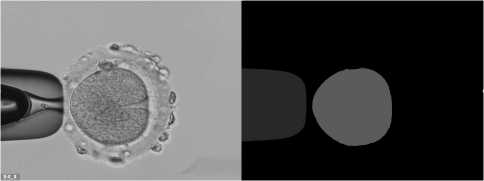
Рис. 4. Исходное изображение и полученная для него маска с помощью нейронной сети resnext с наилучшим качеством
В остальных же случаях, когда проводится сама операция ИКСИ, а именно, инъекция сперматозоида в яйцеклетку с помощью микроинъектора, модель хорошо предсказывает положение объектов и с высоким качеством производит сегментацию изображений (рис. 3– 8). Так, на рис. 3 видно, что нейронная сеть при сегментации хорошо устраняет шум; на рис. 6 можно видеть, что алгоритмом хорошо определена форма и искажения яйцеклетки; на рис. 7 и 8 представлен процесс проникновения иглы в яйцеклетку, при котором яйцеклетка наибольшим образом деформируется.

Рис. 5. Исходное изображение и полученная для него маска
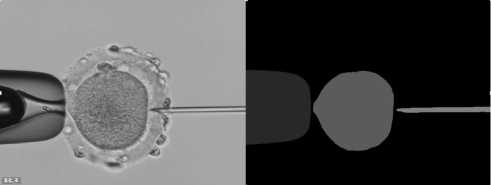
Рис. 6. Исходное изображение и полученная для него маска

Рис. 7. Исходное изображение и полученная для него маска
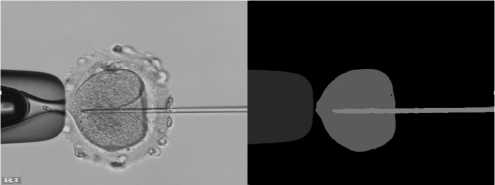
Рис. 8. Исходное изображение и полученная для него маска
Выводы
В данной работе предложен метод автоматической семантической сегментации кадров видеозаписи процесса интрацитоплазматической инъекции сперматозоида на четыре класса: микроинъектор, микропипетка, яйцеклетка и фон. Данный метод основан на использовании глубоких сверточных нейронных сетей. Для обучения нейронных сетей был произведен отбор кадров, выполнена ручная разметка 656 кадров, а также осуществлено разбиение набора данных на обучающую, валидационную и тестовую выборки.
Дополнительную сложность исследованию придавало наличие в данных шума, как генерируемого системой, так и выражающегося в присутствии большого числа артефактов. Кроме того, объекты, отличные от яйцеклетки, часто находились вне фокуса микроскопа, и поэтому их очертания на изображениях были размытыми. Применение аугментаций, имитирующих движение и шумы (использование эффектов размытия, шумов различной природы), позволило улучшить качество работы нейронной сети, а результаты её предсказаний сделать более стабильными. Это также помогло справиться с проблемой переобучения, являющейся типичной при наличии малого числа данных. Несмотря на то, что набор данных изначально был небольшим, использование аугментаций позволило увеличить объем исходной выборки и разнообразить примеры; в итоге это позволило получить модель, которая обладает достаточно хорошей обобщающей способностью.
На основе большого числа экспериментов по выбору оптимальных гиперпараметров нейронной сети, таких как архитектура, кодировщик, оптимизатор, метрика, функция потерь, аугментации, размер пакета данных для одной итерации обучения, были выбраны значения, которые позволили производить сегментацию изображений с наилучшим качеством в режиме реального времени.
Оптимальные результаты были получены с помощью архитектуры FPN с кодировщиком вида resnext с количеством слоёв, равным 101 и наличием 32 разделяемых сверток. Итоговое качество в метрике пересечения над объединением (IoU) составило 0,960592. Помимо этого, для разрешения 512×768, для которого и строились веса модели, нейронная сеть способна производить сегментацию с хорошим качеством в режиме реального времени (15 кадров в секунду, как и в имеющихся видеопоследовательностях).
В дальнейшем планируется использовать полученные сегментационные маски для предсказания результата операции ИКСИ, а также выделять признаки, которые влияют на успех оплодотворения. Это в значительной мере поможет операторам, выполняющим ИКСИ, оптимизировать ход выполнения данной процедуры.
Список литературы Автоматическая сегментация изображений процесса интрацитоплазматической инъекции сперматозоида
- Murid J, Essam M. Intracytoplasmic sperm injection -factors affecting fertilization. In Book: Darwish AMM, ed. Enhancing success of assisted reproduction. Rijeka: IntechOpen; 2012: 117-144.
- Hajiyavand AM, Saadat M, Abena A, Sadak F, Sun X. Effect of injection speed on oocyte deformation in ICSI. micromachines 2019; 10: 226.
- Hafiz P, Nematollahi M, Boostani R, Jahromi BN. Predicting implantation outcome of in vitro fertilization and intracytoplasmic sperm injection using data mining techniques. Int J Fertil Steril 2017; 11(3): 184-190.
- Mostaar A, Sattari MR, Hosseini S, Deevband MR. Use of artificial neural networks and PCA to predict results of infertility treatment in the ICSI method. J Biomed Phys Eng 2019; 9(6): 679-686.
- Rubino P, Vigano P, Luddi A, Piomboni P. The ICSI procedure from past to future: a systematic review of the more controversial aspects. Hum Reprod Update 2015; 22(2): 194-227.
- Tiegs AW, Scott RT. Evaluation of fertilization, usable blastocyst development and sustained implantation rates according to intracytoplasmic sperm injection operator experience. Reprod Biomed Online 2020; 41(1): 19-27.
- Daniel CE, Hickman C, Wilkinson T, Oliana O, Gwinnett D, Trew G, Lavery S. Maximising success rates by improving ICSI technique: which factors affect outcome? Fertil Steril 2015; 104(3): E95-E96.
- Shen S, Khabani A, Klein N, Battaglia D. Statistical analysis of factors affecting fertilization rates and clinical outcome associated with intracytoplasmic sperm injection. Fertil Steril 2003; 79(2): 355-360.
- Ebner T, Moser M, Sommergruber M, Jesacher K, Tews G. Complete oocyte activation failure after ICSI can be overcome by a modified injection technique. Hum Reprod 2004; 19(8): 1837-1841.
- Mirroshandel SA, Ghasemian F. Automated morphology detection from human sperm images. In Book: Palermo GD, Sills ES, eds. Intracytoplasmic sperm injection -Indications, techniques and applications. Springer International Publishing AG; 2018.
- Saadat M, Hajiyavand AM, Singh Bedi AP. Oocyte positional recognition for automatic manipulation in ICSI. Micromachines (Basel) 2018; 9(9): 429.
- Targosz A, Przystalka P, Wiaderkiewicz R, et al. Semantic segmentation of human oocyte images using deep neural networks. Biomed Eng Online 2021; 20: 40.
- Zhao M, Li H, Li R, Li Y, Luo X, Li TC, Lee TL, Wang WJ, Chan DYL. Automated and precise recognition of human zygote cytoplasm: A robust image-segmentation system based on a convolutional neural network. Biomed Signal Process Control 2021; 67: 102551.
- He P, et al. Semantic video segmentation for intracytoplasmic sperm injection procedures. arXiv preprint 2021. Source: ahttps://arxiv.org/abs/2101.01207n.
- Ronneberger O, Fischer P, Brox Th. U-Net: Convolutional networks for biomedical image segmentation. Medical Image Computing and Computer-Assisted Intervention 2015: 234-241.
- Lin T, Dollar P, Girshick R, He K, Hariharan B, Belongie S. Feature pyramid networks for object detection. IEEE Conf on Computer Vision and Pattern Recognition (CVPR) 2017: 936-944.
- Li H, et al. Visualizing the loss landscape of neural nets. arXiv preprint 2017. Source: https://arxiv.org/abs/1712.09913>.
- Szegedy C, Liu W, Jia Y, Sermanet P, Reed S, Anguelov D, Erhan D, Vanhoucke V, Rabinovich A. Going deeper with convolutions. Proc IEEE Conf on Computer Vision and Pattern Recognition 2015: 1 -9.
- Xie S, Girshick R, Dollar P, Tu Z, He K. Aggregated residual transformations for deep neural networks. arXiv preprint 2016. Source: https://arxiv.org/abs/1611.05431v1>.
- Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proc IEEE conf on Computer Vision and Pattern Recognition 2018: 7132-7141.
- Tan M, Le Q. EfficientNet: Rethinking model scaling for convolutional neural networks. Proc 36th Int Conf on Machine Learning 2019: 6105-6114.
- Chollet F. Xception: Deep learning with depthwise separable convolutions. Proc IEEE conf on Computer Vision and Pattern Recognition 2017: 1251-1258.
- Tan C, et al. A survey on deep transfer learning. Int conf on Artificial Neural Networks 2018: 270-279.
- Deng J, Dong W, Socher R, Li L, Li K, Fei-Fei L. ImageNet: A large-scale hierarchical image database. 2009 IEEE Conf on Computer Vision and Pattern Recognition 2009: 248-255.
- Xie Q, et al. Self-training with noisy student improves imagenet classification. Proc IEEE/CVF Conf on Computer Vision and Pattern Recognition 2020: 10687-10698.
- Kingma DP, Ba J. Adam: A method for stochastic optimization. 3rd Int Conf for Learning Representations 2015. Source: https://arxiv.org/abs/1412.6980>.


