Данные по изменчивости региона ITS 1-2 ядерной рибосомальной ДНК Betula turkestanica, B. tianschanica, B. procurva
Автор: Медведева С.О., Черепанова О.Е., Толкач О.В., Пономарев В.И., Малосиева Г.В.
Журнал: Лесохозяйственная информация @forestry-information
Рубрика: Материалы 7-го международного совещания "Сохранение лесных генетических ресурсов"
Статья в выпуске: 2, 2023 года.
Бесплатный доступ
Изложены предварительные результаты анализа генетического полиморфизма региона ITS 1-2 ядерной рибосомальной ДНК Betula turkestanica Litv., B. tianschanica Rupr., B. procurva Litv., выполненного с целью прояснения таксономического статуса, филогенетических взаимоотношений и происхождения этих видов берез. Ареалы B. procurva, B. turkestanica, B. tianschanica охватывают территорию Центральной Азии (Памиро-Алтай), при этом B. procurva встречается изолировано от основного ареала в горной части Урала (зона бореальных лесов и Зауральская лесостепь). Эти виды берез обладают морфологическими признаками B. microphylla Bunge и B. pendula Roth, но имеют и отличительные особенности морфологии. Показан уровень дифференциации и распределение генетического разнообразия между группами гаплотипов, а также приводится генетическая сеть, демонстрирующая взаимоотношения между исследованными образцами. Основываясь на результатах генетического анализа, высказано предположение о происхождении и филогенетических взаимоотношениях B. procurva, B. turkestanica, B. tianshanica. Полученная сеть гаплотипов подтверждает гипотезу о возможном общем предке B. procurva, B. pendula из секции Betula (белые березы) и B. nana из секции Apterocaryon (карликовые березы). Сделан вывод об информативности региона ITS 1-2 ядерной рибосомальной ДНК для дифференциации исследованных видов берез на территории Кыргызстана. При этом наибольшее гаплотипическое разнообразие характерно для образцов B. procurva с дивергентным положением образцов Курганской группы, что свидетельствует о длительном существовании этой дизъюнкции.
Betula, its-маркер, ядерная рибосомальная днк, филогенетический анализ, таксономия
Короткий адрес: https://sciup.org/143180081
IDR: 143180081 | УДК: 575.86 | DOI: 10.24419/LHI.2304-3083.2023.2.10
Текст научной статьи Данные по изменчивости региона ITS 1-2 ядерной рибосомальной ДНК Betula turkestanica, B. tianschanica, B. procurva
Data on Variability of Nuclear Ribosomal DNA ITS 1–2 Markers of Betula turkestanica, B. tianschanica, B. procurva
Svetlana O. Medvedeva 1Olga E. Cherepanova2
Candidate of Biological Sciences
Olga V. Tolkach3
Vasily I. Ponomarev4
Galina V. Malosieva5
Введение
Березы ( Betula L.) – листопадные деревья и кустарники Северного полушария, встречающиеся в диапазоне широт от субтропиков до Арктики и обладающие высокой экологической пластичностью. Они участвуют в формировании экосистем болот, высокогорий, тундры и различных типов бореальных лесов. Некоторые представители рода Betula являются важнейшими лесообразующими видами. Быстрорастущие и легко адаптирующиеся к изменению климата виды берез имеют большой потенциал использования в лесовосстановительных проектах [1].
Род Betula считается одним из самых таксономически сложных родов среди покрытосеменных [2]. Виды, относящиеся к этому роду, характеризуются высокой изменчивостью морфологических признаков, обусловленной интенсивной и широко распространённой гибридизацией, что затрудняет филогенетические исследования и идентификацию видов. В результате этого не существует единого мнения о видовом составе берез. По оценке Ashburner and McAllister [3], р. Betula включает около 65 видов берез, по данным других авторов их значительно больше – около 120 [4, 5].
Важным вопросом систематики рода Betula является таксономическое положение некоторых видов берез, принадлежащих секции Betula , таких как B. procurva, B. turkestanica, B. tianschanica, ареалы которых охватывают территорию Центральной Азии (Памиро-Алтай); при этом B. procurva растет изолировано и в горной части Урала (зона бореальных лесов и Зауральская лесостепь). Эти виды берез обладают морфологическими признаками B. microphylla Bunge и B. pendula Roth, но имеют и отличительные особенности морфологии (беловатая или желто-коричневая шелушащаяся кора, средняя высота дерева, длина и форма сережек, морфология плодов) [3, 12, 13]. В монографии Ashburner и McAllister [3] B. procurva и B. turkestanica считаются синонимами B. tianschanica . Однако другие авторы полагают, что эти виды самостоятельные [13, 14]. Для прояснения таксономического статуса вышеуказанных видов берез мы начали работу по изучению генетического разнообразия и генетической структуры популяций B. procurva, B. turkestanica, B. tianshanica на основе анализа нуклеотидных последовательностей ядерной рибосомальной ДНК (регион ITS 1–2).
Цель исследования – получение предварительных данных о филогенетическом положении и происхождении B. procurva, B. turkestanica,
B. tianschanica , а также оценка информативности региона ITS 1–2 ядерной рДНК в качестве молекулярного маркера для определения таксономического статуса изучаемых видов берез.
Материалы и методы исследования
Сравнительный анализ полиморфизма межгенных спейсеров ядерной рибосомальной ДНК (регион ITS 1–2) проведен на материале, полученном как из высушенных (гербарных), так и из свежих листьев берез. Перечень, количество и географическое положение образцов, использованных в работе, представлены в таблице. Были проанализированы образцы листьев берез B. turkestanica, B. procurva, B. tianshanica, собранные в естественных местообитаниях (Кыргызстан, близ реки Гульча), в Ботаническом саду им. Е.З. Гареева НАН (Кыргызстан, Бишкек), а также гербарные образцы Betula nana L. из Челябинской обл. и B. procurva из Курганской обл. B. nana не входит в секцию Betula и является представителем карликовых берез (секция Apterocaryon). Однако образцы B. nana были использованы в нашем исследовании для прояснения филогенетических закономерностей, в связи с предполагаемым родством этого вида с B. procurva [10,15]. Для построения генетической сети и филогенетического анализа дополнительно включены последовательности региона ITS 1–2 из Генбанка для березы пушистой (Betula pubescens Ehrh.) и березы повислой (B. pendula) – предположительных родственных видов B. turkestanica, B. procurva, B. tianshanica. Образец ольхи серой (Alnus incana (L.) Moench) использовали в качестве внешней группы (см. таблицу)1.
Тотальная ДНК была выделена с использованием CTAВ-метода [16]. Для анализа генетического разнообразия использовали межгенные спейсеры ядерной рибосомальной ДНК (регион ITS 1–2). Амплификацию регионов осуществляли с использованием универсальных праймеров, реакционных условий и температурных режимов, рекомендованных для этих участков [17]. Последующее секвенирование проводили по методу Сэнгера при помощи BrilliantDye v. 3.1 kit (Gene Analyzers 3130; NimaGen, Netherlands). Редактирование и выравнивание полученных последовательностей выполнялось вручную в программах BioEdit7.0 [18] и Mega6 [19].
Для проведения статистического анализа единичные образцы были объединены в группы по географическому признаку (группа B. procurva из Кыргызстана, Курганская группа B. procurva , группа B. tianschanica и B. turkestanica ). Уровень дифференциации и распределение генетического полиморфизма между группами гаплотипов (анализ молекулярной дисперсии – AMOVA) определяли с помощью пакета программ DNASP 6.0
Географическое положение и гаплотипы образцов
|
№ |
Вид |
Географическое положение образцов / Реф. № Генбанка |
Кол-во образцов |
Гаплотипы |
|
1 |
B. turkestanica |
Кыргызстан, с. Гульча 40°18 ′ 49 ″ /73°26 ′ 39 |
1 |
Г3 |
|
2 |
B. procurva |
Кыргызстан, с. Гульча 40°18 ′ 49 ″ /73°26 ′ 39 |
1 |
Г7 |
|
3 |
B. procurva |
Ботанический сад им. Е.З. Гареева НАН, Кыргызстан, Бишкек |
1 |
Г6 |
|
4 |
B. procurva |
Российская Федерация, Курганская область 55°27 ′ /65°20 ′ |
2 |
Г5 |
|
5 |
B. nana |
Российская Федерация, Челябинская область 55°09 ′ /61°24 ′ |
2 |
Г4 |
|
6 |
B. tianschanica |
Кыргызстан, с. Гульча 40°18 ′ 49 ″ /73°26 ′ 39 |
1 |
Г8 |
|
7 |
B. pubescens |
Генбанк реф. № AY761130, Черногория |
1 |
Г2 |
|
8 |
B. pendula |
Генбанк реф. №AY761127, Финляндия |
1 |
Г1 |
|
9 |
A. incana |
Генбанк реф. № AJ251665, Европа |
1 |
Внешняя группа |
1 База данных NCBI. (по состоянию на 15 сентября 2021).
[20]. Значения FST были получены для оценки молекулярной дивергенции между отдельными группами. Филогенетические отношения между гаплотипами реконструированы с помощью NETWORK ver. 4.6.1.2 (метод Reduced Median, RM) [21].
Результаты и обсуждение
В результате секвенирования межгенного спейсера (ITS 1–2) ядерной рДНК различных образцов берез были получены последовательности длиной около 600 п.н. После выравнивания их с последовательностями B. pendula и B. pubescens , загруженными из Генбанка, были идентифицированы 9 гаплотипов, включая внешнюю группу A. incana , отношения между которыми были визуализированы в виде сети (рисунок).
Общее число проанализированных сайтов составило 570 (включая гэпы), количество вариабельных сайтов – 69 (включая гэпы), разнообразие гаплотипов – 0,949, общее количество мутаций – 73. Все виды характеризовались отдельными гаплотипами, при этом у B. procurva было выявлено 3 гаплотипа (см. таблицу).
Нуклеотидные последовательности региона ITS 1–2 ядерной рДНК исследованных образцов оказались достаточно информативными для изучения филогенетических отношений, а также определения филогенетического положения B. procurva, B. tianschanica, B. turkestanica внутри секции Betula . Все изученные образцы имели уникальные наборы мутаций в последовательностях ITS 1–2, образуя различные гаплотипы (см. таблицу и рисунок).
Значения генетических дистанций (FST) между географическими группами образцов (группа B. procurva из Кыргызстана, Курганская группа B. procurva , группа B. tianschanica и B. turkestanica ) варьировали от 0,067 до 0,600. Наибольший показатель генетической дифференциации (FST – 0,600) был отмечен между образцами B. tianschanica , B. turkestanica из Кыргызстана и B. procurva из Курганской обл. Анализ
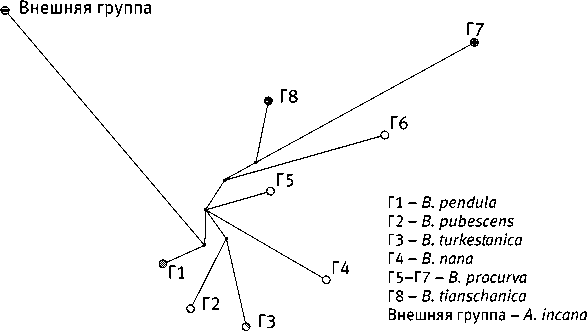
Генетическая сеть, демонстрирующая взаимоотношения между исследуемыми образцами видов рода Betula , которая построена на основе матрицы генетических дистанций Нея для секвенированных последовательностей ядерной рибосомальной ДНК (регион ITS 1–2)*
* Длина отрезков сети соответствует количеству мутационных шагов.
выявил низкую генетическую дифференциацию между образцами B. procurva из различных географических локализаций, что свидетельствует об их гомогенности. Индекс фиксации FST между B. procurva из Курганской обл. и B. procurva из Кыргызстана составил 0,206. Низкие значения генетической дифференциации (FST – 0,067) между группой B. procurva из Кыргызстана и группой B. turkestanica , B. tianschanica могут свидетельствовать об относительно недавней и быстрой дивергенции этих видов от общего предка, а также о высокой степени межвидовой гибридизации.
Пространственная ориентация ITS 1–2 гаплотипов построена, принимая во внимание делеции как пятый тип нуклеотида. Полученная сеть гаплотипов подтверждает гипотезу о возможном общем предке B. procurva, B. pendula и B. nana . На это указывает отхождение эволюционных линий гаплотипов B. procurva, B. nana, B. pendula от одного узла (см. рисунок). Пространственное распределение ITS 1–2 гаплотипов B. procurva согласуется с ранее выявленной географической структурой хлоропластных гаплотипов [22]. При этом в отличие от вышеуказанного исследования для региона ITS 1–2 не было выявлено общности гаплотипов.
Полученная генетическая сеть указывает на раннее расхождение и длительную изоляцию курганских и кыргызских образцов B. procurva .
Заключение
Проведенное молекулярно-генетическое исследование, основанное на маркерах ITS 1–2 ядерной рДНК, позволило сделать вывод об общности происхождения B. procurva, B. turkestanica, B. tianshanica. Регион ITS 1–2 ядерной рибосомальной ДНК оказался достаточно информативным для дифференциации этих близкородственных видов берез на территории Кыргызстана. Наибольшее гаплотипическое разнообразие характерно для образцов B. procurva с дивергентным положением образцов Курганской группы, что свидетельствует о длительном существовании этой дизъюнкции. Полученная сеть гаплотипов подтверждает гипотезу о возможном общем предке B. procurva, B. pendula из секции Betula (белые березы) и B. nana из секции Apterocaryon (карликовые березы). Для подтверждения вышеизложенных гипотез необходимо провести анализ большего количества образцов с использованием дополнительных молекулярных маркеров для более полного изучения филогенетических взаимоотношений B. procurva, B. turkestanica и B. tianschanica.
Список литературы Данные по изменчивости региона ITS 1-2 ядерной рибосомальной ДНК Betula turkestanica, B. tianschanica, B. procurva
- Dubois, H. Potential of Birch (Betula péndula Roth and B. pubescens Ehrh.) for Forestry and Forest-Based Industry Sector within the Changing Climatic and Socio-Economic Context of Western Europe / H. Dubois, E. Verkasalo, H. Claessens // Forests. - 2020. - № 11. - P. 336. DOI: 10.3390/f11030336.
- Phylogenetic relationships of Betula species (Betulaceae) based on nuclear ADH and chloroplast matK sequences / P. Jarvinen, A.E. Palmé, L.O. Morales, M. Lannenpaa, M. Keinanen, T. Sopanen, M. Lascoux // American Journal of Botany. - 2004. - № 91. - P. 1834-1845. Doi: 10.3732/ajb.91.11.1834.
- Ashburner, K. The Genus Betula: A Taxonomic Revision of Birches / K. Ashburner, H.A. McAllister. - London : Kew Publishing, 2016.
- Коропачинский, И.Ю. Естественная гибридизация и проблемы систематики берез Северной Азии / И.Ю. Коропачинский // Сибирский экологический журн. - 2013. - Т. 20. - № 4. - С. 459-479. DOI: 10.1134/ S1995425513040045.
- Ветчинникова, Л.В. Береза: вопросы изменчивости (морфо-физиологические и биохимические аспекты) / Л.В. Ветчинникова. - Москва : Наука, 2004. - 183 с.
- Li, J.H. Phylogenetics of Betula (Betulaceae) inferred from sequences of nuclear ribosomal DNA. - Text : electronic / J.H. Li, S. Shoup, Z.D. Chen // Rhodora. - 2005. - № 107. - P. 69-86. - Режим доступа: https://www. jstor.org/stable/23314506.
- Nagamitsu, T. Endemic dwarf birch Betula apoiensis (Betulaceae) is a hybrid that originated from Betula ermanii and Betula ovalifolia / T. Nagamitsu, T. Kawahara, A. Kanazashi // Plant Species Biology. - 2006. - № 21. - P. 19-29. DOI: 10.1111/j.1442-1984.2006.00147.x.
- Molecular phylogeny and genome size evolution of the genus Betula (Betulaceae) / N. Wang, H.A. McAllister, P.R. Bartlett, R.J.A. Buggs // Ann Bot-London. - 2016. - № 117. - P. 1023-1035. DOI: 10.1093/aob/mcw048.
- Chloroplast DNA variation of Betula humilis Schrk. in Poland and Belarus / K. Jadwiszczak, A. Banaszek, E. Jablonska, O.V. Sozinov // Tree Genetics and Genomes. - 2012. - № 8. - P. 1017-1030. DOI: 10.1007/ s11295-012-0482-y.
- Extensive sharing of chloroplast haplotypes among European birches indicates hybridization among Betula pendula, B. pubescens and B. nana / A.E. Palmé, Q. Su, S. Palsson, M. Lascoux // Molecular Ecology. - 2004. - № 13. - P. 167-178. - DOI: 10.1046/j.1365-294x.2003.02034.x.
- Multispecies genetic structure and hybridization in the Betula genus across Eurasia / Y. Tsuda, V. Semerikov, F. Sebastiani, G.G. Vendramin, M. Lascoux // Molecular Ecology. - 2017. - № 26. - P. 589-605. DOI: 10.1111/ mec.13885
- Комаров, В.И. Флора СССР / В.И. Комаров. - Москва : изд-во Академии наук СССР, 1936.
- Васильев, В.Н. Березы Урала / В.Н. Васильев // Тр. Ин-та экологии растений и животных. - Вып. 69. -Свердловск, 1969. - С. 59-140.
- Skvortsov, A.K. A new system of the genus Betula L. - the birch / A.K. Skvortsov // Bulletin of Moscow Society of Naturalist. - 2002. - № 107. - P. 73-76.
- Vaarama, A. On the taxonomy, biology and origin of Betula tortuosa Ledeb. / A. Vaarama, T. Valanne // Reports of the Kevo Subarctic Research Station. - 1973. - № 10. - P. 70-84.
- Doyle, J.J. Isolation of Plant DNA from Fresh Tissue / J.J. Doyle, and J.L. Doyle // Focus. - 1990. - T. 12. - № 1. - P. 13-15.
- Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics / T.J. White, T. Bruns, S. Lee, T. Taylor // PCR protocols: a guide to methods and applications. - New York : Academic Press, 1990. - P. 315-322.
- Hall, T.A. Bioedit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT / T.A. Hall // Nucl. Acid Symp. - 1999. - № 41. - P. 95-98. DOI: 10.12691/ajmr-3-2-1.
- MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0 / K. Tamura, G. Stecher, D. Peterson, A. Filipski, S. Kumar // Molecular biology and evolution. - 2013. - T. 30. - № 12. - P. 2725-2729. DOI: 10.1093/molbev/mst197.
- DnaSP 6: DNA Sequence Polymorphism Analysis of Large Datasets / J. Rozas, A. Ferrer-Mata, J.C. Sánchez-DelBarrio, S. Guirao-Rico, P. Librado, S.E. Ramos-Onsins, A. Sánchez-Gracia // Mol. Biol. Evol. - 2017. - № 34. -P. 3299-3302. DOI: 10.1093/molbev/msx248.
- Bandelt, H.J. Median-joining networks for inferring intraspecific phylogenies / H.J. Bandelt, P. Forster, A. Röhl // Mol Biol Evol. - 1999. - № 1. - P. 37-48. DOI: 10.1093/oxfordjournals.molbev.a026036.
- TrnL-trnfF cpDNA polymorphism in some representatives of the genus Betula / S. Medvedeva, O. Cherepanova, O. Tolkach, V. Ponomarev, G. Malosieva // BIO Web of Conferences. -2021. - № 35. DOI:10.1051/bioconf/20213500017.


