Дефензины растений: биологическая роль, механизмы действия и методы анализа
Автор: Кулаева Ольга Алексеевна, Клюкова М.С., Повыдыш М.Н., Тихонович Игорь Анатольевич, Жуков В.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры
Статья в выпуске: 1 т.52, 2017 года.
Бесплатный доступ
Одной из актуальных проблем современного сельского хозяйства остается потеря урожая вследствие различных биотических и абиотических факторов. У растений существует многокомпонентная система защиты, включающая создание защитных барьеров, активацию реакции гиперчувствительности и синтез антимикробных пептидов, которые представляют собой низкомолекулярные соединения, проявляющие широкий спектр активности против грибов, бактерий и вирусов. К антимикробным пептидам растений относятся несколько групп соединений, в том числе дефензины - богатые цистеином пептиды, состоящие из 45-54 аминокислот. По-видимому, это один из наиболее распространенных классов антимикробных пептидов, которые участвуют в различных защитных реакциях и обнаруживаются у всех живых существ (B.P. Thomma с соавт., 2002). Гены растительных дефензинов экспрессируются в разных органах растений, где их белковые продукты необходимы при биотическом и абиотическом стрессах, то есть эти пептиды крайне важны с точки зрения селекции сортов, устойчивых к патогенам и абиотическим стрессам. Дефензины характеризуются сильной вариабельностью последовательностей, что, вероятно, коррелирует с разнообразием механизмов действия этих пептидов, способных как проникать внутрь клетки патогена, так и индуцировать гибель клетки, оставаясь на ее поверхности (T.M. Shafee с соавт., 2016). Большинство растительных дефензинов обладают антифунгальной, некоторые - антибактериальной активностью, которая может сочетаться с антимикотическими свойствами. Для небольшого числа дефензинов показано участие в обеспечении устойчивости к тяжелым металлам, холодовому стрессу, засухе, засолению и в процессах онтогенеза. Современные методические подходы позволяют проводить эффективный поиск новых форм активности дефензинов, исследуя дикорастущие немодельные растительные объекты. Интенсивное изучение их транскриптомов стало возможным с развитием секвенирования следующего поколения (Next Generation Sequencing). Для корректного аннотирования последовательностей высоковариабельных пептидов небольшого размера в пределах семейства необходимо применять специальные программы, в частности SPADA (Small Peptide Alignment Discovery Application), алгоритм работы которой заключается в проведении множественного выравнивания последовательностей известных представителей генного семейства и построении предсказательной модели для поиска новых членов этого семейства (P. Zhou с соавт., 2013). При анализе большого числа генов, кодирующих дефензины, предсказание активности вновь выявляемых дефензинов и обнаружение консервативных аминокислот также возможно с использованием методов вычислительной биологии. Подход, основанный на множественном выравнивании последовательностей и кластерном анализе, позволяет разделить дефензины на группы со сходной функциональной активностью (N.L. van der Weerden с соавт., 2013). Таким образом, сочетание современных методов молекулярной и вычислительной биологии существенно повышает эффективность изучения этой группы защитных белков.
Дефензины, пептиды, богатые цистеином, защитные реакции, биотический и абиотический стресс, секвенирование следующего поколения
Короткий адрес: https://sciup.org/142214154
IDR: 142214154 | УДК: 632.938.2:57.023:57.21:57.087.1 | DOI: 10.15389/agrobiology.2017.1.3rus
Текст обзорной статьи Дефензины растений: биологическая роль, механизмы действия и методы анализа
Различные повреждения, вызванные насекомыми, грибами, вирусами или физическими факторами, приводят к потерям урожая. Для борьбы с негативным влиянием окружающей среды растения используют многокомпонентную систему, включающую реакцию гиперчувствительности, укрепление защитных барьеров с использованием компонентов клеточной стенки, активацию продукции защитных белков и антимикробных пептидов (1). Антимикробные пептиды представляют собой низкомолекулярные
Работа О.А. Кулаевой финансово поддержана Российским фондом фундаментальных исследований (грант ¹ 16-34-60132), работа М.С. Клюковой финансово поддержана грантом Президента РФ по поддержке ведущих научных школ (НШ-6759.2016.4), работа И.А. Тихоновича поддержана Российским научным фондом (грант ¹ 14-24-00135), работа В.А. Жукова финансово поддержана Российским научным фондом (грант ¹ 16-16-00118).
соединения (размером 12-95 аминокислот), которые синтезируются в клетках всех живых организмов. Структура и механизмы действия антимикробных пептидов очень разнообразны. Растительные антимикробные пептиды подразделяют на несколько семейств — циклотиды, дефензины, тионины, липидпереносящие белки, гевеиноподобные белки и др. (1-3).
Растительные дефензины — небольшие богатые цистеином пептиды, состоящие из 45-54 аминокислот. Они обнаруживаются у большинства представителей царства Растения. Дефензины растений, насекомых, млекопитающих и грибов структурно и функционально сходны (4-6). Гены растительных дефензинов экспрессируются в разных органах — семенах (7, 8), листьях (9), корнях (10), цветках (11, 12) и симбиотических клубеньках (13). Показана как конститутивная, так и индуцибельная экспрессия генов, кодирующих растительные дефензины (11, 14). Основная функция дефензинов заключается в ингибировании грибного поражения (8, 11, 15), но для некоторых пептидов описана антибактериальная активность (16, 17), ингибирование трипсина (18), а также участие в формировании устойчивости к тяжелым металлам (19), холодовому стрессу (20, 21), засухе (22), засолению (23, 24) и в процессах развития (25).
Структура растительных дефензинов . Современные исследования показывают, что по признаку структурной организации дефензины животных, растений и грибов можно разделить на два суперсемейства (цис- и транс-дефензины) с независимым эволюционным происхождением (26). У цис-дефензинов два параллельных дисульфидных мостика связывают α -спираль с β -тяжами, а у транс-дефензинов аналогичные дисульфидные мостики противоположно ориентированы и связываются с разными структурными элементами. Дефензины растений — основные представители цис-дефензинов. С использованием ядерно-магнитно-резонансной спектроскопии показано, что у растительных дефензинов основу молекулы формирует трехслойный элемент, состоящий из одной α -спирали, соединенной цистеиновыми мостиками с β -тяжами (рис. 1) (7, 8, 27, 28).

Рис. 1. Аминокислотная последовательность зрелого дефензина MtDef4. Линии, соединяющие цистеиновые остатки, — дисульфидные мостики. Спираль и стрелки отмечают расположение соответственно α-спира- ли и β-тяжей. Консервативные остатки цистеина выделены жирным шрифтом.
Все дефензины растений образуются при процессинге белков-предшественников, которые могут быть неодинаковыми у представителей разных видов. В растительных организмах идентифицированы дефензины с 8 или 10 консервативными цистеиновыми остатками. Предполагается, что такие остатки необходимы для поддержания конформации молекулы, тогда как выявленная высокая вариабельность функциональных регионов дефензинов обеспечивает их специфичность по отношению к мишеням и обусловливает разнообразие функций этого белкового семейства (26, 29).
Функциональная характеристика некоторых дефензинов. Для большинства известных дефензинов характерны антифунгальная и (или) бактерицидная активности. Дефензин NaD1 был выделен из внешних клеточных слоев цветка декоративного табака Nicotiana alata, что коррелирует с его защитной функцией в репродуктивных органах. Исследования in vitro показали, что NaD1, представленный в виде димера, подавляет рост Botrytis cinerea и Fusarium oxysporus (11). ω-Хордотионин и γ-хордоти-онин, обнаруженные в эндосперме зерен ячменя, ингибируют трансляци- онную активность как в эукариотических бесклеточных системах млекопитающих (лизаты клеток печени мыши и ретикулоцитов кролика), так и в прокариотических системах (Escherichia coli), но не проявляют того же эффекта по отношению к процессам трансляции в растительных системах (30). AlfAFP из семян Medicago sativa относится к антифунгальным пептидам и обеспечивает устойчивость к грибному патогену Verticillium dahlia в трансгенных растениях картофеля (11, 31). Дефензины из Raphanus sativus (RsAFP) накапливаются по мере созревания семян и высвобождаются после нарушения целостности их оболочек для создания микросреды, подавляющей рост грибов, при этом наблюдается уменьшение элонгации гиф и их усиленное ветвление (8, 32). MsDef1 из семян M. sativa значительно ингибирует рост F. graminearum in vitro (33). Эти дефензины — примеры пептидов с 8 консервативными цистеиновыми остатками. К другому подклассу (с 10 консервативными цистеиновыми остатками) относятся дефензины PhD1 и PhD2 (соответственно Petunia hybrida defensin 1 и 2), выделенные из цветков петунии. В их трехмерной структуре присутствует дополнительная дисульфидная связь (11, 34), которая не изменяет конформацию по сравнению с другими дефензинами, но, вероятно, придает дополнительную термодинамическую стабильность (34). В экспериментах in vitro PhD1 и PhD2 блокировали рост F. oxysporum и в меньшей степени B. cinerea (11).
Помимо антигрибной активности, некоторые растительные дефензины обладают антибактериальными свойствами. Так, Ср-тионин II, идентифицированный в семенах Vigna unguiculata , действует против грамполо-жительных и грамотрицательных бактерий — Staphylococcus aureus , E . coli и Pseudomonas syringae (16). Фабатины, выделенные из бобов Vicia faba , также угнетают рост различных бактерий, но неактивны против дрожжей Sac-charomyces cerevisiae и Candida albicans (17). PsD1 (из Pisum sativum ) — антибактериальный пептид, который локализуется в ядрах обработанных клеток Neurospora crassa и взаимодействует с белками контроля и остановки клеточного цикла, например циклином F (35).
Показано, что 8 генов M. truncatula MtDefNS специфически экспрессируются в клубеньках, образованных при симбиозе с Sinorhizobium meliloti , и (за исключением одного гена) находятся на 8-й хромосоме, что предполагает некоторую специализацию этих последовательностей. Также большинство из них филогенетически кластеризуются и формируют отдельную кладу по отношению к другим классическим дефензинам (13). Однако конкретная функция дефензинов при симбиотических взаимоотношениях к настоящему моменту не известна.
Для небольшого числа дефензинов описано участие в процессах адаптации растений к абиотическим воздействиям. Так, повышенные концентрации NaCl влияли на изменение экспрессии генов, кодирующих дефензины у Arachis hypogaea (23) и Arabidopsis thaliana (24). Влияние дефицита воды на изменение экспрессии генов, кодирующих дефензины, показано у Glycine max (22, 36). Холод также влияет на экспрессию генов дефензинов (20, 21). У озимой пшеницы выявлен ген Tad1 , экспрессия которого индуцируется специфично при акклиматизации к холодовому стрессу (21). У гипераккумулятора цинка и кадмия Arabidopsis halleri выявлен ген, кодирующий дефензин AhPDF1.1, который активно изучается (19, 37-40). Экспрессия этого гена в A. thaliana и S. cerevisiae приводит к росту устойчивости к цинку, но не к кадмию, кобальту, железу или натрию. У A. halleri проявляется цинк-зависимость пула дефензинов как на уровне их транскриптов, так и на уровне пептидов. По сравнению с A. thaliana у A. halleri наблюдается повышение количества дефензинов в побегах (19).
Для некоторых дефензинов показано участие в ответе на несколько стрессоров. У Brassica rapa экспрессия генов, кодирующих дефензины BrDLFP и BrBetvAFP, значительно изменялась при холодовом стрессе, засухе и засолении (41). При изучении влияния предобработки растений арабидопсиса нетоксичными концентрациями кремния и кадмия на инфицирование B. cinerea показано усиление экспрессии гена, кодирующего дефензин PDF1.2 (42). Экспрессия гена дефензина NbDef2.2 у N. bentham-iana повышается не только после инфицирования растений Pseudomonas syringae pv. tabaci, но также при поранении и обработке этиленом (43). Таким образом, растительные дефензины могут быть компонентами перекрывающихся систем ответа на абиотические и биотические воздействия.
Механизмы действия растительных дефензинов. Сильная вариабельность последовательностей растительных дефензинов, видимо, коррелирует с разнообразием механизмов их действия (26). К настоящему моменту в основном хорошо изучены механизмы действия дефензинов с антифунгальной активностью. Растительные дефензины могут взаимодействовать со специфическими сайтами связывания (рецепторами) — компонентами мембраны клетки-мишени, например бактериальным липидом II, сфинголипидами (44) и фосфолипидами грибов (45). При этом разные растительные дефензины взаимодействуют с различными классами сфинголипидов (46-49). Так, RsAFP2 из редиса связывается с гликосфинголипидами Pichia pastoris и C. albicans (50). Дефензин NaD1 взаимодействует с различными фосфолипидами, но не со сфинголипидами (45). Присутствие специфических сфинголипидов на поверхности мембраны необходимо для опосредования клеточной гибели грибов, так как дрожжевые мутанты, дефектные по генам, вовлеченным в синтез сфинголипидов, устойчивы к дефензинам, которые с ними связываются (47, 48, 51). После взаимодействия с рецепторами дефензины либо проникают внутрь грибной клетки и там взаимодействуют с внутриклеточными мишенями, либо остаются на поверхности (например, как MsDef1) и индуцируют клеточную гибель посредством индукции сигнального каскада (рис. 2) (52). Пример дефензина, проникающего внутрь клетки, — MtDef4 (см. рис. 2). В его последовательности идентифицирован RGFRRR мотив, который, по-видимому, служит сигналом транслокации, необходимым для проникновения в клетку гриба. При замене этого мотива на AAAARR или RGFRAA MtDef4 утрачивает способность проникать внутрь клетки (53). Тем не менее, нет свидетельств, что эта последовательность присутствует у других растительных дефензинов, проникающих в клетки гриба (52).
А Б
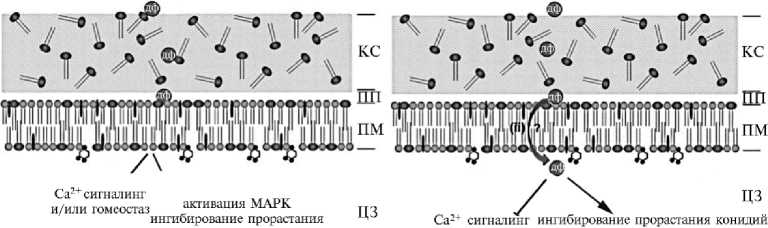
конидий и/или гомеостаз ингибирование слияния клеток гибель гриба гибель триба
Рис. 2. Предполагаемые механизмы антифунгального действия на примере дефензинов MsDef1 (А, не проникает в клетку гриба) и MtDef4 (Б, проникает в клетку гриба) : ДФ — дефензин, КС — клеточная стенка гриба, ПП — периплазматическое пространство, ПМ — плазматическая мембрана, ЦЗ — цитозоль, MAPK — митоген-активируемая протеинкиназа (цит. по 52 с изменениями).
В последнее время появляется все больше сведений о пептидах, которые вызывают гибель клеток, стимулируя выработку активных форм кислорода (АФК) (54-56). Образование АФК и индукцию окислительного стресса наблюдали в клетках-мишенях при обработке дефензинами RsAFP2, DmAMP1(54), HsAFP1 (57) и NaD1 (55).
Пока что остаются неизвестными молекулярные механизмы действия дефензинов при абиотическом стрессе. Это связано с небольшим (относительно роли дефензинов в биотическом стрессе) объемом исследований, сложностью проведения такого рода экспериментов и тем, что дефензины крайне различаются как по структуре, так и по механизмам действия. Большинство работ посвящено изучению активации определенных генов, кодирующих дефензины, и обсуждению предположений об их роли в возникновении устойчивости к определенным факторам (39).
Использование методов секвенирования и биоинформатики для исследования дефензинов. Защитные свойства, проявляемые дефензинами, делают их перспективными целевыми признаками при селекции культур, устойчивых к различным патогенам. Также перспективным представляется поиск новых форм активности дефензинов у дикорастущих растительных объектов, не относящихся к модельным. С развитием методов NGS (next generation sequencing) секвенирования стало возможным быстро выявлять последовательности геномов и (или) транскрипто-мов многих видов растений, в том числе немодельных (58, 59). При этом становится актуальной проблема аннотации и функциональной характеристики полученных при секвенировании последовательностей (60). Из-за высокой степени их дивергенции у дефензинов одной группы и ограниченного числа пептидов, выявленных экспериментально, наиболее распространенные инструменты поиска схожих последовательностей и технологии выявления генов чаще всего не обеспечивают надежную идентификацию в случае столь коротких пептидов (61). Для достоверного выявления генов, кодирующих короткие пептиды, разрабатываются специальные наборы алгоритмов. К их числу относится SPADA (Small Peptide Alignment Discovery Application). Алгоритм работы SPADA заключается в нахождении всех связанных паралогичных генов в пределах генного семейства, множественном выравнивании последовательностей и построении предсказательной модели для поиска. В SPADA применяются несколько подходов к поиску схожих последовательностей — BLAST и HMMER , также инструменты для предсказания генов, например Augustus , что значительно повышает эффективность аннотации генных семейств с большим числом членов. Показано, что с помощью SPADA почти все представители семейств генов, кодирующих короткие богатые цистеином белки у A. thaliana и M. truncatula, аннотируются эффективнее, чем в других программах (61). Таким образом, SPADA может успешно использоваться для выявления генов, кодирующих дефензины, в том числе у немодельных объектов.
С появлением возможности в короткие сроки получать информацию о геноме и (или) транскриптоме изучаемого объекта, а также с разработкой алгоритмов для правильной аннотации число последовательностей, идентифицированных как дефензины, неуклонно растет. При исследовании конкретных групп пептидов необходимо изучать их биологическую активность, однако полный анализ активности дефензинов, выявленных традиционными методами, практически никогда не проводился — изучались лишь отдельные аспекты действия против определенных грибов или бактерий. При анализе большого числа генов дефензинов методы вычис-7
лительной биологии могут использоваться для предсказания активности вновь выявляемых пептидов и обнаружения консервативных аминокислот. Так, подход, основанный на множественном выравнивании последовательностей и последующем кластерном анализе, позволил разделить дефензины разных видов, представленные в базе данных NCBI (National Center for Biotechnology Information, США), на 18 групп (29). Оказалось, что дефензины из одной группы обладают сходной функциональной активностью. Стоит отметить, что из-за низкого подобия последовательностей сравнительный анализ дефензинов, проведенный классическим прямым сравнением последовательностей, может иметь крайне низкую эффективность. Для более корректного выравнивания таких сложных наборов последовательностей следует применять специальные методы, основанные на баркоди-ровании консервативных участков последовательностей цистеинов (62). Это позволяет проводить сравнительный анализ даже сильно различающихся последовательностей, включая дефензины.
Представление результатов разных научных коллективов в базах данных со свободным доступом повышает эффективность исследований. Так, с использованием экспрессионных атласов становится возможным выявление профиля экспрессии определенных групп генов. Это позволяет судить об органо- или тканеспецифичности экспрессии конкретного гена и вовлеченности его в те или иные процессы (63, 64).
Таким образом, дефензины растений (группа пептидов, проявляющих антифунгальную и бактерицидную активность, а также участвующих в разнообразных ответах на абиотические воздействия) крайне важны в связи с проблемой получения устойчивых сортов сельскохозяйственных культур. Представители этой группы пептидов характеризуются крайне низким сходством первичной структуры (за исключением консервативно расположенных цистеиновых остатков), однако современные методы компьютерной биологии в сочетании с технологиями секвенирования ускоряют выявление и анализ последовательностей, кодирующих дефензины, в том числе у немодельных объектов биологии растений.
Список литературы Дефензины растений: биологическая роль, механизмы действия и методы анализа
- Goyal R.K., Mattoo A.K. Multitasking antimicrobial peptides in plant development and host defense against biotic/abiotic stress. Plant Sci., 2014, 228: 135-149 ( ) DOI: 10.1016/j.plantsci.2014.05.012
- Tam J.P., Wang S., Wong K.H., Tan W.L. Antimicrobial peptides from plants. Pharmaceuticals, 2015, 8(4): 711-757 ( ) DOI: 10.3390/ph8040711
- Egorov T.A., Odintsova T.I. Defense peptides of plant immune system. Russian Journal of Bioorganic Chemistry, 2012, 38(1): 1-9 ( ) DOI: 10.1134/S1068162012010062
- Thomma B.P., Cammue B.P., Thevissen K. Plant defensins. Planta, 2002, 216(2): 193-202 ( ) DOI: 10.1007/s00425-002-0902-6
- Stotz H.U., Thomson J., Wang Y. Plant defensins: defense, development and application. Plant Signal. Behav., 2009, 4(11): 1010-1012 ( ) DOI: 10.4161/psb.4.11.9755
- Комалетдинова Ф.М. Роль растительных дефензинов в многокомпонентной системе защиты растений. Сельскохозяйственная биология, 2009, 5: 8-16.
- Bruix M., Jimenez M.A., Santoro J., Gonzalez C., Colilla F.J., Mendez E., Rico M. Solution structure of gamma 1-H and gamma 1-P thionins from barley and wheat endosperm determined by proton NMR: a structural motif common to toxic arthropod proteins. Biochemistry, 1993, 32(2): 715-724 ( ) DOI: 10.1021/bi00053a041
- Fant F., Vranken W., Broekaert W., Borremans F. Determination of the three-dimensional solution structure of Raphanus sativus antifungal protein 1 by 1 H NMR. J. Mol. Biol., 1998, 279(1): 257-270 ( ) DOI: 10.2210/pdb1ayj/pdb
- Do H.M., Lee S.C., Jung H.W., Sohn K.H., Hwang B.K. Differential expression and in situ localization of a pepper defensin (CADEF1) gene in response to pathogen infection, abiotic elicitors and environmental stresses in Capsicum annuum. Plant Sci., 2004, 166(5): 1297-1305 ( ) DOI: 10.1016/j.plantsci.2004.01.008
- Huang G.-J., Lai H.-C., Chang Y.-S., Sheu M.-J., Lu T.-L., Huang S.-S., Lin Y.-H. Antimicrobial, dehydroascorbate reductase, and monodehydroascorbate reductase activities of defensin from sweet potato storage roots. J. Agric. Food Chem., 2008, 56(9): 2989-2995 ( ) DOI: 10.1021/jf072994j
- Lay F.T., Brugliera F., Anderson M.A. Isolation and properties of floral defensins from ornamental tobacco and petunia. Plant Physiol., 2003, 131(3): 1283-1293 ( ) DOI: 10.1104/pp.102.016626
- Urdangarín M.C., Norero N.S., Broekaert W.F., de la Canal L. A defensin gene expressed in sunflower inflorescence. Plant Physiol. Biochem., 2000, 38(3): 253-258 ( ) DOI: 10.1016/S0981-9428(00)00737-3
- Maróti G., Downie J.A., Kondorosi É. Plant cysteine-rich peptides that inhibit pathogen growth and control rhizobial differentiation in legume nodules. Curr. Opin. Plant Biol., 2015, 26: 57-63 ( ) DOI: 10.1016/j.pbi.2015.05.031
- Chiang C.C., Hadwiger L.A. The Fusarium solani-Induced expression of a pea gene family encoding high cysteine content proteins. Mol. Plant-Microbe Interact., 1991, 4(4): 324-331 ( ) DOI: 10.1094/MPMI-4-324
- Lacerda A., Vasconcelos É.A.R., Pelegrini P.B., Grossi-de-Sa M.F. Antifungal defensins and their role in plant defense. Front. Microbiol., 2014, 5: 116 ( ) DOI: 10.3389/fmicb.2014.00116
- Franco O.L., Murad A.M., Leite J.R., Mendes P.A., Prates M.V., Bloch C. Identification of a cowpea γ-thionin with bactericidal activity. FEBS J., 2006, 273(15): 3489-3497 ( ) DOI: 10.1111/j.1742-4658.2006.05349.x
- Zhang Y., Lewis K. Fabatins: new antimicrobial plant peptides. FEMS Microbiol. Lett., 1997, 149(1): 59-64 ( ) DOI: 10.1111/j.1574-6968.1997.tb10308.x
- Melo F.R., Rigden D.J., Franco O.L., Mello L.V., Ary M.B., Grossi de Sá M.F., Bloch C. Inhibition of trypsin by cowpea thionin: Characterization, molecular modeling, and docking. Proteins: Struct., Funct., Bioinf., 2002, 48(2): 311-319 ( ) DOI: 10.1002/prot.10142
- Mirouze M., Sels J., Richard O., Czernic P., Loubet S., Jacquier A., François I.E.J.A., Cammue B.P.A., Lebrun M., Berthomieu P., Marquès L. A putative novel role for plant defensins: a defensin from the zinc hyper-accumulating plant, Arabidopsis halleri, confers zinc tolerance. Plant J., 2006, 47(3): 329-342 ( ) DOI: 10.1111/j.1365-313X.2006.02788.x
- Gaudet D.A., Laroche A., Frick M., Huel R., Puchalski B. Cold induced expression of plant defensin and lipid transfer protein transcripts in winter wheat. Physiologia Plantarum, 2003, 117(2): 195-205 ( ) DOI: 10.1034/j.1399-3054.2003.00041.x
- Koike M., Okamoto T., Tsuda S., Imai R. A novel plant defensin-like gene of winter wheat is specifically induced during cold acclimation. BBRC, 2002, 298(1): 46-53 ( ) DOI: 10.1016/S0006-291X(02)02391-4
- Stolf-Moreira R., Medri M.E., Neumaier N., Lemos N.G., Brogin R.L., Marcelino F.C., De Oliveira M.C.N., Farias J.R.B., Abdelnoor R.V., Nepomuceno A.L. Cloning and quantitative expression analysis of drought-induced genes in soybean. Genet. Mol. Res., 2010, 9(2): 858-867 ( ) DOI: 10.4238/vol9-2gmr701
- Sui J., Jiang D., Zhang D., Song X., Wang J., Zhao M., Qiao L. The salinity responsive mechanism of a hydroxyproline-tolerant mutant of peanut based on digital gene expression profiling analysis. PloS ONE, 2016, 11(9): e0162556 ( ) DOI: 10.1371/journal.pone.0162556
- Nishiyama R., Le D.T., Watanabe Y., Matsui A., Tanaka M., Seki M., Yamaguchi-Shinozaki K., Shinozaki K., Tran L.S.P. Transcriptome analyses of a salt-tolerant cytokinin-deficient mutant reveal differential regulation of salt stress response by cytokinin deficiency. PLoS ONE, 2012, 7(2): e32124 ( ) DOI: 10.1371/journal.pone.0032124
- Okuda S., Tsutsui H., Shiina K., Sprunck S., Takeuchi H., Yui R., Kasahara R.D., Hamamura Y., Mizukami A., Susaki D., Kawano N., Sakakibara T., Namoki S., Itoh K., Otsuka K., Matsuzaki M., Nozaki H., Kuroiwa T., Nakano A., Kanaoka M.M., Dresselhaus T., Sasaki N., Higashiyama T. Defensin-like polypeptide LUREs are pollen tube attractants secreted from synergid cells. Nature, 2009, 458(7236): 357-361 ( ) DOI: 10.1038/nature07882
- Shafee T.M., Lay F.T., Hulett M.D., Anderson M.A. The defensins consist of two independent, convergent protein superfamilies. Mol. Biol. Evol., 2016, 33(9): 2345-2356 ( ) DOI: 10.1093/molbev/msw106
- Almeida M.S., Cabral K.M., Kurtenbach E., Almeida F.C., Valente A.P. Solution structure of Pisum sativum defensin 1 by high resolution NMR: plant defensins, identical backbone with different mechanisms of action. J. Mol. Biol., 2002, 315(4): 749-757 ( ) DOI: 10.1006/jmbi.2001.5252
- Broekaert W.F., Terras F.R., Cammue B.P., Osborn R.W. Plant defensins: novel antimicrobial peptides as components of the host defense system. Plant Physiol., 1995, 108(4): 1353-1358 ( ) DOI: 10.1104/pp.108.4.1353
- van der Weerden N.L., Anderson M.A. Plant defensins: common fold, multiple functions. Fungal Biol. Rev., 2013, 26(4): 121-131 ( ) DOI: 10.1016/j.fbr.2012.08.004
- Méndez E., Rocher A., Calero M., Girbés T., Citores L., Soriano F. Primary structure of omega-hordothionin, a member of a novel family of thionins from barley endosperm, and its inhibition of protein synthesis in eukaryotic and prokaryotic cell-free systems. Eur. J. Biochem., 1996, 239(1): 67-73 ( ) DOI: 10.1111/j.1432-1033.1996.0067u.x
- Gao A.-G., Hakimi S.M., Mittanck C.A., Wu Y., Woerner B.M., Stark D.M., Shah D.M., Liang J., Rommens C.M. Fungal pathogen protection in potato by expression of a plant defensin peptide. Nat. Biotechnol., 2000, 18(12): 1307-1310 ( ) DOI: 10.1038/82436
- Terras F.R., Eggermont K., Kovaleva V., Raikhel N.V., Osborn R.W., Kester A., Rees S.B., Torrekens S., Van Leuven F., Vanderleyden J. Small cysteine-rich antifungal proteins from radish: their role in host defense. Plant Cell, 1995, 7(5): 573-588 ( ) DOI: 10.1105/tpc.7.5.573
- Spelbrink R.G., Dilmac N., Allen A., Smith T.J., Shah D.M., Hockerman G.H. Differential antifungal and calcium channel-blocking activity among structurally related plant defensins. Plant Physiol., 2004, 135(4): 2055-2067 ( ) DOI: 10.1104/pp.104.040873
- Janssen B.J., Schirra H.J., Lay F.T., Anderson M.A., Craik D.J. Structure of Petunia hybrida defensin 1, a novel plant defensin with five disulfide bonds. Biochemistry, 2003, 42(27): 8214-8222 ( ) DOI: 10.1021/bi034379o
- Lobo D.S., Pereira I.B., Fragel-Madeira L., Medeiros L.N., Cabral L.M., Faria J., Bellio M., Campos R.C., Linden R., Kurtenbach E. Antifungal Pisum sativum defensin 1 interacts with Neurospora crassa cyclin F related to the cell cycle. Biochemistry, 2007, 46(4): 987-996 ( ) DOI: 10.1021/bi061441j
- Stolf-Moreira R., Medri M.E., Neumaier N., Lemos N.G., Pimenta J.A., Tobita S., Brogin R.L., Marcelino-Guimarães F.C., Oliveira M.C.N., Farias J.R.B., Abdelnoor R.V., Nepomuceno A.L. Soybean physiology and gene expression during drought. Genet. Mol. Res., 2010, 9(4): 1946-1956 ( ) DOI: 10.4238/vol9-4gmr851
- Mith O., Benhamdi A., Castillo T., Berge M., MacDiarmid C.W., Steffen J., Eide D.J., Perrier V., Subileau M., Gosti F., Berthomieu P., Marques L. The antifungal plant defensin AhPDF1.1b is a beneficial factor involved in adaptive response to zinc overload when it is expressed in yeast cells. MicrobiologyOpen, 2015, 4(3): 409-422 ( ) DOI: 10.1002/mbo3.248
- Oomen R.J., Séveno-Carpentier E., Ricodeau N., Bournaud C., Con-éjéro G., Paris N., Berthomieu P., Marquès L. Plant defensin AhPDF1.1 is not secreted in leaves but it accumulates in intracellular compartments. New Phytol., 2011, 192(1): 140-150 ( ) DOI: 10.1111/j.1469-8137.2011.03792.x
- Shahzad Z., Ranwez V., Fizames C., Marquès L., Martret B., Alassimone J., Godé C., Lacombe E., Castillo T., Saumitou-Laprade P., Berthomieu P., Gosti F. Plant Defensin type 1 (PDF1): protein promiscuity and expression variation within the Arabidopsis genus shed light on zinc tolerance acquisition in Arabidopsis halleri. New Phytol., 2013, 200(3): 820-833 ( ) DOI: 10.1111/nph.12396
- Nguyen N.N., Ranwez V., Vile D., Soulié M.-C., Dellagi A., Expert D., Gosti F. Evolutionary tinkering of the expression of PDF1s suggests their joint effect on zinc tolerance and the response to pathogen attack. Front. Plant Sci., 2014, 5: 70 ( ) DOI: 10.3389/fpls.2014.00070
- Ahmed N.U., Park J.-I., Jung H.-J., Seo M.-S., Kumar T.S., Lee I.-H., Nou I.-S. Identification and characterization of stress resistance related genes of Brassica rapa. Biotechnol. Lett., 2012, 34(5): 979-987 ( ) DOI: 10.1007/s10529-012-0860-4
- Cabot C., Gallego B., Martos S., Barceló J., Poschenrieder C. Signal cross talk in Arabidopsis exposed to cadmium, silicon, and Botrytis cinerea. Planta, 2013, 237(1): 337-349 ( ) DOI: 10.1007/s00425-012-1779-7
- Bahramnejad B., Erickson L.R., Chuthamat A., Goodwin P.H. Differential expression of eight defensin genes of N. benthamiana following biotic stress, wounding, ethylene, and benzothiadiazole treatments. Plant Cell Rep., 2009, 28(4): 703-717 ( ) DOI: 10.1007/s00299-009-0672-8
- Wilmes M., Cammue B.P., Sahl H.-G., Thevissen K. Antibiotic activities of host defense peptides: more to it than lipid bilayer perturbation. Nat. Prod. Rep., 2011, 28(8): 1350-1358 ( ) DOI: 10.1039/c1np00022e
- Poon I.K., Baxter A.A., Lay F.T., Mills G.D., Adda C.G., Payne J.A., Phan T.K., Ryan G.F., White J.A., Veneer P.K., van der Weerden N., Anderson M.A., Kvansakul M., Hulett M.D. Phosphoinositide-mediated oligomerization of a defensin induces cell lysis. Elife, 2014, 3: e01808 ( ) DOI: 10.7554/eLife.01808
- Terras F.R., Schoofs H.M., De Bolle M.F., Van Leuven F., Rees S.B., Van-derleyden J., Cammue B.P., Broekaert W.F. Analysis of two novel classes of plant antifungal proteins from radish (Raphanus sativus L.) seeds. J. Biol. Chem., 1992, 267(22): 15301-15309.
- Thevissen K., Cammue B.P., Lemaire K., Winderickx J., Dickson R.C., Lester R.L., Ferket K.K., Van Even F., Parret A.H., Broekaert W.F. A gene encoding a sphingolipid biosynthesis enzyme determines the sensitivity of Saccharomyces cerevisiae to an antifungal plant defensin from dahlia (Dahlia merckii). PNAS, 2000, 97(17): 9531-9536 ( ) DOI: 10.1073/pnas.160077797
- Thevissen K., Warnecke D.C., François I.E., Leipelt M., Heinz E., Ott C., Zähringer U., Thomma B.P., Ferket K.K., Cammue B.P. Defensins from insects and plants interact with fungal glucosylceramides. J. Biol. Chem., 2004, 279(6): 3900-3905 ( ) DOI: 10.1074/jbc.M311165200
- Thevissen K., Ferket K.K., François I.E., Cammue B.P. Interactions of antifungal plant defensins with fungal membrane components. Peptides, 2003, 24(11): 1705-1712 ( ) DOI: 10.1016/j.peptides.2003.09.014
- Thevissen K., de Mello Tavares P., Xu D., Blankenship J., Vandenbosch D., Idkowiak-Baldys J., Govaert G., Bink A., Rozental S., de Groot P.W.J., Davis T.R., Kumamoto C.A., Vargas G., Nimrichter L., Coenye T., Mitchell A., Roemer T., Hannun Y.A., Cammue B.P.A. The plant defensin RsAFP2 induces cell wall stress, septin mislocalization and accumulation of ceramides in Candida albicans. Mol. Microbiol., 2012, 84(1): 166-180 ( ) DOI: 10.1111/j.1365-2958.2012.08017.x
- Ramamoorthy V., Cahoon E.B., Li J., Thokala M., Minto R.E., Shah D.M. Glucosylceramide synthase is essential for alfalfa defensin-mediated growth inhibition but not for pathogenicity of Fusarium graminearum. Mol. Microbiol., 2007, 66(3): 771-786 ( ) DOI: 10.1111/j.1365-2958.2007.05955.x
- Vriens K., Cammue B., Thevissen K. Antifungal plant defensins: mechanisms of action and production. Molecules, 2014, 19(8): 12280-12303 ( ) DOI: 10.3390/molecules190812280
- Sagaram U.S., El-Mounadi K., Buchko G.W., Berg H.R., Kaur J., Pandurangi R.S., Smith T.J., Shah D.M. Structural and functional studies of a phosphatidic acid-binding antifungal plant defensin MtDef4: identification of an RGFRRR motif governing fungal cell entry. PLoS ONE, 2013, 8(12): e82485 ( ) DOI: 10.1371/journal.pone.0082485
- Aerts A.M., François I.E., Bammens L., Cammue B., Smets B., Winderickx J., Accardo S., De Vos D.E., Thevissen K. Level of M (IP) 2C sphingolipid affects plant defensin sensitivity, oxidative stress resistance and chronological life-span in yeast. FEBS Lett., 2006, 580(7): 1903-1907 ( ) DOI: 10.1016/j.febslet.2006.02.061
- van der Weerden N.L., Lay F.T., Anderson M.A. The plant defensin, NaD1, enters the cytoplasm of Fusarium oxysporum hyphae. J. Biol. Chem., 2008, 283(21): 14445-14452 ( ) DOI: 10.1074/jbc.M709867200
- Mello E.O., Ribeiro S.F., Carvalho A.O., Santos I.S., Da Cunha M., Santa-Catarina C., Gomes V.M. Antifungal activity of PvD1 defensin involves plasma membrane permeabilization, inhibition of medium acidification, and induction of ROS in fungi cells. Curr. Microbiol., 2011, 62(4): 1209-1217 ( ) DOI: 10.1007/s00284-010-9847-3
- Aerts A.M., Bammens L., Govaert G., Carmona-Gutierrez D., Madeo F., Cammue B., Thevissen K. The antifungal plant defensin HsAFP1 from Heuchera sanguinea induces apoptosis in Candida albicans. Front. Microbiol., 2011, 2: 47 ( ) DOI: 10.3389/fmicb.2011.00047
- Zhukov V.A., Kulaeva O.A., Zhernakov A.I., Tikhonovich I.A. «Next generation sequencing» for studying transcriptome profiles of tissues and organs of garden pea (Pisum sativum L.) (review). Agricultural Biology, 2015, 50(3): 278-287 ( ) DOI: 10.15389/agrobiology.2015.3.278eng
- Zhukov V.A., Zhernakov A.I., Kulaeva O.A., Ershov N.I., Borisov A.Y., Tik-honovich I.A. De novo assembly of the pea (Pisum sativum L.) nodule transcriptome. Int. J. Genomics, 2015, 2015: 695947 ( ) DOI: 10.1155/2015/695947
- Conesa A., Madrigal P., Tarazona S., Gomez-Cabrero D., Cervera A., McPherson A., Szcześniak M.W., Gaffney D.J., Elo L.L., Zhang X., Mortazavi A. A survey of best practices for RNA-seq data analysis. Genome Biology, 2016, 17: 13 ( ) DOI: 10.1186/s13059-016-0881-8
- Zhou P., Silverstein K.A., Gao L., Walton J.D., Nallu S., Guhlin J., Young N.D. Detecting small plant peptides using SPADA (small peptide alignment discovery application). BMC Bioinformatics, 2013, 14: 335 ( ) DOI: 10.1186/1471-2105-14-335
- Shafee T.M., Robinson A.J., Weerden N., Anderson M.A. Structural homology guided alignment of cysteine rich proteins. SpringerPlus, 2016, 5: 27 ( ) DOI: 10.1186/s40064-015-1609-z
- Alves-Carvalho S., Aubert G., Carrere S., Cruaud C., Brochot A.-L., Jacquin F., Klein A., Martin C., Boucherot K., Kreplak J., da Silva C., Moreau S., Gamas P., Wincker P., Gouzy J., Burstin J. Full-length de novo assembly of RNA-seq data in pea (Pisum sativum L.) provides a gene expression atlas and gives insights into root nodulation in this species. Plant J., 2015, 84(1): 1-19 ( ) DOI: 10.1111/tpj.12967
- He J., Benedito V.A., Wang M., Murray J.D., Zhao P.X., Tang Y., Udvardi M.K. The Medicago truncatula gene expression atlas web server. BMC Bioinformatics, 2009, 10: 441 ( ) DOI: 10.1186/1471-2105-10-441


