Деструктор бифенила Rhodococcus sp. VR43-1: выделение, молекулярно-биологическая характеристика
Автор: Воронина Анна Олеговна, Плотникова Елена Генриховна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 1, 2019 года.
Бесплатный доступ
В микробном сообществе донных отложений р. Чапаевки, протекающей по территории предприятия «Средне-Волжский завод химикатов» (г. Чапаевск, Самарская обл.), выявлены генетические структуры (bphA1-гены), кодирующие бифенил 2,3-диоксигеназу (БДО), ключевой фермент, ответственный за деструкцию бифенила и полихлорированных бифенилов (ПХБ, токсичные трудноразлагаемые поллютанты). Обнаружено сходство на уровне 95.8-99.7% исследуемых нуклеотидных последовательностей с генами α-субъединиц БДО и других диоксигеназ (подсемейства бензол/толуол ДО) штаммов рода Rhodococcus. С использованием метода накопительного культивирования из образца донных отложений был выделен активный штамм-деструктор бифенила, обозначенный VR43-1. При сравнению нуклеотидных последовательностей генов 16S рРНК с гомологичными последовательностями из базы данных EzBioCloud, штамм VR43-1 имел наибольший уровень сходства (100%) с типовым штаммом R. wratislaviensis NBRC 100605T. Сравнение гена bphA1 штамма VR43-1 с bphA1-генами активных деструкторов бифенила/ПХБ R. jostii RHA1 и штаммов R. wratislaviensis Р13 и Ch628 показало их сходство на уровне 99.1 и 100%, соответственно.
Бифенил, полихлорированные бифенилы, бактерии-деструкторы, bpha1-гены, молекулярное клонирование генов
Короткий адрес: https://sciup.org/147227062
IDR: 147227062 | УДК: 579.222:577.152.199.2 | DOI: 10.17072/1994-9952-2019-1-48-55
Текст научной статьи Деструктор бифенила Rhodococcus sp. VR43-1: выделение, молекулярно-биологическая характеристика
стойких органических загрязнителей (СОЗ) Программой ООН по окружающей среде (ЮНЕП) (URL: http//chm.pops.int). Многочисленные исследования показали, что ПХБ негативно влияют на здоровье человека. Так, при длительном воздействии ПХБ повышается уровень возникновения онкологических заболеваний, происходит нарушение работы жизненно важных систем организма, таких как эндокринная, нервная, репродуктивная и иммунная [Murugan and Vasudeva, 2018].
Разложение ПХБ в природе осуществляется анаэробными и аэробными микроорганизмами [Liang et al., 2014]. Выделено и охарактеризовано большое количество аэробных бактерий-деструкторов бифенила (в том числе штаммов рода Rhodococcus ), способных к утилизации или частичной трансформации ПХБ [Warren et al., 2004; Yang et al., 2004; Taguchi et al., 2007; Xiong et al., 2011]. Аэробный процесс разложения бифенила и ПХБ у бактерий осуществляется одними и теми же ферментными системами и включает четыре этапа [Furukawa, 2000]. Первая реакция – включение двух гидроксильных групп в ароматическое кольцо бифенила, происходит под действием фермента бифенил 2,3-диоксигеназы (БДО) и считается определяющей стадией разложения бифенила/ПХБ [Pieper, Seeger, 2008]. Ген bphA1, кодирующий α-субъединицу БДО, играет ключевую роль в распознавании и связывании субстрата [Piper, 2005] и является важным маркером при исследовании биодеградационного потенциала бактерий [Шум-кова и др., 2015].
В последние годы восстановление загрязнённых территорий с использованием биологического потенциала бактерий является одним из наиболее оптимальных способов утилизации стойких токсичных соединений, как с экологической, так и с экономической точек зрения [Егорова и др., 2014; Sharma et al., 2018]. Поэтому постоянно проводится поиск новых, перспективных для использования в биотехнологических целях, активных бактерий-деструкторов СОЗ.
В течение нескольких десятилетий, до конца 1980-х гг., на территории г. Чапаевска (Самарская обл.) на химическом предприятии «СреднеВолжский завод химикатов» (ОАО «СВЗХ») осуществлялся выпуск различных хлорорганических соединений, в том числе 1,2,4-трихлорбензола, который далее использовался для изготовления содержащей ПХБ технической смеси – «Совтол-10» [Федоров, Оноприенко, 1993]. В результате интенсивной производственной деятельности произошло загрязнение территории предприятия и р. Чапаев-ки, протекающей вдоль территории завода, токсичными поллютантами [Назаров и др., 2016].
Цель работы – определить наличие ключевых генов разложения бифенила (bphA1-генов) в микробном сообществе донных отложений р. Чапаевки (территория химического предприятия ОАО «СВЗХ», г. Чапаевск) и выделить бактерии- деструкторы бифенила (полихлорированных бифенилов).
Материалы и методы исследования
Объекты исследования
В качестве материала для исследований были взяты четыре образца донных отложений р. Чапа-евки, протекающей по территории химического предприятия ОАО «СВЗХ» (Самарская обл.). Пробы отбирались в стерильные пробирки объемом 50 мл и транспортировались к месту исследования (ИЭГМ УрО РАН, г. Пермь) в контейнере при температуре +4°С. Методом хромато-масс-спектро-метрии в образце № 4 были обнаружены хлорированные бифенилы в количестве 0.213 мг/кг, а также эфиры фталевой кислоты (дибутилфталат, диоктилфталат), хлорсодержащие органические загрязнители линдан, алохлор, триалат и имеющие ароматическое строение (хлорбензолы и ДДТ) [Назаров и др., 2016].
Выделение ДНК, ПЦР и клонирование bphA1 -генов
Выделение тотальной ДНК из образцов донных отложений проводили с использованием коммерческого набора реактивов «MP Biomedicals» (США). Концентрацию ДНК определяли на приборе QubitTM Fluorometer, («Invitrogen», США) при применении реактивов производителя.
С матрицы тотальной ДНК проводили амплификацию bphA1- генов с праймерами BPHD-f3 и BPHD-r1 [Iwai et al., 2010], специфичными к гену α-субъединицы БДО, на приборе MyCycler («Bio-Rad Laboratories», США) согласно протоколу. Клонирование ПЦР-фрагменты bphA1- генов и последующий отбор рекомбинантных клонов осуществляли, как описано в статье [Шумкова и др., 2015].
Анализ полиморфизма длин рестрикционных фрагментов bphA1- генов (ПДРФ-анализ) осуществляли с использованием эндонуклеазы рестрикции HhaI («Fermentas», Литва).
Для подготовки к секвенированию клонированных в составе вектора pTZ57R/T фрагментов ДНК проводили амплификацию вставки с использованием стандартных праймеров M13 (М13F 5’-GTTTTCCCAGTCACGAC-3’ и М13R 5’-CAGGAAACAGCTATGAC-3’), для которых имеются сайты связывания в векторе pTZ57R по обеим сторонам от полилинкера. Описание секвенирования и анализа клонированных последовательностей bphA1- генов приведены ниже.
Накопительное культивирование, выделение штаммов-деструкторов бифенила
Для получения накопительной культуры в колбы Эрленмейера объемом 250 мл добавляли 50 мл минеральной среды К1 [Maltseva et al., 1999], об- разец весом 0.5 г и бифенил (1 г/л) в качестве единственного источника углерода и энергии. Культивирование проводилось 1 месяц при температуре 28°С с аэрацией на термошейкере (120 об./мин). Полученную суспензию высевали на ага-ризованную среду К1, бифенил добавляли на крышку перевернутой чашки Петри и культивировали при температуре 28°С до появления колоний. Чистоту культур проверяли при выращивании на богатой среде LB [Маниатис и др., 1984].
Морфо-физиологические признаки бактериальной культуры
Морфологию выделенной чистой культуры изучали по общепринятым методикам [Методы общей бактериологии, 1983]. Рост бактериальной культуры при изменении осмолярности среды изучали как на агари-зованной минеральной среде Раймонда [Розанова, Назина, 1982] с добавлением бифенила в качестве единственного источника углерода и энергии, так и на богатой среде Раймонда (в состав среды входит дрожжевой экстракт (2.5 г/л), триптон (5 г/л)), при концентрациях NaCl (г/л): 0, 30, 50, 70. Культивирование осуществляли в термостате при 28 ° С в течение 7 сут. Оценку роста бактериального штамма проводили при различных температурных режимах (+4 ° С, +10 ° С, +28 ° С, +37 ° С и +45 ° С), на агаризованных средах: К1 (минеральная среда) с добавлением бифенила и LB (богатая среда). Рост колоний учитывали на 7-е сут.
Способность исследуемого штамма разлагать ароматические соединения
Способность разлагать ароматические соединения оценивали путем культивирования в жидкой среде K1 на термошейкере (120 об./мин) и/или на агаризованной среде К1 при 28°С. В качестве источника углерода и энергии использовали следующие субстраты: толуол и фенол – помещали на крышку перевернутой чашки Петри; бифенил, нафталин, фенантрен, а также орто -фталевую, салициловую, пара -оксибензойную, бензойную, про-токатеховую кислоты (в виде натриевых солей в пересчете на количество кислоты), вносили в среду в концентрации 0.5 г/л. Бактериальный рост в жидкой среде на вышеперечисленных субстратах оценивали путем измерения оптической плотности культуры на спектрофотометре UV-Visible BioSpec-mini («Shimadzu», Япония) при λ max 600 нм, на агаризованной среде – оценивали рост бактериальных колоний.
Секвенирование и анализ генов 16S рРНК и bphA1
Выделение ДНК из исследуемого изолята осуществляли общепринятым методом [Short protocols in molecular biology, 1995]. Идентификацию клеточной культуры проводили с использованием стандартных бактериальных праймеров 27F и 1492R [Tiirola et al., 2002] для амплификации гена 16S рРНК. Амплификацию bphA1-генов проводили с праймерами BPHD-f3 и BPHD-r1 [Iwai et al., 2010]. Определение и анализ нуклеотидных последовательностей амплифициро-ванных генов осуществляли с применением набора реактивов Big Dye Terminator Cycle Sequencing Kit v. 3.1 («Applied Biosystems», США) на проборе Genetic Analyser 3500XL («Applied Biosystems», США), в лаборатории молекулярной биологии и генетики при кафедре ботаники и генетики растений, ПГНИУ). Анализ нуклеотидных последовательностей осуществляли с использованием программ Sequence Scanner v 2.0 и MEGA 6.0 (URL: . Поиск гомологичных последовательностей генов bphA1 и 16S рРНК проводили в международных базах данных GenBank [http:/] и EzBioCloud [] соответственно.
Результаты и их обсуждение
Разнообразие bphA1 -генов в микробном сообществе донных отложений р. Чапаевки
Методом накопительного культивирования донных отложений (образца № 4) на бифениле была получена ассоциация микроорганизмов, из которой была выделена тотальная ДНК. С использованием праймеров к гену bphA1 [Iwai et al., 2010] была осуществлена амплификация и получен ПЦР-продукт размером ~500 п.н., который был клонирован в составе вектора pTZ57R в клетках E. coli . Создана библиотека bphA1 -генов, включающая 88 клонов. Методом ПДРФ-анализа у представителей каждой геномогруппы (клоны Ch9, Ch14) были определены нуклеотидные последовательности клонированных участков и проведён филогенетический анализ (таблица, рис. 1).
При анализе клонов Ch9 и Ch14 обнаружено высокое сходство (98.9–99.2%) с генами (bphA1) α-субъединиц бифенил 2,3-диоксигеназы (БДО, бен-зол/толуол подсемейство ДО (Б/Т ДО)) некульти-вируемых бактериальных клонов, а также с bphA1-подобными генами других Б/Т ДО штаммов рода Rhodococcus (таблица). Полученные данные указывают на присутствие в микробном сообществе донных отложений р. Чапаевки генов bphA1 и bphA1-подобных генов. Стоит отметить, что при сравнении нуклеотидных последовательностей двух исследуемых клонов (Ch9 и Ch14) с гомологичными последовательностями в международной базе данных GenBank (URL: http:/, процентное сходство и перечень представителей бактериальных штаммов идентичны (таблица), в то же время между собой участки сравниваемых нуклеотидных последовательностей исследуемых клонов являются гетерогенными.
Наличие в общей ДНК, выделенной из полученной накопительной культуры (НК, образец дон- ных отложений р. Чапаевки), нуклеотидных последовательностей, гомологичных генам α-субъединицы БДО родококов, указывает на присутствие в исследуемом микробном сообществе деструкторов бифенила рода Rhodococcus.
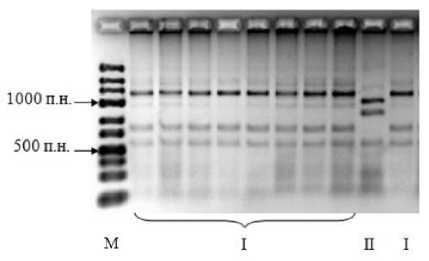
Рис. 1 . Электрофореграмма рестрикционных фрагментов генов bphA1 после обработки эндонуклеазой Hha I (I, II – геномогруппы).
М – маркер молекулярных масс O’GeneRuler™ 100 bp Plus DNA Ladder (“Fermentas”, Литва)
Выделение и характеристика штамма-деструктора бифенила
Из полученной НК был выделен бактериальный штамм (обозначенный VR43-1), способный использовать бифенил в качестве единственного источника углерода и энергии. При выращивании штамма на агаризованной полноценной среде LB образовывались округлые, матовые, светлобежевые колонии размером 1–3 мм. Клетки штамма грамположительные, неподвижные, не образуют спор, каталазоположительные. Штамм VR43-1 способен к росту на агаризованных средах (среда LB и минеральная среда К1 с добавлением бифенила в качестве субстрата) в интервале температур от +4ºС до +37ºС. Ростовые эксперименты при изменении осмолярности среды показали, что штамм VR43-1 растет (как на среде LB, так и на минеральной среде Раймонд с добавлением бифенила) при концентрации NaCl до 70 г/л.
Сравнение клонированных нуклеотидных последовательностей фрагментов генов гидроксилирующих диоксигеназ ( bphA1- генов) с гомологичными последовательностями из базы данных GenBank
|
Клон (геномогруп-па)/ штамм, размер анализируемого фрагмента ДНК |
Гомологичные гены в GenBank |
Номера сравниваемых нуклеотидных последовательностей в GenBank |
Сходство, % |
Ссылка |
Место выделения bphA1 -генов и/или бактерий-деструкторов |
|
Клон Ch9 (I), 387 п.н. / Клон Ch14 (II), |
Гены α-субъединицы бифенил 2,3- диоксигеназы ( bphA1 ); клоны HS7/HS8/NS7/NS8, некультивируемые бактерии |
JN675903.1/ JN675902.1/ JN675900.1/ JN675901.1 |
99.2-98.9 / 99.1-98.6 / < 92 |
н.д.* |
Нет данных |
|
373 п.н. / Штамм VR43-1, 364 п.н. |
Ген α-субъединицы бифенил 2,3-диоксигеназы ( bphA1 ); Rhodococcus wratislavien-sis P13 |
KP972446.1 |
99.7 / 99.7 / 100 |
Шумкова и др., 2015 |
Территория завода ОАО «Галоген», (г. Пермь) |
|
Ген α-субъединицы бензотрифторид 2,3-диоксигеназы ( btfA1 ) ; Rhodococcus sp. 065240 |
AB970510.1 |
99.2 / 98.9 / 99.7 |
Yano et al., 2015 |
Нет данных |
|
|
Ген 1,2-дигидробензол-1,2-диол -дегидрогеназы ( bnzB ) ; Rhodococcus opacus B4 (pROB02) |
AP011117.1 |
99.2 / 98.9 / 99.7 |
Na et al., 2005 |
Территория химических заводов. (Хиросима, Япония) |
|
|
Ген α-субъединицы толуол диоксигеназы ( terpA ); Rhodococcus sp. L4 |
EF527236.1 |
98.9 / 98.6 / 99.7 |
н.д. |
Ил из очистных сооружений химического завода (Таиланд) |
Окончание таблицы
|
Клон (геномогруп-па)/ штамм, размер анализируемого фрагмента ДНК |
Гомологичные гены в GenBank |
Номера сравниваемых нуклеотидных последовательностей в GenBank |
Сходство, % |
Ссылка |
Место выделения bphA1-генов и/или бактерий-деструкторов |
|
Ген α-субъединицы изопропилбензол 2,3-диоксигеназы ( ipbA1 ); Rhodococcus erythropolis BD2 (pBD2) |
AY223810.1 |
98.4 / 98.1 / 100 |
Stecker et al., 2003 |
Нет данных |
|
|
Ген α-субъединицы бифенил 2,3-диоксигеназы ( bphA1 ); Rhodococcus jostii RHA1 |
D32142.1 |
95.8 / 96.2 / 99.1 |
Masai et al., 1995 |
Загрязнённая почва (Япония) |
УрМЗ) Rhodococcus erythropolis BD2 [AY223810.1]
(bphAi) Rhodococcus wratislauiensis Ch628
(bphAT) штамм VR43-1
(terpA) Rhodococcus sp. L4 [EF527236.1]
(btfARj Pdiodococcus sp. 065240 [AB970510.1]
(bnzB) Rhodococcus opacus B4 [AP011117.1]
(bphAZ) Rhodococcus sp. RHA1 [D32142.1]
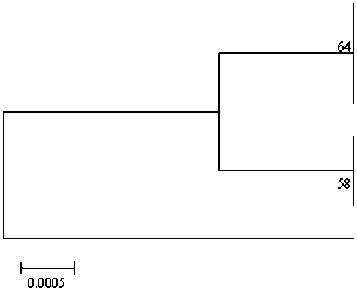
Рис. 2 . Положение bphA1 -генов исследуемого штамма VR43-1 на филогенетическом дереве, построенном на основании сравнительного анализа нуклеотидных последовательностей с использованием метода UPGMA.
Цифрами показана достоверность ветвления, установленная с помощью «bootstrap»-анализа. В качестве аутгруппы использована нуклеотидная последовательность большой субъединицы бифенил 2,3-диоксигназы Rhodococcus jostii RHA1 (D32142.1)
(bphAJ) Rhodococcus wratislaytensis Pl3 [KP972446.1]
Определение филогенетического положения на основании анализа генов 16S рРНК показало, что исследуемый штамм относится к роду Rhodococcus. При сравнении нуклеотидной последовательности гена 16S рРНК штамма VR43-1 с гомологичными последовательностями, имеющимися в базе данных EzBioCloud (URL: сходство с типовым штаммом Rhodococcus wratislaviensis NBRC 100605T(BAWF01000105) составило 100%.
При изучении биодеградационных свойств штамма VR43-1 была определена способность использовать штаммом различные моно- и полиаро-матические соединения в качестве единственного источника углерода и энергии. Помимо способности расти на минеральных средах с добавлением бифенила, штамм VR43-1 демонстрировал хороший рост на агаризованных средах с бензолом, толуолом, фенолом и дибутилфталатом. Активный рост штамма наблюдался в жидкой среде К1 на бифениле, нафталине, рост отсутствовал при вы- ращивании на фенантрене. При культивировании в жидкой среде К1 штамм характеризовался способностью использовать а качестве субстрата моно-ароматические кислоты: орто-фталевую, пара-оксибензойную, протокатеховую, бензойную, но рост не наблюдался на салициловой кислоте.
Анализ нуклеотидной последовательности, ам-плифицированной с использованием праймеров к гену bphA1 [Iwai et al., 2010], штамма VR43-1 показал 100%-ное сходство с генами α-субъединиц бифенил ДО штамма R. wratislaviensis P13 и изопропилбензол ДО штамма R. erythropolis BD2 (таблица). Штамм R. wratislaviensis P13, выделенный из почвы, загрязненной хлорорганическими соединениями (территория предприятия ОАО «Галоген», г. Пермь), является активным деструктором бифенила и хлорированных бифенилов [Шум-кова и др., 2015]. Другой штамм, R. erythropolis BD2, выделенный при культивировании на изопропилбензоле, характеризуется способностью утилизировать широкий спектр моно(поли)аро- матических соединений, таких как бифенил, толуол, бензол, фенол и другие [Dabrock et al., 1994].
Проведенное сравнение амплифицированного фрагмента ДНК ( bphA1 ) штамма VR43-1 с bphA1 -геном активного деструктора бифенила/ПХБ R. jostii RHA1 показало их сходство на уровне 99.1% (таблица). Штамм R. jostii RHA1 обладает исключительно высокой деструктивной активностью по отношению к индивидуальным конгенерам хлорированных бифенилов и различным смесям ПХБ [Furukawa, 2000].
Высокое сходство bphA1- гена штамма VR43-1 (на уровне 99.7%) с генами бензотрифторид ДО ( btfA1 ), толуол ДО ( terpA ) и 1,2-дигидробензол-1,2-диол дегидрогеназы ( bnzB ) может быть обусловлено сходством большинства БДО с другими диоксигеназами подсемейства «бензол/толуол ДО» (к которому принадлежат вышеперечисленные ДО), благодаря чему БДО характеризуются способностью деградировать соединения со сходной химической структурой, обладая широкой субстратной специфичностью [Gibson and Parales, 2000].
Это утверждение подтверждается наличием активного бактериального роста штамма VR43-1 на различных моно(поли)ароматических соединениях (в том числе бензоле, толуоле).
Ранее из загрязненной почвы на территории предприятия ОАО «СВЗХ» (г. Чапаевск) был выделен штамм R. wratislaviensis Ch628, активный деструктор полихлорированных бифенилов и других хлорароматических соединений [Егорова и др., 2014]. Филогенетический анализ показал 100%-ное сходство анализируемых участков bphA1- генов исследуемого штамма VR43-1 и штамма Ch628 (таблица; рис. 2). На основании полученных данных можно предположить, что на загрязненной хлорор-ганическими соединениями территории предприятия ОАО «СВЗХ» (как в почве, так и в донных отложениях) функционируют микробные сообщества, в составе которых доминируют бактерии-деструкторы рода Rhodococcus, содержащие ферментные системы разложения бифенила/ПХБ и других (хлор)ароматических соединений.
Работа выполнена в рамках государственного задания, номер госрегистрации темы: 01201353249.
Список литературы Деструктор бифенила Rhodococcus sp. VR43-1: выделение, молекулярно-биологическая характеристика
- Егорова Д.О. и др. Бактерии-деструкторы полихлорированных бифенилов из почв с различным уровнем загрязнения // Вестник Пермского университета. Сер. Биология. 2014. Вып. 4. C. 64-72.
- Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии // Молекулярное клонирование. М.: Мир. 1984. 390 с.
- Методы общей бактериологии: пер. с англ. / под ред. Ф. Герхардта с соавторами. М.: Мир, 1983. Т. 1-3.
- Назаров А.В. и др. Эколого-микробиологическая оценка грунтов, загрязненных полихлорированными бифенилами // Экология человека. 2016. № 3. С. 3-8.
- Розанова Е.П., Назина Т.Н. Углеводородокисляющие бактерии и их активность в нефтяных пластах // Микробиология. 1982. Т. 51. С. 324-348.
- Федоров Л.А., Оноприенко В.В. Диоксины как экологическая опасность: ретроспектива и перспективы. М.: Наука, 1993. 266 с.
- Шумкова Е.С.и др. Полиморфизм генов bphA бактерий-деструкторов бифенила/полихлорированных бифенилов // Молекулярная биология. 2015. T. 49, № 4. C. 638-648.
- Dabrock B. et al. Identification and characterization of a transmissible linear plasmid from Rhodococcus erythropolis BD2 that encodes isopropylbenzene and trichloroethene catabolism // Applied and environmental microbiology. 1994. Vol. 60, № 3. P. 853-860.
- Furukawa K. Biochemical and genetic bases of microbial degradation of polychlorinated biphenyls (PCBs) // The Journal of general and applied microbiology. 2000. Vol. 46, № 6. P. 283-296.
- Gibson D., Parales R. Aromatic hydrocarbon dioxygenases in environmental biotechnology // Current opinion in biotechnology. 2000. Vol. 11, № 3. P. 236-243.
- Iwai S. et al. Gene-targeted-metagenomics reveals extensive diversity of aromatic dioxygenase genes in the environment // ISME J. 2010. Vol. 4. P. 279-285.
- Liang Y. et al. Potential for polychlorinated biphenyl biodegradation in sediments from Indiana Harbor and Ship Canal // International biodeterioration & biodegradation. 2014. Vol. 89. P. 50-57.
- Maltseva O.V. et al. Degradation of anaerobic reductive dechlorination products of Aroclor 1242 by aerobic bacteria // Biodegradation. 1999. Vol. 10. P. 363-371.
- Masai E. et al. Characterization of biphenyl catabolic genes of gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1 // Applied and Environmental Microbiolog. 1995. Vol. 61, № 6. P. 2079-2085.
- Murugan K., Vasudevan N. Intracellular toxicity exerted by PCBs and role of VBNC bacterial strains in biodegradation // Ecotoxicology and Environmental Safety. 2018. Vol. 157. P. 40-60.
- Na K-S. et al. Isolation and characterization of benzene-tolerant Rhodococcus opacus strains // Journal of Bioscience and Bioengineering. 2005. Vol. 99, № 4. P. 378-382.
- Ohtsubo Y. et al. Strategies for bioremediation of polychlorinated biphenyls // Appl Microbiol Biotechnol. 2004. Vol. 65. P. 250-258.
- Petrić I. et al. Insight in the PCB-degrading functional community in long-term contaminated soil under bioremediation // Journal of soils and sediments. 2011. Vol. 11, № 2. P. 290-300.
- Pieper D.H. Aerobic degradation of polychlorinated biphenyls // Appl Microbiol Biotechnol. 2005. Vol. 67. P. 170-191.
- Pieper D.H., Seeger M. Bacterial metabolism of polychlorinated biphenyls // J Mol Microbiol Biotechnol. 2008. Vol.15. P. 121-138.
- Sharma J.K. et al. Advances and perspective in bioremediation of polychlorinated biphenyl-contaminated soils // Environ. Sci. Pollut. Res. 2018. Vol. 25. P. 16355-16375.
- Short protocols in molecular biology. 3rd ed. / Eds. Ausbel F.M. et al. New York: John Wiley & Sons, 1995. 450 p.
- Stecker C. et al. Complete nucleotide sequence and genetic organization of the 210-kilobase linear plasmid of Rhodococcus erythropolis BD2 // Journal of bacteriology. 2003. Vol. 185, № 17. P. 5269-5274.
- Taguchi K. et al. Polychlorinated biphenyl/biphenyl degrading gene clusters in Rhodococcus sp. K37, HA99, and TA431 are different from well-known bph gene clusters of rhodococci // Bioscience, biotechnology, and biochemistry. 2007. Vol. 71, № 5. P. 1136-1144.
- Takeda H. et al. Characterization of transcriptional regulatory genes for biphenyl degradation in Rhodococcus sp. strain RHA1 // Journal of Bacteriology. 2004. Vol. 186, № 7. P. 2134-2146.
- Warren R. et al. Functional characterization of a catabolic plasmid from polychlorinated-biphenyldegrading Rhodococcus sp. strain RHA1 // Journal of Bacteriology. 2004. Vol. 186, № 22. P. 7783-7795.
- Xiong F. et al. Expression, purification and functional characterization of a recombinant 2,3-dihydroxybiphenyl-1,2-dioxygenase from Rhodococcus rhodochrous // Mol. Biol. Rep. 2011. Vol. 38. P. 4303-4308.
- Yang X. et al. Genome sequence of Rhodococcus sp. strain R04, a polychlorinated-biphenyl biodegrader // Journal of Bacteriology. 2011. Vol. 193, № 18. P. 5032-5033.
- Yano K. et al. Degradation of benzotrifluoride via the dioxygenase pathway in Rodococcus sp. 065240 // Bioscience, Biotechnology, and Biochemistry. 2015. Vol. 79, №. 3. P. 496-504.


