Динамика микробного сообщества типичного чернозема при биодеградации целлюлозы и соломы ячменя
Автор: Чирак Е.Л., Орлова О.В., Аксенова Т.С., Кичко А.А., Чирак Е.Р., Проворов Николай Александрович, Андронов Евгений Евгеньевич
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Зерновые: генетический потенциал, селекционное улучшение, агробиотехнологии
Статья в выпуске: 3 т.52, 2017 года.
Бесплатный доступ
Изучение процессов разложения целлюлозы - важное направление исследований для сельскохозяйственной науки, поскольку солома служит одним из самых доступных органических удобрений. Было проведено большое число работ, направленных на исследование биохимических механизмов разрушения целлюлозы, а также оценку таксономического разнообразия целлюлозолитических микроорганизмов. Однако состав микробного сообщества оценивался только с помощью методов, основанных на культивировании и, как следствие, обнаруживающих небольшую часть почвенного микробиома. С появлением методов секвенирования нового поколения (NGS) открылась возможность анализа сообщества микроорганизмов через обнаруженные в почве копии гена 16S рРНК. Основной задачей представленной работы была реализация комплексного подхода, сочетающего классические агрохимические методы оценки интенсивности процессов биоразложения с современными молекулярными методами (анализ почвенного метагенома 16S рРНК), для изучения динамики микробных сообществ, разлагающих два субстрата, содержащих целлюлозу, - солому и фильтровальную бумагу. С помощью метода высокопроизводительного секвенирования мы впервые показали, что при внесении в почву соломы наблюдаются более глубокие изменения структуры микробного сообщества, чем при внесении химически чистой целлюлозы. Модельный опыт проводили на типичном черноземе, отобранном в Воронежской области на залежных участках с глубины 2-15 см. В почву вносили химически чистую целлюлозу в виде сильно измельченной фильтровальной бумаги и измельченную солому ячменя из расчета 1 г на 100 г почвы. Анализы проводили в день внесения субстратов (0-е сут), а также на 7-е, 14-е, 21-е и 28-е сут. Оценивали выделение СО2, содержание нитратов, аммонийного азота, лабильного углерода, микробной биомассы. Таксономическую структуру микробиома определяли на основании анализа тотальной ДНК. В первые 7 сут эксперимента в варианте с соломой наблюдали прирост биомассы и связанную с этим активизацию дыхательных процессов. В опыте с целлюлозой значительного прироста биомассы не наблюдалось, а дыхание активизировалось с задержкой. Внесение целлюлозы и соломы снизило содержание нитратного азота в почве относительно контроля, причем в варианте с соломой показатель снизился в меньшей степени. В течение опыта были обнаружены таксоны, численность которых как увеличивалась ( Chthoniobacteraceae, Xanthomonadaceae, Chitinophagaceae ), так и уменьшалась ( Gaiellaceae ). Среди первых встречались классические целлюлозолитики ( Chitinophaga и представители семейств Streptosporangiaceae и Micromonosporaceae ) и бактерии, пока не замеченные в процессах биодеградации ( Chthoniobacter, Chitinophaga ). При разложении целлюлозы было обнаружено проявление гомеостаза сообщества - значимые изменения состава микроорганизмов на 14-е сут эксперимента с восстановлением до исходного состояния на 28-е сут. Полученные нами данные об изменениях микробиома при внесении сломы и химически чистой целлюлозы могут служить основой для построения предикативных моделей динамики микробиомов в процессе разложения органического вещества в зависимости от типа разлагаемого субстрата и физико-химических особенностей почвы.
Микробиом, почва, метагеном, солома, целлюлоза, биоразложение
Короткий адрес: https://sciup.org/142214055
IDR: 142214055 | УДК: 631.461:577.2 | DOI: 10.15389/agrobiology.2017.3.588rus
Текст научной статьи Динамика микробного сообщества типичного чернозема при биодеградации целлюлозы и соломы ячменя
Разрушение растительных остатков в почве — одна из важнейших биогеохимических функций почвенных микроорганизмов. Глобальные изменения климата, напрямую влияющие на температурный и водный режим, наряду с нарастающим негативным антропогенным воздействием могут существенно изменить направленность и интенсивность биодеградации органических субстратов. Все больше подтверждений получает гипотеза о зависимости конечного результата разрушения субстрата не только от его химического состава, но и от структуры разлагающего микробного сообщества. Однако закономерности формирования биоразнообразия и функционирования микробного сообщества при разложении соломы остаются не изученными (1). Это связано, в частности, с полифункциональностью микроорганизмов: изменение структуры микробного сообщества не обязательно зависит от типа вносимого субстрата (2).
К важнейшим, в том числе по массе, компонентам растительных остатков относится целлюлоза. Солома — один из наиболее доступных в сельском хозяйстве пожнивных остатков, которые рекомендуется оставлять на полях и вносить почву, она считается хорошим ресурсом для гуму-сонакопления. Ее изогумусовый коэффициент, по разным источникам, равен 0,10-0,25, то есть при оставлении 2-4 т соломы в почве образуется 0,2-1,0 т гумуса на 1 га (3). Мы предположили, что разложение чистой целлюлозы и соломы будет происходить неодинаково из-за их химических особенностей. Солома на 90 % состоит из клетчатки, в состав которой входит 35-45 % целлюлозы, 14-20 % лигнина, 20-30 % пентозанов, 2-3 % кутина и 3-5 % кремниевых солей (3). Как следствие, в разрушении различающихся субстратов будут задействованы разные микроорганизмы, в первую очередь целлюлозоразлагающие, что имеет несомненный практический и теоретический интерес (4, 5).
Изучение микробного сообщества почвы при внесении соломы и чистой целлюлозы молекулярными методами даст возможность более четко показать роль функциональной структуры сообщества, проследить ее связь с таксономическим составом, пространственно-временной изменчивостью перечисленных показателей и агрохимическими параметрами почвы. Этот подход позволит выявить возможную роль микроорганизмов, не учитываемых традиционными методами. Интересно также в динамике рассмотреть различия в таксономической структуре микробных сообществ, ведущих разложение соломы и чистой целлюлозы, и сравнить микробоце-нозы, сложившиеся к концу этого процесса.
С помощью метода высокопроизводительного секвенирования мы впервые показали, что при внесении в почву соломы наблюдаются более глубокие изменения структуры микробного сообщества, чем при внесении химически чистой целлюлозы.
Целью настоящей работы была реализация комплексного подхода, сочетающего классические биохимические методы оценки интенсивности процессов биоразложения с современными молекулярными методами, для изучения динамики микробных сообществ, разлагающих два субстрата, которые содержат целлюлозу, — солому и фильтровальную бумагу.
Методика . В модельном опыте на типичном черноземе, отобранном в Воронежской области (заповедник Каменная степь, 51°01'41.6"N, 40°43'39,3"E) на залежных участках с глубины 2-15 см (Собщ 4,48±0,06 %, С гум. 4,37±0,05 %, N общ. 0,394±0,002 %, рН вод. 6,31±0,01, рН сол. 7,35±0,0, сумма обменных оснований 44,17±0,43 мг-экв/100 г, содержание подвижного фосфора и калия по Мачигину 67,5±8,0 и 12,6±0,8 мг/100 г) вносили химически чистую целлюлозу в виде сильно измельченной фильтровальной бумаги и измельченную солому ячменя (зольность 7,6 %, Nобщ. 1,95 %, Pобщ. 1,1 %, Kобщ. 2,47 %). Контролем служила почва без внесения целлюлозосодержащих субстратов.
Субстрат вносили из расчета 1 г на 100 г почвы. Опыт проводили в чашках Петри, повторность на срок 5-кратная, на последний съем — 7-кратная. Закладывали по 50 г сухой почвы в каждую чашку и по 30 г во флаконы объемом 500 мл (для учета выделения углекислоты, 5-кратная повторность). Почву увлажняли до 50 % полной полевой влагоемкости, помещали под стеклянный колпак вместе с емкостью с водой для сохранения влажности на постоянном уровне и инкубировали в темном поме- щении при температуре 23-25 °С. В день внесения субстратов (0-е сут), а также на 7-е, 14-е, 21-е и 28-е сут оценивали выделение СО2, содержание нитратов, аммонийного азота, лабильного углерода, микробной биомассы. Таксономическую структуру микробиома определяли на основании анализа тотальной ДНК. Все данные получали для смешанного образца в 3-5-кратной повторности.
Агрохимические анализы почвы проводили общепринятыми методами (6). Содержание лабильного углерода в вытяжке по Шульцу (7) определяли на спектрофотометре Ultraspec («LKB», Швеция; λ = 340 нм) (8). Дыхание почвы измеряли на газовом хроматографе Цвет (ЗАО «Цвет», Россия); детектор — катарометр, газ-носитель — гелий. Микробную биомассу в почве учитывали методом субстрат-индуцированного дыхания (9), определяя суммарную биомассу и грибную биомассу (обработка стрептомицином и рифампицином, 16 мг антибиотика на 1 г почвы).
ДНК выделяли из 0,5 г почвы в 3 повторностях для каждого срока (всего 45 образцов). Образцы в течение 1 мин подвергали механическому разрушению в экстрагирующем буфере со стеклянными шариками при максимальной мощности прибора Precellys («Bertin Technologies», Франция). Использовали экстрагирующий буфер следующего состава: 350 мкл раствора А (200 мM натрий-фосфатный буфер, 240 мM изотиоцианат гуанидина; pH 7,0), 350 мкл раствора Б (500 мM Tris-HCl, 1 % SDS, w/v; pH 7,0), 400 мкл смеси фенол: хлороформ (1:1). Полученный препарат центрифугировали при максимальной скорости в течение 5 мин. Отбирали водную фазу и проводили повторную экстракцию хлороформом. ДНК осаждали, добавляя равный объем изопропилового спирта. После центрифугирования осадок промывали 70 % этанолом и растворяли в воде при 65 °С в течении 5-10 мин. Очистку проводили с помощью электрофореза в 1 % агарозном геле с последующим выделением ДНК из геля методом сорбции на оксиде кремния (10, 11).
Очищенный препарат ДНК (10-15 нг) использовали в качестве матрицы в ПЦР (30 с при 95 °С, 30 с при 50 °С, 30 с при 72 °С; всего 30 циклов) с применением полимеразы Encyclo («Евроген», Россия) и универсальных праймеров к вариабельному участку V4 гена 16S рРНК — F515 (5'-GTGCCAGCMGCCGCGGTAA) и R806 (5‘-GGACTACVSGGGTATCT-AAT) (12). Кроме того, в праймеры вводили олигонуклеотидные идентификаторы для каждой пробы и служебные последовательности, необходимые для пиросеквенирования по протоколу фирмы «Roche» (Швейцария). Подготовку проб и секвенирование выполняли на приборе GS Junior («Roche», Швейцария) согласно рекомендациям фирмы-производителя.
Секвенированные последовательности анализировали в программе QIIME 1.8.0 (13). Выполняли разделение библиотек по идентификаторам, проверку качества секвенирования и фильтрацию нуклеотидных последовательностей, объединение последовательностей в операционные таксономические единицы (ОТЕ, аналог вида) по методу de novo на основании 97 % порога сходства генов 16S рРНК (14), отбор репрезентативных последовательностей (по одной от каждой ОТЕ), выравнивание репрезентативных нуклеотидных последовательностей методом Uclust, построение матрицы генетических дистанций и филогенетического древа по методу Fasttree. Из полученных данных были удалены последовательности, представленные во всех образцах менее 2 раз, а также относящиеся к хлоропластам. Таксономическую идентификацию ОТЕ проводили с использованием алгоритма RDP и банка данных Greengenes (15).
По данным таблицы представленности ОТЕ в пробах вычисляли индексы биоразнообразия Шеннона (ChaoI): Sest (ChaoI) = Sobs + a2/2b, где Sest — оцениваемое число ОТЕ; Sobs — наблюдаемое число ОТЕ; a — число ОТЕ, встреченных однократно; b — число ОТЕ, встреченных 2 раза. Кроме того, были построены «кривые накопления», характеризующие динамику увеличения числа ОТЕ в зависимости от количества секвениро-ванных последовательностей. Статистическую обработку проводили в программе QIIME . При учете бактериальной биомассы, содержания нитратного азота в почве и выделения СО2 и рассчитывали средние и стандартные ошибки средних (±SEM). Достоверность различий оценивали по t-критерию Стьюдента (уровень значимости р < 0,05).
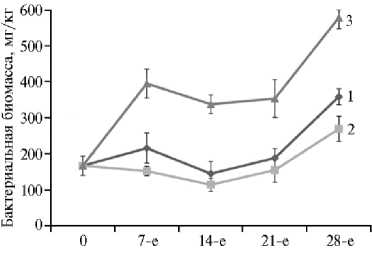
Результаты. Одним из параметров, отражающих повышение микробиологической активности почвы при деструкции сложных органических соединений, служит количество бактериальной биомассы. Наибольшая биомасса была зафиксирована в варианте с внесением в почву соломы (рис. 1, А). Суммарная бактериальная биомасса, накопленная при внесении в почву целлюлозы, статистически не отличалась от контрольной. Причиной этого, по-видимому, был недостаток доступного для микроорганизмов азота, поскольку в химически чистой целлюлозе азот отсутствует, в то время как в соломе он содержится в соотношении с углеродом С:N = 23,7. Использованная для опыта солома, кроме того, содержала значительное количество лабильных органических веществ, что привело к увеличению их доли в почве на 40 % относительно контроля.
А
В
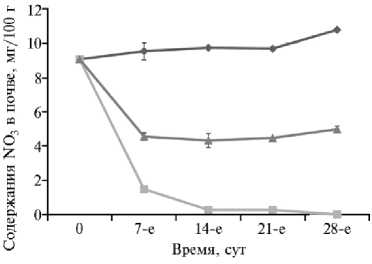
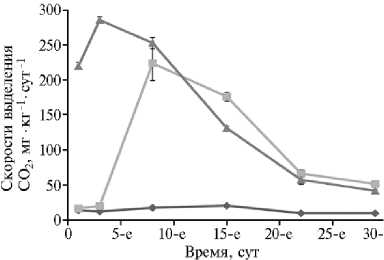
Рис. 1. Динамика бактериальной биомассы (А) , скорости выделения углекислоты (дыхания) (Б) и содержания нитратного азота в почве (В) в модельном опыте по разложению различных субстратов: 1 — контроль (почва без внесения субстратов), 2 — целлюлоза, 3 — солома. Почва — типичный чернозем. Указаны ошибки средних.
Оценка количества выделяемого СО2 показала, что в варианте с соломой биологические процессы наиболее интенсивно шли в первые 3 сут (см. рис. 1, Б) за счет исполь- зования водорастворимых органических соединений, что согласуется с данными литературы (16). В варианте с целлюлозой активизация микроор- ганизмов, сопровождавшаяся выделением углекислого газа, происходила с запаздыванием (наблюдалась лаг-фаза) (17, 18). Начиная с 8-х сут, интенсивность выделения углекислоты из почвы снижалась.
Внесение целлюлозы и соломы уменьшило содержание нитратного азота в почве относительно контроля (см. рис. 1, В). Причем в варианте с соломой показатель снизился в меньшей степени, чем при внесении чистой целлюлозы. Это объясняется уже упомянутым отсутствием азота в фильтровальной бумаге, что в процессе разложения приводит к иммобилизации минерального азота из почвы (3). В варианте с соломой разложение субстрата происходило с использованием азота, содержащегося не только в почве, но и в субстрате.
Наши данные, полученные с помощью классических методов, согласуются друг с другом. В первые 7 сут эксперимента мы наблюдали прирост биомассы и связанную с этим активизацию дыхательных процессов в эксперименте с соломой. В варианте с целлюлозой значительного прироста биомассы не наблюдалось, а дыхание усиливалось с задержкой. Можно предположить, что солома как субстрат стимулировала значительную часть бактериального сообщества, включая R-стратегов, активно утилизи- рующих легкодоступные питательные вещества из растительных остатков
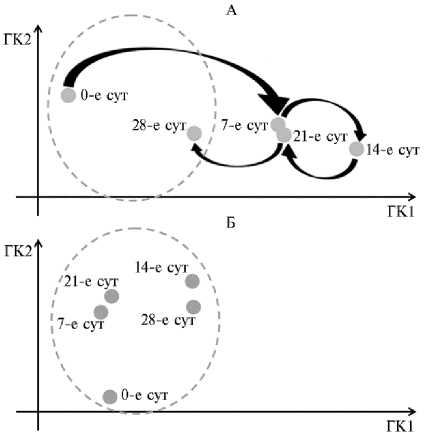
Рис. 2. Результаты анализа сходства микробных сообществ (анализ β -разнообразия) с помощью метода главных компонент (ГК) при внесении в почву соломы (А) и целлюлозы (В) (модельный опыт, почва — типичный чернозем) . Кружками отмечены усредненные для повторностей образцы (на каждые сутки по 3 повторности). Пунктиром выделены области, в которых находились контрольные образцы (почва без внесения субстратов).
менным вне зависимости от вносимого
(18). Целлюлоза активизировала лишь небольшое число микроорганизмов, узкоспециализированных на разрушении этого субстрата. Наблюдаемая динамика количества азотсодержащих соединений была связана с тем, что растворимые формы азота в экспериментах с внесением целлюлозосодержащих субстратов вовлекались в их биодеградацию. Поскольку солома сама по себе содержит азот в органической форме, в первую очередь бактерии будут потреблять именно его.
В результате секвенирования ДНК, выделенной из почвы, было получено 87563 последовательностей, принадлежащих к 408 ОТЕ. ОТЕ относились к 22 бактериальным и 1 археотной филам, а также к 265 семействам прокариот. Биоразнообразие почвенного сообщества бактерий оставалось неиз-субстрата (индексы Шеннона и
ChaoI варьировали в пределах соответственно 7,4-6,9 и 611-528).
Солома, внесенная в почву, влияла на состав бактериального сообщества в большей степени, чем фильтровальная бумага. По данным высокопроизводительного секвенирования, как и агрохимического анализа, самые значительные изменения в составе сообщества, наблюдались на 14-е сут эксперимента с последующим восстановлением состава сообщества до исходного состояния (контроль, рис. 2), что указывает на гомеостаз сообщества, поддержание которого привело к восстановлению исходной структуры в процессах разложения целлюлозосодержащих субстратов. Надо отметить, что были обнаружены семейства бактерий, доля которых в сообществе как уменьшалась (Gaiellaceae), так и увеличивалась
( Chthoniobacteraceae , Xanthomonadaceae , Chitinophagaceae ) (рис. 3). На графике, построенном с помощью метода главных компонент, в варианте с внесением в почву целлюлозы все образцы (вне зависимости от времени отбора проб) находились в непосредственной близости от контрольных. Следовательно, целлюлоза оказывала слабое воздействие на микробное сообщество почвы на начальных этапах биоразложения.
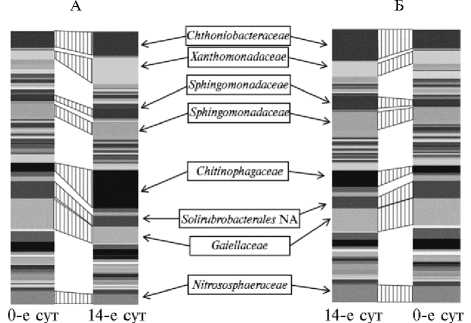
Рис. 3. Состав бактериального сообщества почвы на 0-е и 14-е сут эксперимента при внесении соломы (А) и целлюлозы (Б) (модельный опыт, почва — типичный чернозем) .
В опыте с соломой мы наблюдали значительное увеличение числа бактерий (в некоторых случаях в 88 раз), принадлежащих к роду Chitinophaga, семействам Streptosporangiaceae и Micromonosporaceae, на 28-е сут (рис. 4). Представители этих таксонов, по современным данным, способны разрушать целлюлозу (1921). На 28-е сут при внесении в почву фильтровальной бумаги было зафиксировано (рис. 4) увеличение численности бактерий родов Promicromonospora и Dev- osia, которые также способны вырабатывать целлюлозолитические ферменты (22, 23). При этом обнаружили только одну ОТЕ, численность которой возрастала как в опыте с соломой, так и с фильтровальной бумагой. Выявленная ОТЕ принадлежала роду Chthoniobacter филы Verrucomicrobia. Бактерии упомянутой филы еще очень слабо изучены (один культивируемый представитель), поэтому данные об их способности разлагать целлюлозу пока отсутствуют (24). Все обнаруженные археи относились к роду Nitro-sosphaersceae, широко представленному в почвах России (25). Однако эта ОТЕ не демонстрировала значимой динамики на протяжении всего эксперимента (см. рис. 2).
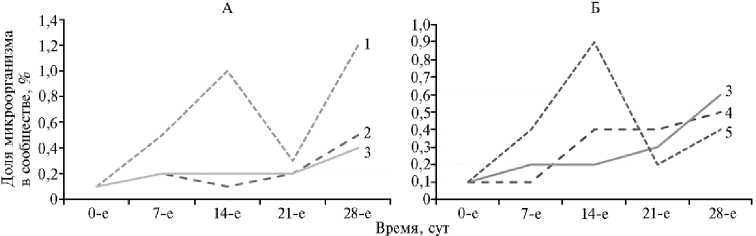
Рис. 4. Динамика численности бактерий семейств Streptosporangiaceae (1) и Micromonosporaceae (2) и родов Chtoniobacter (3) , Promicromonospora (4) , Devosia (5) при внесении в почву соломы (А) и целлюлозы (В) (модельный опыт, почва — типичный чернозем) .
Поскольку мы впервые применили методику метагеномного анализа гена 16S рРНК, сравнение данных, полученных нами, с результатами других работ затруднительно. Большинство авторов исследовали видовую принадлежность обнаруженных целлюлозолитиков (26-28), мы оперировали таксономическими единицами. На уровне более высоких таксономических рангов (семейств и порядков) в нашем исследовании под- твердились полученные ранее данные, поскольку в большинстве работ указывается на то, что актинобактерии активно вовлекаются в разложение целлюлозы. Однако мы обнаружили также ОТЕ, принадлежащие к родам, участие которых в разрушении целлюлозы в почве еще не было показано (Chthoniobacter, Chitinophaga).
Нужно также отметить, что все выявленные закономерности проверены только для изучаемого типа почвы (типичный чернозем) и требуют подтверждения на почвах других типов (дерново-подзолистая, серая лесная и др.). Дальнейшее изучение процессов разложения целлюлозы в почве будет связано с анализом ферментной системы гидролиза целлюлозы, представленной у почвенных бактерий сложным комплексом белков, которые способны разрушать β -1,4-глюкозидные связи внутри молекулы целлюлозы (эндоглюканазы), с редуцирующего и нередуцирующего конца (целлобиогидролаза), а также в димере глюкозы — целлобиозе (целлобиаза). Требуется также молекулярно-генетическое исследование микроорганизмов (включая геномное секвенирование), ответственных за разрушение целлюлозосодержащих субстратов.
Таким образом, с помощью классических агрохимических методов и высокопроизводительного секвенирования нами получены сходные результаты. При внесении в почву соломы в ее разложение вовлекалась значительная часть бактериального сообщества, поскольку такой субстрат был более сбалансированным по составу основных биогенных элементов и более естественным для почвы. В результате мы наблюдали усиление дыхания микроорганизмов, рост их биомассы к 14-м сут и дальнейшее снижение этих показателей к 28-м сут. Лишь небольшая часть бактериального сообщества оказывалась способна к разрушению чистой целлюлозы, существенных различий по составу микробных сообществ в динамике для этого варианта не обнаружили. Полученные нами данные об изменениях микробиома при внесении сломы и химически чистой целлюлозы могут служить основой для построения предикативных моделей динамики микробиомов в процессе разложения органического вещества в зависимости от типа разлагаемого субстрата и физико-химических особенностей почвы. Построенные в будущем предиктивные модели позволят изучить связь таксономической и функциональной структуры почвенного микробиома, а также оптимизировать состав микробных препаратов и методы их внесения в почву.
Список литературы Динамика микробного сообщества типичного чернозема при биодеградации целлюлозы и соломы ячменя
- Nannipieri P., Ascher J., Ceccherini M.T., Landi L., Pietramellara G., Renella G. Microbial diversity and soil functions. Eur. J. Soil Sci., 2003, 54(4): 655-670 ( ) DOI: 10.1046/j.1351-0754.2003.0556.x
- Marschner M., Middlestead P., Clark I.D. Using a simple high-performance liquid chromatography separation and fraction collection methodology to achieve compound-specific isotopic analysis for dissolved organic compounds. Rapid Commun. Mass Sp., 2005, 19(2): 2610269 ( ) DOI: 10.1002/rcm.1780
- Минеев В.Г. Агрохимия. М., 2004.
- Halsall D.M., Gibson A.H. Cellulose decomposition and associated nitrogen fixation by mixed cultures of Cellulomonas gelida and Azospirillum species or Bacillus macerans. Appl. Environ. Microb., 1985, 50(4): 1021-1026.
- Tomme P., Warren R.A.J., Gilkes N. Cellulose hydrolysis by bacteria and fungi. Adv. Microb. Physiol., 1995, 37: 1-81 ( ) DOI: 10.1016/S0065-2911(08)60143-5
- Poll C., Marhan S., Ingwersen J., Kandeler E. Dynamics of litter carbon turnover and microbial abundance in a rye detritusphere. Soil Biol. Biochem., 2008, 40: 1306-1321 ( ) DOI: 10.1016/j.soilbio.2007.04.002
- Шульц Э., Кершенс М. Характеристика разлагаемой части органического вещества почв и ее трансформации при помощи экстракции горячей водой. Почвоведение, 1998, 7: 890-894.
- Паников Н.С., Горбенко А.Ю., Светлов С.В. Способ определения суммарного содержания водорастворимых органических веществ в почве. А.C. ¹ 3949440/30-15. Опубл. 1987. Бюл. ¹ 23.
- West A.W., Sparling G.P. Modification to the substrate-induced respiration method to permit measurement of microbial biomass in soils of different water contents. J. Microbiol. Meth., 1986, 5: 177-189 ( ) DOI: 10.1016/0167-7012(86)90012-6
- Diaferia G., Saltini G., del Vecchio E., Rossi-Bernardi L., Biunno I., Malferrati G., Monferinin P., De Blasio P. High-quality genomic DNA from Human whole blood and mononuclear cells. BioTechniques, 2002, 33(6): 1128-1130.
- Андронов Е.Е., Петрова С.Н., Чижевская Е.П., Коростик Е.В., Ахтемова Г.А., Пинаев А.Г. Влияние внесения генетически модифицированного штамма Sinorhizobium meliloti Ach_5 на структуру почвенного сообщества микроорганизмов. Микробиология, 2009, 78(4): 525-534.
- Bates S.T., Berg-Lyons J.G., Caporaso W.A., Walters W.A., Knight R., Fierer N. Examining the global distribution of dominant archaeal populations in soil. ISME J., 2010, 5: 908-917 ( ) DOI: 10.1038/ismej.2010.171
- Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Huttley G.A. QIIME allows analysis of high-throughput community sequencing data. Nat. Methods, 2010, 7(5): 335-336 ( ) DOI: 10.1038/nmeth.f.303
- Edgar R.C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26: 2460-2461 ( ) DOI: 10.1093/bioinformatics/btq461
- DeSantis T.Z., Hugenholtz P., Larsen N., Rojas M., Brodie E.L., Keller K., Andersen G.L. Green genes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Appl. Environ. Microbiol., 2006, 72: 5069-5072 ( ) DOI: 10.1128/AEM.03006-05
- Мишустин Е.Н. Использование соломы как органического удобрения. М., 1980.
- Паников Н.С., Афремова В.Д., Асеева И.В. Кинетика разложения целлюлозы в почвах. Почвоведение, 1984, 1: 56-62.
- Кожевин П.А. Микробные популяции в природе. М., 1989.
- Chung E.J., Park T.S., Jeon C.O., Chung Y.R. Chitinophaga oryziterrae sp. nov., isolated from the rhizosphere soil of rice (Oryza sativa L.). Int. J. Syst. Evol. Microbiol., 2012, 112: 345-351 ( ) DOI: 10.1099/ijs.0.036442-0
- Zhang L.P., Jiang C.L., Chen W.X. Streptosporangium yunnanense sp. nov. and Streptosporangium purpuratum sp. nov., from soil in China. Int. J. Syst. Evol. Microbiol., 2005, 55: 719-724 ( ) DOI: 10.1099/ijs.0.02565-0
- De Menezes A.B., McDonald J.E., Allison H.E., McCarthy A.J. Importance of Micromonospora spp. as colonizers of cellulose in freshwater lakes as demonstrated by quantitative reverse transcriptase PCR of 16S rRNA. Appl. Environ. Microbiol., 2012, 78(9): 495-505 ( ) DOI: 10.1128/AEM.07314-11
- Cazemier A.E., Verdoes J.C., Reubsaet F.A., Hackstein J.H., van der Drift C., Den Camp H.J.O. Promicromonospora pachnodae sp. nov., a member of the (hemi)cellulolytic hindgut flora of larvae of the scarab beetle Pachnoda marginata. Antonie Van Leeuwenhoek, 2003, 83(2): 135-148 ( ) DOI: 10.1023/A:1023325817663
- Yoo S.H., Weon H.Y., Kim B.Y., Hong S.B., Kwon S.W., Cho Y.H., Stackebrandt E. Devosia soli sp. nov., isolated from greenhouse soil in Korea. International Journal of Systematic and Evolutionary Microbiology, 2006, 56: 2689-2692 ( ) DOI: 10.1099/ijs.0.64214-0
- Sangwan P., Chen X., Hugenholtz P., Janssen P.H. Chthoniobacter flavus gen. nov., sp. nov., the first pure-culture representative of subdivision two, Spartobacteria classis nov., of the phylum Verrucomicrobia. Appl. Environ. Microbiol., 2004, 70(10): 5875-5881 ( ) DOI: 10.1128/AEM.70.10.5875-5881.2004
- Чирак Е.Л., Першина Е.В., Дольник А.С., Кутовая О.В., Василенко Е.С., Когут Б.М., Мерзлякова Я.В., Андронов Е.Е. Таксономическая структура микробных сообществ в почвах различных типов по данным высокопроизводительного секвенирования библиотек гена 16s-pPHK. Сельскохозяйственная биология, 2013, 3: 100-109 ( ) DOI: 10.15389/agrobiology.2013.3.100rus
- Eichorst S.A., Kuske C.R. Identification of cellulose-responsive bacterial and fungal communities in geographically and edaphically different soils by using stable isotope probing. Appl. Environ. Microbiol., 2012, 78(7): 2316-2327 ( ) DOI: 10.1128/AEM.07313-11
- Vries M., Schöler A., Ertl J., Xu Z., Schloter M. Metagenomic analyses reveal no differences in genes involved in cellulose degradation under different tillage treatments. FEMS Microbiol. Ecol., 2015, 91(7): fiv069 ( ) DOI: 10.1093/femsec/fiv069
- Hess M., Sczyrba, A., Egan R., Kim T.W., Chokhawala H., Schroth G., Luo S., Clark D.S., Chen F., Zhang T., Mackie R. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen. Science, 2011, 331: 463-467 ( ) DOI: 10.1126/science.1200387


