Доместикация как частный случай эволюции: об универсальности принципов и механизмов (обзор)
Автор: Глазко В.И., Косовский Г.Ю., Глазко Т.Т.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры, проблемы
Статья в выпуске: 5 т.58, 2023 года.
Бесплатный доступ
Высокая скорость роста изменений состояния биосферы, в которой существенную роль выполняет превращение деятельности человека в геологическую силу (В.И. Вернадский, 1944), приводит к необходимости особого внимания к поискам механизмов управления генетическими потоками у доместицированных видов растений и животных как основы жизнеобеспечения современного человечества. Н.И. Вавилов отмечал, что доместикация представлять собой экспериментальную эволюцию, для управления которой необходимо историческое понимание эволюционного процесса (Н.И. Вавилов, 1987). Основной механизм естественного и искусственного отборов заключается в преимущественном воспроизводстве особей, наиболее приспособленных к предложенным условиям отбора, то есть имеющих репродуктивный успех и обеспечивающих сохранение потомства, что лежит в основе так называемого самоодомашнивания некоторых видов млекопитающих, включая человека (L. Raviv с соавт., 2023). Внутривидовая и межвидовая дифференциация и кооперация для выполнения задач воспроизводства популяций описана и у растений (R. Sharifi, C.M. Ryu, 2021), и у животных (A.M.M. Rodrigues с соавт., 2023; M.A. Zeder, 2012). На примере прокариот показано, что культивирование в разных условиях приводит к существенной дифференциации по характеристикам образования колоний (регуляция поведения) у потомства, причем эти изменения необратимы (невозможность «обратной» эволюции, так называемая дискриминация родственников) (O. Rendueles с соавт., 2015). У многоклеточных организмов существенный вклад в необратимость эволюции вносят симбионты - «кооперанты» на клеточном (E. Rosenberg с соавт., 2010) и геномном (M. Ramakrishnan с соавт., 2021) уровнях. У доместицированных растений и животных большая часть генома занята мобильными генетическими элементами - транспозонами (ТЕ) и продуктами их рекомбинаций (D. Almojil с соавт., 2021). Автономные TE - это потомки экзогенных вирусов и источники основных компонент сетей регуляции профилей генной экспрессии, влияющих на последствия гибридизации, стресс-реактивность (K. Mukherjee, L.L. Moroz, 2023). Обнаруживается определенное сходство ТЕ по участию в основных биологических процессах у растений и животных, несмотря на существенные различия в доминировании вариантов ТЕ, их вовлеченности в организацию архитектоники интерфазного ядра, участии в формировании мотивов для факторов регуляции транскрипции (Y. Qiu, C. Köhler, 2020). Следует отметить, что ранние этапы эволюции основаны на дифференциации и кооперации протобиополимеров (РНК, белки, липиды, углеводы) (Y. Shi и соавт., 2023), прокариот и эукариот (J. Brueckner, W.F. Martin, 2020; C. Al Jewari, S.L. Baldauf, 2023). Наколенные данные свидетельствуют о том, что дифференциация и кооперация - универсальные механизмы эволюционного процесса. В эволюции многоклеточных организмов ключевую роль играют мутуалистические взаимоотношения между такими организмами, микробиотами, вирусами и их потомками. Это обстоятельство необходимо учитывать при поиске методов управления генетическими потоками у доместицированных человеком видов для их сохранения и улучшения.
Репродуктивный успех, мутуализм, доместикация, транспозоны, микробиота, генная экспрессия, регуляторные сети
Короткий адрес: https://sciup.org/142239853
IDR: 142239853 | УДК: 631:575.8:573.7 | DOI: 10.15389/agrobiology.2023.5.821rus
Текст обзорной статьи Доместикация как частный случай эволюции: об универсальности принципов и механизмов (обзор)
Исключительный научный вклад одного из выдающихся ученых XX века Николая Ивановича Вавилова составляют работы по теоретическим основам селекции (переизданы в 1987 году) (1). По определению Н.И. Вавилова, селекция культурных растений и домашних животных по существу представляет собой экспериментальную эволюцию, и для того чтобы понимать и до известной степени управлять ею, необходимо историческое понимание эволюционного процесса (1). Выражение «эволюция, управляемая человеком» введено Н.И. Вавиловым в научную лексику. Основная цель его работ заключалась в том, чтобы «научиться управлять процессом развития органического мира» — эволюцией домашних животных и культурных растений. В апреле 1932 года на заседании Академии наук СССР, Коммунистической академии и Всесоюзной академии сельскохозяйственных наук им. В.И. Ленина, посвященном памяти Ч. Дарвина, Н.И. Вавилов говорил о том, что исследовательская работа с растением и животным должна быть пронизана идеями эволюционизма, на основе которых возможно «овладение организмами» (2).
В настоящем обзоре мы обобщили и проанализировали данные о генных и геномных механизмах доместикации, драйверах эволюции, изменениях организмов в связи с доместикацией, сходстве и различиях естественного и искусственного отборов у представителей разных таксономических групп и роли этих процессов в условиях современных антропогенных изменений биосферы.
Антропоцен как фактор влияния на биосферу. Необходимость эволюционного подхода к выявлению механизмов доместикации приобрела особое значение в конце XX века, когда пришло осознание стремительного сокращения биоразнообразия в глобальном масштабе и стало понятно, что из-за высокой скорости трансформации биосферы вследствие деятельности человека наступила новая геологическая эпоха, получившая название антропоцен (3).
Впервые представления о наступлении такой эпохи сформулировал Владимир Иванович Вернадский. В своей последней опубликованной статье В.И. Вернадский писал о едином историческом процессе, охватывающем всю биосферу планеты (4). В этой статье он отмечал, что уже в начале XX века Алексей Петрович Павлов (1854-1929) — русский и советский ученый -геолог, палеонтолог и стратиграф в последние годы своей жизни, указывая на геологическую роль человека, говорил об антропогенной эре, которую переживает человечество.
Как и любая эпоха, антропоцен продолжает эволюционировать. Совсем недавно относительно неповрежденные природные экосистемы занимали примерно 12 % поверхности Земли, однако в настоящее время на их долю приходится только 1,4 % (5). Приблизительно 9 % населения мира недоедает, и, по прогнозам, к 2030 году это число вырастет до 9,8 %, когда более 850 млн человек столкнутся с голодом (6). Более того, аграрная цивилизация дошла до предела экстенсивного развития, занимая 38 % земной поверхности и потребляя примерно 70 % мировых запасов пресной воды при уровне энергозатрат 1,2 % (7).
Особое значение темпы и биосферные последствия развития аграрной цивилизации приобрели в последние времена. Ярким примером служат данные об изменении биомассы мегафауны (видов животных с массой более 10 кг) после ее последнего крупного вымирания на Земле в период от 50 000 до 3000 лет назад, когда две трети родов млекопитающих и половина видов исчезли (8). После катастрофы глобальная экосистема постепенно восстановилась, затем скорость накопления ее биомассы резко возросла (главным образом за счет сельскохозяйственных видов животных), но повышенная скорость накопления биомассы наблюдается только для популяции человека (8). Предполагается, что массовое вымирание животного мира в четвертичный период эры кайнозоя связано с деятельностью современного человека как основной движущей силы глобальных потерь мегафауны в течение позднего четвертичного периода (9). Но только с появлением техносферы в результате интеллектуальной активности человека возникла глобальная угроза его существованию.
Принято считать, что аграрная цивилизация затрагивает последние 12 тыс. лет (10) — короткий срок по сравнению с несколькими миллиардами лет эволюции биосферы. Тем не менее в настоящее время доместици-рованные виды по массе конкурируют со своими дикими предшественниками (11). Выполненный Y.M. Bar-On с соавт. (11) анализ распределения биомасс в разных царствах живых объектов и разных частях планеты показал, что из ≈ 550 гигатонн (Гт) С на растения приходилось ≈ 450 Гт С (доминирующее царство, занимают главным образом наземные территории, в то время как животные (≈ 2 Гт С) в основном представлены морскими обитателями, а бактерии (≈ 70 Гт С) и археи (≈ 7 Гт С) преимущественно занимают ниши глубоко под поверхностью. Наземная биомасса примерно на два порядка превышает морскую. Анализ показывает, что глобальная пирамида морской биомассы содержит больше консументов, чем продуцентов. Оказалось также, что масса населения Земли, исторически влияющая на глобальную биомассу известных таксонов, включая млекопитающих, рыб и растения, на порядок превышает суммарную массу всех диких млекопитающих (11).
Доместикация как феномен. Трансформация биосферы, сокращение биоразнообразия привела к тому, что все большее внимание исследователей привлекают вопросы, связанные с «эволюцией руками человека» — доместикацией. Доместикация растений и животных — это ключевое событие в формировании аграрной цивилизации. Сокращение биоразнообразия, в том числе сельскохозяйственных видов (12, 13), и истощение плодородных почв в глобальном масштабе (5) привели к пониманию необходимости уделять особое внимание сельскохозяйственным видам как основе существования современного общества. По-видимому, именно этим объясняется резкий рост числа научных публикаций, посвященных анализу процессов и особенностей доместикации. Так, в 2015 году M.A. Zeder (10) приводит данные о том, что в 2013 году опубликовано только 811 статей в 350 различных журналах, включая 42 статьи в PNAS , в которых рассматриваются проблемы, связанные с доместикацией. По нашим данным, в 2021 году число таких научных статей достигло 11 077. Однако до сих пор нет четкого определения, что же подразумевается под термином «доместикация», хотя еще Ч. Дарвин рассматривал процессы доместикации как ускоренную эволюцию под влиянием искусственного отбора (14). И тем более остается открытым вопрос, каковы генетические механизмы этого процесса (15).
Доместикационный синдром у одомашненных животных и культурных растений включает ряд признаков, отличающих их от близкородственных диких видов. По сути он отражает широко распространенные в естественных условиях мутуалистические (симбиотические) межвидовые взаимоотношения, которые при доместикации определяются особенностями не только доместицируемого вида, но и доместикатора — человека (5). Именно поэтому определение доместикационного синдрома до сих пор остается дискуссионным даже для видов млекопитающих (16). Единственное, что объединяет и не вызывает споров, — это то, что доместицированные формы растений, животных и грибов обладают высоким фенотипическим разнообразием по сравнению с близкородственными дикими видами.
Обобщающее определение доместикации было дано M.A. Zeder (17), в котором подчеркивается, что единственным отличием от мутуалистических (симбиоических) взаимоотношений, наблюдаемых в дикой природе, является присутствие доместикатора — человека и то, что доместицируемые должны обладать возможностями вписываться в его нишу (17).
Наиболее выраженным изменением у доместицированных животных принято считать уменьшение размера мозга, прежде всего за счет гиппокампа, гипоталамуса, гипофиза и миндалевидного тела, регулирующих эндокринную функцию и вегетативную нервную систему, что влияет на такие поведенческие реакции, как агрессия и ответ на стресс, вызванный окружающей средой. Резкое уменьшение размеров этих областей у домашних животных может быть напрямую связано с повышением порогов проявления агрессии и страха (17).
В то же время изменения мозга, вызванные одомашниванием, происходят мозаично и отражают определенные адаптации к требованиям особой экологической ниши одомашнивания. Они остаются стабильными после одичания и восстановления диких популяций. Даже по прошествии длительных периодов времени и многих поколений в диких условиях не возникает вторичной эволюционной тенденции к увеличению размера мозга (18). Как и в случае других признаков, уменьшение размеров мозга у доме-стицированных животных происходит видоспецифично. У свиней области мозга, контролирующие обонятельные и слуховые функции, менее редуцированы, чем зрительные структуры или структуры, обеспечивающие двигательные функции; то же самое справедливо для овец. У крыс и норок области мозга, контролирующие двигательные функции, обнаруживают большую степень редукции, чем контролирующие зрительные или обонятельные функции. Уменьшение размера частей мозга, которые контролируют двигательные функции, у норок, выращенных в клетках, почти на 11 % больше, чем у зверьков в открытом вольере, и на 20 % больше, чем у одичавших. Некоторые участки мозга могут увеличиваться в размерах при одомашнивании, что отмечалось для участков мозга, отвечающих за память и обучение у почтовых голубей (17).
Такая же мозаичность и видоспецифичность изменений обнаруживается у растений по признакам, которые служат мишенями естественного и искусственного отборов (19, 20). Наглядным примером видоспецифично-сти может быть сравнение мутации гена Waxy ( Wx ), кодирующего крахмальную синтазу, у кукурузы и риса. У риса отбор по этой мутации сопровождается снижением изменчивости в районе ∼ 250 kb в области локализации гена Wx на 6-й хромосоме, причем снижение генетической изменчивости распространяется примерно на 39 генов, локализованных в этом районе. У кукурузы полигенные системы, включающие гены ae1 , bt2 и su1 , участвующие в путях биосинтеза крахмала, находятся под прямым давлением искусственного отбора (21). Но у кукурузы геном намного больше, чем у риса, при этом плотность локализации структурных генов относительно низкая, что ограничивает эффекты «путешествия автостопом» и задевает только небольшое число генетически сцепленных генов.
Интересно, что в некоторых случаях один и тот же ген лежит в основе параллельной эволюции и сходной фенотипической изменчивости у разных видов растений. Например, клейкий рис возникает из-за дефекта донора сплайсинга в Waxy гене, мутации в этом гене также лежат в основе фенотипов клейких злаков у ячменя, кукурузы и бусенника (травянистое многолетнее растение Иововы слезы) (22). Мутации гена VIRESCENS , кодирующего фактор регуляции транскрипции, приводит к изменению цвета плодов у финиковых и масличных пальм, винограда, яблок, какао и цитрусовых (23-25).
Аналогичная закономерность наблюдается и у фенотипов домашних животных. Например, аллельная изменчивость KIT (tyrosine kinase receptor) — рецептора, критического для миграции популяций стволовых клеток, включая предшественники меланоцитов, и/или MC1R (рецептор мелано-кортина) вызывает изменение окраски у коз, свиней, лошадей, крупного рогатого скота и кур (7).
Имеются также примеры множественного независимого одомаш- нивания одного и того же вида по очень разным путям в разных регионах, например у свиней (26) и сортов сои (27).
В целом результаты исследований наглядно свидетельствуют о коэволюции видов, взаимодействия с которыми лежат в основе формирования этносов и аграрной цивилизации человека, и его культурных традиций. В основе такой мутуалистической кооперации лежат разные гены метаболических путей, кооперация которых может быть специфична и для разных сортов, пород и видов (28).
Интересно отметить, что в геноме можно выделить эволюционно консервативные гены, которые, как правило, ассоциированы с фенотипическими признаками, характерными для ранга крупных филогенетических таксонов (уровня отрядов, семейств), и альтернативные варианты, полиморфизм которых типичен для изменчивости более мелких групп, включая широкое внутривидовое разнообразие доместицированных видов (29). Так, у американской норки эволюционно консервативны гены, позволяющие отнести ее к водным и полуводным млекопитающим (30). В то же время использование результатов полногеномного секвенирования и мононуклео-тидных полиморфизмов (Genome-Wide Association Studies, GWAS) у полу-доместицированной (подвергавшейся искусственному отбору) американской норки позволило обнаружить гены, полиморфизм которых ассоциирован с качеством шкурки и меха (110 генов для характеристик шерсти, 163 — для показателей качества меха и 98 — для размера шкурки), 194 гена для цветотипов, а также выявить 19 районов на 3-й, 4-й, 5-й, 6-й, 7-й, 8-й, 9-й и 10-й хромосомах, которые содержали 33 гена-кандидата контроля качества меха, функций волосяных фолликулов и особенностей размера шкурки. Выявлено множество генов, связанных с процессом цикла роста волос и линьки, развитием эпидермиса, с сигнальным путем Wnt (один из внутриклеточных путей, регулирующий эмбриогенез, дифференцировку клеток, развитие злокачественных опухолей) и развитием мышц (31, 32).
Для растений характерны те же тенденции. Повышенным полиморфизмом обладают, как правило, гены-мишени искусственного отбора. Признаки доместикации у растений можно разделить на три группы. Первая связана с урожайностью, эти признаки влияют на размножение, форму и размер растений (более длинные и жесткие боковые побеги у картофеля, неосыпаемость семян у зерновых культур, образование нераскрывающихся стручков у бобовых, увеличение размера плодов у культурных видов деревьев). Вторая группа признаков связана со снижением химической и физической защиты, типичной для близкородственных диких видов, которая облегчает их расселение в дикой природе (например, потеря горечи в культивируемом миндале, потеря/уменьшение ости у риса и пшеницы). Признаки, относящиеся к третьей группе, касаются прорастания семян (33).
Накопленные данные свидетельствуют о том, что доместицирован-ные виды отличаются от близкородственных диких повышенной изменчивостью мишеней доместикации — определенных комплексов фенотипических признаков и, соответственно, полиморфизмом вовлеченных в их формирование геномных элементов. По-видимому, можно выделить совокупность геномных элементов, образующих «субгеном», зеркально отражающий особенности доместикатора и являющийся его основной мишенью.
Различия между искусственным и естественным отбором. Мишенью вариантов отбора прежде всего служит преимущественное воспроизводство особей с желательными признаками по сравнению с альтернативными вариантами. Именно условия воспроизводства, включая скре- щивания, отличают искусственный отбор от естественного. Не удается получить надежные свидетельства того, что в дикой природе не встречаются единичные особи, несущие признаки, типичные для доместицированных форм. Более того, накапливаются наблюдения о признаках самоодомашни-вания у ряда видов — у человека, бонобо, слона. Сам главный доместика-тор — человек как вид уникален своей сложной культурой и социальными структурами, разнообразием языков и широким использованием инструментов. Согласно гипотезе самоодомашнивания человека, этот уникальный набор черт может быть результатом эволюционного процесса самоиндуциро-ванного одомашнивания, в ходе которого человек эволюционировал, чтобы быть менее агрессивными и более приспособленными к кооперации в целях сохранения потомства. Сходные процессы описаны у предкового вида человека — бонобо, а также слона. Внутривидовое самоодомашнивание обеспечило отчетливое сходство когнитивных, поведенческих и физиологических характеристик между людьми, бонобо и слонами, направленных прежде всего на сохранение потомства (34).
У животных выделяют четыре основные формы общих изменений репродуктивной функции при доместикации: исключение конкуренции между самцами и ограниченность числа самок для скрещиваний, у самок — увеличение пищевых ресурсов, защита от хищников и снижение материнского стресса . В генетический контроль таких изменений вовлечены процессы, связанные с развитием нервного гребня (NCC). Это способствует формированию общих признаков синдрома доместикации у разных таксонов (35).
Но и в естественных условиях внедрение в новую нишу приводит к увеличению поведенческой активности, влияя на взаимоотношения между самцами и самками. Динамика иерархической и поведенческой структуры популяций‐основателей в новых условиях может оказывать решающее влияние на внутри- и межвидовые взаимоотношения, приводить к их долговременным изменениям и формированию новых поведенческих реакций. Для популяций-основателей, которые расширяются в новую нишу, отделенную от родительской популяции, необходимость установления связей с незнакомцами, отсутствие четких территорий и первоначально новые кормовые ресурсы могут вызвать изменение исходной иерархической структуры, к которой адаптируются последующие поколения. Гипотеза «социальности основателя» сформирована на наблюдениях об относительно более сильных связях у более подвижного пола (самцы у млекопитающих) по сравнению с менее подвижным (самки у млекопитающих), относительном уменьшении осваиваемых территорий и повышенной толерантности при внутри- и межвидовых взаимодействиях в новых условиях (36). Естественный отбор благоприятствует выживанию популяции через альтруизм (посредством влияния на репродуктивный успех популяции как целого). Репродуктивная ценность определяет реализацию альтруизма в зависимости от индивидуальных различий в возрасте, поле (самцы, самки) и общем состоянии, внутренние взаимодействия в группе могут изменять репродуктивную ценность особей, чтобы стимулировать альтруизм и обеспечить выживание популяции (37).
Человек доместицировал генетически далекие грибы для аналогичных целей — ферментации продуктов, богатых липидами и сахаром, для придания им привлекательного внешнего вида, запаха и ароматизации, а также для увеличения срока годности и безопасности продукта. Многочисленные независимые события одомашнивания также произошли внутри вида. Получены 826
свидетельства фенотипической конвергенции при одомашнивании грибов для производства сыра ( Saccharomyces cerevisiae , Penicillium roqueforti , P. ca-memberti и Geotrichum candidum ) и для производства вяленого мяса ( P. nal-giovense и P. salamii ). Конвергенция, последовавшая за адаптацией к сходным экологическим нишам, затронула особенности формирования колоний (пушистость и цвет), липолиз, протеолиз, выработку летучих соединений и конкурентоспособность в борьбе с вредителями пищевых продуктов. Описаны сходства в потере генетического разнообразия в одомашненных популяциях и в вырождении неиспользуемых признаков, таких как выработка токсинов и половое размножение. Фенотипическая конвергенция иногда происходила благодаря сходным механизмам геномной адаптации, в частности горизонтальному переносу и потере генов (38).
Накоплены данные, свидетельствующие о том, что социокультурные особенности ниш, созданных человеком, вносят относительно больший вклад во внутривидовую дифференциацию культурных растений и домести-цированных животных, чем экологические факторы (39). То есть главные отличия естественного отбора от искусственного определяются человеком как доместикатором, его поведением, особенностями создаваемой им ниши и контролируемых человеком мутуалистических взаимоотношений между разными видами.
Дифференциация и кооперация — от протобиополимеров до человека. Принято связывать появление жизни на Земле с мутуализмом (кооперацией) протобиомолекул и конкуренцией между ними по признаку стабильности (40).
Древний мир представлен сообществами РНК, обладающих разнообразными каталитическими свойствами и реплицируемых рибозимами-полимеразами. Начало отбора связано с поиском баланса между двумя крайностями — смесью нуклеотидов с низкой упорядоченностью и низкой сложностью и кристаллами с высокой упорядоченностью, но низкой сложностью. Предполагается, что поиск такого баланса приводит к появлению продукта эволюционного процесса с высоким порядком (множество цепочек с общими определенными последовательностями), высокой сложностью (длина последовательности) и, соответственно, высокой функциональностью (изменчивостью), что становится началом отбора (41). Аминокислоты служат катализаторами, структурными стабилизаторами, а также посредниками, обеспечивающими связь между РНК и липидами при формировании протоклетки.
Конкуренция между протоклетками опосредована действием мембраностабилизирующего пептида MSP, который синтезируется по механизму прямого участия РНК. РНК с характерными последовательностями специфически связывает аминокислоты и объединяет их, образуя первично дипептиды. MSP может проникать в мембрану и препятствовать выходу амфифилов (жирных кислот или подобных молекул) из мембраны. В результате обмена амфифилами между мембраной и окружающей средой протоклетка без MSP потеряет амфифилы и сократится, в то время как протоклетка, содержащая MSP, вырастет до более крупных размеров (42).
Кооперация между археями и протеобактериями лежит в основе возникновения эукариот, появления митохондрий. В среднем у эукариот бактериальные гены составляют в целом 56 %, без пластид — 53 %, у фотосин-тетизирующих линий эукариот, у которых цианобактериальный предок пластид внес дополнительные гены, — 61 % (43).
Внутриклеточные паразиты, которые подвергаются редуктивной эволюции в процессе адаптации к богатой питательными веществами среде зараженных ими клеток, отказываются от бактериальных генов контроля метаболических процессов. Такая потеря адаптивных генов наиболее выражена у паразита человека Encephalitozoon intestinalis, у которого 86 % генов архейского и 14 % — бактериального происхождения. У эукариот наиболее изучен геном риса, содержащий 67 % бактериальных и 33 % архейных генов. Функциональная дихотомия, первоначально описанная для дрожжей, когда гены архей участвуют в обработке генетической информации, а бактериальные — в метаболических процессах, сохраняется во всех супергруппах эукариот (43).
Взаимодействия геномов архей и протеобактерий привело к появлению большого числа химерных генов (рис. 1). Выявлен целый класс ядер-ных химерных генов, сформировавшихся в процессе эндосимбиоза (S-гены, 282 семейства) с последующей эволюцией эукариотических линий (44).
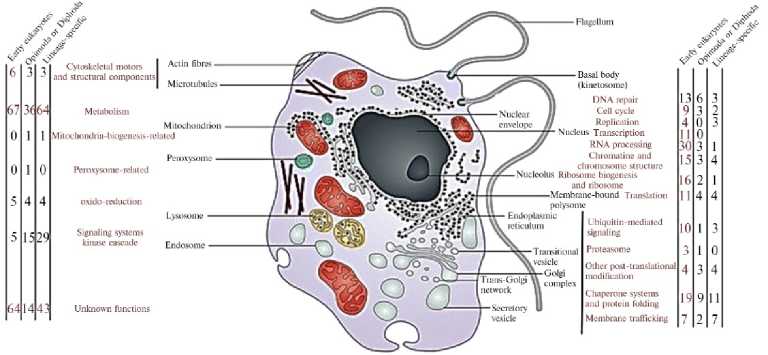
Рис. 1. Функции 573 S-генов в эукариотической клетке. Цифры, выделенные красным, соответствуют функциям, содержащим важные S-гены в дрожжах (44).
В настоящее время в домене Eukaryota выделяют группу Excavata . Филогенетическое дерево у основания разветвляется: первая крупная ветвь эукариот — Parabasalia , за которой последовательно отделяются Fornicata , Preaxostyla и Discoba . Отсутствие аэробных митохондрий у Parabasalia , For-nicata и Preaxostyla свидетельствует о том, что современные эукариоты возникли в аноксических условиях (45). Предполагается, что появление митохондрий происходило в два этапа, которые были связаны с двумя эн-досимбиотическими событиями: с формированием промитохондриальных структур, затем митохондриальных, первое наиболее вероятно связано с γ - и/или δ-протеобактериями ( γ -/δ-proteo), второе — с α-протеобактери-ями (α-proteo) (45).
У прокариот изменения факторов окружающей среды приводят к возникновению новых межклеточных взаимоотношений и культуральноморфологических свойств (рис. 2) (46). В исследовании в качестве абиотического фактора использовали плотную агаризованную среду (1,5 %) CTT-HA либо полужидкий агар (0,5 %) CTT-SA. Биотическим фактором служил газон либо Bacillus subtilis, либо Escherichia coli на 1,5 % агаре с питательной смесью CTT. Без нее выращивали на плотном агаре B. subtilis-TPM и E. coli-TPM. При такой обработке для M. xanthus питательные вещества были доступны только из B. subtilis-TPM или E. coli-TPM. Размах фенотипической изменчивости (см. рис. 2, красные кружки) увеличивается на плотном агаре и при относительном голодании (на бактериальных газонах) (46).
Важно отметить, что при воздействии факторов окружающей среды у прокариот наблюдается дифференциация близких родственников (рис. 3), у разных биообъектов часто отмечают так называемую дискриминацию родственников, при этом эволюция происходит только в направлении большей адаптивности организма (47).
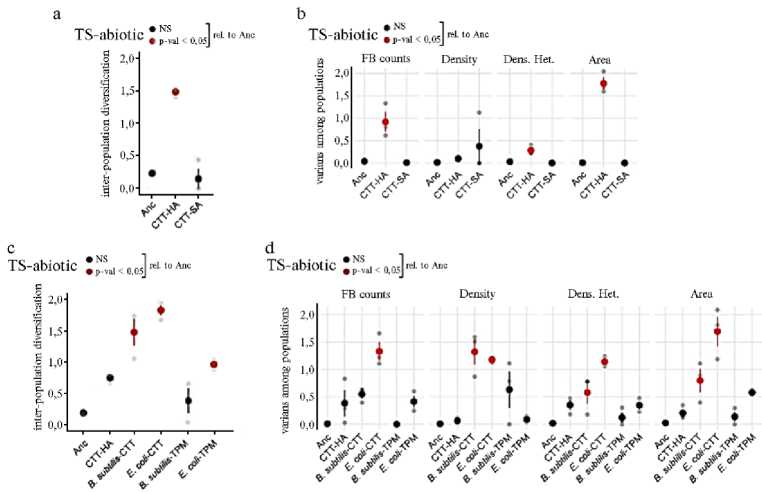
Рис. 2. Пример изменчивости колониально-морфологических свойств бактерий Myxococcus xanthus при разных обработках (TS). Характеристики популяций: число колоний, плотность колоний, гетерогенность колоний по плотности, площадь (46).
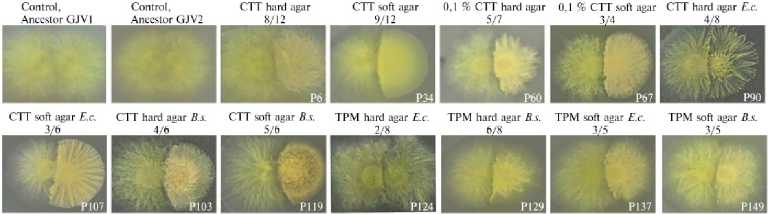
Рис. 3. Примеры изменений взаимоотношений между колониями Myxococcus xanthus в результате выращивания на плотном (hard) или полужидком (soft) агаре, на полноценной среде или на газонах Bacillus subtilis- TPM и Escherichia coli- TPM. На двух первых фотографиях показаны фенотипы самоконтроля при слиянии колоний для двух предковых вариантов. B.s. — B. subtilis ; CTT — полноценная питательная среда, содержащая казитон; E.c. — E. coli ; TPM — среда без кази-тона (голодная) (47).
Внутривидовая дифференциация близкородственных форм хорошо известна, в некоторых случаях она имеет очевидную генетическую основу (например, между подвидами европейского кролика). Несмотря на неотличимость кариотипов этих двух подвидов, предположительно центральная область Х-хромосомы (перицентромерные районы) вовлечена в репродуктивную изоляцию между ними (нарушения конъюгации гомологов в мейозе), что приводит к ограниченности генетических потоков между подвидами. Предковой для домашнего кролика является французская форма Oryctolagus c. cuniculus (48). Эта ограниченность настолько велика, как в
Австралии, куда были также завезены представители испанского подвила ( O. c. algirus ), и домашний кролик. Остров был захвачен именно испанским подвидом и какого-либо существенного появления их помесей с домашним кроликом не наблюдалось (49). При внутри- (как у подвидов европейского кролика) и межвидовой гибридизации ведущую роль выполняет дискриминация хромосом (элементов генома близких родственников), как правило, связанная с тем, что у видов с половой дифференциацией в хромосомах (в частности, в перицентромерных районах) накапливаются мобильные генетические элементы (транспозоны), нарушающие прохождение мейотиче-ских делений при формировании гамет. Систематический обзор исследований по межвидовой гибридизации у млекопитающих свидетельствует о том, что общая частота негативных последствий (49 %) выше, чем положительных (13 %) и нейтральных (38 %) (50).
У растений показано, что алло- и автополиплоидизация приводит к существенному изменению профилей генной экспрессии, активации транспозонов, транскрипции длинной некодирующей РНК, микроРНК, массовым структурным перестройкам в геномах (51-53).
Мобильные генетические элементы, регуляторные сети и эволюция. Транспозоны рассматриваются в качестве драйверов эволюции и источников различных элементов, вовлекаемых в регуляцию профилей генной экспрессии (53-55). В эволюции идет последовательный процесс увеличения размеров геномов, но такое увеличение не сопровождается соответствующим ростом числа генов, кодирующих белки (рис. 4) (56), что можно рассматривать как указание на взрастающее значение регуляторных сетей. Симбиоз с транспозонами (в большинстве своем потомками ДНК-содержащих вирусов и ретровирусов) у высших таксонов занимает половину генома.
5. cerevisiae
II. sapiens 7?. norvegicus M. mnscuins
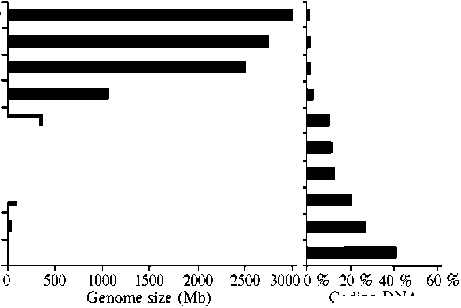
Coding DNA
Рис. 4. Размер генома эукариот и доля некодирующей аминокислотные последовательности ДНК от низших к высшим эукариотам. Минимальный размер генома последовательно увеличивается от простых эукариот (например, дрожжей) до сложных позвоночных (в частности, человека). Доля кодирующей ДНК в более крупных геномах очень мала, то есть большая часть генома, приобретенного этими организмами, является некодирующей (56).
G. gallus
F. rubripes C intestinaiis ■
D. melanogaster ■ C. elegans ■ N. crassa
Представленность транспозонов (TE) существенно варьирует в геномах разных видов. В ряде исследований обнаружено, что TE служат важным источником цис-регуляторных мотивов в форме сайтов связывания факторов регуляции транскрипции (TF), дистального энхансера, супрессора или изолятора. Обнаружено, что значительная доля TF в геномах млекопитающих происходит из TE (54, 57). Известны TF, генерируемые TE, которые могут влиять на трехмерную организацию хроматина и таким образом вовлечены в регуляцию активности генов, которые не находятся в непосредственной близости. Примером этого у позвоночных служит последовательность, с которой связывается регулятор транскрипции — белок CTCF с 11 цинковыми пальцами, он участвует в образовании петель и топологически ассоциированных доменов и представляет собой производное от TE. Следует отметить, что TE вносят значительный вклад в формирование видоспецифичных петель в ДНК человека и мыши за счет привнесения новых мотивов для TF CTCF (58). Мотивы связывания TF CTCF выявлены и у ряда ретровирусов млекопитающих, в частности у провируса бычьего лейкоза (BLV) (59). TF CTCF вовлечен как в регуляцию транскрипционной активности BLV, так и в дерегуляцию трехмерной (3D) архитектуры хроматина хозяина, окружающей сайт интеграции BLV. В провирусной ДНК BLV выявлено in vivo три консервативных мотива связывания CTCF. В геноме BLV последовательность, с которой связывается CTCF, локализована в областях модификаций гистонов, вовлеченных в регуляцию транскрипции, и участвует в подавлении активности промотора 5´-длинного концевого повтора (LTR), что приводит к латентности вируса, но в то же время способствует активности промотора 3´LTR, вовлеченного в транскрипцию соответствующей длинной некодирующей РНК (lncRNA). Интеграция BLV нарушает регуляцию организации трехмерного хроматина клетки-хозяина, изменяя образование петель хроматина в местах интеграции провирусной ДНК в геном хозяина. Нередко последовательности автономных TE характеризуются высокой изменчивостью метилирования ДНК, что также сказывается на их вовлеченности в регуляцию генной экспрессии.
У растений фактор TF CTCF отсутствует, но сходное влияние TE на пространственную организацию генома обнаружено. Например, у риса и сорго миниатюрные транспонируемые элементы с инвертированными повторами ассоциированы с наличием областей прикрепления к ядерному матриксу и служат «якорями» для петель (60, 61).
Генная регуляторная сеть — это система взаимодействий между молекулярными регуляторами (например, факторами транскрипции) и субстратами (например, сайтами связывания факторов транскрипции), которые управляют экспрессией генов, участвующих в сложных биологических процессах (62). Растет число исследований о непосредственной роли TE в эволюции генных регуляторных сетей, их вовлеченности в ответы на действие биотических и абиотических факторов окружающей среды (54, 63-65).
В геномах растений ретротранспозоны с длинными концевыми повторами (LTR-ретротранспозоны, эндогенные ретровирусы — ERV) представляют наиболее распространенную группу мобильных генетических элементов, они вовлечены в организацию геномной архитектуры и в проявление фенотипической изменчивости. Общая структура ретротранспозонов и доменов, ответственных за различные фазы их репликации, высококонсервативны у всех эукариот. В хромосомах высших растений присутствуют два основных суперсемейства LTR-ретротранспозонов — Ty1/Copia и Ty3/Gypsy. Число копий членов этих суперсемейств может увеличиваться, часто они активируются различными биотическими и абиотическими стрессами из-за всплесков ретротранспозиции. ERV — важные факторы видовой изменчивости, характеризуются большим разнообразием структуры, размеров и механизмов транспозиций, что делает их важными участниками эволюции геномов (66). Кроме того, ERV влияют на паттерн экспрессии соседних генов, вовлекаясь в формирование малой интерферирующей РНК (siRNA) и РНК-направленное метилирование ДНК (RdDM) (67).
TE занимают 60-80 % генома у кукурузы, 29 % у риса и 17 % у арабидопсиса (68, 69). Арабидопсис (119,1 Mb) и рис (373,8 Mb) обладают одними из самых маленьких геномов среди соответственно двудольных и однодольных растений, и их геномы считаются эталонными в своих классах , для кукурузы характерен один из самых длинных геномов среди однодольных (2,3 Gb) (70).
Стресс-реактивность растений тесно связана с факторами регуляции транскрипции. Так, у кукурузы суперсемейство фактора регуляции транскрипции AP2/ERF, специфичное для растений, включает 229 AP2 / ERF генов, и их экспрессия меняется под влиянием целого спектра факторов экологического стресса. Кроме того, некоторые представители этого суперсемейства вовлечены в половую дифференциацию растений и в регуляцию разных стадий онтогенеза (71). Суперсемейство TF AP2/ERF, наряду с вовлеченностью в ответы на стрессирующие факторы окружающей среды, непосредственно участвует в реализации действия ряда гормонов растений (72). То есть регуляторные системы стресс-реактивности растений тесно связаны с ключевыми системами регуляции онтогенеза.
Выполнено сравнение вклада ТЕ в регуляцию транскрипционной активности (по их влиянию на экспрессию близлежащих генов) в качестве регуляторных мотивов при реакции на стресс. Исследовали связь между присутствием суперсемейств TE выше, ниже или внутри интронов близлежащих генов и дифференциальной экспрессией этих генов в различных стрессовых условиях у организмов, контрастных по числу ТЕ в геномах, — бедного TE Arabidopsis thaliana и богатого TE Solanum lycopersicum . Обнаружено, что в ответе на стресс участвуют гены, которые расположены недалеко от представителей различных суперсемейств TE, в частности SINE при протеотоксическом стрессе, Copia и Gypsy — при тепловом у A. thaliana , EPRV и hAT при инфекции, а также Harbinger, LINE при световом стрессе у S. lycopersicum . Эти же авторы составили карту TE-опосредованных сетей регуляции ответа на стресс у растений на основе профилей экспрессии генов стрессореактивности у двух контрастирующих по этому признаку видов. Была изучена регулирующая роль, которую TE выполняют в ответе на стресс, позволяя растениям быстрее адаптироваться к новым условиям окружающей среды (73).
В геноме пшеницы примерно 85 % последовательностей занято ТЕ. Обнаружено, что около 36 % из 70 818 генов содержат по меньшей мере одну вставку TE внутри гена, в основном в трех гомеологах (триадах). Вставки TE внутри экзонов или в нетранслируемых областях (UTRS) одного или нескольких гомеологов в триаде связаны с различиями в их экспрессии. Обнаружена статистически значимая корреляция между наличием/отсутствием вставок TE, принадлежащих к шести надсемействам и 17 подсемействам TE, и подавлением одного гомеологичного гена. Выявлена прямая связь между наличием вставок TE из определенных суперсемейств и экспрессией генов, связанных с реакциями на биотический и абиотический стресс. То есть TE у пшеницы выполняет важную роль в контроле экспрессии генов специфичным для генома путем (74).
ТЕ широко распространены и разнообразны в геномах хвойных деревьев. У некоторых видов хвойных в TE идентифицированы потенциально важные мотивы последовательностей, которые могли бы связывать дополнительные регуляторные факторы, способствуя формированию регуляторной сети (75). Показано вовлечение ТЕ в ответ на биотические факторы экологического стресса у растений, например при инокуляции проростков хвойных двумя видами грибов, что приводило к глобальному изменению метилирования геномной ДНК и экспрессии ряда ТЕ (76).
Интересно отметить, что один и тот же принцип кооперации между биообъектами для увеличения пластичности в ответ на действия факторов окружающей среды в царствах животных и растений реализуется неодинаково. В геномах животных среди ТЕ доминируют короткие неавтономные ретротранспозоны, у растений — эндогенные ретровирусы (54); у животных, по крайней мере позвоночных, ключевым элементом в архитектонике интерфазного ядра, по-видимому, служит мотив посадки TF CTCF, у растений — сложный комплекс мотивов, вовлекаемых в межбелковые взаимодействия (77). Существенно различаются и семейства ТЕ, последовательности которых составляют основу для формирования факторов регуляции транскрипции TF у многоклеточных (78).
Один из ярких примеров участия TE в приобретении новых признаков у животных — формирование и эволюция плаценты млекопитающих, связанные с активностью эндогенных ретровирусов, многие семейства которых были одомашнены с последующей генерацией новых регуляторных генов или регуляторных элементов (79). Из TE, ассоциированных с плацентой, две различные группы ретровирусных LTR-ретротрансферазонов — THE1B и RLTR13D5 способствовали расселению сотен специфичных для плаценты регуляторных элементов соответственно у человекообразных приматов и мышей.
Интересно, что в конвергентной эволюции эндосперма (питательная ткань у цветковых растений) также участвовали ТЕ-транспозиции. Специфичный для эндосперма фактор транскрипции MADS-box I типа PHERES1 (PHE1) у Arabidopsis thaliana взаимодействует с двумя видами мотивов, один из которых — связанный с MADS-box канонический мотив CArG, другой представляет собой частично модифицированный мотив CArG, причем оба они обогащены элементами ДНК транспозона Helitron (80). Многие гены-мишени с мотивами связывания PHE1, полученными из Helitron, имеют высокую экспрессию в эндосперме, что отражает специфическую для эндосперма регуляцию PHE1. Предполагается, что распределение мотивов, связывающих фактор транскрипции MADS-box I типа с помощью Helitron, способствовало эволюции эндосперма, позволив вовлечь важнейшие гены развития в общую транскрипционную сеть аналогично тому, что происходило при участии ТЕ в формировании плаценты млекопитающих (81).
В совокупности накопленные данные свидетельствует о том, что именно ТЕ служат основным источником элементов сетей, регулирующих профили генной экспрессии на уровне ДНК, с последующим вовлечением в эти процессы в качестве инструментов прямого действия некодирующих РНК, факторов регуляции транскрипции, межбелковых взаимодействий. Вся иерархия этой регуляторной системы у многоклеточных организмов высокочувствительна к внешним воздействиям и тесно взаимодействует с микробиомом, непосредственно отвечая на изменчивость последнего.
Микробное сообщество, связанное с растениями (микробиом), играет важную роль в коммуникациях между ними. Передача сигналов между растениями без их физического контакта может быть опосредована микоризными гифами и стеблями паразитических форм, возможно использование летучих веществ и корневых экссудатов, участие субстанций, выделяемых микробами или животными. Это позволять растению, генерирующему сигнал, влиять на адаптацию микробиома у растения, принимающего сигнал, с помощью стимулирующих или конкурентных механизмов (82). Пластичность развития создает возможность для одного генома формировать разные фенотипы в зависимости от внешних сигналов. Такая пластичность обеспечивает фенотипические диапазоны, в пределах которых животные и растения будут приспосабливаться к вызовам окружающей среды
(например, к климатическим изменениям), и способствует тому, что организм создает новую экологическую нишу, изменяя окружающую среду. Предполагается, что элементарной единицей эволюции служит холобионт — многоклеточный организм с его микробиотой (83, 84).
Интеграция исследований по биологии развития и экологии в эволюционную теорию привела к возникновению относительно новой области знаний — экологической эволюционной биологии развития (ecological evolutionary developmental biology, Eco-Evo-Devo) (85). В целом принципы эволюции биосферы одинаковы — дифференциации и кооперации, и доместикация — частный случай этого процесса. Выживание и адаптация касаются не только индивидуального организма. Это концепции, которые охватывают различные иерархические уровни — от нуклеиновых кислот и белков, генов и клеток до экосистем и социальных структур. Становится очевидным, что одним из универсальных путей биологической эволюции служит реализуемый на разных уровнях организации биологических объектов скачкообразный переход от дифференциации (включая половую, социальную дифференциацию) к кооперации.
ДНК-маркеры и селекция. Одно из новых направлений поиска ДНК-маркеров, необходимых для управления генетическими потоками до-местицированных видов, — поиск регуляторных сетей и их основных элементов, контроль и коррекция которых может существенно уменьшить сроки селекционной работы. У разных видов к настоящему времени выполнено полногемномое секвенирование и поиск связей между мононуклеотидными полиморфизмами (SNP) и изменчивостью фенотипических признаков. В частности, организован крупнейший на сегодняшний день ресурс Zoonomy (86) по сравнительной геномике млекопитающих. На основе сопоставления геномов у 240 видов млекопитающих выявлены SNP, полиморфизмы которых, вероятно, влияют на фенотипические характеристики и изменяют риск заболеваний. По меньшей мере 332 млн нуклеотидов в геноме человека (примерно 10,7 % генома, что более чем в 5 раз превышает число нуклеотидов, кодирующих белки) и других видов необычно консервативны по сравнению с последовательностями, относительно нейтральными по скорости эволюции, а 4552 нуклеотида ультраконсервативны (почти идеально консервативны). Из 101 млн консервативных SNP 80 % находятся вне экзонов, кодирующих белок (86).
У доместицированных видов млекопитающих, например у овец, в большинстве случаев SNPs (около 90 %), ассоциированные с изменчивостью хозяйственно ценных признаков, локализованы в некодирующих последовательностях генома. Это приводит к предположению о том, что именно перестройка экспрессии генов служит ведущим фактором фенотипического разнообразия у доместицированных видов (87-89) и человека, что связано со сложностью изменяющихся признаков и особенностями распространения болезней (90).
Итак, становится очевидным, что «симбиотические» взаимоотношения между человеком и сельскохозяйственными видами растений и животных требуют определенного изменения в адресности стратегии и тактики поисков геномных элементов, контроль которых мог бы существенно повлиять на управляемость, скорость и эффективность селекционной работы. В конечном итоге популяционно-генетические особенности представителей доместицируемых видов являются результатом ответа на баланс интенсивности действия факторов естественного и искусственного отборов. В этом ответе участвуют ключевые гены метаболических путей, связанных с устойчивостью к биотическим и абиотическим стрессам и с процессами форми-834
рования желательных фенотипов по хозяйственно ценным признакам, а также иерархия регуляторных элементов, обусловленная полиморфизмом внутригеномных факторов (таких как транспозоны) и пластичностью структуры микробиоты.
Список литературы Доместикация как частный случай эволюции: об универсальности принципов и механизмов (обзор)
- Вавилов Н.И. Теоретические основы селекции. М., 1987.
- Ревенкова А.И. Николай Иванович Вавилов: 1887-1943. М., 1962.
- Глазко В.И., Иваницкая Л., Чешко В. Антропоцен. Философия биотехнологии. Стабильная адаптивная стратегия Homo sapiens, эволюционный риск и эволюционная семантика. М., 2018.
- Вернадский В.И. Несколько слов о ноосфере. Успехи современной биологии, 1944, 18(2): 113-120.
- Guo Z., Zhang L., Li Y. Increased dependence of humans on ecosystem services and biodiversity. PLoS ONE, 2010, 5(10): e13113 (doi: 10.1371/journal.pone.0013113).
- FAO, IFAD, UNICEF, WFP, WHO. The state of food security and nutrition in the world 2020. Transforming food systems for affordable healthy diets. FAO, Rome, 2020 (doi: 10.4060/ca9692en).
- Andersson L., Purugganan M. Molecular genetic variation of animals and plants under domesti-cation. PNAS USA, 2022, 119(30): e2122150119 (doi: 10.1073/pnas.2122150119).
- Barnosky A.D. Megafauna biomass tradeoff as a driver of Quaternary and future extinctions. PNAS USA, 2008, 105(Suppl 1): 11543-11548 (doi: 10.1073/pnas.0801918105).
- Sandom C., Faurby S., Sandel B., Svenning J.C. Global late Quaternary megafauna extinctions linked to humans, not climate change. Proc. Biol. Sci., 2014, 281(1787): 20133254 (doi: 10.1098/rspb.2013.3254).
- Zeder M.A. Core questions in domestication research. PNAS USA, 2015, 112(11): 3191-3198 (doi: 10.1073/pnas.1501711112).
- Bar-On Y.M., Phillips R., Milo R. The biomass distribution on Earth. PNAS USA, 2018, 115(25): 6506-6511 (doi: 10.1073/pnas.1711842115).
- FAO. The state of the world’s animal genetic resources for food and agriculture /B. Rischkowsky, D. Pilling (eds.). FAO, Rome, 2007.
- Hazell P., Wood S. Drivers of change in global agriculture. Philos. Trans. R. Soc. Lond. B Biol. Sci., 2008, 363(1491): 495-515 (doi: 10.1098/rstb.2007.2166).
- Дарвин Ч. Изменения домашних животных и культурных растений. Т. 4. М.-Л., 1951. 15. Purugganan M.D. What is domestication? Trends Ecol. Evol., 2022, 37(8): 663-671 (doi: 10.1016/j.tree.2022.04.006).
- Johnsson M., Henriksen R., Wright D. The neural crest cell hypothesis: no unified explanation for domestication. Genetics, 2021, 219(1): iyab097 (doi: 10.1093/genetics/iyab097).
- Zeder M.A. Pathways to animal domestication. In: Biodiversity in agriculture: domestication, evolution, and sustainability. Cambridge University Press, Cambridge, 2012: 227-259 (doi: 10.1017/CBO9781139019514.013).
- Kruska D.C. On the evolutionary significance of encephalization in some eutherian mammals: effects of adaptive radiation, domestication, and feralization. Brain Behav. Evol., 2005, 65(2): 73-108 (doi: 10.1159/000082979).
- Li L.F., Olsen K.M. To have and to hold: selection for seed and fruit retention during crop domestication. Curr. Top. Dev. Biol., 2016, 119: 63-109 (doi: 10.1016/bs.ctdb.2016.02.002).
- Mueller N.G., Horton E.T., Belcher M.E., Kistler L. The taming of the weed: developmental plasticity facilitated plant domestication. PLoS ONE, 2023, 8(4): e0284136 (doi: 10.1371/journal.pone.0284136).
- Whitt S.R., Wilson L.M., Tenaillon M.I., Gaut B.S., Buckler E.S. Genetic diversity and selection in the maize starch pathway. PNAS USA, 2002, 99(20): 12959-12962 (doi: 10.1073/pnas.202476999).
- Purugganan M.D. Evolutionary insights into the nature of plant domestication. Curr. Biol., 2019, 29(14): R705-R714 (doi: 10.1016/j.cub.2019.05.053).
- Hazzouri K.M., Flowers J.M., Visser H.J., Khierallah H.S.M., Rosas U., Pham G.M., Meyer R.S., Johansen C.K., Fresquez Z.A., Masmoudi K., Haider N., El Kadri N., Idagh-dour Y., Malek J.A., Thirkhill D., Markhand G.S., Krueger R.R., Zaid A., Purugganan M.D. Whole genome re-sequencing of date palms yields insights into diversification of a fruit tree crop. Nat. Commun., 2015, 6: 8824 (doi: 10.1038/ncomms9824).
- Singh R., Low E.T., Ooi L.C., Ong-Abdullah M., Nookiah R., Ting N.C., Marjuni M., Chan P.L., Ithnin M., Manaf M.A., Nagappan J., Chan K.L., Rosli R., Halim M.A., Azizi N., Budiman M.A., Lakey N., Bacher B., Van Brunt A., Wang C., Hogan M., He D., MacDon-ald J.D., Smith S.W., Ordway J.M., Martienssen R.A., Sambanthamurthi R. The oil palm VI-RESCENS gene controls fruit colour and encodes a R2R3-MYB. Nat. Commun., 2014, 5: 4106 (doi: 10.1038/ncomms5106).
- Allan A.C., Hellens R.P., Laing W.A. MYB transcription factors that colour our fruit. Trends Plant Sci., 2008, 13(3): 99-102 (doi: 10.1016/j.tplants.2007.11.012).
- Ottoni C., Flink L.G., Evin A., Geörg C., De Cupere B., Van Neer W., Bartosiewicz L., Linder-holm A., Barnett R., Peters J., Decorte R., Waelkens M., Vanderheyden N., Ricaut F.X., Cakirlar C., Cevik O., Hoelzel A.R., Mashkour M., Karimlu A.F., Seno S.S., Daujat J., Brock F., Pinhasi R., Hongo H., Perez-Enciso M., Rasmussen M., Frantz L., Megens H.J., Crooijmans R., Groenen M., Arbuckle B., Benecke N., Vidarsdottir U.S., Burger J., Cucchi T., Dobney K., Larson G. Pig domestication and human-mediated dispersal in western Eurasia revealed through ancient DNA and geometric morphometrics. Mol. Biol. Evol., 2013, 30(4): 824-832 (doi: 10.1093/molbev/mss261).
- Lee G.A., Crawford G.W., Liu L., Sasaki Y., Chen X. Archaeological soybean (Glycine max) in East Asia: does size matter? PLoS ONE, 2011, 6(11): e26720 (doi: 10.1371/journal.pone.0026720).
- Глазко В.И. Генные и геномные подписи доместикации. Сельскохозяйственная биология, 2018, 53(4): 659-672 (doi: 10.15389/agrobiology.2018.4.659rus).
- Glazko G.V., Koonin E.V., Rogozin I.B. Molecular dating: ape bones agree with chicken entrails. Trends Genet., 2005, 21(2): 89-92 (doi: 10.1016/j.tig.2004.12.006).
- Wang L., Zhou S., Lyu T., Shi L., Dong Y., He S., Zhang H. Comparative genome analysis reveals the genomic basis of semi-aquatic adaptation in American mink (Neovison vison). Animals (Basel), 2022, 12(18): 2385 (doi: 10.3390/ani12182385).
- Valipour S., Karimi K., Do D.N., Barrett D., Sargolzaei M., Plastow G., Wang Z., Miar Y. Genome-wide detection of selection signatures for pelt quality traits and coat color using whole-genome sequencing data in American Mink. Genes (Basel), 2022, 13(11): 1939 (doi: 10.3390/genes13111939).
- Cai Z., Villumsen T.M., Asp T., Guldbrandtsen B., Sahana G., Lund M.S. SNP markers associ-ated with body size and pelt length in American mink (Neovison vison). BMC Genet., 2018, 19(1): 103 (doi: 10.1186/s12863-018-0688-6).
- Martínez-Ainsworth N.E., Tenaillon M.I. Superheroes and masterminds of plant domestication. Comptes Rendus Biologies, 2016, 339(7-8): 268-273 (doi: 10.1016/j.crvi.2016.05.005).
- Raviv L., Jacobson S.L., Plotnik J.M., Bowman J., Lynch V., Benítez-Burraco A. Elephants as an animal model for self-domestication. PNAS USA., 2023, 120(15): e2208607120 (doi: 10.1073/pnas.2208607120).
- Gleeson B.T., Wilson L.A.B. Shared reproductive disruption, not neural crest or tameness, explains the domestication syndrome. Proc. Biol. Sci., 2023, 290(1995): 20222464 (doi: 10.1098/rspb.2022.2464).
- Brooks J., Yamamoto S. The founder sociality hypothesis. Ecol. Evol., 2021, 11(21): 14392-14404 (doi: 10.1002/ece3.8143).
- Rodrigues A.M.M., Gardner A. Reproductive value and the evolution of altruism. Trends Ecol. Evol., 2022, 37(4): 346-358 (doi: 10.1016/j.tree.2021.11.007).
- Ropars J., Giraud T. Convergence in domesticated fungi used for cheese and dry-cured meat maturation: beneficial traits, genomic mechanisms, and degeneration. Curr. Opin. Microbiol., 2022, 70: 102236 (doi: 10.1016/j.mib.2022.102236).
- Colino-Rabanal V.J., Rodríguez-Díaz R., Blanco-Villegas M.J., Peris S.J., Lizana M. Human and ecological determinants of the spatial structure of local breed diversity. Sci. Rep., 2018, 8: 6452 (doi: 10.1038/s41598-018-24641-3).
- El-Maarouf-Bouteau H. The seed and the metabolism regulation. Biology (Basel), 2022, 11(2): 168 (doi: 10.3390/biology11020168).
- Mayer C. Order and complexity in the RNA world. Life (Basel), 2023, 13(3): 603 (doi: 10.3390/life13030603).
- Shi Y., Yu C., Ma W. Towards an RNA/peptides world by the direct RNA template mechanism: the emergence of membrane-stabilizing peptides in RNA-based protocells. Life (Basel), 2023, 13(2): 523 (doi: 10.3390/life13020523).
- Brueckner J., Martin W.F. Bacterial genes outnumber archaeal genes in eukaryotic genomes. Genome Biol. Evol., 2020, 12(4): 282-292 (doi: 10.1093/gbe/evaa047).
- Méheust R., Bhattacharya D., Pathmanathan J.S., McInerney J.O., Lopez P., Bapteste E. For-mation of chimeric genes with essential functions at the origin of eukaryotes. BMC Biol., 2018, 16(1): 30 (doi: 10.1186/s12915-018-0500-0).
- Al Jewari C., Baldauf S.L. An excavate root for the eukaryote tree of life. Sci. Adv., 2023, 9(17): eade4973 (doi: 10.1126/sciadv.ade4973).
- La Fortezza M., Rendueles O., Keller H., Velicer G.J. Hidden paths to endless forms most wonderful: ecology latently shapes evolution of multicellular development in predatory bacteria. Commun. Biol., 2022, 5(1): 977 (doi: 10.1038/s42003-022-03912-w).
- Rendueles O., Zee P.C., Dinkelacker I., Amherd M., Wielgoss S., Velicer G.J. Rapid and wide-spread de novo evolution of kin discrimination. PNAS USA, 2015, 112(29): 9076-9081 (doi: 10.1073/pnas.1502251112).
- Geraldes A., Ferrand N., Nachman M.W. Contrasting patterns of introgression at X-linked loci across the hybrid zone between subspecies of the European rabbit (Oryctolagus cuniculus). Genet-ics, 2006, 173: 919-933 (doi: 10.1534/genetics.105.054106).
- Alves J.M., Carneiro M., Day J.P., Welch J.J., Duckworth J.A., Cox T.E., Letnic M., Strive T., Ferrand N., Jiggins F.M. A single introduction of wild rabbits triggered the biological invasion of Australia. PNAS USA, 2022, 119(35): e2122734119 (doi: 10.1073/pnas.2122734119).
- Adavoudi R., Pilot M. Consequences of hybridization in mammals: a systematic review. Genes (Basel), 2021, 13(1): 50 (doi: 10.3390/genes13010050).
- Singh A., At V., Gupta K., Sharma S., Kumar S. Long non-coding RNA and microRNA land-scape of two major domesticated cotton species. Comput. Struct. Biotechnol. J., 2023, 21: 3032-3044 (doi: 10.1016/j.csbj.2023.05.011).
- Jin S., Han Z., Hu Y., Si Z., Dai F., He L., Cheng Y., Li Y., Zhao T., Fang L., Zhang T. Structural variation (SV)-based pan-genome and GWAS reveal the impacts of SVs on the speci-ation and diversification of allotetraploid cottons. Mol. Plant., 2023, 16(4): 678-693 (doi: 10.1016/j.molp.2023.02.004).
- Ramakrishnan M., Satish L., Kalendar R., Narayanan M., Kandasamy S., Sharma A., Emamver-dian A., Wei Q., Zhou M. The dynamism of transposon methylation for plant development and stress adaptation. Int. J. Mol. Sci., 2021, 22(21): 11387 (doi: 10.3390/ijms222111387).
- Almojil D., Bourgeois Y., Falis M., Hariyani I., Wilcox J., Boissinot S. The Structural, functional and evolutionary impact of transposable elements in eukaryotes. Genes (Basel), 2021, 12(6): 918 (doi: 10.3390/genes12060918).
- Глазко В.И., Косовский Г.Ю., Глазко Т.Т. Геномные источники разнообразия как драй-веры доместикации. Сельскохозяйственная биология, 2022, 57(5): 832-851 (doi: 10.15389/agrobiology.2022.5.832rus).
- Kumar R.P., Senthilkumar R., Singh V., Mishra R.K. Repeat performance: how do genome packaging and regulation depend on simple sequence repeats? Bioessays, 2010, 32(2): 165-174 (doi: 10.1002/bies.200900111).
- Jordan I.K., Rogozin I.B., Glazko G.V., Koonin E.V. Origin of a substantial fraction of human regulatory sequences from transposable elements. Trends Genet., 2003, 19(2): 68-72 (doi: 10.1016/s0168-9525(02)00006-9).
- Choudhary M.N., Friedman R.Z., Wang J.T., Jang H.S., Zhuo X., Wang T. Co-opted transpos-ons help perpetuate conserved higher-order chromosomal structures. Genome Biol., 2020, 21(1): 16 (doi: 10.1186/s13059-019-1916-8).
- Bellefroid M., Rodari A., Galais M., Krijger P.H.L., Tjalsma S.J.D., Nestola L., Plant E., Vos E.S.M., Cristinelli S., Van Driessche B., Vanhulle C., Ait-Ammar A., Burny A., Ciuffi A., de Laat W., Van Lint C. Role of the cellular factor CTCF in the regulation of bovine leukemia virus latency and three-dimensional chromatin organization. Nucleic Acids Res., 2022, 50(6): 3190-3202 (doi: 10.1093/nar/gkac107).
- Avramova Z., Tikhonov A., Chen M., Bennetzen J.L. Matrix attachment regions and structural colinearity in the genomes of two grass species. Nucleic Acids Res., 1998, 26(3): 761-767 (doi: 10.1093/nar/26.3.761).
- Argentin J., Bolser D., Kersey P.J., Flicek P. Comparative analysis of repeat content in plant genomes, large and small. Front. Plant Sci., 2023, 14: 1103035 (doi: 10.3389/fpls.2023.1103035).
- Davidson E., Levin M. Gene regulatory networks. PNAS USA, 2005, 102(14): 4935 (doi: 10.1073/pnas.0502024102).
- Chuong E.B., Elde N.C., Feschotte C. Regulatory activities of transposable elements: from con-flicts to benefits. Nat. Rev. Genet., 2017, 18(2): 71-86 (doi: 10.1038/nrg.2016.139).
- Bourque G., Leong B., Vega V.B., Chen X., Lee Y.L., Srinivasan K.G., Chew J.L., Ruan Y., Wei C.L., Ng H.H., Liu E.T. Evolution of the mammalian transcription factor binding repertoire via transposable elements. Genome Res., 2008, 18(11): 1752-1762 (doi: 10.1101/gr.080663.108).
- Zattera M.L., Bruschi D.P. Transposable elements as a source of novel repetitive DNA in the eukaryote genome. Cells, 2022, 11(21): 3373 (doi: 10.3390/cells11213373).
- Papolu P.K., Ramakrishnan M., Mullasseri S., Kalendar R., Wei Q., Zou L.H., Ahmad Z., Vinod K.K., Yang P., Zhou M. Retrotransposons: how the continuous evolutionary front shapes plant genomes for response to heat stress. Front. Plant. Sci., 2022, 13: 1064847 (doi: 10.3389/fpls.2022.1064847).
- Niu X., Chen L., Kato A., Ito H. Regulatory mechanism of a heat-activated retrotransposon by DDR complex in Arabidopsis thaliana. Front. Plant Sci., 2022, 13: 1048957 (doi: 10.3389/fpls.2022.1048957).
- Le Q.H., Wright S., Yu Z., Bureau T. Transposon diversity in Arabidopsis thaliana. PNAS USA, 2000, 97(13): 7376-7381 (doi: 10.1073/pnas.97.13.7376).
- Bennetzen J.L. Transposable elements, gene creation and genome rearrangement in flowering plants. Curr. Opin. Genet. Dev., 2005, 15(6): 621-627 (doi: 10.1016/j.gde.2005.09.010).
- Zhou S., Wei F., Nguyen J., Bechner M., Potamousis K., Goldstein S., Pape L., Mehan M.R., Churas C., Pasternak S., Forrest D.K., Wise R., Ware D., Wing R.A., Waterman M.S., Livny M., Schwartz D.C. A single molecule scaffold for the maize genome. PLoS Genet., 2009, 5(11): e1000711 (doi: 10.1371/journal.pgen.1000711).
- Cheng C., An L., Li F., Ahmad W., Aslam M., Ul Haq M.Z., Yan Y., Ahmad R.M. Wide-range portrayal of AP2/ERF transcription factor family in maize (Zea mays L.) development and stress responses. Genes (Basel), 2023, 14(1): 194 (doi: 10.3390/genes14010194).
- Xiong R., Chu Z., Peng X., Cui G., Li W., Dong L. Transcript-wide identification and expression pattern analysis to comprehend the roles of AP2/ERF genes under development and abiotic stress in Trichosanthes kirilowii. BMC Plant Biol., 2023, 23(1): 354 (doi: 10.1186/s12870-023-04362-0).
- Deneweth Jю, Van de Peer Yю, Vermeirssen V. Nearby transposable elements impact plant stress gene regulatory networks: a meta-analysis in A. thaliana and S. lycopersicum. BMC Genomics, 2022, 23(1): 18 (doi: 10.1186/s12864-021-08215-8).
- Bariah I., Gribun L., Kashkush K. Transposable elements are associated with genome-specific gene expression in bread wheat. Front. Plant Sci., 2023, 13: 1072232 (doi: 10.3389/fpls.2022.1072232).
- Voronova A., Rendón-Anaya M., Ingvarsson P., Kalendar R., Ruņģis D. Comparative study of pine reference genomes reveals transposable element interconnected gene networks. Genes (Basel), 2020, 11(10): 1216 (doi: 10.3390/genes11101216).
- Voronova A. Retrotransposon expression in response to in vitro inoculation with two fungal path-ogens of Scots pine (Pinus sylvestris L.). BMC Res. Notes, 2019, 12(1): 243 (doi: 10.1186/s13104-019-4275-3).
- Yang T., Wang D., Tian G., Sun L., Yang M., Yin X., Xiao J., Sheng Y., Zhu D., He H., Zhou Y. Chromatin remodeling complexes regulate genome architecture in Arabidopsis. The Plant Cell, 2022, 34(7): 2638-2651 (doi: 10.1093/plcell/koac117).
- Mukherjee K., Moroz L.L. Transposon-derived transcription factors across metazoans. Front. Cell. Dev. Biol., 2023, 11: 1113046 (doi: 10.3389/fcell.2023.1113046).
- Dunn-Fletcher C.E., Muglia L.M., Pavlicev M., Wolf G., Sun M.A., Hu Y.C., Huffman E., Tumukuntala S., Thiele K., Mukherjee A., Zoubovsky S., Zhang X., Swaggart K.A., Lamm K.Y.B., Jones H., Macfarlan T.S., Muglia L.J. Anthropoid primate-specific retroviral element THE1B controls expression of CRH in placenta and alters gestation length. PLoS Biol., 2018, 16(9): e2006337 (doi: 10.1371/journal.pbio.2006337).
- Batista R.A., Moreno-Romero J., Qiu Y., van Boven J., Santos-González J., Figueiredo D.D., Köhler C. The MADS-box transcription factor PHERES1 controls imprinting in the endosperm by binding to domesticated transposons. eLife, 2019, 8: e50541 (doi: 10.7554/eLife.50541).
- Qiu Y., Köhler C. Mobility connects: transposable elements wire new transcriptional networks by transferring transcription factor binding motifs. Biochem. Soc. Trans., 2020, 48(3): 1005-1017 (doi: 10.1042/BST20190937).
- Sharifi R., Ryu C.M. Social networking in crop plants: wired and wireless cross-plant communi-cations. Plant Cell Environ., 2021, 44(4): 1095-1110 (doi: 10.1111/pce.13966).
- Rosenberg E., Sharon G., Atad I., Zilber-Rosenberg I. The evolution of animals and plants via symbiosis with microorganisms. Environ. Microbiol. Rep., 2010, 2(4): 500-506 (doi: 10.1111/j.1758-2229.2010.00177.x).
- Guerrero R., Margulis L., Berlanga M. Symbiogenesis: the holobiont as a unit of evolution. Int. Microbiol., 2013, 16(3): 133-143 (doi: 10.2436/20.1501.01.188).
- Feng L., Dong T., Jiang P., Yang Z., Dong A., Xie S.Q., Griffin C.H., Wu R. An ECO-EVO-DEVO genetic network model of stress response. Hortic. Res., 2022, 9: uhac135 (doi: 10.1093/hr/uhac135).
- Christmas M.J., Kaplow I.M., Genereux D.P., Dong M.X., Hughes G.M., Li X., Sullivan P.F., Hindle A.G., Andrews G., Armstrong J.C., Bianchi M., Breit A.M., Diekhans M., Fanter C., Foley N.M., Goodman D.B., Goodman L., Keough K.C., Kirilenko B., Kowalczyk A., Law-less C., Lind A.L., Meadows J.R.S., Moreira L.R., Redlich R.W., Ryan L., Swofford R., Valen-zuela A., Wagner F., Wallerman O., Brown A.R., Damas J., Fan K., Gatesy J., Grimshaw J., Johnson J., Kozyrev S.V., Lawler A.J., Marinescu V.D., Morrill K.M., Osmanski A., Paulat N.S., Phan B.N., Reilly S.K., Schäffer D.E., Steiner C., Supple M.A., Wilder A.P., Wirthlin M.E., Xue J.R., Zoonomia Consortium, Birren B.W., Gazal S., Hubley R.M., Koepfli K.P., Marques-Bonet T., Meyer W.K., Nweeia M., Sabeti P.C., Shapiro B., Smit A.F.A., Springer M.S., Teel-ing E.C., Weng Z., Hiller M., Levesque D.L., Lewin H.A., Murphy W.J., Navarro A., Paten B., Pollard K.S., Ray D.A., Ruf I., Ryder O.A., Pfenning A.R., Lindblad-Toh K., Karlsson E.K. Evolutionary constraint and innovation across hundreds of placental mammals. Science, 2023, 380(6643): eabn3943 (doi: 10.1126/science.abn3943).
- Davenport K.M., Massa A.T., Bhattarai S., McKay S.D., Mousel M.R., Herndon M.K., White S.N., Cockett N.E., Smith T.P.L., Murdoch B.M. Characterizing genetic regulatory ele-ments in ovine tissues. Front. Genet., 2021, 12: 628849 (doi: 10.3389/fgene.2021.628849).
- Xiang R., Berg I.V.D., MacLeod I.M., Hayes B.J., Prowse-Wilkins C.P., Wang M., Bolormaa S., Liu Z., Rochfort S.J., Reich C.M., Mason B.A., Vander Jagt C.J., Daetwyler H.D., Lund M.S., Chamberlain A.J., Goddard M.E. Quantifying the contribution of sequence variants with regula-tory and evolutionary significance to 34 bovine complex traits. PNAS USA, 2019, 116(39): 19398-19408 (doi: 10.1073/pnas.1904159116).
- Naval-Sanchez M., Nguyen Q., McWilliam S., Porto-Neto L.R., Tellam R., Vuocolo T., Re-verter A., Perez-Enciso M., Brauning R., Clarke S., McCulloch A., Zamani W., Naderi S., Re-zaei H.R., Pompanon F., Taberlet P., Worley K.C., Gibbs R.A., Muzny D.M., Jhangiani S.N., Cockett N., Daetwyler H., Kijas J. Sheep genome functional annotation reveals proximal regula-tory elements contributed to the evolution of modern breeds. Nat. Commun., 2018, 9(1): 859 (doi: 10.1038/s41467-017-02809-1).
- Albert F.W., Kruglyak L. The role of regulatory variation in complex traits and disease. Nat. Rev. Genet., 2015, 16(4): 197-212 (doi: 10.1038/nrg3891).


