Фенотипический и молекулярно-генетический скрининг устойчивости образцов томата к фитофторозу
Автор: Хунвану В.Н., Монахос Г.Ф., Монахос С.Г.
Журнал: Овощи России @vegetables
Рубрика: Селекция, семеноводство и биотехнология растений
Статья в выпуске: 5 (79), 2024 года.
Бесплатный доступ
Актуальность. Фитофтороз томата, вызываемый Phytophthora infestans, в прохладных и влажных условиях среды может приводить к потере практически 100% урожая в условиях открытого грунта. При этом генетические особенности P. infestans позволяют со временем преодолевать генетическую устойчивость растений-хозяев, что требует от селекционеров искать новые гены устойчивости к фитофторозу и получать новые сорта, обладающие сразу несколькими генами устойчивости.
Томат, фитофтороз, маркер-опосредованный отбор, устойчивость, генетическая коллекция
Короткий адрес: https://sciup.org/140307821
IDR: 140307821 | УДК: 635.64-047.37:632.444.2 | DOI: 10.18619/2072-9146-2024-5-12-17
Текст научной статьи Фенотипический и молекулярно-генетический скрининг устойчивости образцов томата к фитофторозу
Оригинальная статья / Original article УДК: 635.64-047.37:632.444.2
Ф итофтороз, вызываемый оомицетом
Phytophthora infestans (Mont.) de Bary, – одна из основных болезней представителей паслёновых, в том числе и томата (Solanum lycopersicum L.), по всему миру, особенно в регионах с относительно низкими температурами и высокой влажностью [1]. При этом потери урожая томата из-за поражения фитофторио-зом могут достигать от 41 до 100% в открытом грунте и от 12 до 65% в открытом грунте при использовании фунгицидов [2]. Единственным способом эффективного и устойчивого контроля за распространением P. infestans прежде всего остаётся использование созданных ранее и создание новых устойчивых сортов томата, дополненное селективным применением фунгицидов, удаление поражённых растений и рядом других агротехнических приёмов [3, 4].
Ряд других генов, например, R1 и R3a , обеспечивающих устойчивость к P. infestan s, известных для паслёновых в целом, лучше всего были охарактеризованы у картофеля ( S. tuberosum L.) и его близких родственников, таких как, например, S. demissum Lindl., хотя и их устойчивость была частично преодолена [7-9].
Преодоление устойчивости в создаваемых сортах томата P. infestans обусловлено генетическими особенностями её генома [5]. Способность преодолевать защитные механизмы растения-хозяина обеспечивается изменением плоидности, масштабным делециям, которые могут удалять из генома факторы, распознаваемые растением-хозяином, изменениями в паттернах экспрессии генов и высокой активностью транспозонов [10]. В связи с этим, одним из направлений селекции томата на устойчивость к фитофторозу представляется накопление в растении множества генов устойчивости, которые в совокупности позволяют лучше защитить растение, что уже было продемонстрировано на примере взаимного действия Ph-2 и Ph-3 генов [11].
В России, хотя фитофтороз является одним из самых вредоносных заболеваний томата, выращиваемого в открытом грунте, научные работы, посвященные поиску генов-маркеров устойчивости томата к фитофторозу, практически отсутствуют. В связи с этим, целью настоящей работы стало тестирование ряда известных молекулярных маркеров, ассоциированных с генами устойчивости к фитофторозу, на возможность их дальнейшего использования в маркер-опосредован-ной селекции, фенотипический и молекулярно-генетический скрининг генетической коллекции томата.
Материалы и методы
Генетическая коллекция образцов томата была получена из ООО «Селекционная станция имени Н.Н. Тимофеева». Для проведения исследования были использованы 12 линий томата, которые были высажены и выращены в открытом грунте на территории Селекционно-семеноводческого центра овощных культур ФГБОУ ВО РГАУ – МСХА имени К.А.Тимирязева в 2023 году. В общей сложности было изучено 335 растений.
Для создания искусственного инфекционного фона были собраны образцы P. infestans в естественной среде. Пораженные P. infestans листья собирали и выдерживали при температуре +4°C в течение 4 часов. Затем зараженные листья переносили в дистиллированную воду на 15 минут для создания суспензии инокулята. Полученную суспензию ооспор P. infestans использовали для двукратной инокуляции растений томата.
Раствор инокулята ооспор P. infestans наносили на все растения томата около 10 часов утра за несколько дней до цветения. Оценку устойчивости растений проводили только на основании факта наличия или отсутствия поражения фитофторозом по стандартной методике [12]. Симптомы поражения определяли визуально по характерным признакам: наличие пятен на листьях и плодах томата с белым мицелием, которые со временем увеличивались в размерах и становились черными, наличие коричневой мраморности на плодах.
Для проведения молекулярно-генетического скрининга устойчивости к P. infestans была отобрана линия томата Kp6 с чёткой фенотипической дифференциацией растений на пораженные фитофторозом растения и растения без симптомов поражения. С каждого растения было собрано 100-150 мг молодых листьев для выделения ДНК методом CTAB (цетилтриметиламмония бромид) [13]. Концентрацию и качество выделенной ДНК оценивали на спектрофотометре NanoPhotometer N50 (Implen, Германия).
Выделенную ДНК использовали для проведения ПЦР реакции по стандартной методике с 0,25 ед. ДНК-полимеразы Taq (Евроген, Россия) в соответствии с рекомендациями производителя в амплификаторе Bio-Rad T100 (Bio-Rad, США) по программе, описанной Mullis [14].
Для выявления наличия гена Ph-3 в исследуемых образцах использовали разработанный прежде для картофеля CASP (Cleaved Amplified Polymorphic Sequences) молекулярный маркер TG328 [12]. Для амплификации маркера TG328 размером 500 п.н. использовали прямой праймер 5-` GGTGATCTGCTTATAGACTTGGG-3` и обратный 5-` AAGGTCTAAAGAAGGCTGGTGC-3.`Гидролиз ампликонов проводили в течение 16 часов с использованием 0,9 ед. на 10 мкл ПЦР-продукта эндонуклеазы рестрикции BstNI. Ожидаемые фрагменты ДНК после успешного гидролиза продуктов аплификации составляют 260 и 240 п.н.
Наличие R-генов R1 и R3а в исследуемых растениях определяли при помощи разработанных ранее SCAR (Sequence-Characterized Amplified Region) молекулярных маркеров [8, 15]. Для выявления гена R1 использовали молекулярные маркеры R1 -1400 (прямой праймер 5-` CACTCGTGACAT-ATCCTCACTA-3;` обратный 5-` CAACCCTGGCATGCCACG-3)`и R1-1250 (прямой праймер 5-` CACTCGTGACATATCCTCACTA-3; обратный 5-` GTAGTACCTATCT-TATTTCTGCAAGAAT-3)` с ожидаемыми размерами продуктов амплификации 1400 п.н. и 1250 п.н. соответственно [8]. Для выявления гена R3а использовали молекулярные маркеры RT-R3a (прямой праймер 5-`ATCGTTGT-CATGCTATGAGATTGTT-3;` обратный 5-` CTTCAAGGTAGTGGGCAG-TAGCTT-3)` и R1380 (прямой праймер 5-` TCCGACATGTATTGATCTCCCTGAGCCA-3;` обратный 5-`CTTCAGCTTCTTACAGTAGG -3)`с ожидаемыми размерами продуктов амплификации 1000 п.н. и 1380 п.н. соответственно [16].
Продукты амплификации окрашивали интеркали-рующим красителем GelRed и разделяли методом электрофореза в 1,5% агарозном геле по стандартной методике. В качестве маркера молекулярных весов использовали ДНК-маркер Step100 (Биолабмикс, Россия), содержащий фрагменты ДНК длиной от 100 до 1000 п.н.
Результаты
Оценка фенотипического проявления признака устойчивости к фитофторозу на искусственном инфекционном фоне
Изучение фенотипического проявления устойчивости среди изучаемых 12 линий растений томата выявило одну линию томата, в которой растения не демонстрировали признаков поражения P. infestans, 6 линий, в которых наблюдались как пораженные P. infestans растения, так и растения без признаков поражения, и 5 линий, в которых все растения демонстрировали признаки поражения P. infestans (см. рис. 1; см. табл. 1).
Среди 6 расщепляющихся линий, в которых встречались как пораженные P. infestans растения, так и растения без следов поражения, фенотипическое расщепление по признаку отсутствия или наличия поражения патогеном варьировало от 1,6:1 до 4,4:1 (см. табл. 1). Лишь в одной линии, Кр4, соотношение соответствует 3:1. При этом стоит отметить, что после инокуляции патогеном поражение растений происходило не одновременно. В ряде случаев отдельные растения начинали демонстрировать признаки поражения P. infestans только в конце вегетационного периода.
Молекулярно-генетический скрининг генетической коллекции томата на наличие генов устойчивости к фитофторозу
Для проведения молекулярно-генетического скрининга растений томата на наличие устойчивости к фитофторозу были отобраны 12 растений линии Кр6, часть которых характеризовалась наиболее ярким фенотипическим проявлением симптомов поражения P. infestans , а другая часть демонстрировала полное отсутствие поражения патогеном.
Амплификация выделенной ДНК из растений линии Кр6 с праймерами маркера TG328, ассоциированного с геном Ph-3 , показала наличие фрагмента длиной 500 п.н. у всех исследованных растений (см. рис. 2а). Последующая обработка рестриктазой BstNI выявила наличие сайта рестрикции в амплифицированных фрагментах ДНК у растений Кр6-1, Кр6-2, Кр6-3 и Кр6-4. В этих образцах присутствовал как фрагмент 500 п.н., так и фрагмент длиной 240–260 п.н. (см. рис. 2б).

Рис.1.Симптомы поражения фитофторозом растений томата.а – растения с симптомами поражения;б – растения без симптомов поражения;в – пораженные листья,г,д – пораженные фитофторозом плоды (снизу)и плоды без симптомов поражения (сверху).Размерные линейки:5 см
Fig.1.Symptoms ofLate Blight oftom ato plants.a – plants with disease symptoms;b – plants withoutsymptoms;c – affected leaves,d,e – fruits affected by Late Blight (from below)and fruits without symptoms (from above).Size ranges:5 cm
Таблица 1. Результаты фенотипической оценки коллекции образцов томата на устойчивость к фитофторозу на искусственном инфекционном фоне
Table 1. Results of a Late Blight disease assay of a collection of tomato accessions
|
Название линии |
Общее число растений |
Число растений |
Соотношение растений с признаками поражения и без признаков поражения |
|
|
Без признаков поражения |
С признаками поражения |
|||
|
Kр1 |
24 |
0 |
24 |
- |
|
Kр2 |
28 |
0 |
28 |
- |
|
Kр4 |
32 |
24 |
8 |
3 : 1 |
|
Kр5 |
28 |
0 |
28 |
- |
|
Kр6 |
28 |
18 |
10 |
1,8 : 1 |
|
Kр7 |
32 |
26 |
6 |
4,3 : 1 |
|
Kр8 |
24 |
0 |
24 |
- |
|
Kр9 |
26 |
16 |
10 |
1,6 : 1 |
|
Kр10 |
32 |
20 |
12 |
1,67 : 1 |
|
Kр11 |
24 |
0 |
24 |
- |
|
Kр12 |
27 |
17 |
10 |
1,7 : 1 |
|
Kр14 |
30 |
30 |
0 |
- |
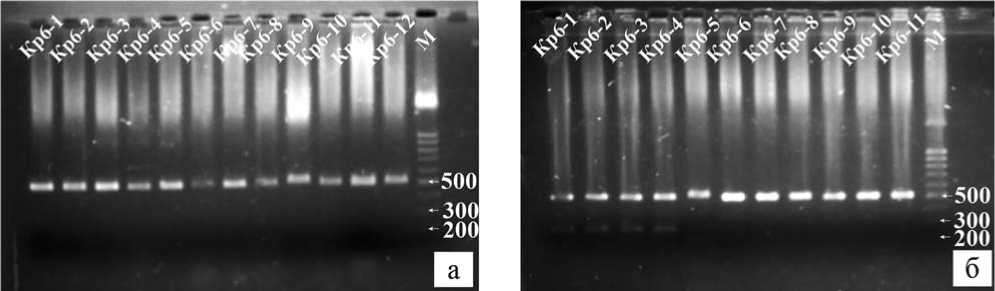
Рис.2.Результаты молекулярно-генетического скрининга на присутствие маркера TG328в линии Кр6.а – до обработки рестриктазой BstNI;б – после обработки рестриктазой BstNI.Сверху указаны номера образцов,справа отмечены длины фрагментов ДНК,М – маркер длин фрагментов ДНК
Fig.2.Results ofmoleculargeneticscreening for the presence ofthe TG328markerin the Kr6 line.a – before treatmentwith BstNI restrictase;b – after treatmentwith BstNIrestrictase.The accession numbers are shown above,the lengths ofDNA fragments are m arked on the right,and M is a m arkerfor the lengths ofDNA fragments
ПЦР с праймерами маркера R1-1400, ассоциированного с геном R1, привела к получению двух фрагментов, длинами 1400 п.н. и 1000 п.н (см. рис. 3а) у всех исследованных растений, кроме Кр6-3, Кр6-7 и Кр6-9. У растения Кр6-3 выявлялся только фрагмент 1000 п.н. В то же время амплификация с праймерами маркера R11205 привела к получению фрагмента длиной около 190 п.н. (см. рис. 3б).
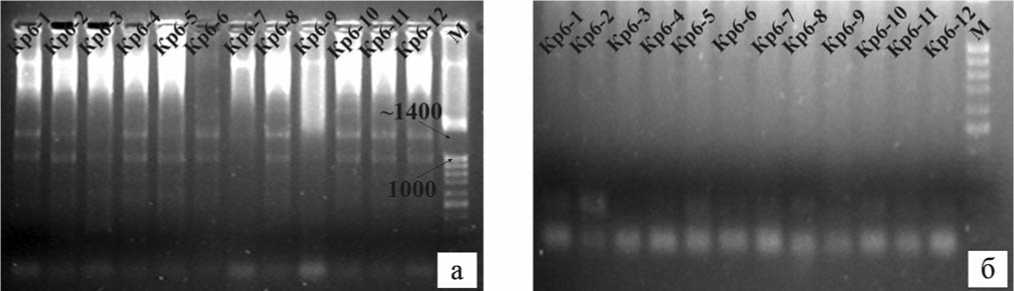
Рис.3.Результаты молекулярно-генетического скрининга на присутствие маркеров R1-1400 (а) и R1-1205 (б) генов устойчивости к фитофторозу.Сверху указаны номера образцов,M – маркер длин фрагментов ДНК
Fig.3.Results ofmoleculargeneticscreening for the presence ofmarkers R1-1400 (a)and R1-1205 (b)ofLate Blightresistance genes. The accession numbers are shown above,and M is a m arker for the lengths ofDNA fragments
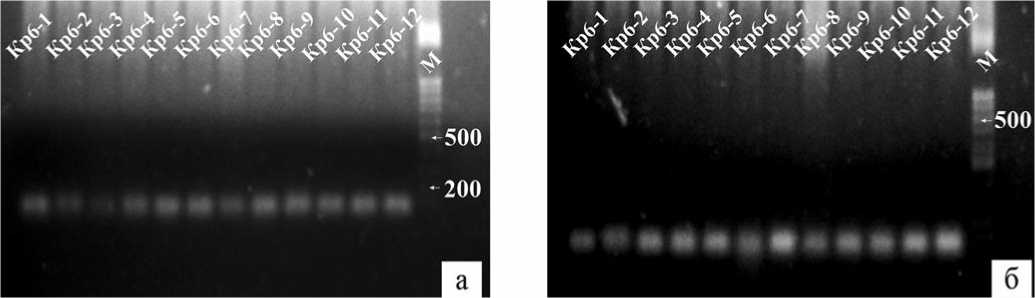
Рис.4.Результаты молекулярно-генетического скрининга на присутствие маркеров R3-1380 (в),RT-R3a (г).Сверху указаны номера образцов,M – маркер длин фрагментов ДНК
Fig.4.Results ofmolecular geneticscreening for the presence ofmarkers R3-1380 (b),RT-R3a (d).Acsession numbers are indicated above,M is a m arker for the lengths ofDNA fragments
В результате амплификации ДНК с праймерами маркеров R3-1380 и RT-R3a целевых фрагментов обнаружено не было (см. рис. 4).
Обсуждение
Наши результаты показали, что ген устойчивости Ph-3 является доминантным. Эти результаты согласуются с результатами, полученными Hansona P. et al. , которые показали в 2016 г., что ген Ph-3 устойчивости к фитофторозу является доминантным [17]. Однако к концу вегетации количество полностью устойчивых растений в расщепленных популяциях снизилось у линий, полученных от гетерозиготных родителей типа Kp6. По нашему мнению, только линии, полученные от гомозиготных доминантных родителей, таких как линия Kp14, проявили полную устойчивость к фитофторозу. Из этих анализов следует, что эффект доминирования, наблюдаемый в этом гене, не является полным доминированием, а скорее неполным. Это подтверждает результаты, полученные в 2002 году Chunwongse J. et al. , показали, что этот ген Ph-3 представляет собой ген с неполным доминированием, но чья устойчивость превосходит таковую у генов Ph-1 и Ph-2 [3].
Наличие фрагмента размером 500 п.н. указывает на отсутствие сайта рестрикции на этом фрагменте. Эта последовательность соответствует рецессивному аллелю гена устойчивости Ph-3 к фитофторозу. Наличие фрагментов размером 240 /260 п.н. свидетельствует о существовании на этом фрагменте сайта рестрикции BstN1 . Эта последовательность соответствует аллелю устойчивости. Эти два фрагмента примерно одинаковы по размерам и очень близки друг к другу (см. рис. 3). Аналогичные результаты были получены M. Mutschler и представлены R. Shekasteband et al. , в 2015 г., которые показали, что этот маркер не разделяет четко два фрагмента, образующихся в результате расщепления эндонуклеазой рестрикции [18]. Однако само наличие сайта рестрикции на этой последовательности гена свидетельствует об эффективности маркера в идентификации этого гена.
Растения Kp6-1, Kp6-2, Kp6-3 и Kp6-4 несут аллели устойчивости и восприимчивости гена Ph-3 , поэтому имеют гетерозиготный генотип (P/T). С другой стороны, растения Кр6-5, Кр6-6, Кр6-7, Кр6-8, Кр6-9, Кр6-10, Кр6-11 и Кр6-12 являются двойными носителями восприимчивос-го аллеля гена Ph-3 . Они имеют гомозиготный генотип (T/T). Хотя полная устойчивость была достигнута, когда ген Ph-3 был высоко экспрессирован под его нативным промотором, абсолютная корреляция между уровнем экспрессии гена и устойчивостью не была обнаружена.
Наличие дополнительных, еще не определенных гипостатических генов, необходимо для обеспечения полной устойчивости. Кроме того, было установлено, что существуют новые изоляты P. infestans , которые способны преодолеть ген устойчивости Ph-3 [17].
Образование нескольких продуктов в ПЦР реакции на наличие маркера R1 -1400 может говорить о присутствии нескольких аллельных вариантов этого маркера, в одном из которых произошла делеция. Отсутствие других маркеров генов R1 и R3a можно связать с невозможностью применения этих маркеров для видов вне секции Petota, к которым относятся представители рода Solanum, образующие клубни.
Подтвержденная эффективность маркера TG328 в обнаружении гена Ph-3 устойчивости томата к фитофторозу является важным преимуществом, которое может быть использовано в будущих программах селекции томата на устойчивость к этому заболеванию. Однако следует подчеркнуть, что для эффективной борьбы со всеми расами этого заболевания необходимо создавать сорта томата, сочетающие в своем геноме гены Ph-2 и Ph-3 , и по возможности другие гены устойчивости.
Заключение
Устойчивость растений томата к фитофторозу, являющегося одним из наиболее вредоносных заболеваний томата, в настоящее время может идентифицирован как на искусственном инфекционном фоне, так и с помощью молекулярных маркеров на известные гены устойчивости. Нами было показано, что маркер TG328 позволяет эффективно выявлять наличие гена Ph-3 у образцов исследованной генетической коллекции, которые могут быть в дальнейшем использованы в селекционных программах на устойчивость, основанных на использовании маркер-опо-средованного отбора. Фенотипическая оценка устойчивости томата к фитофторозу на искусственном инфекционном фоне выявила 1 линию растений без симптомов поражения заболеванием, 5 линий растений, полностью пораженных фитофторозом, и 6 линий, в которых наблюдали расщепление по проявлению симптомов поражения. Молекулярно-генетическое исследование выявило, что устойчивые растения были гетерозиготами по гену Ph-3 . Кроме того, большая часть исследованных растений имела ген R1 , который, однако, при отсутствии гена Ph-3 , не обеспечивал устойчивость растений к фитофторозу.
Таким образом, эти растения представляют ценный генетический материал для использования в селекционных программах на устойчивость томата к фитофторозу.
Об авторах:
ООО «Селекционная станция имени Н.Н. Тимофеева»,
SPIN-код: 3741-6845, –0002–6603–6933
Сократ Григорьевич Монахос – д.с.-х.н., профессор, заведующий кафедрой ботаники, селекции и семеноводства садовых растений, ФГБОУ ВО РГАУ-МСХА имени К.А. Тимирязева, SPIN-код: 7130-9663, –0001–9404–8862, Researcher ID: L-5962–2013
Sokrat G. Monakhos – Dr. Sci. (Agriculture), Professor, Head of the Botany, Plant Breeding and Seed Technology Department, Russian State Agrarian University – Moscow Timiryazev Agricultural Academy, SPIN-code: 71309663, –0001–9404–8862, Researcher ID: L-5962–2013
ISSN 2618-7132 (Online) Овощи России №5 2024 [ 17 ] Vegetable crops of Russia №5 2024 ISSN 2072-9146 (Print)
Список литературы Фенотипический и молекулярно-генетический скрининг устойчивости образцов томата к фитофторозу
- Andrivon D. Biology, ecology and epidemiology of the potato late blight pathogen Phytophthora infestans in soil. Phytopathology. 1995;85:1053-1056.
- Nowicki M., Foolad M.R., Nowakowska M., Kozik E.U. Potato and tomato late blight caused by Phytophthora infestans: an overview of pathology and resistance breeding. Plant Disease. 2012;96:1–17.
- Chunwongse J., Chunwongse C., Black L., Hanson P. Molecular mapping of the Ph-3 gene for late blight resistance in tomato. J. Hort. Sci. Biotechnol. 2002;77:281–286.
- Poudel A., Pandey M., Shah K., Acharya B., Shrestha J. Evaluation of fungicides for management of late blight (Phytophthora infestans) of potato. Agrica. 2020;9(1):10–17. https://doi.org/10.5958/2394-448X.2020.00004.8
- Mazumdar P., Singh P., Kethiravan D., Ramathani I., Ramakrishnan N. Late blight in tomato: insights into the pathogenesis of the aggressive pathogen Phytophthora infestans and future research priorities. Planta. 2021;253:119. https://doi.org/10.1007/s00425-021-03636-x
- Wang Y.Y., Chen C.H., Hoffmann A., Hsu Y.C., Lu S.F., Wang J.F., Hanson P. Evaluation of the Ph-3 gene specific marker developed for marker assisted selection of late blight resistant tomato. Plant Breeding. 2016; 135(5):636-642. https://doi.org/10.1111. pbr.12395
- Khavkin E., Sokolova E., Beketova M., Pankin A., Kuznetsova M., Kozlovskaya I., Spiglazova S., Statsyuk N., Yashina I., Potato resistance to late blight as related to the R1 and R3 genes introgressed from S. demissum. In: Schepers HTAM (ed.) PPO-Special Report no. 14. Wageningen, DLO Foundation. 2010. pp. 231-238.
- Sokolova E., Pankin A., Beketova M., Rogozina E., Kuznetsova M., Spiglazova S., Yashina I., Khavkin E. SCAR markers of the Rgenes and germplasm of wild Solanum species for breeding late blight-resistant potato cultivars. Plant Genetic Resources. 2011;9(2):309–312. https://doi.org/10.1017/ S1479262111000347
- Paluchowska P, ´Sliwka J, Yin Z. Late blight resistance genes in potato breeding. Planta. 2022;255(6):127.
- Matson M.E.H., Liang Q., Lonardi S., Judelson H.S. Karyotype variation, spontaneous genome rearrangements affecting chemical insensitivity, and expression level polymorphisms in the plant pathogen Phytophthora infestans revealed using its first chromosome-scale assembly. PLoS Pathog. 2022;18(10):e1010869. https://doi.org/10.1371/journal.ppat.1010869
- Merk H.L., Ashrafi H., Foolad M.R. Selective genotyping to identify late blight resistance genes in an accession of the tomato wild species Solanum pimpinellifolium. Euphytica. 2012;187(1):63–75. https://doi.org/10.1007/s10681-012-0729-6
- Robbins M.D., Masud M.A.T., Panthee D.R., Gardner R.G., Francis D.M., Stevens M.R. Marker-assisted selection for coupling phase resistance to Tomato spotted wilt virus and Phytophthora infestans (late blight) in tomato. Hortic. Sci. 2010;45:1424–1428.
- Doyle J. DNA Protocols for Plants. In: Hewitt, G.M., Johnston, A.W.B., Young, J.P.W. (Eds). Molecular Techniques in Taxonomy. Berlin, Heidelberg: Springer. 1991. 283-293. https://doi.org/10.1007/978-3-642-83962-7_18
- Mullis K.B. The unusual origin of the polymerase chain reaction. Sci Am. 1990;262:56–61.
- Ballvora A., Ercolano M.R., Weiss J., Meksem K., Bormann C.A., Oberhagemann P., Salamini F., Gebhardt C. The R1 gene for potato resistance to late blight (Phytophthora infestans) belongs to the leucine zipper/NBS/LRR class of plant resistance genes. Plant J. 2002;30(3):361-71. https://doi.org/10.1046/j.1365-313x.2001.01292.x
- Huang, S., Van Der Vossen, E.A.G., Kuang, H., Vleeshouwers, V.G.A.A., Zhang, N., Borm, T.J.A., Van Eck, H.J., Baker, B., Jacobsen, E. and Visser, R.G.F. Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato. The Plant Journal. 2005;42:251-261. https://doi.org/10.1111/j.1365-313X.2005.02365.x
- Hansona P., Lua S.-F., Wanga J.-F., Chena W., Kenyona L., Tana C.-W., Teeb K.L., Wanga Y.-Y., Hsua Y.-C., Schafleitnera R., Ledesmaa D., Yanga R.-Y. Conventional and molecular markerassisted selection and pyramiding of genes for multiple disease resistance in tomato. Scientia Horticulturae. 2016;201:346-354. https://doi.org/10.1016/j.scienta.2016.02.020
- Shekasteband R., Hutton S.F., Scott J.W. Designing new DNA markers and determining the effective size of Ph-2 and Ph-3 introgressions for late blight resistance stacking purposes in tomato. TGC REPORT. 2015;65:22-31.


