Филогенетический анализ штаммов рода Rhizobium, выделенных из клубеньков Vavilovia formosa (Stev.) Fed
Автор: Кимеклис А.К., Сафронова В.И., Кузнецова И.Г., Сазанова А.Л., Белимов А.А., Пинаев А.Г., Чижевская Е.П., Пухаев А.Р., Попов К.П., Андронов Е.Е., Проворов Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Микробно-растительные системы
Статья в выпуске: 5 т.50, 2015 года.
Бесплатный доступ
Из пяти родов трибы Fabeae самым малоизученным остается Vavilovia Fed., содержащий единственный вид Vavilovia formosa (Stev.) Fed. Ареал вавиловии прекрасной расположен на высокогорье Центрального и Восточного Кавказа, известны всего несколько популяций на территориях Армении, Дагестана и Северной Осетии, Азербайджана, Ирана, Ирака, Сирии и Турции, а ее произрастание ограничено условиями среды обитания. Единственное филогенетическое исследование изолятов ризобий из клубеньков вавиловии Североосетинской популяции показало достаточно широкое видовое и генетическое разнообразие как быстрорастущих, так и медленнорастущих микросимбионтов (V.I. Safronova с соавт., 2014). Было установлено, что все быстрорастущие изоляты, принадлежащие к виду Rhizobium leguminosarum, служат носителями гена nodX. Вавиловию прекрасную и ее микросимбионтов можно рассматривать как перспективные модельные объекты для исследования механизмов эволюции специфичности бобово-ризобиального симбиоза. В результате трех экспедиций в горные регионы Армении, Дагестана и Северной Осетии были найдены и собраны образцы эндемичного бобового растения Vavilovia formosa (Stev.) Fed. с клубеньками, из которых впоследствии получены изоляты клубеньковых бактерий (ризобий). Мы выбрали 19 штаммов быстрорастущих ризобий, выделенных из клубеньков 10 растений, чтобы определить их видовую принадлежность, выявить географическую изоляцию, а также охарактеризовать генетическую обособленность выделенных штаммов по отношению к ризобиям, имеющим других растений-хозяев из трибы Fabeae. Для этого были просеквенированы один из фрагментов ITS (internally transcribed spacer) длиной 1000-1300 п.н. и фрагмент гена nodA длиной 666 п.н., а также проведен скрининг штаммов на наличие гена nodX, контролирующего хозяйскую специфичность ризобий. На основании полученных последовательностей были рассчитаны генетические расстояния между группами ризобий: изолятами из разных регионов (Армения, Дагестан, Северная Осетия) и от разных растений-хозяев (вавиловия, горох, клевер). Результаты секвенирования фрагмента ITS показали, что все изученные штаммы принадлежат к виду Rhizobium leguminosarum (биовар viciae ). ITS-дендрограмма выявила относительно высокую гетерогенность внутри группы изолятов, однако на nodA -дендрограмме они, напротив, формировали очень компактную группу. Несовпадение этих дендрограмм позволяет предположить, что ген nodA, сцепленный с генами хозяйской специфичности у ризобий, может активно переноситься в популяциях R. leguminosarum, обеспечивая свободное комбинирование специфичности к вавиловии с различными вариантами бактериальной хромосомы. Сравнение генетических расстояний по ITS для изолятов из трех регионов указывает на дивергенцию микросимбионтов вавиловии, связанную с географической изоляцией. Данные по генетическим расстояниям для гена nodA, а также присутствие гена nodX у всех изученных штаммов R. leguminosarum указывают на наличие хозяйской обособленности изученных штаммов внутри биовара viciae. Полученные корреляции происхождения штаммов со структурой гена nodA, по всей видимости, отражают наличие высокоспецифичных взаимодействий каждой группы штаммов R. leguminosarum со своими растениями-хозяевами, тогда как корреляции со структурой локуса ITS свидетельствуют об адаптации ризобий к почвенной среде обитания.
Бобово-ризобиаль-ный симбиоз
Короткий адрес: https://sciup.org/142133629
IDR: 142133629 | УДК: 579.64:631.461.52:577.2 | DOI: 10.15389/agrobiology.2015.5.655rus
Текст научной статьи Филогенетический анализ штаммов рода Rhizobium, выделенных из клубеньков Vavilovia formosa (Stev.) Fed
Триба Fabeae (син. Vicieae ) — одна из наиболее представительных в семействе Бобовые ( Fabaceae Endl.). Она состоит более чем из 300 видов, причем ее таксономическая структура до сих пор подвергается существенным изменениям. Многие виды этой трибы имеют важное сельскохозяйственное значение. В ее состав входят одни из самых древних культивируемых видов растений — горох посевной ( Pisum sativum L.) и вика посев-
Работа поддержана грантом Российского научного фонда 14-26-00094. Исследования выполняли с использованием оборудования ЦКП «Геномные технологии, протеомика и клеточная биология» (ФГБНУ Всероссийский НИИ сельскохозяйственной микробиологии, г. Санкт-Петербург—Пушкин).
ная ( Vicia sativa L.) (1). Из пяти родов трибы Fabeae ( Lathyrus L., Vicia L., Lens Mill., Pisum L. и Vavilovia Fed.) самым малоизученным остается последний, содержащий единственный вид Vavilovia formosa (Stev.) Fed. Ареал вавиловии расположен на высокогорье Центрального и Восточного Кавказа, известны всего несколько популяций на территориях Армении (2), Дагестана и Северной Осетии (3), Азербайджана (4), Ирана, Ирака, Сирии и Турции (5), а ее произрастание ограничено условиями среды обитания (6). Поэтому само растение и его микросимбионты до недавнего времени оставались неизученными.
Недавно полученные данные по таксономическому положению ва-виловии прекрасной, основанные на филогенетически значимых генах matK , trnL-F и trnS-G , а также фрагменте ITS (internally transcribed spacer), свидетельствуют, что она наиболее близка к родам Pisum и Lathyrus , однако формирует отдельный кластер внутри трибы Fabeae (7). Единственное филогенетическое исследование изолятов ризобий из клубеньков вавило-вии североосетинской популяции показало достаточно широкое видовое и генетическое разнообразие как быстрорастущих, так и медленнорастущих микросимбионтов (8).
В той же работе было установлено, что все быстрорастущие изоля-ты, принадлежащие к виду R. leguminosarum , несут ген nodX , характерный для симбионтов Pisum sativum сорта Afghanistan (9). Все известные растения трибы Fabeae формируют группу перекрестной инокуляции и образуют клубеньки с бактериями этого же вида, причем для формирования симбиоза наличие гена nodX в большинстве случаев не обязательно. Однако в геноме P. sativum cv. Afghanistan (так называемого афганского гороха) присутствует аллель sym2A , кодирующий рецептор, который специфично связывается только с Nod-фактором, декорированным дополнительной ацетильной группой, присоединенной под контролем гена nodX (10).
В настоящее время направление по изучению роли гена nodX в обеспечении индивидуального взаимодействия между бобовым растением и ризобиальными бактериями развивается очень активно (11). Представленные выше данные позволяют считать вавиловию прекрасную и ее микросимбионтов перспективными модельными объектами для исследования механизмов эволюции специфичности в бобово-ризобиальном симбиозае.
Благодаря удачным экспедициям в различные регионы Кавказа, наша исследовательская группа располагает образцами растений вавило-вии прекрасной и штаммов ризобий, выделенных из их клубеньков.
Целью настоящей работы было определение видовой принадлежности быстрорастущих изолятов ризобий из клубеньков вавиловии прекрасной на основании последовательностей фрагмента ITS, а также выявление особенностей хозяйской специфичности, которые могут быть оценены при помощи филогенетического анализа генов nodA и nodX . Мы также провели сравнение генетических расстояний, оцененных по последовательностям ITS и nodA в генотипе штаммов ризобий, выделенных из клебеньков разных растений-хозяев в различных географических регионах, для выявления экологически значимых показателей полиморфизма изучаемых изолятов.
Методика. Растения вавиловии прекрасной с клубеньками были собраны в 2012-2013 годах в Северо-Осетинском заповеднике (Северная Осетия, Алагирский р-н), Армении и Дагестане на высоте более 1500 м. В настоящей работе были изучены 19 штаммов ризобий из клубеньков 10 растений вавиловии, принадлежавших к трем разным популяциям.
Штаммы ризобий изолировали из клубеньков V. formosa с исполь- зованием стандартной методики (12). Из каждого клубенька получали по одному штамму, который выращивали на питательной среде ¹ 79 (13) при 28 °С. Все изоляты были депонированы в Ведомственную коллекцию полезных микроорганизмов сельскохозяйственного назначения (ВКСМ) и хранятся на станции низкотемпературного автоматизированного хранения биологических образцов при -80 °С («Liconic Instruments», Лихтенштейн) (14). Информация об этих штаммах доступна online в базе данных ВКСМ .
Для филогенетического анализа из штаммов ризобий выделяли геномную ДНК по стандартному протоколу (15) и амплифицировали фрагмент ITS (intergenic transcribed spacer) длиной 1000-1300 п.н. с помощью праймеров FGPS1490-72 5'-TGCGGCTGGATCCCCTCCTT-3' и FGPL132'-38 5'-CCGGGTTTCCCCATTCGG-3' (16, 17), а также фрагмент гена nodA длиной 666 п.н. с помощью праймеров nodA-1 5'-GCRGTGGAARNTRN-NCTGGGAAA-3' и nodA-2 5'-GGNCCGTCRTCRAASGTCARGTA-3' (18). ПЦР проводили в объеме 25 мкл, содержащем 150 мкМ смеси dNTPs («Heli-con», Россия), 1 ед. Taq-полимеразы («Евроген», Россия), по 10 пМ каждого из праймеров, а также по 10-20 нг очищенной ДНК в качестве матрицы. Амплификацию осуществляли на автоматическом амплификаторе T100 («Bio-Rad», США) при следующем температурном режиме: стартовая денатурация — 2 мин при 95 °С; 35 циклов денатурации — 30 с при 94 °С, 30 с отжига при 50 °С (праймеры FGPS1490-72/FGPL132'-38) или 49 °С (праймеры nodA-1/nodA-2), 1 мин элонгации при 72 °С; финальная элонгация — 3 мин при 72 °С. Результаты ПЦР визуализировали с помощью электрофореза в 1 % агарозном геле («Amresco», США) в 0,5½ TAE, маркер молекулярной массы — ДНК-маркер 100 bp + 1,5 Kb + 3 Kb («Сибэнзим», Россия). Очистку ПЦР-продуктов проводили согласно стандартной методике (19). Последовательности ITS и гена nodA секвенировали на генетическом анализаторе ABI PRISM 3500xl («Applied Biosystems», США), для их первичной обработки применяли программу UGENE («Unipro», Россия) (20). Для полученных последовательностей были найдены схожие последовательности из баз данных GeneBank при помощи анализа BLAST (Basic logical alignment search tool) в NCBI (National Center for Biotechnology Information, . На основе этих данных с использованием метода ближайших соседей в программе MEGA v. 5.0 были сконструированы два филогенетических дерева (21). Для оценки эволюционных расстояний использовали метод p-distance, для статистической поддержки кластеров — метод bootstrap с 1 тыс. реплик.
Наличие гена nodX в изолятах проверяли с помощью ПЦР с праймерами oMP199-F 5'-CCATGGGACCATCCAATGAAC-3' и oMP196-R 5'-TTAAGCGACGGAAAGCCTTC-3' (22). Состав реакционной смеси и температурный режим были аналогичны описанным выше, за исключением температуры отжига, равной 53 °С. В качестве положительного контроля использовали штамм R. leguminosarum bv. viciae A1 (22), обладающий способностью формировать клубеньки на афганских формах гороха.
Для последовательностей фрагментов ITS и гена nodA вычисили генетические расстояния как внутри разных таксономических и географических групп ризобий, так и между ними. Сравнение генетических расстояний для последовательностей ITS и nodA проводили между двумя разными наборами групп микроорганизмов. Это корреляция штаммов R. legu-minosarum bv. viciae (ризобии из гороха), R. leguminosarum bv. trifolii (ризо-бии из клевера) и исследуемых штаммов с растением-хозяином, а также корреляция изолятов из вавиловии с их географическим происхождением.
Были проведены как внутри-, так и межгрупповые сравнения генетических расстояний. Генетические расстояния оценивали по параметру p-distance в программном пакете MEGA v. 5.0 (21).
Результаты . Фрагменты ITS удалось амплифицировать и секвени-ровать полностью или частично для всех изученных штаммов, при этом у изолятов из Дагестана в геноме оказалось как минимум две разные копии рибосомального оперона, содержащие фрагменты ITS разного размера. На дендрограмме, построенной на основе секвенирования ITS (рис. 1), эти последовательности помечены литерами l (long) и s (short).
В таблице 1 указаны длины полученных фрагментов и их сходство с ITS трех типовых штаммов внутри вида R. leguminosarum , один из которых (штамм ТОМ) содержит ген nodX . Геномы этих штаммов секвениро-ваны и доступны online в базе данных NCBI. Примечательно, что по ITS основная часть штаммов, выделенных из вавиловии, обладает большим сходством с биоваром viciae , чем с биоваром trifolii : средние коэффициенты сходства с последовательностями типовых штаммов составляли 83±0,9 % для штамма 3841, 89±0,8 % — для ТОМ и лишь 80±1,0 % — для WSM2304.
1. Происхождение изолятов из клубеньков вавиловии прекрасной Vavilovia formosa (Stev.) Fed. и сходство последовательностей их ITS-фрагментов с последовательностями референтных штаммов из баз данных GenBank
|
Штамм |
¹ растения |
Длина фрагмента ITS, п.н. |
Сходство с последовательностями типовых штаммов, % |
|||
|
Rhizobium legumino-sarum bv. viciae 3841 |
Rhizobium legumino- |
Rhizobium leguminosa-rum bv. trifolii WSM2304 |
||||
|
sarum bv. |
viciae TOM |
|||||
|
Северная Осетия |
||||||
|
Vaf-01 |
1 |
565 |
96 |
86 |
90 |
|
|
Vaf-09 |
613 |
75 |
90 |
75 |
||
|
Vaf-10 |
3 |
1206 |
83 |
95 |
81 |
|
|
Vaf-12 |
1184 |
85 |
89 |
88 |
||
|
Vaf-23 |
4 |
565 |
99 |
88 |
100 |
|
|
Vaf-25 |
649 |
80 |
99 |
72 |
||
|
Vaf-26 |
5 |
1113 |
91 |
94 |
91 |
|
|
Армения |
||||||
|
Vaf-45 |
7 |
1199 |
82 |
93 |
80 |
|
|
1010 |
78 |
90 |
79 |
|||
|
Vaf-72 |
1199 |
82 |
93 |
80 |
||
|
1010 |
78 |
90 |
79 |
|||
|
Vaf-46 |
8 |
1199 |
82 |
93 |
80 |
|
|
1010 |
78 |
90 |
79 |
|||
|
Vaf-51 |
1199 |
82 |
93 |
80 |
||
|
1010 |
78 |
90 |
79 |
|||
|
Дагестан |
||||||
|
VD1/1k |
1D |
1105 |
92 |
86 |
87 |
|
|
VD1/9m |
1287 |
86 |
94 |
75 |
||
|
VD3/2(1) |
3D |
1032 |
82 |
83 |
77 |
|
|
VD3/7k |
1032 |
82 |
83 |
77 |
||
|
VD6/12k |
6D |
1032 |
82 |
83 |
77 |
|
|
VD6/13m |
1032 |
82 |
83 |
77 |
||
|
VD7/1 |
7D |
1032 |
82 |
83 |
77 |
|
|
VD7/20m |
1032 |
82 |
83 |
77 |
||
ITS-дендрограмма показывает, что все исследуемые штаммы относились к семейству Rhizobiaceae и попадали в две группы бактериальных видов, несущие разные типы рибосомального оперона (кластеры I и II). Причем принадлежность к одной или другой группе не зависела от вида штамма или географического распространения. Виды R. leguminosarum bv. viciae , R. leguminosarum bv. trifolii , R. etli присутствовали в обеих группах, как и разные изоляты из вавиловии прекрасной армянского и дагестанского происхождения. Возможно, такое деление было обусловлено наличием лишь нескольких типов хромосом, обеспечивающих адаптацию к среде и жизнеспособность бактерии.
Штаммы ризобий, выделенные из клубеньков вавиловии, не фор-
VD3/2(1) VD3/7k
99 VD6/12k VD6/13m VD7/1 VD7/20m Vaf-45s Vaf-51s Vaf-72s Vaf-46s
---Mesorhizobium sp. WSM4349
-
- Bradyrhizobium japonicum USDA 6
Bradyrhizobium sp. BTAil
---Sinorhizobium mediate WSM419
----Sinorhizobium meliloti 2011
-
— Sinorhizobium fredii USDA 257

----Rhizobium leguminosarum bv. viciae 248 ------------Rhizobium sp. LPU83 ----------Rhizobium etli CFN 42
Rhizobium leguminosarum bv. viciae WSM1455
—Rhizobium leguminosarum bv. trifolii WSM2012
-
- Rhizobium gallicum bv. gallicum R602 /
--------Neorhizobium galegae bv. officinalis НАМ ВI A Rhizobium leguminosarum bv. trifolii WSM597 -Rhizobium leguminosarum bv. trifolii WSM2304 ■Rhizobium etli bv. mimosae Mimi
Vaf-451
Vaf-461
Vaf-721
Vaf-511
2i *-VDl/lk ___iVaf-01 WVaf-12
62г Rhizobium leguminosarum bv. viciae TOM*
87J-Vaf-25
65 1 VDl/9m
| Северная Осетия ^ Дагестан
I
II
Северная Осетия
------Rhizobium leguminosarum bv. trifolii WSM2297
—Rhizobium leguminosarum bv. phaseoli 4292
^—Rhizobium leguminosarum bv. viciae 3841
20[
-Rhizobium leguminosarum bv. viciae WSM1481
■iRhizobium leguminosarum bv. trifolii WSM1325
Vaf-26
Vaf-23
^j Северная Осетия
0,05
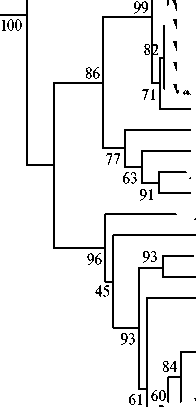
Рис. 1. Филогенетическое дерево, построенное с использованием метода ближайших соседей на основе фрагмента последовательности ITS (758 п.н.) и отражающее таксономическое положение исследованных штаммов ризобий из клубеньков вавиловии прекрасной ( Vavilovia formosa (Stev.) Fed.) разного географического происхождения. На ветвях указаны значения bootstrap > 45 (1000 реплик). I, II — статистически достоверные кластеры. Звездочкой помечен штамм-носитель гена nodX ; s (short) и l (long) — штаммы из Армении, несущие две разные копии фрагмента ITS. Жирным шрифтом выделены изученные штаммы.
мировали отдельного кластера, но распадались на группы. По составу этих групп можно было судить о тенденции изучаемых изолятов к географической изоляции. Все штаммы из Армении, а также 6 из 8 штаммов из Дагестана образовывали кластеры с высокой статистической поддержкой (> 82 %) и входили в I группу. Фрагменты ITS штаммов вавило-вии из Северной Осетии встречались только во II группе, и эти штаммы объединялись со штаммами из своего региона (Vaf-01 и Vaf-12, Vaf-09 и
Vaf-10, Vaf-23 и Vaf-26). Исключением стал штамм Vaf-25, который образовывал одну группу со штаммами VD1/9m из Дагестана и R. leguminosa-rum bv. viciae TOM (носителем гена nodX ) при уровне поддержки 87 %. Подобная кластеризация была показана ранее только для изолятов из Северной Осетии (8). В связи с этим мы проверили изоляты из регионов Дагестана и Армении на наличие гена nodX (рис. 2). Все они оказались носителями этого гена.
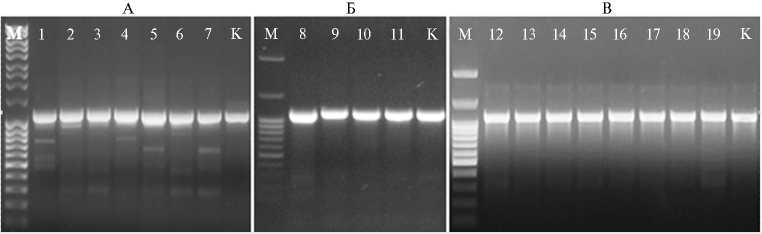
Рис. 2. Результат ПЦР анализа североосетинских (А) , армянских (Б) и дагестанских (В) изо-лятов ризобий из клубеньков вавиловии прекрасной Vavilovia formosa (Stev.) Fed. с праймерами omP196/omP199 : 1 — Vaf-01, 2 — Vaf-09, 3 — Vaf-10, 4 — Vaf-12, 5 — Vaf-23, 6 — Vaf-25, 7 — Vaf-26, 8 — Vaf-45, 9 — Vaf-72, 10 — Vaf-46, 11 — Vaf-51, 12 — VD1/1k, 13 — VD1/9m, 14 — VD3/2(1), 15 — VD3/7k, 16 — VD6/12k, 17 — VD6/13m, 18 — VD7/1, 19 — VD7/20m. Контроль (К) — ДНК штамма Rhizobium leguminosarum bv. viciae А1. Маркеры молекулярной массы (М) — MassRuler (Thermo Scientific, США) (А) и 100 bp + 1,5 Kb + 3Kb («Сибэнзим», Россия) (Б, В).
Важно отметить, что последовательность ITS, которая представляет коровую часть генома, дает нам понимание только таксономической принадлежности изучаемых штаммов. Вопрос о хозяйской специфичности ри-зобий можно решить, изучая гены, вовлеченные в образование бобово-ризобиального симбиоза. К ним относятся гены из nod оперонов (23). В нашем исследовании мы рассматриваем ген nodA , который, хоть и не участвует в непосредственном контроле специфичности симбиоза, сцеплен с генами, кодирующими эти факторы специфичности. Преимущество использования именно этого гена заключается в том, что в геномах ризобий он представлен только одной копией (18).
Дендрограммы, построенные на основе последовательностей ITS-региона и гена nodA , существенно различались между собой (рис. 1, 3). На nodA -дендрограмме все исследуемые изоляты формировали единый кластер при уровне поддержки 100 % со штаммами, принадлежащими R. leguminosarum bv. viciae , что позволяет отнести их к группе перекрестной инокуляции гороха. На основании анализа гена nodA внутри этого кластера изоляты были разделены на три группы, не соответствующие ITS-группам. Так, изоляты из Северной Осетии и Дагестана совместно сформировали группы I и III, а Армянские изоляты вошли только в группу II (см. рис. 3). Стоит отметить, что на nodA -дендрограмме штамм R. legumi-nosarum bv. viciae TOM оказался наиболее близкородственным по отношению к изучаемым изолятам. Еще одна немаловажная особенность, позволяющая судить об обособленности изучаемой группы штаммов, состоит в том, что во всех трех кластерах, содержащих изоляты из вавиловии, отсутствовали штаммы из других таксономических групп.
По данным сравнения фрагментов ITS, связь с хозяйской специфичностью у разных групп ризобий не прослеживалась, поскольку значения внутригрупповых и межгрупповых генетических расстояний достоверно не различались (табл. 2). Напротив, можно делать выводы о тенденции 660
к географической изоляции изучаемых микросимбионтов вавиловии.
IQOl
loot
XQQ] Bradyrhizobium japonicum USDA 6 ' Bradyrhizobium sp. CCBAU 41267
---------Mesorhizobium sp. WSM4349
\Rhizobium leguminosarum bv. phaseoli 4292 ^^Rhizobium etli CFN 42
-Rhizobium gallicum bv. gallicum R602
--------------Neorhizobium galegae bv. officinalis НАМ ВI
BOI Sinorhizobium meliloti 2011
---Sinorhizobium medicae WSM419
—Rhizobium leguminosarum bv. trifolii WSM2297
—Rhizobium leguminosarum bv. trifolii WSM2012
—Rhizobium leguminosarum bv. trifolii WSM1325
-----Rhizobium leguminosarum bv. trifolii SVSM597
----Rhizobium leguminosarum bv. trifolii WSM2304
12\Bbizobium leguminosarum bv. viciae WSM1481
100 'Rhizobium leguminosarum bv. viciae WSM1455 \Rhizobium leguminosarum bv. viciae 248
VyRhizobium leguminosarum bv. viciae 3841
---Rhizobium leguminosarum bv. viciae TOM*
Vaf-23
Vaf-26
Vaf-10
Vaf-09
__Vaf-01
100 VD7/20m
VD7/1
VD6/13k
VD3/2m
VD6/12m
Vaf-46
Vaf-45
Vaf-72
Vaf-51
Vaf-12
VDl/9m
100 VDl/lk
-Vaf-25
I
II
III
0,05
Рис. 3. Филогенетическое дерево, построенное с использованием метода ближайших соседей на основе частичной последовательности гена nodA (531 п.н.) и отражающее таксономическое положение исследованных штаммов ризобий из клубеньков вавиловии прекрасной Vavilovia formosa (Stev.) Fed. разного географического происхождения. На ветвях указаны значения bootstrap > 45 (1000 реплик). I, II, III — статистически достоверные кластеры. Звездочкой помечен штамм-носитель гена nodX . Жирным шрифтом выделены изученные штаммы.
2. Генетические расстояния по последовательностям ITS и nodA у ризобий, выделенных от разных растений-хозяев в различных географических регионах
|
Штаммы, происхождение |
Генетические |
расстояния |
|
ITS |
nodA |
|
|
Корреляция последовательносте |
й ДНК с растение |
м-хозяином |
|
Сравнения внутри групп: |
||
|
из вавиловии ( Rhizobium leguminosarum bv. viciae ) |
0,082±0,008 |
0,036±0,005 |
|
из гороха ( Rhizobium leguminosarum bv. viciae ) |
0,115±0,010 |
0,016±0,003 |
|
из клевера ( R. leguminosarum bv. trifolii ) Межгрупповые сравнения: |
0,089±0,008 |
0,119±0,010 |
|
вавиловия—горох |
0,096±0,008 |
0,045±0,006 |
|
вавиловия—клевер |
0,099±0,008 |
0,252±0,015 |
|
горох—клевер |
0,100±0,008 |
0,254±0,016 |
Продолжение таблицы 2
|
Корреляция последовательностей ДНК c географическим происхождением |
||
|
Сравнение внутри групп: Армения |
0,000±0,000 |
0,001±0,001 |
|
Дагестан |
0,054±0,007 |
0,040±0,005 |
|
Северная Осетия |
0,050±0,006 |
0,040±0,005 |
|
Межгрупповые сравнения: Армения—Дагестан |
0,116±0,012 |
0,039±0,006 |
|
Армения—Северная Осетия |
0,060±0,007 |
0,039±0,006 |
|
Дагестан—Северная Осетия |
0,111±0,011 |
0,034±0,005 |
В то же время при сравнении генетических расстояний по гену nodA выявлялись четкие корреляции с хозяйской специфичностью, однако связь с географическим происхождением изолятов вавиловии отсутствовала. Поскольку ген nodA не участвует в контроле хозяйской специфичности (он кодирует общий для всех ризобий этап синтеза Nod-фактора — присоединение жирнокислотного остатка к олигохитиновой цепи), данная корреляция может быть обусловлена сцеплением nodA с генами хозяйской специфичности (эти гены образуют на Sym -плазмидах R. leguminosarum компактные кластеры размером около 20 т.п.н.). Полученные корреляции, по всей видимости, отражают наличие высокоспецифичных взаимодействий каждой группы штаммов R. leguminosarum со своими растениями-хозяевами.
Несовпадение дендрограмм, построенных по nodA и ITS, позволяет предположить, что находящиеся на плазмидах nod -гены могут активно переноситься в популяциях R. leguminosarum , обеспечивая свободное комбинирование бактериальных генов специфичности к вавило-вии и различных вариантов хромосом у штаммов биовара viciae , а в некоторых случаях и биовара trifolii (последнее подтверждается 100 % сходством ITS-локуса одного из изолятов вавиловии с типовым штаммом WSM2304 и соответствует ранее опубликованным данным о возможности активного переноса Sym -плазмид между этими биоварами в природных популяциях).
Таким образом, на основании данных по последовательности ITS показано, что все изученные штаммы ризобий, выделенные из клубеньков вавиловии прекрасной, принадлежат к виду Rhizobium leguminosarum . Принимая во внимание результаты филогенетического анализа локуса ITS, а также гена nodA , можно утверждать, что эти штаммы относятся к биовару viciae . Согласно представленным генетическим расстояниям, все они образуют статистически обособленную группу, внутри которой прослеживается тенденция к географической изоляции и узкой хозяйской специфичности. Формирование обособленных от других штаммов R. legumi-nosarum bv. viciae кластеров, а также наличие гена нодуляции афганского гороха nodX у всех изолятов, выделенных из Vavilovia formosa , могут послужить основой для дальнейшего изучения этой группы клубеньковых бактерий и ее возможного выделения в обособленную таксономическую группу внутри вида R. leguminosarum.
Список литературы Филогенетический анализ штаммов рода Rhizobium, выделенных из клубеньков Vavilovia formosa (Stev.) Fed
- Ljuština M., Miki A. Archaeological evidence for the domestication of lentil (Lens culinaris) and its distribution in Europe. J. Lentil Res., 2010, 4: 26-29.
- Габриелян Е.Т. Род Vavilovia Fed. В кн.: Флора Армении. Т. 4/Под ред. А.Л. Тахтаджяна. АН ССР Армения, Ереван, 1962.
- Дзюбенко Н.И., Дзюбенко Е.А. Vavilovia formosa (Stev.) An. Fed. Агроэкологический атлас России и сопредельных стран: экономически значимые растения, их вредители, болезни и сорные растения/Под ред. А.Н. Афонина, С.Л. Грина, Н.И. Дзюбенко, А.Н. Фролова. 2008 (online http://www.agroatlas.ru/ru/content/related/Vavilovia_formosa/).
- Карягин И.И. Флора Азербайджана. Т. 5. Баку, 1954.
- Maxted N., Ambrose M. Peas (Pisum L.). In: Plant genetic resources of legumes in the Mediterranean/N. Maxted, S.J. Bennett (eds.). Kluwer, Dordrecht, 2001.
- Makasheva R.K., Drozd A.M., Adamova O.P., Golubev A.A. Perennial pea. Bull. Appl. Bot. Gen. Plant Breed., 1973, 51: 44-56.
- Kenicer G., Smýkal P., Vishyakova M., Mikić A. Vavilovia formosa, an intriguing Pisum relative. Grain Legumes, 2009, 51: 8-12.
- Safronova V.I., Kimeklis A.K., Chizhevskaya E.P., Belimov A.A., Andronov E.E., Pinaev A.G., Pukhaev A.R., Popov K.P., Tikhonovich I.A. Genetic diversity of rhizobia isolated from nodules of the relic species Vavilovia formosa (Stev.) Fed. Antonie van Leeuwenhoek, 2014, 105: 389-399 ( ) DOI: 10.1007/s10482-013-0089-9
- Ma S.W., Iyer V.N. New field isolates of Rhizobium leguminosarum biovar viciae that nodulate the primitive pea cultivar Afghanistan in addition to modern cultivars. Appl. Environ. Microbiol., 1990, 56: 2206-2212.
- Kozik A., Heidstra R., Horvath B., Kulikova O., Tikhonovich I., Ellis T.H.N., van Kammen A., Lie T.A., Bisseling T. Pea lines carrying syml or sym2 can be nodulated by Rhizobium strains containing nodX; sym1 and sym2 are allelic. Plant Sci., 1995, 108: 41-49 ( ) DOI: 10.1016/0168-9452(95)04123-C
- Zhukov V., Radutoiu S., Madsen L.H., Rychagova T., Ovchinnikova E., Borisov A., Tikhonovich I., Stougaard J. The pea Sym37 receptor kinase gene controls infection-thread initiation and nodule development. MPMI, 2008, 21(12): 1600-1608 ( ) DOI: 10.1094/MPMI-21-12-1600
- Novikova N., Safronova V. Transconjugants of Agrobacterium radiobacter harbouring sym genes of Rhizobium galegae can form an effective symbiosis with Medicago sativa. FEMS Microbiol Lett., 1992, 93: 261-268 ( ) DOI: 10.1111/j.1574-6968.1992.tb05107.x
- Allen O.N. Experiments in soil bacteriology. Burgess Publishing Co., Minneapolis, 3d ed., 1959: 117.
- Safronova V.I., Tikhonovich I.A. Automated cryobank of microorganisms: Unique possibilities for long-term authorized depositing of commercial microbial strains. In: Microbes in applied research: current advances and challenges/A. Mendez-Vilas (ed.). World Scientific Publishing Co., 2012: 331-334 ( ) DOI: 10.1142/9789814405041_0066
- Румянцева М.Л., Симаров Б.В., Онищук О.П., Андронов Е.Е., Чижевская Е.П., Белова В.С., Курчак О.Н. Биологическое разнообразие клубеньковых бактерий в экосистемах и агроценозах: теоретические основы и методы. СПб-Пушкин, 2011.
- Normand P., Ponsonnet C., Nesme X., Neyra M., Simonet P. ITS analysis of prokaryotes. In: Molecular microbial ecology manual/A.D.L. Akkermans, J.D. van Elsas, F.J. de Bruijn (eds.). Kluwer Academic Publishers, Dordrecht, Netherlands, 1996: 1-12.
- Ponsonnet C., Nesme X. Identification of Agrobacterium strains by PCR-RFLP analysis of pTi and chromosomal regions. Arch. Microbiol., 1994, 161: 300-309 ( ) DOI: 10.1007/BF00303584
- Haukka K., Lindstrom K., Young J.P. Three phylogenetic groups of nodA and nifH genes in Sinorhizobium and Mesorhizobium isolates from leguminous trees growing in Africa and Latin America. Appl. Environ. Microbiol., 1998, 64: 419-426.
- Андронов Е.Е., Пинаев А.Г., Першина Е.В., Чижевская Е.П. Научно-методические рекомендации по выделению высокоочищенных препаратов ДНК из объектов окружающей среды. СПб, 2011.
- Okonechnikov K., Golosova O., Fursov M., the UGENE team. Unipro UGENE: a unified Bioinformatics toolkit. Bioinformatics, 2012, 28: 1166-1167 ( ) DOI: 10.1093/bioinformatics/bts091
- Tamura K., Peterson D., Peterson N., Stecher G., Nei M., Kumar S. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol., 2011, 28: 2731-2739 ( ) DOI: 10.1093/molbev/msr121
- Ovtsyna A.O., Rademaker G.-J., Esser E., Weinman J., Rolfe B.G., Tikhonovich I.A., Lugtenberg B.J.J., Thomas-Oates J.E., Spaink H.P. Comparison of characteristics of the nodX genes from various Rhizobium leguminosarum strains. MPMI, 1999, 12: 252-258 ( ) DOI: 10.1094/MPMI.1999.12.3.252
- Laguerre G., Nour S.M., Macheret V., Sanjuan J., Drouin P., Amarger N. Classification of rhizobia based on nodC and nifH gene analysis reveals a close phylogenetic relationship among Phaseolus vulgaris symbionts. Microbiology, 2001, 147: 981-993 ( ) DOI: 10.1099/00221287-147-4-981


