Филогенетическое положение галотолерантного штамма Brevibacterium sp. U1, выделенного из засоленной почвы, в системе рода Brevibacterium
Автор: Ананьина Людмила Николаевна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Генетика
Статья в выпуске: 4, 2019 года.
Бесплатный доступ
Исследовано филогенетическое положение галотолерантного штамма Brevibacterium sp. U1, выделенного ранее из почвы района промышленной соледобычи (г. Березники, Пермский край), в системе рода Brevibacterium на основе анализа нуклеотидных последовательностей ect-генов, кодирующих ферменты биосинтеза осмопротекторного вещества - этоина, и 16S рДНК. Анализ нуклеотидных последовательностей генов ectB и ectC согласовался с результатами анализа 16S рДНК. Однако было выявлено значительное эволюционное расстояние нуклеотидных последовательностей ectC-гена штамма Brevibacterium sp. U1 от филогенетически близкородственного вида B. aurantiacum.
16s рднк, ect-гены, филогения
Короткий адрес: https://sciup.org/147227112
IDR: 147227112 | УДК: 579.871.8 | DOI: 10.17072/1994-9952-2019-4-459-463
Текст научной статьи Филогенетическое положение галотолерантного штамма Brevibacterium sp. U1, выделенного из засоленной почвы, в системе рода Brevibacterium
Представители рода Brevibacterium семейства Brevibacteraceae класса Actinobacteria, выделены из молокосодержащих продуктов, включая сыры, встречаются у людей и птиц в качестве комменсалов или условно-патогенных микроорганизмов; населяют морские и почвенные экосистемы [Ivanova et al., 2004]. Особое внимание исследователи уделяют ассоциированным с созреванием сыра бревибактериям. Поскольку в процессе производства сыров их поверхность солят или погружают в насыщенный рассол, изучена осмоадапация бревибактерий к условиям высокой осмолярности внешней среды. На примере биотехнологически значимого вида B. linens было показано, что преобладающим осмопротектором является эктоин [Bernard et al., 1993].
Последующее изучение геномов представителей рода Brevibacterium показало наличие генов, кодирующих биосинтез эктоина у большинства исследованных видов, за исключением видов, ассоциированных с человеком [Pham et al., 2017]. В других работах выявлена колинеарность эволюционных расстояний между нуклеотидными последовательностями 16S рДНК и аминокислотными последовательностями ферментов синтеза эктоина близкородственных организмов (эволюционное расстояние менее 0.15) [Cai et al., 2011; Widderich et al., 2014].
Ранее из почвы района солеразработок Верхнекамского месторождения солей был выделен гало-толерантный грамположительный бактериальный штамм U1, способный к активному росту по сравнению с другими исследованными в этой работе штаммами в полноценной среде Раймонда в присутствии 12%-ного NaCl. На основе морфологиче-
ских характеристик штамм был отнесен к роду Brevibacterium [Плотникова и др., 2006].
Цель настоящей работы – определение филогенетического положения штамма Brevibacterium sp. U1 в системе рода Brevibacterium на основе анализа последовательностей 16S рДНК и генов, кодирующих ферменты биосинтеза эктоина.
Материалы и методы исследования
Объекты исследования
Brevibacterium sp. U1 был выделен из почвы района солеразработок Верхнекамского месторождения солей [Плотникова и др., 2006].
Среды и условия культивирования
Минеральная среда Раймонда (г/л деионизированной воды): NH4NO3 - 2.0, MgSO4 × 7H2O - 0.2, KH2PO4 - 2.0, Na2HPO4 - 3, CaCl2 × 6H2O - 0.01, Na2CO3 - 0.1, pH - 7.0 [Raymond, 1961].
Агаризованная богатая среда Раймонда следующего состава (г/л среды Раймонда): триптон - 5, дрожжевой экстракт - 2.5, NaCl - 30, агар - 15.
Культивирование штамма проводили на агари-зованной богатой среде Раймонда в термостатируемом шкафу ТС-1/80 СПУ (Россия) при температуре 28оС.
Молекулярно-генетические и биоинформационные методы исследования
Геномную ДНК из бактериальных клеток выделяли SDS-CTAB методом [Wilson, 1995].
С препаратов ДНК амплифицировали фрагмент гена 16S рРНК согласно методике, описанной ранее [Anan’ina et al., 2011].
Амплификацию и детекцию ect -генов на матрице ДНК выполняли, используя систему праймеров G2F/G2R согласно протоколу, описанному в статье [Ананьина, Шестакова, Плотникова, 2018].
Секвенирование генов проводили с помощью соответствующих праймеров и Big Dye Terminator Ready Reaction Kit v3.1 («Thermo Fisher Scientific», «Fisher Scientific», США), следуя инструкциям фирмы-производителя на приборе Genetic Analyzer 3500xl в лаборатории молекулярной биологии и генетики при кафедре ботаники и генетики растений ПГНИУ.
Биоинформационное обеспечение
Сервис публичной базы данных Национального центра биотехнологической информации США (NCBI) – Blastn . Пакет программ Mega v. 5.0, позволяющий выравнивать нуклеотидные последовательности, рассчитывать эволюционные расстояния с последующим графи- ческим представлением результатов.
Результаты и обсуждение
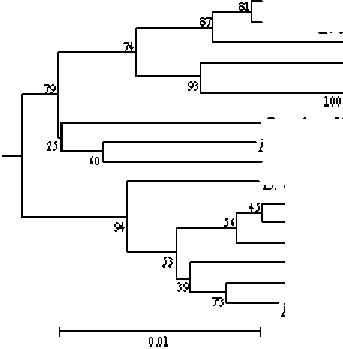
Сравнительный анализ нуклеотидной последовательности гена 16S рРНК длиной 1425 п.н. штамма Brevibacterium sp. U1 с помощью программы Blastn выявил высокий уровень сходства с нуклеотидными последовательностями генов представителей рода Brevibacterium, что подтвердило результат предварительной идентификации штамма на основе морфо-физиологических характеристик. На филогенетическом дереве штамм входил в кластер Brevibacterium sensu stricto, формируя подкластер с видами B. aurantiacum и B. antiquum, уровень сходства нуклеотидных последовательностей гена 16S рРНК составил 99.29 и 97.8%, соответственно. Следует отметить, что уровень 16S рДНК сходства между видами кластера Brevibacterium sensu stricto находился в пределах 93.5-99.2%. Максимальное значение сходства 99.2% отмечено между видами B. linens и B. iodinum. Таким образом, уровень сходства штамма Brevibacterium sp. U1 и представителя вида B. aurantiacum сопоставим с межвидовым для группы Brevibacterium sensu stricto (рис. 1). Кроме того, типовой штамм вида B. aurantiacum VKM Ac-2111Т был выделен из сыра сорта Camambert . Современное исследование показало доминирование представителей вида B. aurantiacum в пяти различных сырах, созревающих в рассоле [Cogan et al., 2014]; в то время как исследуемый штамм Brevibacterium sp. U1 был выделен из засоленной почвы, загрязненной отходами соледобывающего предприятия [Плотникова и др., 2006]. Принимая во внимание вышесказанное, становится очевидной необходимость дополнительного исследования для уточнения таксономического (филогенетического) положения штамма Brevibacterium sp. U1 с применением других генов.
Использование разработанной нами ранее системы праймеров [Ананьина, Шестакова, Плотникова, 2018] позволило получить ПЦР-продукт ожидаемой длины около 800 п.н., включающий фрагменты ectB- и ectC-генов, на ДНК-матрице исследуемого штамма Brevibacterium sp. U1. Предварительный скрининг в публичных базах данных нуклеотидной последовательности фрагмента ect-оперона с помощью программы Blastn выявил его гомологию с ect-генами бактерий рода Brevibacterium.
В настоящее время только у 6 из 15 видов группы Brevibacterium sensu stricto представлены нуклеотидные последовательности ect-генов в публичной базе данных Национального центра биотехнологической информации США. Уровни сход- ства нуклеотидных последовательностей ectB-и ectC-генов типовых штаммов видов группы Brevibacterium sensu stricto находились в пределах 64.2-93.2 и 74.6-97.6%, соответственно. Результаты свидетельствуют о более высоком уровне дивергенции ect-генов по сравнению с геном 16S рРНК (рис. 2а, б). При этом ectC-ген имел меньшие значения эволюционного расстояния между видами группы Brevibacterium sensu stricto, чем ectB-ген (рис. 2а, б). Наиболее высокий уровень сходства нуклеотидных последовательностей ectB-и ectC-генов, 97.3 и 94.3%, соответственно, исследуемый штамм проявил с видом B. aurantiacum (рис. 2а, б). Примечательно, что значение сходства нуклеотидной последовательности ectB-гена штамма U1 было выше уровня межвидового сход- ства. В то время как для ectC-гена это значение находилось в вычисленном интервале, а на филогенетическом дереве штамм формировал отдельную ветку в группе B. aurantiacum/ B. antiquum (рис. 2б). При этом положение видов относительно друг друга в кластере Brevibacterium sensu stricto на «ectB»- и «ectC»-деревьях отличалось, что может быть следствием горизонтального переноса генов. Современные исследования геномов штаммов, близкородственных виду B. aurantiacum, выявили большое количество транспозаз и интеграз, а также фрагменты гомологичных последовательностей ДНК, что предполагает горизонтальный перенос генов, который, по мнению авторов, обеспечивает приспособление к условиям экологической ниши [Levesque et al., 2019].
-----------UI
"B. aurantiac™ (X76566.1)
-----B. anftguum VKM Ac-2118* (AY243344.1)
-B. marinum HFW-26Y(AM4218071)
rB. picturae LMG 220611 (AJ620364.1)
1--------------------------B. sandarakinum 01-Je-003x (FN293377.1)
В. секте KMM 3637* (AY228463.1)
B. casei DSM 20657* (AJ251418.1)
----------------B. ammonubficum Al* (JF937067.1)
B. ocean! BBH7*(AM158906.2)
---------------------------------В. slhguriense МВ18*(АМ937247.2)
--------В. epidermidis NCDO 2286* (Х76565.1)
----В. permense VKM Ac-22801 (AY243343.1)
-В. sedimims CGMCC 1.15472’ (KX356313.1)
йюДпшп (X83813.1)
Рис. 1 . Филогенетическое дерево, построенное на основе анализа нуклеотидных последовательностей 16S рДНК с использованием метода «ближайшего соседа»; масштаб соответствует 1 замене на каждые 100 нуклеотидов
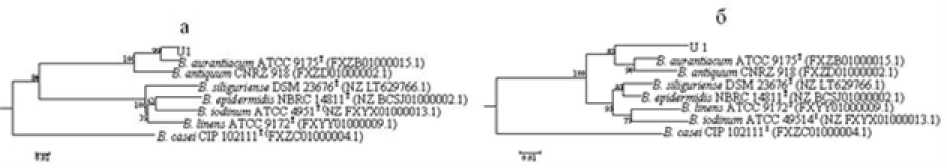
Рис. 2. Филогенетическое дерево, построенное на основе анализа нуклеотидных последовательностей генов (а) ect B и (б) ect C с использованием метода «ближайшего соседа»; масштаб соответствует 1 замене на каждые 100 нуклеотидов
Заключение
Полученные в ходе исследования данные выявили высокий уровень сходства нуклеотидных последовательностей гена 16S рРНК и генов, кодирующих ферменты биосинтеза эктоина, штамма Brevibacterium sp. U1 с видом B. aurantiacum. Тем не менее, выявлено значительное эволюционное расстояние нуклеотидных последовательностей ectC-гена исследованного штамма и близкородственного вида B. aurantiacum. В связи с вышеска- занным можно предположить, что исследуемый штамм может быть представителем нового вида.
Работа выполнена в рамках государственного задания, номер госрегистрации темы: 01201353247.
Список литературы Филогенетическое положение галотолерантного штамма Brevibacterium sp. U1, выделенного из засоленной почвы, в системе рода Brevibacterium
- Ананьина Л.Н., Шестакова Е.А., Плотникова Е.Г. Детекция генов, кодирующих ферменты биосинтеза эктоина, у актинобактерий, выделенных из почвы района разработки Верхнекамского соленосного бассейна // Вестник Пермского университета. Сер. Биология. 2018. Вып. 3. С. 264-269.
- Плотникова Е.Г. и др. Характеристика микроорганизмов, выделенных из техногенных почв Прикамья // Экология. 2006. № 4. С. 261-268.
- Anan'ina L.N. et al. Naphthalene-degrading bacteria of the genus Rhodococcus from the Verkhnekamsk salt mining region of Russia // Antonie Van Leeuwenhoek. 2011. Vol. 100. P. 309-316.
- Bernard T. et al. Ectoine accumulation and osmotic regulation in Brevibacterium linens // J. Gen. Microbiol. 1993. Vol. 139. P. 129-136.
- Cai L. et al. Comparative genomics study of polyhydroxyalkanoates (PHA) and ectoine relevant genes from Halomonas sp. TD01 revealed extensive horizontal gene transfer events and co-evolutionary relationships // Microb. Cell Fact. 2011. Vol. 1. P. 88.
- Cogan T. M. et al. Biodiversity of the surface microbial consortia from Limburger, Reblochon, Livarot, Tilsit, and Gubbeen cheeses // Microbiol. Spectr. 2014. Vol. 2.
- DOI: 10.1128/microbiolspec.CM-0010-2012
- Ivanova E.P. et al. Brevibacterium celere sp. nov., isolated from degraded thallus of a brown alga // Int. J. Syst. Evol. Microbiol. 2004. Vol. 54. P. 2107-2111.
- Levesque S. et al. Mobilome of Brevibacterium aurantiacum sheds light on its genetic diversity and its adaptation to smear-ripened cheeses // Front. Microbiol. 2019. Vol. 10. P. 1270.
- Pham N.P. et al. Comparative genomic analysis of Brevibacterium strains: insights into key genetic determinants involved in adaptation to the cheese habitat // BMC Genomics. 2017. Vol. 7. P. 955.
- Raymond R.L. Microbial oxidation of n-paraffinic hydrocarbons // Develop. Ind. Microbiol. 1961. Vol. 2. P. 23-32.
- Widderich N. et al. Biochemical properties of ectoine hydroxylases from extremophiles and their wider taxonomic distribution among microorganisms // PLoS One. 2014. Vol. 8.
- DOI: 10.1371/journal.pone.0093809
- Wilson K. Preparation of genomic DNA from bacteria // Ausubel F.M., Brent R., Kingston R.E., Moore D.D., Seidman J.G., Smith J.A., Struhl K. (eds). Current protocols in molecular biology. 3rd edn. Wiley; New York, 1995.


