Филогенетическое разнообразие бактерий семейства Micrococcaceae, выделенных из биотопов с различным антропогенным воздействием
Автор: Ястребова О.В., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 4, 2020 года.
Бесплатный доступ
Проанализированы данные по систематической принадлежности и распространению бактерий сем. Micrococcaceae, выделенных из экосистем с различной степенью антропогенной нагрузки (г. Пермь, Березники, Соликамск, Оса; Пермский край). Показано филогенетическое разнообразие и широкое распространение представителей семейства Micrococcaceae в различных биотопах района разработок Верхнекамского месторождения солей. Разнообразие видов бактерий семейства Micrococcaceae выявлено в почве и ризосфере растений, произрастающих вблизи солеотвалов. На основании анализа гена 16S рРНК исследуемые штаммы семейства Micrococcaceae были отнесены к семи родам: Arthrobacter, Pseudoarthrobacter, Glutamicibacter, Micrococcus, Kocuria, Nesterenkonia, Rothia. Исследуемые культивируемые бактерии семейства Micrococcaceae, выделенные из экотопов с экстремальными условиями среды, являются перспективными для дальнейшего их использования в биотехнологических целях.
Micrococcaceae, идентификация, ген 16S рРНК, засоление, загрязненные экотопы
Короткий адрес: https://sciup.org/147229668
IDR: 147229668 | УДК: 579:873:579.222.2 | DOI: 10.17072/1994-9952-2020-4-321-333
Текст научной статьи Филогенетическое разнообразие бактерий семейства Micrococcaceae, выделенных из биотопов с различным антропогенным воздействием
Micrococcaceae – семейство порядка Actinomycetales , включающее роды грамположи-тельных, облигатно и факультативно аэробных бактерий, имеющих широкое распространение в почвенных и водных экосистемах. Типовым для семейства является род Micrococcus , впервые описанный Cohn F. в 1872 г. Семейство Micro-coccaceae , предложенное E. Pribram [1929], было в дальнейшем изменено E. Stackebrandt с коллегами
-
[1997] на основе последовательностей гена 16S рРНК. Помимо типового рода Micrococcus , в семейство были включены роды Arthrobacter [Conn, Dimmick, 1947], Kocuria [Koch, Schumann, Stackebrandt, 1995], Nesterenkonia [Stackebrandt et al., 1995], Renibacterium [Sanders, Fryer, 1980], Rothia [Georg, Brown, 1967], Stomatococcus [Ber-gan, Kocur, 1982], Citricoccus [Altenburger et al., 2002], Acaricomes [Pukall et al., 2006], Zhihen-gliuella [Zhang et al., 2007], Yaniella [Li, Zhi, Euzéby, 2008], Sinomonas [Zhou et al., 2009], Auri-
© Ястребова О. В., Плотникова Е. Г., 2020
tidibacter [Yassin et al., 2011] и Enteractinococcus [Cao et al., 2012]. В 2016 г. виды рода Arthrobacter были реклассифицированы в отдельные 5 родов семейства: Paenarthrobacter, Pseudoarthrobacter, Glutamicibacter, Paeniglutamicibacter, Pseudoglu-tamicibacter [Busse, 2016] .
Представители семейства Micrococcaceae встречаются в морских экосистемах, в тканях растений, на поверхности кожи млекопитающих, а также являются типичными обитателями почвенных экосистем. Бактерии данного семейства присутствуют как в загрязненных, так и в неподверженных антропогенному воздействию почвах, где представляют значимую часть культивируемых микроорганизмов. Среди представителей семейства Micrococcaceae описаны деструкторы устойчивых, токсичных органических соединений, в том числе полициклических ароматических углеводородов и фталатов [Hu, Yang, Wang, 2015; Vandera et al., 2015]. В семействе Micrococcaceae обнаружены галофильные виды, принадлежащие к родам Nesterenkonia и Kocuria, имеющие оптимум роста в средах с повышенным содержанием соли, а также галотолерантные бактерии рода Arthrobacter [Kushner, 1978; Hamedi, Mohammadipanah, Vento- sa, 2013].
Цель работы – филогенетическая характеристика бактерий семейства Micrococcaceae , выделенных из разных экотопов Пермского края.
Материалы и методы исследования
Из рабочей коллекции микроорганизмов Лаборатории молекулярной микробиологии и биотехнологии Института экологии и генетики микроорганизмов УрО РАН отобран 41 штамм бактерий, идентифицированных ранее как представители семейства Micrococcaceae . Исследуемые штаммы были выделены из различающихся по физикохимическим условиям среды экотопов: образцов калийных солей (каменная соль, карналлит) из Верхнекамского месторождения калийно-магние-вых солей (ВКМС); техногенно-минеральных образований (ТМО); почв, грунтов и донных отложений, загрязненных отходами химических и соледобывающих производств (г. Березники, Соликамск, Пермский край), из почвы сельскохозяйственного использования (г. Оса, Пермский край), активного ила очистных сооружений г. Перми (табл. 1).
Таблица 1
Штаммы и источники выделения бактерий сем. Micrococcaceae
|
Источник выделения |
Штамм |
|
Калийные соли (ВКМС) |
|
|
Каменная соль (глубина 239.7–239.8 м) |
38-1 |
|
Карналлит (глубина 254.2–254.4 м) |
43-1 |
|
Каменная соль (глубина 372.2–372.4 м) |
69-6 |
|
Каменная соль (глубина 411.5-411.6 м) |
72-1 |
|
72-2 |
|
|
72-4 |
|
|
Глина (мергель, глубина 326.9–327.0 м) |
YKS63 |
Соледобывающие предприятия (г. Березники)
|
ТМО, шламохранилище (глубина 0.5 м) |
BO19 |
|
ТМО, шламохранилища (глубина 0.2 м) |
ВО37 |
|
ТМО2, шламохранилище (глубина 0.2 м) |
BO25 |
|
ТМО2, шламохранилище (глубина 0.4 м) |
BO34-1 |
|
ТМО2, шламохранилище (глубина 0.3м) |
BO21 |
|
Почва на расстоянии 500 м от солеотвала |
B1 |
|
Почва на расстоянии 5 м от солеотвала |
SN17 (ВКМ Ас-2065) |
|
Почва на расстоянии 500 м от солеотвала |
SMB11 (ВКМ Ас-2552) |
|
SMB145 (ВКМ Ас-2551) |
|
|
SF27 (ВКМ Ас-2063) |
|
|
Донные отложения р. Зырянка, вблизи солеотвала |
DF14 (ВКМ Ас-2064) |
|
Почва/грунт на расстоянии 3‒10 м от солеотвала |
B901 ВКМ Ac-2548 |
|
B904 (ВКМ Ac-2549) |
|
|
B905 (ВКМ Ac-2550) |
|
|
Донные отложения промышленных стоков, «Промканал» |
PD13 |
|
Ризосферная почва бескильницы расставленной ( Puccinellia distans ), около солеотвала |
PB8-1 |
|
Окончание табл. 1 |
|
|
Источник выделения |
Штамм |
|
Соледобывающие предприятия (г. Соликамск) |
|
|
Ризосферная почва бескильницы расставленной ( Puccinellia distans ), 4 м от рассолосборника, около солеотвала |
BR3-22(1) |
|
BR3-22(2) |
|
|
BR4-2 |
|
|
Ризосферная почва ежи сборной ( Dactylis glomerata ), около солеотва-ла (загрязнение дизельным топливом) |
NDT16 |
|
NDT18 |
|
|
EDT13 |
|
|
Ризосферная почва мятлика лугового ( Poa pratensis ), 5‒10 м от со-леотвала |
M56-102 |
|
104 |
|
|
ML 17 |
|
|
М87-1 |
|
|
М46-10 |
|
|
M45-1N |
|
|
Ризосферная почва мятлика лугового ( Poa pratensis ), 20 м от солеот-вала |
72 |
|
M55-2N |
|
|
M55-5N |
|
|
ООПТ «Осинская лесная дача» (г. Оса) |
|
|
Почва |
WD25 |
Биологические очистные сооружения (г. Пермь)
|
Активный ил |
SA7 |
|
SA14 |
Для уточнения таксономического положения отобранных штаммов были проведены амплификация, секвенирование и филогенетический анализ гена 16S рРНК. Чистые культуры бактерий выращивали на агаризованной богатой среде Раймонда [Raymond, 1961] с добавлением 5 г/л триптона и 2.5 г/л дрожжевого экстракта в качестве ростовых субстратов.
Выделение ДНК из биомассы колоний, выросших на агаризованной среде, осуществляли методом щелочного лизиса. Единичная колония культуры помещалась в пробирку «эппендорф», содержащую 100 мкл 0.05N NaOH. Смесь инкубировали при температуре 95 о С 15 мин., затем охлаждали в течение 15 мин. при температуре 20°С и центрифугировали при 12 000 об/мин 2 мин. Для проведения амплификации генов 16S рРНК отбирали 1 мкл супернатанта лизата.
Амплификацию гена 16S рРНК проводили с использованием бактериальных праймеров 27F (5`-AGAGTTTGATC(A/C)TGGCTCAG-3`) и 1492R (5`-АСGG(C/T)TACCTTGTTACGACTT-3`) на приборе My Cycler “Bio-Rad Laboratories” (США) согласно описанию [Weisburg et al., 1991]. Определение нуклеотидных последовательностей проводили с применением набора реактивов Big Dye Terminator Cycle Sequencing Kit v. 3.1 на автоматическом секвенаторе Genetic Analyser 3500XL “Applied Biosystems” (США) согласно рекомендациям производителя.
Филогенетический анализ полученных нуклеотидных последовательностей 16S рДНК осуществляли с использованием программ CLUSTAL W , Sequence Scanner v
2.0. Поиск гомологичных последовательностей осуществляли при использовании баз данных GenBank и EzTaxon . Филогенетическое дерево строили с помощью программы MEGA7 c использованием метода «neighbor joining» [Kumar, 2016].
Результаты и их обсуждение
Штаммы бактерий, изолированные из различных экотопов Пермского края (табл. 1), были идентифицированы на основании анализа гена 16S рРНК, как представители класса Actinobacteria, порядка Micrococcales , семейства Micrococcaceae. Исследуемые штаммы были отнесены к родам: Ar-throbacter, Pseudoarthrobacter, Glutamicibacter, Micrococcus, Kocuria, Nesterenkonia, Rothia .
Бактерии рода Arthrobacter
Девять штаммов бактерий рода Arthrobacter были изолированы из образцов почвы и донных отложений водоемов, отобранных в районе разработок ВКМС (табл. 1, 2). Восемь штаммов были близкородственны виду А. crystallopoietes (сходство с типовым штаммом вида по генам 16S рРНК составляло 99.7–99.89%) (табл. 2).
Штамм M56-102 наиболее близок виду A. echini (сходство по генам 16S рРНК – 98.05%) (табл. 2).
На дендрограмме, показывающей филогенетические отношения исследуемых бактерий родов Arthro-bacter, Glutamicibacter и Pseudoarthrobacter, показано, что данные штаммы разделяются на четыре кластера, имеющих отдельные ветви. Восемь штаммов рода Arthrobacter группируются с типовым штаммом вым штаммом вида A. echini, (рис. 1).
вида A. сrystallopoietes , а штамм M56-102 – с типо-
Таблица 2
Результаты филогенетического анализа штаммов рода Arthrobacter
|
Штамм |
Типовой штамм ближайшего родственного вида, номер в базе данных GenBank |
Количество сравниваемых нуклеотидов |
Сходство генов 16S рРНК, % |
|
B1 |
Arthrobacter crystallopoietes DSM 20117 T (Х80738) |
512 |
99.61 |
|
SMB11 B901 B904 B905 SMB145 |
Arthrobacter crystallopoietes DSM 20117 T (Х80738) |
1400 |
99.70 |
|
SF27 |
Arthrobacter crystallopoietes DSM 20117 T (Х80738) |
1400 |
99.85 |
|
DF14 |
Arthrobacter crystallopoietes DSM 20117 T (Х80738) |
940 |
99.89 |
|
M56-102 |
Arthrobacter echini AM23 T (NR 148833) |
871 |
99.00 |
Ранее было установлено, что выделенные штаммы Arthrobacter spp. обладают широкой субстратной специфичностью и способны к росту на моно- и поли-ароматических углеводородах, а также фталатах при культивировании в присутствии 3%-ного NaCl. Все штаммы использовали орто -фталат ( орто -ФК) и дибутилфталат (ДБФ), протокатеховую кислоту, а также нафталин в качестве единственного источника углерода и энергии; два штамма (SF27 и DF14) растут также на фенантрене [Плотникова и др., 2011; Гагар-ских, Корсакова, 2014].
Бактерии рода Pseudoarthrobacter
Четыре штамма, изолированных из шламохра-нилища соледобывающего предприятия (штаммы BO21, BO19, BO34-1) и из донных отложений промышленных стоков г. Березники (штамм PD13), ранее были идентифицированы как представители рода Arthrobacter, однако согласно новой классификации, основанной на данных хемо-таксономических характеристик и филогенетическом анализе [Busse, 2016], были отнесены к новому роду Pseudoarthrobacter (табл. 3).
Таблица 3
Результаты филогенетического анализа штаммов рода Pseudoarthrobacter
|
Штамм |
Типовой штамм ближайшего родственного вида и номер в базе данных GenBank |
Количество сравниваемых нуклеотидов |
Сходство генов 16S рРНК, % |
|
PD13 |
Pseudoarthrobacter siccitolerans 4J27 (NR108849) |
850 |
99,03 |
|
BO21 |
Pseudoarthrobacter oxydans DSM 20119 T |
1388 |
99,20 |
|
BO19 |
Pseudoarthrobacter oxydans DSM 20119 T (Х83408) |
1233 |
100 |
|
BO34-1 |
Pseudoarthrobacter oxydans DSM 20119 T (Х83408) |
1401 |
100 |
Штаммы BO21, BO19 BO34-1 были близкородственны штамму P. oxydans DSM 20119 T (сходство по гену 16S рРНК 99,20–100%), штамм PD13 показал сходство на уровне 99,03% с типовым штаммом вида P. siccitolerans . Филогенетический анализ, основанный на сравнении нуклеотидных последовательностей гена 16S рРНК, показал, что исследуемые бактерии рода Pseudoarthrobacter находятся в одном кластере со штаммами видов P. oxydans, P. phe-nantrenivorans и P. siccitolerans (рис. 1).
Как было показано ранее, выделенные штаммы рода Pseudoarthrobacter способны к утилизации ароматических субстратов, в частности орто- ФК, ДБФ, нафталина, салицилата, бензоата [Корсакова, Пьянкова, Плотникова, 2013; Гагарских, Корсакова, 2014; Гагарских, Ястребова, 2017].
Бактерии рода Glutamicibacter
Девять штаммов, выделенных из образцов почв района солеразработок: почвы вблизи солеотвала
(штаммы SN17, PB8-1, NDT16, NDT18, EDT13), шламохранилища (штамм BO25), рассолосборника (штаммы BR3-22(1), BR3-22(2), BR4-2), были ранее отнесены к роду Arthrobacter [Плотникова и др. 2011; Гагарских, Корсакова 2014]. Однако на основании новой классификации, предложенной Busse [Busse, 2016], штаммы были реклассифицированы как представители рода Glutamicibacter .
Штаммы SN17 и PB8-1 показали наибольшее сходство с типовым штаммом вида G. arilaitensis (100%-ное сходство по гену 16S рРНК), штаммы BO25, BR4-2, NDT16, NDT18 и EDT13 близкородственны G. nicotianae DSM 20123T (сходство -98,85–99,25%), штаммы BR3-22(1) и BR3-22(1) наиболее близки G. halophytocola KLBMPT (сходство 99,00 -99,41%) (табл. 4). В то же время, на дендрограмме (рис. 1), показывающей филогенетические отношения исследуемых бактерий (ген 16S рРНК), видно, что штаммы BR3-22(1), BR3-22(2), EDT13, NDT16, NDT18, BR4-2, BO25 рас- полагаются на одной ветви (составляют один кластер) со штаммом вида G. halophytocola. Штаммы
PB8-1, SN17 хорошо кластеризуются с типовым штаммом вида G. arilaitensis (рис. 1).
BR3-!2(H BR3-22P)
fflt/tommiKria-hofo^ytocdaKLEMP 5180' (JX993762)
EDT13
64 --МОП 6
BR4-2
MDT18
ВО25
---- Gi utamicibac ter п/го &№ DS M 2 01231 (N R 0 2619 0)
iPB8-l
;i\ii7
G/ cftom/riboc ter onto tenss R e ll 7 (N R 0 7 4 6 03)
G/ иtomtritoc terbergere/ Cal06' (NR 025612)
13/vtom/ri tor ter pro top tom? toe DSM 20183' (NR 026195)
*■ throb Deter gtosery/so// DCV811 (KF212463)
■ ^throbDctergtob/Vbrmrs J CM 1332' (NR 112192) ’sectoorthrobartersc^n/rormpsALL1 (NR 025084) iA-throbDCtero/ytoeKV-eSl1 (NR 041545)
A-throbDCterpDJcer?3 DSM 2 0 545' (NR 0 2 6191)
ВС21
0034-1
PD13
’sevd od г thro be c ter p b его nttosr? г того/? 5Sphe3'(NR 0 7 47 70) ’sevdoorttoobDcteroxytoosDSM 201 "191 (NR 026236)
’seydoDrttoobDctorricritoterms 4J2 7 (NR 108849)
М 56-102
-A-thrabD/rterechYm AM231 (NR 148833)
62 '---jArthrabDCterp/tiocDDVDeTpl1 (NR 13 39 69) ^toroborter raseos C№9O' (NR 028925)
thro batter koreensis CAI 5-8' (NR 0 2 5 665)
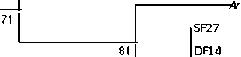
SMB11, Bl
0905, B901,. 0904
SMB145
A-thrabc c tore/уз totopo fetesDSM20117 (NR 026189)
кипу
Рис. 1 . Дендрограмма, отображающая филогенетическое положение штаммов родов Arthrobacter , Pseudoarthrobacter и Glutamicibacter
Таблица 4
Результаты филогенетического анализа штаммов рода Glutamicibacter
|
Штамм |
Типовой штамм ближайшего родственного вида и номер в базе данных GenBank |
Количество сравниваемых нуклеотидов |
Сходство генов 16S рРНК, % |
|
SN17 |
Glutamicibacter arilaitensis Re117 T (NR_074608) |
823 |
100 |
|
PB8-1 |
856 |
||
|
NDT16 |
Glutamicibacter nicotianae DSM 20123 T (NR_026190) |
873 |
98.85 |
|
NDT18 |
916 |
||
|
BR4-2 |
Glutamicibacter nicotianae DSM 20123 T (NR_026190) |
847 |
99.00 |
|
BO25 |
Glutamicibacter nicotianae DSM20123 T (NR_026190) |
823 |
99.10 |
|
EDT13 |
Glutamicibacter nicotianae DSM 20123 T (NR_026190) |
1340 |
99.25 |
|
BR3-22(1) |
Glutamicibacter halophytocola KLBMP T (JX993762) |
850 |
99.00 |
|
BR3-22(2) |
Glutamicibacter halophytocola KLBMP T (JX993762) |
848 |
99.41 |
Исследуемые штаммы Glutamicibacter spp. , как было установлено ранее, способны к деструкции фталатов ( орто- ФК, ДБФ), бензоата, а штаммы SN17,
BO25 – к разложению нафталина и салицилата [Плотникова и др., 2011; Корсакова, Пьянкова, Плотникова, 2013; Гагарских, Корсакова, 2014].
Бактерии рода Micrococcus
Восемь штаммов бактерий рода Micrococcus выделены из разных биотопов района солеразработок: шесть штаммов – из образцов калийных солей, отобранных с глубины 254.2–411.6 м; штамм
BO37 – из образца ТМО шламохранилища, а штамм 104 – из ризосферной почвы мятлика лугового (табл. 1). Все штаммы, согласно анализу генов 16S рРНК, близкородственны Micrococcus lu-teus ATCC 4698 T на уровне 99.13–100% (табл. 5, рис. 2).
Таблица 5
Результаты филогенетического анализа штаммов рода Micrococcus
|
Штамм |
Типовой штамм ближайшего родственного вида и номер в базе данных GenBank |
Количество сравниваемых нуклеотидов |
Сходство генов 16S рРНК, % |
|
69-6 |
Micrococcus luteus NCTC 2665 T (CP001628) |
814 |
99.13 |
|
38-1 |
Micrococcus luteus NCTC 2665 T (CP001628) |
1383 |
99.57 |
|
43-1 |
Micrococcus luteus NCTC 2665 T (CP001628) |
1237 |
99.59 |
|
YKS63 |
Micrococcus luteus NCTC 2665 T (CP001628) |
752 |
99.73 |
|
72-1 |
Micrococcus luteus NCTC 2665 T (CP001628) |
739 |
100 |
|
72-4 |
784 |
||
|
ВО37 |
500 |
||
|
104 |
886 |
BO37
YKS63
38-1
43-1
94 69-6
72-1, 72-4
Micrococcus luteus ATCC 4698T (CP 035298)
Micrococcus aloeverae AE-6T (NR 134088)
Micrococcus yunnanensis YIM 65004T (NR 116578)
Micrococcus lylae DSM 20315T (NR 026200)
Micrococcus endophyticus YIM 56238T (NR 044365)
Citricoccus nitrophenolicus PNP1T (NR 117546)
Micrococcus terreus V3M1T (NR 116649)
59 Micrococcus cohnii WS4601T (NR 117194)
Micrococcus antarcticus T2T (NR 025285.1)
Micrococcus flavus LW4T (NR 043881)
Рис. 2. Дендрограмма, отображающая филогенетическое положение штаммов рода Micrococcus
Установлено, что все исследуемые штаммы рода Micrococcus способны к утилизации бензоата, а штаммы YKS63, 43-1 и 72-1 растут также на орто- ФК, но не способны к деструкции полиарома-тических углеводородов [неопубликованные данные].
Бактерии рода Kocuria
Штаммы рода Kocuria были выделены из ризосферы мятлика лугового, произрастающего на за- соленной почве (5 штаммов), один штамм ‒ из глубинной породы каменной соли и один штамм ‒ из почвы агроэкосистемы (табл. 1). Выделенные штаммы близкородственны (по гену 16S рРНК) c типовыми штаммами трех видов рода Kocuria: K. arsenatis, K. rosea, K. polaris (табл. 6, рис. 3).
Филогенетический анализ, основанный на сравнении нуклеотидных последовательностей гена 16S рРНК, показал, что исследуемые бактерии рода Kocuria разделяются на 3 кластера: штамм ML17 находится в одном кластере с видами K. ar- senatis и K. rhizophila, штамм M45-1N – на одной ветви со штаммом K. polaris, а штаммы М46-10, 72 и 72-2 – в кластере с видом K. rosea (рис. 3). Штамм M87-1 представлен на отдельной ветви, имеет небольшой процент сходства (98.94%) с типовым штаммом вида Kocuria arsenatis, в дальнейшем может представлять интерес для описания нового таксона (вида) рода Kocuria.
Таблица 6
Результаты филогенетического анализа штаммов рода Kocuria
|
Штамм |
Типовой штамм ближайшего родственного вида и номер в базе данных GenBank |
Количество сравниваемых нуклеотидов |
Сходство генов 16S рРНК, % |
|
WD25 |
Kocuria arsenatis CM1E1 T (KM874399) |
918 |
99,78 |
|
М87-1 |
Kocuria arsenatis CM1E1 T (KM874399) |
941 |
98,94 |
|
ML17 |
Kocuria arsenatis CM1E1 T (KM874399) |
895 |
100 |
|
M45-1N |
Kocuria polaris CMS 76or T (AJ278868) |
743 |
99,70 |
|
М46-10 |
Kocuria rosea DSM 20447 T (X87756) |
892 |
99,66 |
|
72-2 |
Kocuria rosea DSM 20447 T (X87756) |
721 |
99,77 |
|
72 |
Kocuria rosea DSM 20447 T (X87756) |
860 |
100 |
Как показано ранее, штаммы Kocuria sp. WD25 и ML17 обладают широкой субстратной специфичностью и способны к деструкции ряда моно- и полиароматических углеводородов: фенантрена, нафталина, бензоата, а также орто -ФК и ДБФ [Юдин, Ястребова, Плотникова, 2019].
Бактерии рода Nesterenkonia
Два штамма M55-5N и M55-2N рода Nesteren-konia были выделены из ризосферы мятлика лугового, произрастающего на засоленной почве (табл. 1). На основании анализа гена 16S рРНК штаммы отнесены к виду N. halotolerans со сходством 100% с типовым штаммом вида. На дендрограмме данные штаммы находятся в одном кластере с типовым штаммом вида N. halotolerans (рис. 4).
Показано, что исследуемые штаммы рода Nesterenkonia не обладают деградативной активностью в отношении моно- и полиароматических углеводородов и фталатов [неопубликованные данные].
Бактерии рода Rothia
Два штамма рода Rothia были выделены из образца активного ила биологических очистных сооружений предприятия ОАО «Сибур-Химпром» (г. Пермь).
Как показал филогенетический анализ, исследуемые штаммы SA7 и SA14 находятся в одном кластере с типовым штаммом вида R. amarae (рис. 5). Сходство по генам 16S рРНК составляет 99.88% (штамм SA7) и 100% (штамм SA14).
Установлено, что выделенные штаммы Rothia sp. SA7 и SA14 растут на бензоате и ортоФК, но не способны к росту на сложных эфирах фталевой кислоты [Ястребова, Юдин, Плотникова, 2019].
Заключение
В результате проведенной работы были исследованы бактерии семейства Micrococcaceae (порядок Actinomycetales ), выделенные из различных биотопов Пермского края. Показано филогенетическое разнообразие и широкое распространение представителей семейства Micrococcaceae : штаммы были выделены из образцов почв и ризосферы растений, образцов шламов, активного ила очистных сооружений, а также соленосной толщи Верхнекамского месторождения (породы карналлита и каменной соли). Наибольшее разнообразие видов бактерий семейства Micrococcaceae было выявлено в почве и ризосфере растений, произрастающих вблизи солеотвалов. На основании анализа гена 16S рРНК исследуемые штаммы семейства Micrococcaceae были отнесены к семи родам: Arthrobacter (9 штаммов), Pseudoarthrobacter (4 штамма), Glutamicibacter (9 штаммов), Micrococcus (8 штаммов), Kocuria (7 штаммов), Nesterenkonia (2 штамма), Rothia (2 штамма). Среди исследованных бактерий сем. Micrococcaceae наибольшей субстратной специфичностью по отношению к моно- и полиароматическим углеводородам и фталатам обладали штаммы родов Arthro-bacter и Glutamicibacter.
Полученные данные расширяют наши представления о бактериях семейства Micrococcaceae – представителях микрофлоры экосистем с повышенным засолением среды и высокой промышленной нагрузкой. Штаммы бактерий данного семейства являются перспективными для разработки на их основе биотехнологий очистки загрязненных почв и промышленных стоков.
Kocuria rosea DSM 20447T (MN840035)
M46-10
72-2
Kocuria salina Hv14bT (NR 159199)
M45-1N
60 Kocuria polaris CMS 76orT (KX959605) Kocuria himachalensis JCM 13326T (LC113906)
Paenarthrobacter nitroguajacolicus G2-1T (MK424299)
Kocuria aegyptia YIM 70003T (NR 043511)
Kocuria dechangensis NEAU-ST5-33T (NR 137239)
Kocuria oceani FXJ8.095T (NR 156033)
Kocuria flava HO-9041T (CP013254)
Kocuria sediminis FCS-11T(NR 118222)
Kocuria turfanensis HO-9042T (MN826469)
78 Kocuria turfanensis HO-9042T (CP014480)
Kocuria subflava YIM 13062T(NR 144586)
100 Kocuria assamensis S9-65T (KT989850)
Kocuria palustris TAGA27T (NR 026451)
Kocuria halotolerans YIM 90716T (NR 044025)
Kocuria koreensis P31T (NR 116745)
Kocuria uropygioeca 257T (NR 157676)
85 Kocuria uropygialis 36T (NR 157675)
Kocuria marina KMM 3905T (NR 025723)
Kocuria indica NIO-1021T (NR 133767)
Kocuria pelophila RS-2-3T (NR 151938)
89 Kocuria carniphila CCM 132T (NR 027193)
Kocuria atrinae P30T (KT989856)
Kocuria gwangalliensis SJ2T (NR 116266)
70 M87-1
Kocuria tytonicola 489T (MG 547560)
78 Kocuria salsicia 104T (NR 117299)
Kocuria varians ATCC 15306T (NR 114674)
WD25
ML17
Kocuria arsenatis CM1E1T (NR 148610)
Kocuria rhizophila DSM 11926T(KJ476722)
0.002
Рис. 3. Дендрограмма, отображающая филогенетическое положение штаммов рода Kocuria
M55-5N
Nesterenkonia halotolerans YIM70084T(NR 029073)
M55-2N
Nesterenkonia sandarakina YIM 70009T(NR 029119)
Nesterenkonia jeotgali JG-241T (NR 043279)
Nesterenkonia lutea YIM 70081T(NR 029120)
85 Nesterenkonia luteus YIM 70081T(AY588278)
Nesterenkonia aurantiaca CK5T (NR 148812)
Citricoccus lacusdiani JXJ CY21Т (KT318581)
Nesterenkonia halobia DSM 20541Т (NR 026197)
100 Nesterenkonia halophile YIM 70179Т (NR 043205)
Nesterenkonia xinjiangensis YIM70097Т (NR 029075)
Nesterenkonia suensis Sua-BAC020Т (NR 116866)
Nesterenkonia aethiopica DSM 17733Т (NR 042989)
Nesterenkonia lacusekhoensis EL-30Т (NR 028928)
Nesterenkonia cremea 10CТ (NR 156955)
Nesterenkonia flava CAAS 251T (NR 044353)
Nesterenkonia alba CAAS 252Т (NR 044527)
Nesterenkonia massiliensis NP1Т (NR 144709)
100 Nesterenkonia rhizosphaerae EGI 80099Т (NR 134096)
0.005
Рис. 4 . Дендрограмма, отображающая филогенетическое положение штаммов рода Nesterenkonia
SA7
0.1
8С
SA14
Rothia amarae J18 T (NR 029045)
Rothia aerolata CCM 8669 T (MT760050)
Rothia kristinae DSM 20032 Т (NR 026199)
8f L Kocuria subflava YIM 13062 Т (NR 144586)
Rothia terrae L-143 T (NR 043968)
БЕ
Rothia nasimurium CCUG 35957 Т (NR 025310)
Rothia marina JSM 078151 Т (FJ425908)
99 Rothia endophytica YIM 67072 Т (NR 109752)
Rothia mucilaginosa DSM 20746 Т (NR 044873)
Rothia dentocariosa ATCC 17931 Т (NR 074568)
Rothia aeria A1-17B Т (AP017895)
Рис. 5 . Дендрограмма, отображающая филогенетическое положение штаммов рода Rothia
Работа выполнена в рамках государственного задания, номер госрегистрации темы: АААА-А19-119112290008-4, и при финансовой поддержке РФФИ и Министерства образования и науки Пермского края в рамках научного проекта19-44-590011 р_а.
Список литературы Филогенетическое разнообразие бактерий семейства Micrococcaceae, выделенных из биотопов с различным антропогенным воздействием
- Гагарских О.Н., Корсакова Е.С. Галотолерантные бактерии рода Arthrobacter, выделенные из загрязненных почв и отходов соледобывающих предприятий Пермского края // Симбиоз -Россия 2014: материалы VII Всероссийского Конгресса молодых биологов. Екатеринбург, 2014. С. 105-107.
- Гагарских О.Н., Ястребова О.В. Характеристика галотолерантого штамма-деструктора фталатов Arthrobacter sp. PD13-12 // Симбиоз - 2017: X Всерос. конгресс молодых ученых-биологов: сб. тез. Казань, 2017. C. 75-76.
- Корсакова Е.С., Пьянкова А.А., Плотникова Е.Г. Бактерии-деструкторы стойких органических загрязнителей - эфиров фталевой кислоты, как основа для создания новых экобиотехнологий // Известия Самарского научного центра РАН. 2013. Т. 15, № 3 (5). С. 1633-1636.
- Плотникова Е.Г. и др. Галотолерантные бактерии рода Arthrobacter - деструкторы полициклических ароматических углеводородов // Экология. 2011. № 6. С. 459-466.
- Юдин Д.С., Ястребова О.В., Плотникова Е.Г. Характеристика бактерий рода Ko^ria - активных деструкторов фталатов // VI Международная конференция молодых ученых: биофизиков, биотехнологов, молекулярных биологов и вирусологов: сб. тез. Новосибирск, 2019. С. 236-239.
- Ястребова О.В., Юдин Д.С., Плотникова Е.Г. Выделение и характеристика смешанных культур и бактерий-деструкторов фталатов // Утилизация и рециклинг отходов производства и потребления: инновационные подходы и технологии: I Всерос. науч.-практ. конф. Киров, 2019. С. 160-162.
- Altenburger P. et al. Citricoccus muralis gen. nov., sp. nov., a novel actinobacterium isolated from a medieval wall painting // International Journal of Systematic and Evolutionary Microbiology. 2002. Vol. 52. P. 2095-2100.
- Bergan T., Kocur M. Stomatococcus mucilagi-nosus gen. nov., sp. nov., ep. rev., a member of the family Micrococcaceae // International journal of systematic bacteriology. 1982. Vol. 32. P. 374-377.
- Busse H.J. Review of the taxonomy of the genus Arthrobacter, emendation of the genus Arthrobacter sensu-lato, proposal to reclassify selected species of the genus Arthrobacter in the novel genera Glutamicibac-ter gen. nov., Paeniglutamicibacter gen. nov., Pseudoglutamicibacter gen. nov., Paenarthrobacter gen. nov. and Pseudarthrobacter gen. nov., and emended description of Arthrobacter roseus // International Journal of Systematic and Evolutionary Microbiology. 2016. Vol. 66. P. 9-37.
- Cao Y.R. et al. Enteractinococcus coprophilus gen. nov., sp. nov., of the family Micrococcaceae, isolated from Panthera tigris amoyensis faeces, and transfer of aniella fodinae Dhanjal et al. 2011 to the genus Enteractinococcus as Enteractinococ-cus fodinae comb. nov. // International Journal of Systematic and Evolutionary Microbiology. 2012. Vol. 62. P. 2710-2716.
- Cohn F. Untersuchungen über Bakterien // Beiträge zur Biologie der Pflanzen.1872. Vol. 1 (Heft II). P. 127-224.
- Conn H.J., Dimmick I. Soil bacteria similar in morphology to Mycobacterium and Corynebacterium // Journal of Bacteriology. 1947. Vol. 54. P. 291-303.
- Hamedi J., Mohammadipanah F., Ventosa A. Systematic and biotechnological aspects of halophilic and halotolerant actinomycetes // Extremophiles. 2013. Vol. 17. P. 1-13.
- Hu J., Yang Q., Wang J.L. Biodegradation of di-n-butyl phthalate in sequencing batch reactor bio-augmented with Micrococcus sp. and the bacterial community analysis // International Journal of Environmental Science and Technology. 2015. Vol. 12. P. 2819-2828.
- Georg L.K., Brown J.M. Rothia, gen. nov. an anaerobic genus of the family Actinomycetaceae // International journal of systematic bacteriology. 1967. Vol. 17. P. 79-88.
- Koch C., Schumann P., Stackebrandt E. Reclassification of Micrococcus agilis (Ali-Cohen 1889) to the genus Arthrobacter as Arthrobacter agilis comb. nov., and emendation of the genus Arthro-bacter // International journal of systematic bacteriology. 1995. Vol. 45. P. 837-839.
- Kumar S. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Molecular Biology and Evolution. 2016. Vol. 33. P. 1870-1874.
- Kushner D. Life in high salt and solute concentrations // Microbial life in extreme environments. 1978. P. 317-368.
- Li W.J., ZhiX.Y., Euzeby J.P. Proposal of Yaniellace-ae fam. nov., Yaniella gen. nov. and Sinobaca gen. nov. as replacements for the illegitimate pro-karyotic names Yaniaceae //. International Journal of Systematic and Evolutionary Microbiology. 2008. Vol. 58. P. 525-527.
- Pribram E. A contribution to the classification of microorganisms // Journal of Bacteriology. 1929. Vol. 18. P. 361-394.
- Pukall R. et al. Acaricomes phytoseiuli gen. nov., sp. nov., isolated from the predatory mite Phytosei-ulus persimilis // International Journal of Systematic and Evolutionary Microbiology. 2006. Vol. 56. P. 465-469.
- Raymond R.L. Microbial oxidation of n-paraffinic hydrocarbons // Developments in Industrial Microbiology. 1961. Vol. 2, № 1. P. 23-32.
- Sanders J.E., Fryer J.L. Renibacterium salmoninar-um gen. nov., sp. nov., the causative agent of bacterial kidney disease in salmonid fishes // International journal of systematic bacteriology. 1980. Vol. 30. P. 496-502.
- Stackebrandt E. et al. Taxonomic dissection of the genus Micrococcus: Kocuria gen. nov., Nesteren-konia gen. nov., Kytococcus gen. nov., Derma-coccus gen. nov., and Micrococcus Cohn 1872 gen. emend. // International journal of systematic bacteriology. 1995. Vol. 45. P. 682-692.
- Stackebrandt E., Rainey F.A., Ward-Rainey N.L. Proposal for a new hierarchic classification system, Ac-tinobacteria classis nov. // International journal of systematic bacteriology. 1997. Vol. 47. P. 479-491.
- Vandera E. et al. Comparative proteomic analysis of Arthrobacter phenanthrenivorans Sphe3 on phe-nanthrene, phthalate and glucose // Journal of Proteomics. 2015. Vol. 113. P. 73-89.
- Weisburg W.G. et al. 16S ribosomal DNA amplification for phylogenetic study // Journal of Bacteriology. 1991. Vol. 173. P. 697-703.
- Yassin A.F. et al. Auritidibacter ignavus gen. nov., sp. nov., a novel bacterium of the family Micro-coccaceae isolated from ear swab of a man with otitis externa, transfer of the family Yaniellaceae Li et al. 2008 to the family Micrococcaceae and emended description of the suborder Micrococ-cineae // International Journal of Systematic and Evolutionary Microbiology. 2011. Vol. 61. P. 223-230.
- Zhang Y.Q. et al. Zhihengliuella halotolerans gen. nov., sp. nov., a novel member of the family Mi-crococcaceae // International Journal of Systematic and Evolutionary Microbiology. 2007. Vol. 57. P. 1018-1023.
- Zhou Y. et al. Proposal of Sinomonas flava gen. nov., sp. nov., and description of Sinomonas atrocyanea comb. nov. to accommodate Arthrobacter atrocy-aneus // International Journal of Systematic and Evolutionary Microbiology. 2009. Vol. 59. P. 259-263.


