Филогенетическое разнообразие бактерий в каменной соли Верхнекамского месторождения (Пермский край)
Автор: Пьянкова А.А., Белоглазова Ю.А., Корсакова Е.С., Бачурин Б.А., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 4, 2021 года.
Бесплатный доступ
С использованием молекулярно-генетических методов получены новые данные о филогенетическом разнообразии бактерий в галогенных отложениях Верхнекамского месторождения солей (Пермский край). Анализ генов 16S рРНК, обнаруженных в тотальной ДНК образца каменной соли (глубина отбора 239.7-239.8 м), позволил установить присутствие бактерий, принадлежащих классам Actinobacteria (близкородственных родам Rhodococcus, Demequina), Gammaproteobacteria (родам Pseudomonas, Serratia, Shigella), Betaproteobacteria (роду Ralstonia) и Alphaproteobacteria (роду Phyllobacterium). Кроме того, выявлены два филотипа класса Alphaproteobacteria (клон 66ВА (GenBank MH410136) и клон 12ВА (GenBank MH410128), проявляющие низкий уровень сходства по генам 16S рРНК (98.46%) с ближайшими типовыми штаммами рода Mesorhizobium ( M. alhagi CCNWXJ12-2T) и рода Chelativorans ( C. multitrophicus DSM9103T), которые могут представлять новые таксономические единицы.
Верхнекамское месторождение солей, некультивируемые бактерии, клонирование, секвенирование, ген 16s ррнк
Короткий адрес: https://sciup.org/147236785
IDR: 147236785 | УДК: 579.26 | DOI: 10.17072/1994-9952-2021-4-256-262
Текст научной статьи Филогенетическое разнообразие бактерий в каменной соли Верхнекамского месторождения (Пермский край)
Верхнекамское месторождение солей (ВКМС) является одним из крупнейших по запасам калийно-магниевой руды в мире. Месторождение расположено на северо-востоке Пермского края и занимает площадь порядка 4 000 км2. Наиболее распространенными породами пермской галогенной толщи ВКМС являются каменная соль, сильвинитовые и карналлитовые породы. Каменная соль представляет собой почти мономинеральную породу, сложенную на 90–98% галитом (NaCl) с включениями глинистого и органического вещества, гипса, KCl, MgCl 2 и др. [Кудряшов 2001; Барях, Асанов, Паньков, 2008]. Анализ геологической среды ВКМС предполагает возможность выживания в таких экстремальных условиях уникальных галофильных и галотолерантных микроорганизмов. Интерес к данной группе микроорганизмов обусловлен их высоким биотехнологическим потенциалом, поскольку они могут быть использованы для получения осмопротекторных соединений, биополимеров и ферментов, активных при высокой осмолярности среды. Галофильные и галотолерантные микроорганизмы могут применяться в сельскохозяйственной и пищевой промышленности, природоохранных технологиях [Margesin, Schinner, 2001].
Месторождения солей распространены по всему миру. Микробные изоляты и нуклеотидные последовательности 16S рРНК были извлечены из галитовых отложений в Северной и Южной Америке, Европе и Восточной Азии. Жизнеспособные археи и бактерии были выделены из галита возрастом до 280 млн лет. Тем не менее, микробные сообщества глубинных подземных экосистем, в том числе соляных пород, изучены крайне недостаточно [Jaakkola et al., 2016].
В настоящее время для более глубокого изучения разнообразия микроорганизмов все чаще используются культурально-независимые подходы, поскольку предполагается, что менее 1% микроорганизмов, которые представлены в природных местообитаниях, подлежат культивированию [Alain, Querellou, 2009; Равин и др. 2015].
Цель исследования – оценка филогенетического разнообразия бактерий в галите (каменной соли) ВКМС культурально-независимыми методами.
Материалы и методы исследования
Образец для исследований . В качестве материала для исследований был использован образец каменной соли с глубины 239.7–239.8 м скважины № 704/1 Половодовского участка, расположенной в северной части ВКМС (N 56°57ʹ21.60ʺ, E 59°44ʹ24ʺ). Образец соли предоставлен сотрудниками Горного института УрО РАН (Пермь, Россия). Общая минерализация образца каменной соли определялась согласно протоколу [Практикум …, 2001]. Содержание ионов Na+, K+, Ca2+ определяли методом водной вытяжки (приготовленной согласно ГОСТу 26423-85) с последующей детекцией на атомноабсорбционном спектрофотометре АА-6300 («Shimadzu», Япония), как описано [Корсакова и др., 2013].
Рассеянное органическое вещество экстрагировали из образца каменной соли хлороформом [Бачурин, Одинцова, Хохрякова, 2016]. Фракции углеводородов и гетеросоединений в экстрактах хлороформа (хлороформный битум, ХБ) количественно определяли с помощью газового хроматографа-масс-спектрометра Agilent Technologies 6890N MSD 5975 («Agilent», США) путем сопоставления спектров с библиотекой масс-спектров NIST98.
Поверхностную стерилизацию образца каменной соли проводили путем полного погружения в 10M раствор NaOH, а затем в 70%-ный раствор этанола и выдерживали под ультрафиолетовым излучением в ламинарном шкафу в течение 30 мин.
Накопительное культивирование. При получении накопительной культуры (НК) из образца каменной соли была использована среда ATCC 213 «Halobacterium medium» ( ATCCAdvancedCatalogSearch), содержание хлорида натрия в среде составляло 200 г/л. Один грамм образца соли (из толщи керна, отобранный в стерильных условиях) помещали в 100 мл среды и инкубировали в течение 4 недель при температуре 28°С.
Выделение ДНК, ПЦР-амплификация и клонирование генов 16S рРНК . Выделение ДНК из полученной НК проводили с применением набора реактивов Fast DNA spin kit for soil («MP Biomedicals», Франция). Концентрацию выделенной ДНК измеряли с использованием прибора Qubit® Fluorometer 257
(«Invitrogen», США) и набора реактивов Quant-iT™ dsDNA BR Assay Kit («Invitrogen», США). Амплификацию фрагментов бактериальных генов 16S рРНК проводили при использовании универсальных бактериальных праймеров 27F/1492R [Lane, 1991] на приборе C1000 TouchTM Thermal Cycler («Bio-Rad Laboratories», США). Продукты реакции разделяли методом электрофореза в 1%-ном агарозном геле при напряжении 10 В/см, окрашивали раствором бромистого этидия (5 мкг/мл) и фотографировали в УФ-свете с использованием системы гель-документирования Gel DocTM XR («Bio-Rad Laboratories», США).
Для получения библиотек клонов бактериальных генов 16S рРНК полученные ПЦР-фрагменты клонировали в клетках Е . сoli JM109 в составе вектора pTZ57R/T. Для трансформации компетентных клеток Е . сoli JM109 использовали набор реактивов Thermo Scientific InsTAclone PCR Cloning Kit («Thermo Scientific», Литва). Последующий отбор рекомбинантных клонов осуществляли на основании «белоголубого» теста, высевая трансформанты на среду Luria-Bertrani (LB) [Short protocols in molecular biology, 1995], содержащую 50 мкг/мл ампициллина, 100 мМ IPTG, 20 мкг/мл X-gal («Fermentas», Литва). С ДНК-матрицы отобранных рекомбинантных клонов проводили амплификацию фрагментов бактериальных 16S рРНК генов с праймерами 27F/1492R [Lane, 1991]. Анализ полиморфизма длин рестрикционных фрагментов 16S рРНК генов (ПДРФ-анализ) осуществляли с использованием эндонуклеазы рестрикции Hha I («Fermentas», Литва). Для подготовки к секвенированию клонированных в составе вектора pTZ57R/T фрагментов ДНК проводили амплификацию вставки с использованием стандартных праймеров M13 (М13F 5’-GTTTTCCCAGTCACGAC-3’ и М13R 5’-CAGGAAACAGCTATGAC-3’), для которых имеются сайты связывания в векторе pTZ57R по обеим сторонам от полилинкера.
Секвенирование и анализ генов 16S рРНК. Определение нуклеотидных последовательностей проводили с применением набора реактивов Big Dye Terminator Cycle Sequencing Kit v. 3.1 («Applied Biosystems», США) на автоматическом секвенаторе Genetic Analyser 3500XL («Applied Biosystems», США) в Пермском государственном национальном исследовательском университете (кафедра ботаники и генетики растений) согласно рекомендациям производителя. Поиск гомологов генов 16S рРНК осуществляли по международным базам данных GenBank (http:/ и EzBioCloud . Анализ нуклеотидных последовательностей выполняли с использованием алгоритма BLAST (http:/. Множественное выравнивание нуклеотидных последовательностей и построение филогенетического дерева проводили с использованием программы MEGA 7.0 . При построении филогенетического дерева применяли кластерный метод «neighbor-joining». Оценку статистической достоверности ветвления («bootstrap»-анализ) осуществляли на основе 1000 альтернативных деревьев.
Данные о нуклеотидных последовательностях фрагментов генов 16S рРНК исследованных бактериальных клонов депонированы в международной базе данных GenBank под номерами MH399229, MH410126-45.
Результаты и их обсуждение
Химический анализ водной вытяжки исследованного образца каменной соли показал, что ее общая минерализация составила 3.96%; концентрация ионов Na+, K+, Ca2+ – 4.13 г/л, 5.2 г/л, 20.95 г/л, соответственно.
Содержание хлороформного битума (ХБ) в образце составило 68.65 мг/кг. Газохроматический и масс-спектрометрический анализ хлороформных экстрактов показал присутствие углеводородной фракции (3.02%), представленной только алифатическими соединениями. Гетеросоединения (96.98%) являются доминирующими в ХБ и представлены кислородсодержащими структурами (41.07%), в основном – окисями, спиртами, эфирами (38.68%), а также кислотами и их производными (10.09%).
Из образца каменной соли путем прямого высева на агаризованную среду ATCC 213 «Halobacterium medium» не удалось выделить чистые культуры микроорганизмов, поэтому дальнейшие исследования проводились с использованием молекулярно-генетических методов.
Из накопительной культуры, полученной при культивировании образца каменной соли в жидкой среде ATCC 213 «Halobacterium medium», была выделена тотальная ДНК. Концентрация выделенной ДНК составляла 7.12 мкг/мл. При амплификации генов 16S рРНК бактерий с матрицы тотальной ДНК получен ПЦР-продукт искомого размера (около 1400 п.н.). В результате клонирования была создана библиотека генов 16S рРНК, состоящая из 120 бактериальных клонов с покрытием 87.2%, что указывает на то, что большая часть оцененного микробного разнообразия была взята в анализ.
На основе анализа полиморфизма длин рестрикционных фрагментов (ПДРФ-анализ) полученных ампликонов с использованием рестриктазы Hha I, исследуемые клоны были подразделены на 24 группы. На рисунке 1 представлена электрофореграмма рестрикционных фрагментов клонированных генов 16S рРНК восемнадцати произвольно выбранных клонов.
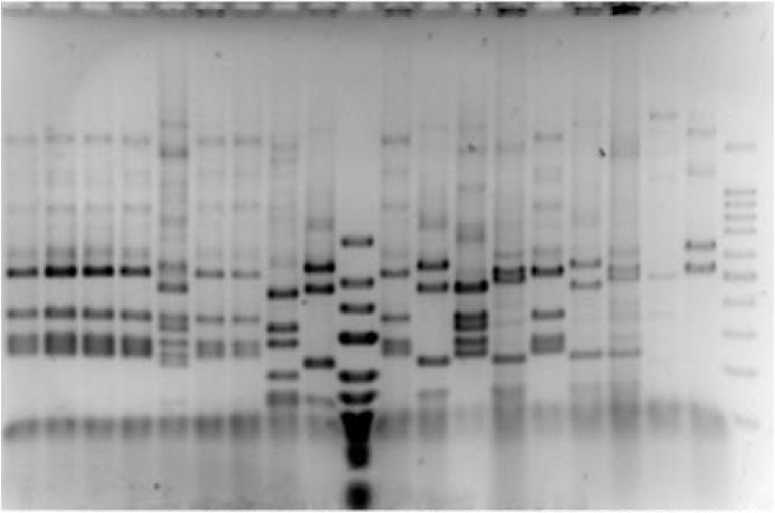
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
Рис. 1 . Электрофореграмма рестрикционных фрагментов клонированных генов 16S рРНК после обработки эндонуклеазаой HhaI :
10 – маркер длин ДНК GeneRulerTM Low Range DNA Ladder («Thermo Scientific», США); 20 – маркер длин ДНК 100+ bp DNA Ladder («Евроген», Россия); 1–9 и 11–19 – рекомбинантные клоны
[Electrophoregram of restriction fragments of cloned 16S rRNA genes after treatment with HhaI endonuclease:
10 - DNA length marker GeneRulerTM Low Range DNA Ladder (Thermo Scientific, USA); 20 - DNA length marker 100+ bp DNA Ladder (Eurogen, Russia); 1-9 and 11-19 - recombinant clones]
Далее, у представителей каждой рестрикционной группы были определены нуклеотидные последовательности фрагментов генов 16S рРНК. По результатам проведенного биоинформационного анализа, последовательности клонированных 16S рДНК были подразделены на 9 групп (операционных таксономических единиц, OTЕ). Индекс Симпсона (D) составлял 0.14, индекс разнообразия Шеннона (H) – 1.42.
Девять ОТЕ на филогенетическом дереве (рис. 2) формировали кластеры, соответствующие двум фи-лумам: « Proteobacteria » и « Actinobacteria ». Филум « Proteobacteria » включал 7 филотипов, относящихся к классам Alpha - Beta - и Gammaproteobacteria . Большая часть клонов (61.74% от всех бактериальных клонов) была отнесена к роду Pseudomonas (класс Gammaproteobacteria ). Наибольший процент сходства (99.73%) клоны имели с типовым штаммом вида Pseudomonas edaphica , изолированного из ризосферы многолетнего кустарника Cistus ladanifer L. (Испания) [Ramirez-Bahena et al., 2019].
Также были выявлены клоны, близкородственные (уровень сходства по генам 16S рРНК составлял 99.34–100%) представителям родов Serratia и Shigella (семейство Enterobacteriaceae , класс Gammaproteobacteria ) и некультивируемым бактериальным клонам генов 16S рРНК, обнаруженных в различных природных источниках, в том числе в арктических донных осадках (FJ849472) [Larouche et al., 2012], в микробиоме северного калифорнийского морского льва (JQ207675), заболоченной почве в Китае (KR148991) (рис. 2).
В исследованном образце каменной соли были обнаружены гены 16S рРНК (филотипы), проявляющие наибольшее сходство с представителями почвенных (ризосферных) бактерий рода Ralstonia (класс Betaproteobacteria ) и рода Phyllobacterium (класс Alphaproteobacteria ) (рис. 2). Кроме того, выявлены два филотипа класса Alphaproteobacteria (клон 66ВА и клон 12ВА), проявляющие низкий уровень сходства по генам 16S рРНК (98.46%) с типовыми штаммами узаконенных видов родов Mesorhizobium ( Mesorhizo-bium alhagi CCNWXJ12-2T) и Chelativorans ( Chelativoran smultitrophicus DSM9103T) [Zhou et al., 2012; Doronina et al., 2010], и которые, вероятно, представляют новые таксономические единицы.
В пределах филума « Actinobacteria » встречались только представители класса Actinobacteria . Около 20% бактериальных клонов от общего их числа включают последовательности генов 16S рРНК с уровнем сходства 99.72% с Rhodococcus qingshengii JCM15477T. Другая группа клонов (0.87% клонов) принадлежит минорному филотипу и содержит последовательность, близкую таковой типового штамма вида
Demequina aestuarii (уровень сходства 99.55%), который был изолирован из донных морских отложений в Корее [Hamada et al., 2015] (рис. 2).
я
<1
IM
|&ттдИа;^м11й1:аБК2(КЯ035070) '
KiKHSedNCB15(FJ849472)
Кпеи92ВА(МН410140)
Клон 77ВА (МН4К130> Клеи ЗОВА (МН4К130> Клеи 119ВА(МН4К14<
ти
---- Клеи 53ВА (MH4K135> Клеи КОВА (МН4К141)
SfogeIIas™si СЕСТ4887’ (FR870445 ™ S6igftt2sp. CH-42 (KR148991) KudhSItlRc 02 0SL4aFA(JQ207675)
Клеи ИВА (МН4К127)
Клеи 121ВА (MH4K145JL Кши 7В А (МН4К126)
Psexdomoxa^ A7ap6i:aKD25’ (LC272923)
Gammaproteobacferia
Psesdraaxm oart^enmns OLsAul(MF598585) Клин S1-037 (KF182781) -Клеи87ВА(МН410139)
-Клеи 1KB A (MH4KU43X Knot 6 В A (МН3992Э)
- №гаа;И АТС C 27511’ (10VLO1000020)
Клои39ВА(МН4К131)
JWsiasia sp. IWLA2 (MN515074)
JMs}asiasp.MI16(LC385700)
-Кп$иК5ВА(МН4К142)
Клон8У71112-1 (KM269680)
<7
Ki™10UB(K¥968992)
Ki™JR42(KC207921) «liWtii™ И 8658’ (KC577468)
Клои71ВА(МН410137)
е СЬе&Штогаа! жибйгирМст DSM9103T (EF457243) СЬеЬЩ-югш» xiiiititrc^!ii:iis LPM-410(FJ167675)
Кп<«12ВА(™410128)
Aiphaproteobacieria
' pKmHSIHA746N9D0 168 В (LN568107)
J |—Клои66ВА(МН4К136)
^7 tesrbizsii™ al&igi CCNWKJ12-2’ (АНАМ) 1000052)
ii
iifcssrtozsMim sp. L CD31 (MH59602) К1юн№игб™#шм sp.T15 (LC070335)
РЫосмта: sp.Toyi4Cthlll (MT626151)
Phsdococcus degra^ixs CsDWL-128 (MT415127)
Phsdococcus qixgsiexgii JCM 15477’(LKRJO1000016)
Клеи 25В А (MH410129X Клеи SOBA (MH4K132> Клеи 52BA (MH4K134)
I---КлоноЫТО! (EFO34304)
TuTil К™™ 51В А (МН410133)
SwItw^aiaaa^jaaniNBRC 106260’ (BBRD01000001) L Demeqxilia sp. ЕЫ 80499 (КТ734858)
Aitmobacts™
ijiji
Рис. 2 . Филогенетическое дерево, построенное с использованием метода neighbor-joining, показывающее положение исследуемых бактериальных клонов, основанное на сравнении нуклеотидных последовательностей гена 16S рРНК. Эволюционные расстояния рассчитаны по методу Jukes-Cantor. Масштаб соответствует 2 нуклеотидным заменам на каждые 100 нуклеотидов. Номера в базе данных GenBank указаны в скобках. Клоны с идентичными генами 16S рРНК перечислены через запятую
[A phylogenetic tree constructed using the neighbor-joining method, showing the position of the studied bacterial clones, based on a comparison of the nucleotide sequences of the 16S rRNA gene. The evolutionary distances are calculated using the Jukes-Cantor method. The scale corresponds to 2 nucleotide substitutions for every 100 nucleotides. The numbers in the GenBank database are shown in parentheses. Clones with identical 16S rRNA genes are comma-separated]
Заключение
В результате проведенных исследований получены новые данные о разнообразии бактерий солей ВКМС. Установлено, что в образце каменной соли, отобранном с глубины 239 м, присутствуют некуль-тивируемые бактерии различных таксонов: молекулярными методами было выявлено девять ОТЕ, принадлежащих классам Actinobacteria, Alpha -, Beta - и Gammaproteobacteria . Выявлены филотипы, проявляющие низкий уровень сходства по генам 16S рРНК с типовыми штаммами узаконенных видов, которые могут представлять новые таксономические единицы.
Список литературы Филогенетическое разнообразие бактерий в каменной соли Верхнекамского месторождения (Пермский край)
- Барях А.А., Асанов В.А., Паньков И.Л. Физико-механические свойства соляных пород Верхнекамского калийного месторождения: учеб. пособие. Пермь, 2008. 199 с.
- Бачурин Б.А., Одинцова Т.А., Хохрякова Е.С. Углеводородные маркеры органического вещества солей Верхнекамского месторождения // Проблемы минералогии, петрографии и металлогении. Научные чтения памяти П.Н. Чирвинского. 2016. № 19. С. 315-323.
- Корсакова Е.С. и др. Разнообразие бактерий семейства Halomonadaceae района разработок Верхнекамского месторождения солей // Микробиология. 2013. Т. 82, № 2. С. 247-250.
- Кудряшов А.И. Верхнекамское месторождение солей. Пермь, 2001. 429 с.
- Практикум по агрохимии: учеб. пособие / под ред. В.Г. Минеева и др. М.: Изд-во МГУ, 2001. 689 с.
- Равин Н.В. и др. Метагеномика как инструмент изучения «некультивируемых» микроорганизмов // Генетика. 2015. Т. 51, № 5. С. 519-528.
- Alain K., Querellou J. Cultivating the uncultured: limits, advances and future challenges // Extremophiles: Life Under Extreme Conditions. 2009. Vol. 13, № 4. P. 583-594.
- Ausbel F.M. et al. Short protocols in molecular biology. Third edition. N.Y.: John Wiley and Sons, 1995. 450 p.
- Doronina N.V. et al. Chelativorans multitrophicus gen. nov., sp. nov. and Chelativorans oligotrophicus sp. nov., aerobic EDTA-degrading bacteria // Int. J. Syst. Evol. Microbiol. 2010. Vol. 60 (Pt 5). P. 1044-1051.
- Fierer N. et al. Assessment of soil microbial community structure by use of taxon-specific quantitative PCR assays // Applied and Environmental Microbiology. 2005. Vol. 71, № 7. P. 4117-4120.
- Hamada M. et al. Draft genome sequences of eight type strains of the genus Demequina // Genome An-nounc. 2015. Vol. 3, № 2. P. e00281-15.
- Jaakkola S.T. et al. Buried alive: microbes from ancient halite // Trends Microbiol. 2016. Vol. 24, № 2. P. 148-160.
- Lane D.J. 16S/23S rRNA sequencing. In: Nucleic acid techniques in bacterial systematics / Eds. E. Stackebrandt, M. Goodfellow. N.Y.: John Wiley and Sons, 1991. P. 115-175.
- Larouche J.R. et al. Microbial biogeography of arctic streams: exploring influences of lithology and habitat // Front Microbiol. 2012. Vol. 3. P. 309.
- Margesin R., Schinner F. Potential of halotolerant and halophilic microorganisms for biotechnology // Extremophiles. 2001. Vol. 5, № 2. P. 73-83.
- Ramirez-Bahena M.H. et al. Pseudomonas edaphica sp. nov., isolated from rhizospheric soil of Cistus ladanifer L. in Spain // Int. J. Syst. Evol. Microbiol. 2019. Vol. 69. P. 3141-3147.
- Zhou M. et al. Draft genome sequence of Mesorhizobium alhagi CCNWXJ12-2T, a novel salt-resistant species isolated from the desert of northwestern China // J. Bacteriol. 2012. Vol. 194, № 5. P. 1261-1262.


