Генетическая характеристика поволжских татар по данным об однородительских маркерах
Автор: Трофимова Наталья Вадимовна, Литвинов Сергей Сергеевич, Хусаинова Рита Игоревна, Ахметова Вита Леоновна, Ахатова Фарида Сериковна, Виллемс Рихард, Хуснутдинова Эльза Камилевна
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Экология
Статья в выпуске: 3-5 т.15, 2013 года.
Бесплатный доступ
Проведен анализ полиморфизма митохондриальной ДНК (мтДНК) и Y хромосомы в двух субпопуляциях татар, проживающих на территории Волго-Уральского региона России. Анализ полиморфизма мтДНК показал, что большинство обнаруженных гаплогрупп мтДНК характерны для народов Западной Евразии. Доля восточно-евразийского компонента у казанских и туймазинских татар составила 11,4% и 23% соответственно. В субпопуляциях татар были выявлены ранее не обнаруженные гаплогруппы B4, F. Показатель генного разнообразия по изученному набору гаплогрупп оказался выше у казанских татар по сравнению с туймазинскими татарами. Проведен анализ распределения частот гаплогрупп Y хромосомы, который показал, что мажорными гаплогруппами Y-хромосомы в обеих субпопуляциях являются R1a-Page07, N-M231 и I-M170. При этом у туймазинских татар, в отличие от казанских, была с относительно высокой частотой обнаружена гаплогруппа R1b-M269, что указывает, по всей видимости, на эффект основателя в этой субпопуляции.
Митохондриальная днк, y-хромосома, филогеография, поволжские татары
Короткий адрес: https://sciup.org/148202021
IDR: 148202021 | УДК: 575.174:599.9
Текст научной статьи Генетическая характеристика поволжских татар по данным об однородительских маркерах
Татары – одна из самых крупных народностей Российской Федерации. По численности в стране они уступают только русским. По данным переписи населения 2010 г., в общей сложности в России проживает около 5 млн 364 тыс татар. Наиболее многочисленная группа татар проживает в Республике Татарстан – 2 млн 43 тыс чел., в Башкортостане их насчитывается более 1 млн 132 тыс чел. Помимо Республики Татарстан, крупные по численности группы татарского населения присутствуют в Челябинской, Свердловской, Ульяновской, Самарской, Кировской областях, Пермском крае, Башкортостане, Удмуртии, Мордовии, Чувашии, Марий Эл [1]. Следует отметить, что татары расселены очень дисперсно, и более или менее многочисленные группы татарского населения имеются практически во всех субъектах Российской Федерации. Язык поволжских татар относится к кипчакским (кыпчакским) языкам тюркской группы алтайской семьи, к которой также причисляются башкиры [2].
По мнению Р.К. Кузеева [3], татары произошли от тюркоязычных племен, которые расселились в раннем средневековье на территории Средней Волги и Нижнего Прикамья, включив, по всей видимо-
сти, в свой состав более раннее финно-угорское и скифо-сарматское население этих территорий Восточной Европы.
В литературе имеются данные о генетическом разнообразии в субпопуляциях татар из Альметьевского, Елабужского [4-5], а также Азнакаевского и Буинского районов [6] Республики Татарстан, поэтому целью этой работы является получение представления о генетическом разнообразии поволжских татар Туймазинского района Республики Башкортостан, а также дисперсной выборки из различных районов Татарстана. При этом мы использовали комплексный подход, который включал в себя анализ полиморфизма как митохондриальной ДНК (мтДНК), так и нерекомбинирующей области Y-хромосомы, что позволяет оценить генетическую структуру популяций как по женской, так и по мужской линии.
Материалом для исследования служили образцы ДНК, выделенные из цельной крови неродственных индивидов, представляющих две субпопуляции татар: казанские татары из различных районов Татарстана (N=53) и татары, проживающие на территории Туймазинского района Республики Башкортостан (N=48). Несмотря на то, что в тексте обсуждаются туймазинские и казанские татары, данное разделение условно, поскольку указывает только на географическое различие, причем казанские татары, как было отмечено, представляют собой дисперсную выборку. Этническая принадлежность, происхождение и родственные отношения уточнялись путем анкетирования, учитывая данные до третьего поколения. Все обследованные дали информационное согласие на участие в исследовании.
Выделение тотальной ДНК проводилось стандартным фенол-хлороформным методом [7]. Проанализированы нуклеотидная последовательность гипервариабельного сегмента 1 (ГВС1) мтДНК длиной 377 п.н.(16024-16400), а также мутации в кодирующем регионе. Определение нуклеотидных последовательностей мтДНК осуществлялось на автоматическом секвенаторе Applied Biosystems (ABI) 3730XL DNA Analyzer. Последовательности были выровнены и проанализированы с помощью программы Sequencer 5.1. Определение гаплогрупп и гаплотипов мтДНК проводилось путем совместного анализа статуса диагностических сайтов рестрикции в кодирующем регионе мтДНК и мотивов ГВСI [8-10].
Показатель генного разнообразия рассчитывали по формуле [11]:
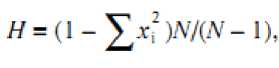
где x i - частота каждой гаплогруппы мтДНК в субпопуляции, N – объем выборки.
Анализ диаллельных маркеров нерекомбинирующей области Y-хромосомы проводился по следующим 14 локусам: M9, M89, YAP (M1), M20, M231, 92R7, M207, M306 (M173), Page07, M269, M170, 12f2, M175, M130. Гаплогруппы Y-хромосомы определялись согласно обновленной классификации консорциума по изучению Y-хромосомы (YCC) [12-14]. Исследование полиморфизма 14 диаллельных локусов Y-хромосомы производилось с использованием ПЦР и последующего ПДРФ анализа. Последний проводился с помощью эндонуклеаз рестрикции «Fermentas» (Литва) и «СибЭнзим» (Россия). Полиморфизм локусов YAP и 12f2 анализировали без проведения рестрикции.
В проведенном исследовании гаплогрупп мтДНК, выявленных у казанских татар и татар из Туймазинского района Республики Башкортостан, было показано, что в анализируемых субпопуляциях западно-евразийские линии преобладают над восточно-евразийскими. В таблице приведены частоты основных митохондриальных гаплогрупп, обнаруженных в ходе исследования.
У туймазинских татар наблюдалось более низкое значение индекса генетического разнообразия (Н) по проанализированному набору гаплогрупп мтДНК (0,92) по сравнению с казанскими татарами, у которых он составлял 0,94.
Западноевразийский компонент в изученных популяциях представлен гаплогруппами H, HV1, I1a, J1, K1, N, R2, T1a, T2, U1, U2, U4, U5, U5a, U5b, U8, V, V7, V13, W1, W3, W6, X2, Y1. Из них с максимальной частотой встречается гаплогруппа Н, относящаяся к числу самых распространенных гап-логрупп мтДНК в Евразии. Её частота составляет 40-50% всех гаплогрупп в большинстве стран
Таблица. Частоты гаплогрупп мтДНК у поволжских татар
|
Казанские татары |
Туймазинские татары |
|||
|
N |
Частота гаплогрупп, % |
N |
Частота гаплогрупп, % |
|
|
Западно-евразийский компонент |
||||
|
H |
11 |
20,8 |
12 |
25,2 |
|
HV1 |
0 |
0 |
1 |
2,1 |
|
I1a |
1 |
1,9 |
0 |
0 |
|
J1 |
7 |
13,2 |
4 |
8,3 |
|
K1 |
3 |
5,7 |
1 |
2,1 |
|
N |
3 |
5,7 |
0 |
0 |
|
R2 |
1 |
1,9 |
0 |
0 |
|
T1a |
2 |
3,8 |
4 |
8,3 |
|
T2 |
3 |
5,7 |
2 |
4,2 |
|
U1 |
1 |
1,9 |
0 |
0 |
|
U2 |
2 |
3,8 |
0 |
0 |
|
U4 |
1 |
1,9 |
4 |
8,3 |
|
U5 |
1 |
1,9 |
0 |
0 |
|
U5a |
2 |
3,8 |
2 |
4,2 |
|
U5b |
2 |
3,8 |
1 |
2,1 |
|
U8 |
1 |
1,9 |
2 |
4,2 |
|
V |
0 |
0 |
1 |
2,1 |
|
V7 |
1 |
1,9 |
1 |
2,1 |
|
V13 |
2 |
3,8 |
0 |
0 |
|
W1 |
0 |
0 |
1 |
2,1 |
|
W3 |
1 |
1,9 |
0 |
0 |
|
W6 |
0 |
0 |
1 |
2,1 |
|
X2 |
1 |
1,9 |
0 |
0 |
|
Y1 |
1 |
1,9 |
0 |
0 |
|
Всего |
47 |
89,1 |
37 |
77,4 |
|
Восточно-евразийский компонент |
||||
|
A |
1 |
1,9 |
1 |
2,1 |
|
B4 |
1 |
1,9 |
1 |
2,1 |
|
B5 |
0 |
0 |
1 |
2,1 |
|
C4 |
0 |
0 |
1 |
2,1 |
|
D4 |
3 |
5,7 |
4 |
8,3 |
|
F1a |
0 |
0 |
1 |
2,1 |
|
G2a |
0 |
0 |
2 |
4,2 |
|
M3a |
1 |
1,9 |
0 |
0 |
|
Всего |
6 |
11,4 |
11 |
23 |
Европы, и около 20-30% гаплогрупп Ближнего Востока и Кавказа [15-18].
У казанских татар ее частота составляет 20,8 %, а у туймазинских 25,2%. Данная картина в принципе соответствует распределению гаплогруппы H на территории Евразии.
В изученных нами выборках татар была обнаружена гаплогруппа V с частотой 5,7% у казанских татар и 4,2% у туймазинских. Как было показано ранее, данная гаплогруппа распространена в Западной и Восточной Европе с пиками в стране Басков [15] и в популяции саамов [19]. Возраст ее коалесценции варьирует от 16,3±4,8 тыс лет назад в Западной Европе до 8,5 ± 2,3 тыс лет назад на Востоке Европы [15, 20]. Кроме того, у казанских татар была обнаружена редко встречающаяся субгапло-группа V13, характеризующаяся заменой G12795A относительно rCRS [21]. Данная субгаплогруппа ранее была обнаружена в популяции татар [22]. Кроме того, она встречается в финно-угорских и тюркских популяциях Волго-Уральского региона [наши неопубликованные данные].
Восточно-евразийский компонент в изученных популяциях татар представлен гаплогруппами A, B4, B5, C4, D4, F1a, G2a, M3a и составляет 11,4% митохондриального пула у казанских татар и 23% у туймазинских. Отдельный интерес представляют гаплогруппы В4 и В5. Гаплогруппа B4 присутствует в генофондах практически всех этнических групп Северной и Восточной Азии, за исключением восточных эвенков, чукчей, коряков и эвенов. Максимальная частота гаплогруппы B4 зарегистрирована у корейцев (20,4%) и монголов (14,9%), а в Северной Азии ее частота изменяется в диапазоне от 3,1% у якутов до 11,3% у теленгитов [23]. Также было показано, что эти гаплогруппы встречаются у тувинцев и тофалар с частотой 5,3% и 4,3% соответственно [24]. Интересно, что гаплогруппы В4 и С4 были обнаружены при анализе древней ДНК из двух археологических памятников в Западной Сибири: Сайгатинский 6 (с VIII по XI вв.) и Зеленый Яр (XVIII в.) [25]. Ранее гаплогруппа B в ВолгоУральском регионе была обнаружена только у башкир [4]. Результаты нашего исследования показывают наличие этой гаплогруппы и в субпопуляции поволжских татар, причем гаплотип ГВС1 у проанализированных нами образцов для субгапло-группы B4b1 идентичен и представлен мотивом 16136, 16183C, 16189, 16217, 16256, 16519, что может свидетельствовать в пользу недавней дивергенции субпопуляций поволжских татар. С другой стороны, малая частота данной гаплогруппы в изученных популяциях не позволяет однозначно трактовать полученные результаты.
Гаплогруппы B5, C4, F1a, G2a были обнаружены у туймазинских татар и отсутствовали у казанских. Гаплогруппа G2 c высокой частотой встречается в популяциях бурят (11%), монголов (8,5%) [24], а в целом гаплогруппа G присутствует также в Волго-Уральском регионе – у коми (2,4%), татар (1,6%), чувашей (0,7%) и башкир (4,8%) [4].
В результате анализа Y-хромосомы нами было обнаружено различие в генетической структуре изученных субпопуляций. Несмотря на то, что пик частот в обеих субпопуляциях приходится на мажорные гаплогруппы R1a-Page07, N-M231 и I-M170, у казанских татар не было обнаружено гап-логруппы R1b-M269, в то время как у туймазин-ских ее доля составляет 15,7%. Даже учитывая, что в настоящий момент микросателлитное разнообразие внутри данной гаплогруппы у туймазинских татар не было проанализировано, обнаруженное различие весьма интересно. Однако можно предположить, что в данной субпопуляции имел место эффект основателя.
Таким образом, изученные субпопуляции татар в целом демонстрируют распределение гаплогрупп мтДНК, характерное для популяций Евразии с преобладающей в митохондриальном пуле гаплогруп-пой H. При этом восточно-евразийский компонент имеет большую встречаемость у туймазинских татар, хотя в целом его доля в изученных субпопуляциях существенно ниже по сравнению с западноевразийским. Мажорными гаплогруппами Y-хромосомы в обеих субпопуляциях являются R1a-Page07, N-M231 и I-M170. При этом у туймазин-ских татар, в отличие от казанских, была с относительно высокой частотой была обнаружена гапло-группа R1b-M269, что указывает, по всей видимости, на эффект основателя в этой субпопуляции.
-
1. Всероссийская перепись населения 2010 года. URL: http://www.perepis-2010.ru
-
2. Белых С.К. История народов Волго-Уральского региона: Учебное пособие. Ижевск, 2006. 54 с.
-
3. Кузеев Р.Г. Народы Среднего Поволжья и Южного Урала: этногенетический взгляд на историю. М.: Наука, 1992. 345 с.
-
4. Бермишева М.А., Тамбетс К., Виллемс Р., Хуснутдинова Э.К. Разнообразие гаплогрупп митохондриальной ДНК в этнических группах Волго-Уральского региона // Мол. биология. 2002. Т. 36. С. 990-1001.
-
5. Орехов В.А . Характеристика митотипов представителей трех этнических групп европейской части России: Авто-реф. дис. … канд. биол. наук. М.: ИОГен РАН, 2002. 22 с.
-
6. Денисова Г.А., Малярчук Б.А., Деренко М.В., Кравцова О.А. Популяционная структура поволжских татар по данным о разнообразии митохондриальной ДНК // Генетика. 2011. Т. 47. № 3. С. 387-393.
-
7. Mathew C.C . The isolation of high molecular weight eucari-otic DNA // Methods in molecular biology/ Ed. Walker J.M. New York, 1984. P. 31-34.
-
8. Macaulay V., Richards M., Hickey E. et al . The emerging tree of West Eurasian mtDNAs: a sinthesis of control region sequenses and RFLPs // Am. J. Genet. 1999. V. 64. P. 232249.
-
9. Richards M., Macaulay V., Hickey E. et al. Tracing European founder lineages in the Near eastern mtDNA pool // Am. J. Hum. Genet. 2000. V. 67. P. 1251-1276.
-
10. Oven M. van, Kayser M. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation // Hum. Mutat. 2009. V. 30. N. 2. P. 386-394.
-
11. Nei M. Molecular Evolutionary Genetics. New York: Columbia Univ. Press, 1988.
-
12. Y Chromosome Consortium. A nomenclature system for the tree of human Y-chromosomal binary haplogroups // Genome Res. 2002. V. 12. P. 339-348.
-
13. Karafet T.M., Mendez F.L., Meilerman M.B., Underhill P.A., Zegura S.L., Hammer M.F . New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree // Genome Res. 2008. V. 5. P. 830-838.
-
14. Myres N.M., Rootsi S., Lin A.A. et al. A major Y-chromosome haplogroup R1b Holocene era founder effect in Central and Western Europe // Eur. J. Hum. Genet. 2011. V. 19. P. 95-101.
-
15. Álvarez-Iglesias V., Mosquera-Miguel A., Cerezo M. et al . A. New Population and Phylogenetic Features of the Internal Variation within Mitochondrial DNA Macro-Haplogroup R0 // PLoS ONE. 2009. 4(4): e5112.
-
16. Achilli A., Rengo C., Magri C. et al. The molecular dissection of mtDNA haplogroup H confirms that the Franco-Cantabrian glacial refuge was a major source for the European gene pool // Am. J. Hum. Genet. 2004. V. 75. P. 910918.
-
17. Loogvali E.L., Roostalu U., Malyarchuk B.A. et al . Disuniting uniformity: a pied cladistic canvas of mtDNA haplo-group H in Eurasia // Mol. Biol. Evol. 2004. V. 21. P. 20122021.
-
18. Roostalu U., Kutuev I., Loogvali E.L. et al. Origin and expansion of haplogroup H, the dominant human mitochondrial DNA lineage in West Eurasia: the Near Eastern and Caucasian perspective // Mol. Biol. Evol. 2007. V. 24. P. 436448
-
19. Tambets K., Rootsi S., Kivisild T. et al. The Western and Eastern roots of the Saami – the story of genetic “outliers” told by mitochondrial DNA and Y-chromosomes // Am. J. Hum. Genet. 2004. V. 74. P. 661-682.
-
20. Torroni A., Bandelt H.-J., Macaulay V., Richards M., Cru-ciani F. et al . A signal, from human mtDNA, of postglacial recolonization in Europe // Am. J. Hum. Genet. 2001. V. 69.
-
21. Andrews R.M., Kubacka I., Chinnery P.F. et al . Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA // Nature Genet. 1999. V. 23. P. 147.
-
22. Malyarchuk B., Derenko M., Denisova G., Kravtsova O . Mi-togenomic diversity in Tatars from the Volga-Ural region of Russia // Mol. Biol. and Evolution. 2010. V. 27. P. 22202226.
-
23. Деренко М.В., Малярчук Б.А . Молекулярная филогеография населения Северной Евразии по данным об изменчивости митохондриальной ДНК / Oтв. ред. И.А. За-харов-Гезехус. Магадан: СВНЦ ДВО РАН, 2010. 120 с.
-
24. Starikovskaya E.B., Sukernik R.I., Derbeneva O.A. et al . Mitochondrial DNA diversity in indigenous populations of the southern extent of Siberia, and the origins of Native American haplogroups // Ann. Hum. Genet. 2005. V. 69. P. 67-89.
-
25. Sato A., Razhev D., Amano T., Masuda R . Genetic features of ancient West Siberian people of the Middle Ages, revealed by mitochondrial DNA haplogroup analysis // J. Human Genetics. 2011. V. 56. P. 602-608.
P. 844-852.
GENETIC CHARACTERISTIC
ON UNIPARENTAL
OF VOLGA TATARS BASED MARKERS DATA
Список литературы Генетическая характеристика поволжских татар по данным об однородительских маркерах
- Всероссийская перепись населения 2010 года. URL: http://www.perepis-2010.ru
- Белых С.К. История народов Волго-Уральского региона: Учебное пособие. Ижевск, 2006. 54 с.
- Кузеев Р.Г. Народы Среднего Поволжья и Южного Урала: этногенетический взгляд на историю. М.: Наука, 1992. 345 с.
- Бермишева М.А., Тамбетс К., Виллемс Р., Хуснутдинова Э.К. Разнообразие гаплогрупп митохондриальной ДНК в этнических группах Волго-Уральского региона//Мол. биология. 2002. Т. 36. С. 990-1001.
- Орехов В.А. Характеристика митотипов представителей трех этнических групп европейской части России: Автореф. дис. … канд. биол. наук. М.: ИОГен РАН, 2002. 22 с.
- Денисова Г.А., Малярчук Б.А., Деренко М.В., Кравцова О.А. Популяционная структура поволжских татар по данным о разнообразии митохондриальной ДНК//Генетика. 2011. Т. 47. № 3. С. 387-393.
- Mathew C.C. The isolation of high molecular weight eucariotic DNA//Methods in molecular biology/Ed. Walker J.M. New York, 1984. P. 31-34.
- Macaulay V., Richards M., Hickey E. et al. The emerging tree of West Eurasian mtDNAs: a sinthesis of control region sequenses and RFLPs//Am. J. Genet. 1999. V. 64. P. 232-249.
- Richards M., Macaulay V., Hickey E. et al. Tracing Euro pean founder lineages in the Near eastern mtDNA pool//Am. J. Hum. Genet. 2000. V. 67. P. 1251-1276.
- Oven M. van, Kayser M. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation//Hum. Mutat. 2009. V. 30. N. 2. P. 386-394.
- Nei M. Molecular Evolutionary Genetics. New York: Columbia Univ. Press, 1988.
- Y Chromosome Consortium. A nomenclature system for the tree of human Y-chromosomal binary haplogroups//Genome Res. 2002. V. 12. P. 339-348.
- Karafet T.M., Mendez F.L., Meilerman M.B., Underhill P.A., Zegura S.L., Hammer M.F. New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree//Genome Res. 2008. V. 5. P. 830-838.
- Myres N.M., Rootsi S., Lin A.A. et al. A major Y-chromosome haplogroup R1b Holocene era founder effect in Central and Western Europe//Eur. J. Hum. Genet. 2011. V. 19. P. 95-101.
- Álvarez-Iglesias V., Mosquera-Miguel A., Cerezo M. et al. A. New Population and Phylogenetic Features of the Internal Variation within Mitochondrial DNA Macro-Haplogroup R0//PLoS ONE. 2009. 4(4): e5112.
- Achilli A., Rengo C., Magri C. et al. The molecular dissection of mtDNA haplogroup H confirms that the Franco-Cantabrian glacial refuge was a major source for the European gene pool//Am. J. Hum. Genet. 2004. V. 75. P. 910-918.
- Loogvali E.L., Roostalu U., Malyarchuk B.A. et al. Disuniting uniformity: a pied cladistic canvas of mtDNA haplogroup H in Eurasia//Mol. Biol. Evol. 2004. V. 21. P. 2012-2021.
- Roostalu U., Kutuev I., Loogvali E.L. et al. Origin and expansion of haplogroup H, the dominant human mitochondrial DNA lineage in West Eurasia: the Near Eastern and Caucasian perspective//Mol. Biol. Evol. 2007. V. 24. P. 436-448
- Tambets K., Rootsi S., Kivisild T. et al. The Western and Eastern roots of the Saami -the story of genetic “outliers” told by mitochondrial DNA and Y-chromosomes//Am. J. Hum. Genet. 2004. V. 74. P. 661-682.
- Torroni A., Bandelt H.-J., Macaulay V., Richards M., Cruciani F. et al. A signal, from human mtDNA, of postglacial recolonization in Europe//Am. J. Hum. Genet. 2001. V. 69. P. 844-852.
- Andrews R.M., Kubacka I., Chinnery P.F. et al. Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA//Nature Genet. 1999. V. 23. P. 147.
- Malyarchuk B., Derenko M., Denisova G., Kravtsova O. Mitogenomic diversity in Tatars from the Volga-Ural region of Russia//Mol. Biol. and Evolution. 2010. V. 27. P. 2220-2226.
- Деренко М.В., Малярчук Б.А. Молекулярная филогеография населения Северной Евразии по данным об изменчивости митохондриальной ДНК/Oтв. ред. И.А. Захаров-Гезехус. Магадан: СВНЦ ДВО РАН, 2010. 120 с.
- Starikovskaya E.B., Sukernik R.I., Derbeneva O.A. et al. Mitochondrial DNA diversity in indigenous populations of the southern extent of Siberia, and the origins of Native American haplogroups//Ann. Hum. Genet. 2005. V. 69. P. 67-89.
- Sato A., Razhev D., Amano T., Masuda R. Genetic features of ancient West Siberian people of the Middle Ages, revealed by mitochondrial DNA haplogroup analysis//J. Human Genetics. 2011. V. 56. P. 602-608.


