Генетические маркеры: сравнительная классификация молекулярных маркеров
Автор: Чесноков Ю.В.
Журнал: Овощи России @vegetables
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 3 (41), 2018 года.
Бесплатный доступ
С развитием молекулярных маркеров, позволяющих производить анализ генотипов на уровне первичного носителя генетической информации - ДНК, появился наиболее разнообразный и наиболее многочисленный класс маркеров на сегодня. Это связано с тем, что каждая отдельно взятая нуклеотидная последовательность уникальна по своей структуре. Совокупность молекулярногенетических методов, получившая название ДНК-фингерпринтинг, наиболее широко используется в современных исследованиях для решения самыхразных задач в различных биологических дисциплинах. В этой связи необходимость в сравнительной классификации существующих на сегодня молекулярно-генетических маркеров представляется актуальной. Исходя из опубликованного литературного материала, приводятся данные по предлагавшимся классификациям молекулярных маркеров. Дается определение термину «маркер » в генетике и селекции. Приводятся свойства и отличительные особенности генетических маркеров. Определяется что такое «хороший» генетический маркер, а также виды, категории, вариации и типы наследования молекулярных маркеров. Выявляемый с помощью молекулярных маркеров полиморфизм подразделяется на полиморфизм самой последовательности (включая нуклеотидные замены и инсерции-делеции) и полиморфизм числа тандемно повторяющихся последовательностей в повторяющихся районах. Кроме того, молекулярные маркеры можно классифицировать на две вариации: анонимные, т.е. те, в которых нуклеотидная последовательность не известна и для проявления молекулярного маркера ее установление не требуется (например, RAPD, AFLP, RFLP), и анонсированные (или детерминированные), т.е. те, в которых нуклеотидная последовательность известна либо выявляется в процессе проведения анализа (например, SNP, CAPS, STS). Однако, каким бы ни было предполагаемое применение молекулярных маркеров, выбор метода исследования также будет зависеть и от изучаемого вида растений. Последующее влияние методов молекулярно-генетического анализа на генетику и практическую селекцию растений будет зависеть от результатов, которые будут получены, в частности, от выявления возможности или невозможности генотипирования особи по одному генетическому маркеру, и от экономической цены получаемых информативных данных.
Генетические маркеры, молекулярные маркеры, характерные свойства и отличия, классификация
Короткий адрес: https://sciup.org/140223782
IDR: 140223782 | УДК: 575.21
Текст научной статьи Генетические маркеры: сравнительная классификация молекулярных маркеров
Споявлением методологий, позволяющих оперировать с ДНК (основным носителем генетической информации на молекулярном уровне), внимание исследователей в значительной степени переключилось на нуклеиновые кислоты как источник информативного полиморфизма. Это связано с тем, что каждая отдельно взятая нуклеотидная последовательность уникальна по своей структуре [1]. Последовательность ДНК может быть использована для любых исследований генетического разнообразия и взаимодействия между организмами [2]. В последние десятилетия был разработан широкий спектр идентификации полиморфизма нуклеиновых кислот. Результатом таких работ стала разработка молекулярных методов исследования организмов с использованием ДНК-маркеров.
Молекулярные маркеры – наиболее разнообразный и наиболее многочисленный класс маркеров на сегодня. В целом, молекулярные маркеры, исходя из методической основы реализации маркерного подхода, можно разделить на три основные группы – маркеры, основанные на:
-
1) молекулярно-генетической блот-гибридизации – Саузерн-, Вестерн- и дот-гибридизации; к этой группе можно отнести и маркеры, разработанные на основе ДНК-чип технологий, по своей сути являющихся не чем иным, как современной, технологически усовер-
- шенствованной модификацией метода блот-гибридизации;
-
2) методе полимеразной цепной реакции (ПЦР);
-
3) методах секвенирования (прочтения последовательности) нуклеиновых кислот.
Следует отметить, что современные методы секвенирования нуклеиновых кислот базируются на использовании как методов биохимической реакции пошагового отщепления нуклеотидов, так и на методах ПЦР и блот-гибридиза-ции. В этом смысле секвенирование можно рассматривать как некое логическое развитие и совершенствование методов блот-гибридизации и ПЦР. Однако принципиальное отличие состоит в разрешающих способностях методологии секвенирования, которые позволяют выделить молекулярные маркеры, разработанные на ее основе, в самостоятельную группу. Секвенирование можно применять для прочтения не только нуклеотидных, но и аминокислотных последовательностей отдельных пептидов и белков. В этом смысле секвенирование можно рассматривать как некий современный подход, в равной степени применимый для анализа как нуклеиновых кислот, так и белков.
Совокупность молекулярно-генетических методов, получившая название ДНК-фингерпринтинг, наиболее широко используется в современных исследованиях для решения самых разных задач в различных биологических дисциплинах. Благодаря многообразию составляющих эту совокупность методов исследователь может не только маркировать геномы и генотипы, но и оценивать генетическое разнообразие внутри и между различными популяциями как естественного, так и искусственного происхождения вплоть до установления полиморфизма на уровне единичного нуклеотида. Однако во избежание некорректного смыслового понимания употребляемых терминов следует рассмотреть современное определение словосочетания «генетический маркер».
Что такое «генетический маркер»?
Как правило, термин «маркер» в генетике и селекции используется в значении «генетический маркер». Он также является синонимом словосочетания «маркерный локус». Маркерный локус – это полиморфный локус, который маркирует (метит):
-
• генотип несущего его индивидуума (как правило, словосочетание «маркерный локус» в этом значении используется в популяционной генетике);
-
• генотип одного или нескольких генетических локусов, связанных с маркером (это определение «маркерного локуса» обычно используется как при идентификации и клонировании генов или фрагментов ДНК, так и в маркерной селекции растений).
В целом генетический маркер может быть определен как: 1) хромосомная метка или аллель, позволяющие проследить специфичный район ДНК; 2) специфичный фрагмент ДНК с известной позицией в геноме; 3) ген, чья фенотипическая экспрессия обычно легко отличима и может быть использована для идентификации несущей ее особи или клетки (ткани, органа) либо как зонд для мечения клеточных органелл (ядра, митохондрий, хлоропластов), хромосом или хромосомных локусов.
Согласно установленной терминологии, генетические маркеры обычно подразделяют на три основных класса: морфологические маркеры (выявляемые на уровне фенотипа организма), молекулярные маркеры (выявляемые на уровне нуклеиновых кислот) и биохимические маркеры (различные белки, в том числе ферменты, и метаболиты). Последний термин, к сожалению, не вполне определен, поскольку в некоторых контекстах он обозначает молекулы, присутствие которых маркирует (метит) различные стадии дифференциации или какую-нибудь физиологическую стадию. К генетическим маркерам можно отнести и цитогенетические маркеры. Они занимают особое место, обладая одновременно свойствами как биохимических, так и молекулярных маркеров (табл. 1). Специфика выявления цитогенетических маркеров на цитологическом уровне делает их ограничено доступными, а также зависимыми от морфо-анатомического строения изучаемого организма, специального дорогостоящего оборудования и химре-активов. Вследствие этого цитогенетические маркеры, по сравнению с другими классами маркеров, не получили широкого распространения и возможности массового применения.
Что такое «хороший» генетический маркер?
«Идеальный» генетический маркер должен быть:
-
• полиморфным, поскольку «исходным материалом» генетика и селекционера является изменчивость, а полиморфизм – ее отражение или проявление;
-
• мультиаллельным, так как благодаря этому увеличивается частота и спектр полиморфности;
-
• кодоминантным, потому что в этом случае гетерозиготный гибрид одновременно проявляет свойства обоих родителей, что позволяет различать обе гомозиготы как друг от друга, так и от гетерозиготы; • неэпистатичным, поскольку проявление маркера индивидуального генотипа может быть выявлено визуально независимо от месторасположения выбранного маркера в геноме индивидуума (кодоми-нирование и неэпистатическое проявление признака могут быть определены как отсутствие внутри- и межлокусного взаимодействия, соответственно);
-
• «нейтральным», так как замена аллелей в маркерном локусе не имеет фенотипического или селективного эффекта (полиморфизм на молекулярном уровне ДНК почти всегда нейтрален);
-
• нечувствительным к воздействию окружающей среды, что должно проявляться
Таблица 1. Генетические маркеры, их свойства и отличительные особенности Table 1. Genetic markers, their characteristics and distinctive features
Морфологические маркеры практически не соответствуют этим критериям. Они недостаточно полиморфны и в большинстве случаев либо рецессивны, либо доминантны. Кроме того, они часто подвержены влиянию других свойств или признаков и могут быть зависимы от воздействия окружающей среды. В то же время, большинство биохимических и молекулярных маркеров отвечают вышеизложенным требованиям. Основное ограничение для изоферментов – это малое число локусов в геноме, которое может быть ими (изоферментами) маркировано или определено. Например, очень трудно выявлять 30-40 изоферментных маркеров одновременно в исследуемых расщепляющихся популяциях. Кроме того, не все ферменты активны или присутствуют во всех тканях или органах растений. Полиморфизм разного рода белков, выявляемый не только одномерным, но и двумерным электрофорезом, может быть более многочисленным и информативным, но также зависит от органа или тканей растений, из которых выделяли белки. В противоположность этому генетические маркеры на уровне ДНК практически бесчисленны и не зависят от специфичности анализируемых частей растений, а также от стадии развития организма, поскольку ДНК во всех органах и тканях данного растения по своему химическому составу и структуре всегда одна и та же. Более того, молекулярные маркеры, как и ДНК исследуемого организма, могут быть непосредственно использованы либо задействованы в дальнейших молекулярно-биологических исследованиях.
Молекулярные методы оценки и изучения генетического разнообразия у растений
Целый ряд молекулярно-генетических методов исследования может быть применен для изучения и анализа генетического разнообразия, существующего в коллекциях генетических ресурсов растений и ином селекционно-значимом материале, что имеет непосредственную ценность для проведения селекционного отбора. Эти методы уже широко применяют в генетике и селекции растений и многие из них довольно подробно описаны (см., например, [3-6]).
Виды, категории, вариации и типы наследования молекулярных маркеров
ДНК-маркеры особенно привлекательны тем, что с их помощью можно выявить различия между двумя особями одного или разных видов, что зачастую не удается сделать с помощью иных классов маркеров. Маркеры, позволяющие найти отличия между анализируемыми особями, называются полиморфными, тогда как
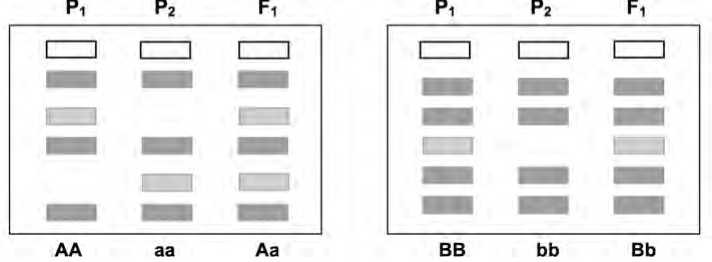
Рисунок. Сравнение кодоминантного (А) и доминантного (Б) полиморфных маркеров:
AA, aa, Aa, BB, bb, Bb – генотипы маркерных локусов; маркерные аллели: полиморфные – показаны более светлым затемнением, мономорфные – затемнены сильнее.
Figure. Comparison codomnant (A) and dominant (Б) polymorphic markers: AA, aa, Aa, BB, bb, Bb - marker loci genotypes; marker alleles: polymorphic - shown by more light shadowing, monomorphic - shadowing is dark.
маркеры, не позволяющие сделать это (например, электрофоретические полосы одного размера, присутствующие у всех исследуемых образцов), определяют как мономорфные. Такое деление справедливо и для биохимических, и морфологических маркеров. Кроме того, полиморфные молекулярные и биохимические маркеры по типу наследования можно разделить на кодоминантные и доминантные (рис.).
Такое разделение базируется на способности маркера отличать гетеро- и гомозиготы друг от друга. С помощью кодоминантных маркеров можно различать гомо- и гетерозиготы, в то время как доминантные маркеры этого делать не позволяют. Как правило, кодоминатные молекулярные и биохимические маркеры проявляют различия в размере электрофоретических полос, в то время как доминантные — в наличии или отсутствии таких полос. Строго говоря, различные формы проявления используемого молекулярного маркера (например, электрофоретические полосы различающихся размеров) являются «аллелями» данного ДНК-маркера. Кодоминантные маркеры могут иметь
Таблица 2. Классификация и сравнительные характеристики основных монолокусных и мультилокусных ДНК-маркеров, наиболее часто используемых при изучении генома растений (по: [7]; с изменениями)
Table 2. Classification and characterictics of main monolocus and multilocus DNA-markers most offen used upon genome investigation of plants (on [7], with changing)
В то же время с молекулярной точки зрения выявляемый полиморфизм можно подразделить на две категории: 1) полиморфизм самой последовательности (включая нуклеотидные замены и инсер-ции-делеции) и 2) полиморфизм числа тандемно повторяющихся последовательностей в повторяющихся районах. В настоящее время не существует подходов, специально разработанных для определения инсерционно-делеционного полиморфизма, который в любом случае устанавливается методами, позволяющими выявлять нуклеотидные замены. Кроме того, молекулярные маркеры можно классифицировать на две вариации: анонимные, т.е. те, в которых нуклеотидная последовательность не известна и для проявления молекулярного маркера ее установление не требуется (например, RAPD, AFLP, RFLP), и анонсированные (или детерминирован- ные), т.е. те, в которых нуклеотидная последовательность известна либо выявляется в процессе проведения анализа (например, SNP, CAPS, STS).
Вместе с тем с генетической точки зрения маркеры, удовлетворяющие критериям определения «хорошего» генетического маркера (см. выше), можно разделить на индивидуально определяемые (т.е. характеризующие индивидуальный локус), как правило, кодоминантные, и на множественные (т.е. характеризующие множество локусов одновременно), как правило, доминантные маркеры. Е.К. Хлесткина и Е.А. Салина [11, 23] подразделяют такие маркеры на монолокусные и мультилокус-ные (табл. 2). Эти различия, конечно же, являются упрощенными: например, при желании можно найти локус-специфичный и доминантный маркер. Тем не менее подразделение маркеров на категории моно-и мультилокусных отражает степень геномного полиморфизма, выявляемого данными маркерами, и потому представляется оправданным. Следует отметить, что монолокусные маркеры чаще всего используют для анализа отдельных особей, т.к. на молекулярном уровне они, как правило, характеризуют полиморфизм самой последовательности , в то время как мультилокусные применяют для оценки значительного количества особей или их разновидностей, поскольку на молекулярном уровне такие маркеры выявляют полиморфизм числа тандемно повторяющихся последовательностей в повторяющихся районах.
Какие маркеры для каких целей пригодны
Общие характеристики основных классов маркеров приведены в табл. 3. Безусловно, она имеет лишь справочный характер, поскольку при рассмотрении результатов использования методов молекулярно-генетического фингерприн-тинга иногда на получаемых электрофореграммах можно найти аллельные маркерные полосы, а RFLP обычно позволяет выявлять несколько доминантных локусов. Кроме того, как это показано выше, RAPD или AFLP маркеры можно преобразовать в однолокусные кодоминантные маркеры. Но поскольку рассматриваемая нами классификация маркеров основывается преимущественно на генетических критериях, то можно видеть, что такое подразделение отвечает различным потребностям молекулярно-генетического анализа живых организмов. Если исходить из этого, то становится очевидным, что методы генетического фингерпринтинга наиболее пригодны в трех случаях: • при исследовании биоразнообразия, т.е. установлении внутри- и межпопуляционной структуры, генетических дистанций, кластеризации;
-
• при позиционном клонировании, так как молекулярный фингерпринтинг позволяет получать высокие плотности маркеров в особых районах генома, в частности около гена, который должен быть клонирован;
-
• при быстром насыщении генетических карт (у видов которые слабо изучены, для которых пробы пока не разработаны; мар-
- Таблица 3. Сравнение наиболее часто используемых маркеров (по: [25]) Table 3. Comparison most offen used markers (on [25])
|
Q. ф ^ Q. m 2 |
1— о о X с; го о. 1— )S ф X |
о 1— о ф 3 с; о ^ |
1— о о X 1X го X о о ^ |
т S с о о о о 1 о с: |
5 о. о с; о с |
S ЕС го н о X го о. о |
Ф о ф 3 |
1— 2 ф It о о с |
|
Морфологические |
Нет |
Ограничено |
Редко |
Да |
Низкий |
Да |
Низкая |
- |
|
Изоферменты |
Нет |
≤ 40 |
Да |
Да |
Низкий |
Да |
Низкая |
Да |
|
Различные белки (двумерный э/ф) |
Нет |
≤ 100 |
Да |
Да |
Низкий |
Да |
Средняя |
Да |
|
RFLP |
Да |
Практически не ограничено |
Да |
Да |
Высокий |
Нет |
Высокая |
Да или нет |
|
SSR |
Да |
Тысячи |
Да |
Да |
Очень высокий |
Нет |
Высокая3 |
Нет |
|
MAAP |
Да |
Практически не ограничено |
Нет |
Нет |
Очень высокий |
Нет |
Низкая |
Да или нет |
|
SSR |
Да |
Практически не ограничено |
Не оценено |
Нет |
Очень высокий |
Нет |
Низкая |
Нет |
|
AFLP |
Да |
Практически не ограничено |
Нет |
Нет |
Очень высокий |
Нет |
Средняя |
Да или нет |
|
STS |
Да |
Ограничено |
Да |
Да |
Средний |
Нет |
Высокая3 |
Да |
|
SNP |
Да или нет |
Ограничено |
Да |
Да |
Средний |
Нет |
Высокая |
Да или нет |
Примечание: 1– «Да» для методов, выявляющих маркерные локусы индивидуально (или для очень небольшого количества); «Нет» для методов, выявляющих профили множественных локусов;
-
2 – маркеры могут быть выявлены только в некоторых органах или на некоторых стадиях развития;
-
3 – для SSR и STS получение праймеров для большого числа информативных маркерных локусов технически трудно и дорого, но после проведения предварительных анализов они могут быть выполнены быстрее, чем RFLP
керы, получаемые с помощью ПЦР).
С другой стороны, кодоминантные маркеры, выявляемые индивидуально, используют для:
-
• построения консенсусных карт видов и всякий раз, когда должен быть произведен обмен маркерами между лабораториями;
-
• получения лучшего разрешения на картах, если популяции были картированы, включая гетерозиготные особи;
-
• сравнительного картирования;
-
• картирования локусов количественных признаков, если должно быть оценено их доминирование;
-
• построения новых генетических карт;
-
• исследований в популяционной генетике.
Каким бы ни было предполагаемое применение маркеров, выбор метода исследования также будет зависеть и от изучаемого вида растений. Микросателлиты или AFLP, например, являются источниками маркеров, которые очень полезны для видов с низким уровнем полиморфизма. С другой стороны, для видов, имеющих высокий уровень полиморфизма, а также клонированные кДНК, гены или анонимные пробы, в большинстве случаев более пригодны RFLP или STS маркеры. Для голосеменных, у которых гаплоидные мегагаметофиты позволяют проводить построение генетических карт индивидуально, скорее могут быть рекомендованы методы типа MAAP или STS нежели RFLP, требующие количества ДНК, несовместимые с размером анализируемого органа или ткани, и слишком трудные для анализа значительного количества особей. Различные способы практического применения этих подходов были рассмотрены нами ранее [6, 24] и здесь обсуждаться не будут.
Заключение
Таким образом, генетические маркеры могут применяться в различных фазах и на различных этапах селекционных программ, например, при оптимизации сохранения генетических ресурсов растений, при выборе родительских пар, при идентификации желаемых аллелей и при получении генотипов с такими аллелями [6]. С их помощью становится возможным не только идентифицировать и локализовать на группах сцепления локусы хромосом и гены, определяющие изучаемые признаки, но и устанавливать их взаиморасположение [2, 24, 26]. Некоторые из существующих на сегодня маркерных подходов уже стали повседневными. Анализ генетической изменчивости с целью организации отобранных генотипов в группы (популяции) или оценка уже существующих групп, а также проведение возвратного скрещивания на основе применения молекулярно-генетических маркеров довольно широко используются как в частных, так и в государственных селекционных программах. Остальные подходы, в которых используются генетические маркеры, пока еще остаются по большей части прерогативой генетики, но не селекции растений. Однако, несмотря на существующие ограничения технического характера, проблема генетического анализа хозяйственно ценных признаков может быть полем деятельности для дальнейшей методической оптимизации и «наведения мостов» между классической и молекулярной генетикой и селекцией растений. Последующее влияние методов ДНК-фингерпринтинга на генетику и практическую селекцию растений будет зависеть от результатов, которые будут получены, в частности, от выявления возможности или невозможности генотипирования особи по одному генетическому маркеру, и от экономической цены получаемых информативных данных. Тем не менее применение генетических маркеров уже несомненно обогатило и развило целый ряд методов по выявлению и использованию генотипической изменчивости, заключенной в генетическом разнообразии растений [3-6].
-
• Литература
-
1. Чесноков Ю.В. Аспекты структурной организации нуклеиновых кислот. СПб.: АФИ. - 2008. - 64 с.
-
2. Чесноков Ю.В. Молекулярные маркеры и управление генетическими ресурсами растений // Идентифицированный генофонд растений и селекция. СПб.: ВИР. - 2005. - С. 240-250
-
3. Karp A., Kresovich S., Bhat K.V., Ayad W.G., Hodgkin T. Molecular tools in plant genetic resources conservation: a guide to the technologies // IPGRI Technical Bulletin. - 1997. - No. 2. – 25 p.
-
4. Phillips R.L., Vasil I.K. DNA-Based Markers in Plants. 2nd Edition. Kluwer Academic Publishers. Dordrecht, Boston, London.- 2001.
-
5. Spooner D., Van Treuren R., De Vicente M.C. Molecular marker for gene bank management // IPGRI Technical Bulletin. - 2005. - No.10. - 127 p.
-
6. Чесноков Ю.В. Генетические ресурсы растений и современные методы ДНК-типирования. СПб.: ВИР. - 2007а. - 80 с.
-
7. Хлесткина Е.К. Молекулярные методы анализа структурно-функциональной организации генов и геномов высших растений // Вавиловский журнал генетики и селекции. - 2011. - Т.15, № 4. - С. 757768
-
8. Botstein D., White R.L., Skolnick M., Davis R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms // Am. J. Hum. Genet. - 1980. – Vol. 32. – P. 314-331
-
9. Jeffreys A.J., Wilson V., Thein S.L. Hypervariable ‘minisatellite’ regions in human DNA // Nature. - 1985. - Vol. 314. - P. 67–73
-
10. Tautz D., Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes // Nucl. Acids Res. - 1984. Vol. 12. – P. 4127–4138
-
11. Olson M., Hood L., Cantor C., Dotstein D. A common language for physical mapping of the human genome // Science. - 1989. - Vol. 245. – P. 1434–1435
-
12. Paran I., Michelmore R.W. Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce // Theor. Appl. Genet. - 1993. - Vol. 85. – P. 985–993
-
13. Orita M., Iwahana H., Kanasawa H., Hayashi K., Sekiya T. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphisms // Proc. Natl. Acad. Sci. USA. - 1989. – Vol. 86. – P. 2766–2770
-
14. Konieczny A., Ausubel F.M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers // Plant J. - 1993. – Vol. 4. – P. 403–410
-
15. Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A., Tingey S.V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers // Nucl. Аcids Res. - 1990. – Vol. 18. – P. 6531–6535
-
16. Welsh J., Chada K., Dalal S.S., Cheng R., Ralph D., McClelland M. Arbitrarily primed PCR fingerprinting of RNA // Nucl. Acids Res. - 1992. – Vol. 20. – P. 4965–4970
-
17. Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification // Genomics. - 1994. – Vol. 20. – P. 176–183
-
18. Kalendar R., Schulman A.H. IRAP and REMAP for retrotransposon-based genotyping and fingerprinting //Nat. Protoc. - 2006. – Vol. 1. – P. 2478–2484
-
19. Vos P., Hogers R., Bleeker M., Reijans M., van de Lee T., Hornes M., Frijters A., Pot J., Peleman J., Kupier M., Zabeau M. AFLP: a new technique for DNA fingerprinting // Nucl. Acids Res. - 1995. – Vol. 23. – P. 4407–4414
-
20. Waugh R., McLean K., Flavell A.J., Pearce S.R., Kumar A., T. Thomas B.B., Powell W. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP) //Mol. Gen. Genet. - 1997. – Vol. 253. – P. 687–694
-
21. Wang D.G., Fan J.B., Siao C.J., Berno A., Young P., Sapolsky R., Ghandour G., Perkins N., Winchester E., Spencer J., Kruglyak L., Stein L., Hsie L., Topaloglou T., Hubbell E., Robinson E., Mittmann M., Morris M.S., Shen N., Kilburn D., Rioux J., Nusbaum C., Rozen S., Hudson T.J., Lipshutz R., Chee M., Lander E.S. Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome // Science. - 1998. – Vol. 280. – P. 1077–1082
-
22. Jaccoud D., Peng K., Feinstein D., Kilian A. Diversity arrays: a solid-state technology for sequence information independent genotyping // Nucl. Acids Res. - 2001. – Vol. 29. – P. e25
-
23. Хлесткина Е.К., Салина Е.А. SNP-маркеры: методы анализа, способы разработки и сравнительная характеристика на примере мягкой пшеницы // Генетика. - 2006. - T. 42. - C. 725–736
-
24. Чесноков Ю.В. Картирование локусов количественных признаков у растений. ВИР, СПб. - 2009. - 100 с.
-
25. De Vienne D. Marquerurs moleculares en genetique et biotechnologies vegetales. INRA, Paris. - 2002. 26. Чесноков Ю.В. Разновидности сцепления генетических маркеров с целевым геном и локусами хромосом // Агрофизика. - 2018. (в печати)
-
-
• References
-
1. Chesnokov Yu.V. Aspects of Structural Organization of Nucleic Acids. SPb.: AFI. - 2008. - 64 p.
-
2. Chesnokov Yu.V. Molecular markers and management of plant genetic resources // Identified Genefound of Plants and Breeding. SPb.: VIR. - 2005. - P. 240-250
-
3. Karp A., Kresovich S., Bhat K.V., Ayad W.G., Hodgkin T. Molecular tools in plant genetic resources conservation: a guide to the technologies // IPGRI Technical Bulletin. - 1997. - No. 2. – 25 p.
-
4. Phillips R.L., Vasil I.K. DNA-Based Markers in Plants. 2nd Edition. Kluwer Academic Publishers. Dordrecht, Boston, London. - 2001.
-
5. Spooner D., Van Treuren R., De Vicente M.C. Molecular marker for gene bank management // IPGRI Technical Bulletin. - 2005. - No.10. - 127 p.
-
6. Chesnokov Yu.V. Genetic Resources of Plants and Modern methods of DNA-typing. SPb.:VIR. -2007a. - 80 p.
-
7. Khlestkina E.K. Molecular methods of structural and functional organization analisis of genes and genomes of high plants // Vavilov journal of genetics and breeding. - 2011. - V.15, No.4. - P. 757-768 8. Botstein D., White R.L., Skolnick M., Davis R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms // Am. J. Hum. Genet. - 1980. - Vol. 32. – P. 314-331
-
9. Jeffreys A.J., Wilson V., Thein S.L. Hypervariable ‘minisatellite’ regions in human DNA // Nature. -1985. - Vol. 314. - P. 67–73
-
10. Tautz D., Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes // Nucl. Acids Res. - 1984. - Vol. 12. – P. 4127–4138
-
11. Olson M., Hood L., Cantor C., Dotstein D. A common language for physical mapping of the human genome // Science. - 1989. – Vol. 245. – P. 1434–1435
-
12. Paran I., Michelmore R.W. Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce // Theor. Appl. Genet. - 1993. – Vol. 85. – P. 985–993
-
13. Orita M., Iwahana H., Kanasawa H., Hayashi K., Sekiya T. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphisms // Proc. Natl. Acad. Sci. USA. - 1989. – Vol. 86. – P. 2766–2770
-
14. Konieczny A., Ausubel F.M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers // Plant J. - 1993. – Vol. 4. – P. 403–410
-
15. Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A., Tingey S.V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers // Nucl. Аcids Res. - 1990. – Vol. 18. - P. 6531–6535
-
16. Welsh J., Chada K., Dalal S.S., Cheng R., Ralph D., McClelland M. Arbitrarily primed PCR fingerprinting of RNA // Nucl. Acids Res. - 1992. – Vol. 20. – P. 4965–4970
-
17. Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification // Genomics. - 1994. – Vol. 20. – P. 176–183
-
18. Kalendar R., Schulman A.H. IRAP and REMAP for retrotransposon-based genotyping and fingerprinting //Nat. Protoc. - 2006. – Vol. 1. – P. 2478–2484
-
19. Vos P., Hogers R., Bleeker M., Reijans M., van de Lee T., Hornes M., Frijters A., Pot J., Peleman J., Kupier M., Zabeau M. AFLP: a new technique for DNA fingerprinting // Nucl. Acids Res. - 1995. – Vol.23. – P. 4407–4414
-
20. Waugh R., McLean K., Flavell A.J., Pearce S.R., Kumar A., T. Thomas B.B., Powell W. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequencespecific amplification polymorphisms (S-SAP) //Mol. Gen. Genet. - 1997. – Vol. 253. – P. 687–694
-
21. Wang D.G., Fan J.B., Siao C.J., Berno A., Young P., Sapolsky R., Ghandour G., Perkins N., Winchester E., Spencer J., Kruglyak L., Stein L., Hsie L., Topaloglou T., Hubbell E., Robinson E., Mittmann M., Morris M.S., Shen N., Kilburn D., Rioux J., Nusbaum C., Rozen S., Hudson T.J., Lipshutz R., Chee M., Lander E.S. Large-scale identification, mapping, and genotyping of singlenucleotide polymorphisms in the human genome // Science. - 1998. – Vol. 280. – P. 1077–1082
-
22. Jaccoud D., Peng K., Feinstein D., Kilian A. Diversity arrays: a solid-state technology for sequence information independent genotyping // Nucl. Acids Res. - 2001. – Vol. 29. – P. e25
-
23. Khlestkina E.K., Salina E.A. SNP markers: methods of analysis, mode of development and comparative characteristics on the example of soft wheat // Genetika. - 2006. - Vol.42. - P. 725-736
-
24. Chesnokov Yu.V. Mapping of Quantitative Trait Loci in Plants. SPb.: VIR. - 2009. - 100 p.
-
25. De Vienne D. Marquerurs moleculares en genetique et biotechnologies vegetales. INRA, Paris. -2002.
-
26. Chesnokov Yu.V. Varieties of marker genetic linkage with target gene and chromosome loci // Agrophysika. - 2018. (in press)
-
Список литературы Генетические маркеры: сравнительная классификация молекулярных маркеров
- Чесноков Ю.В. Аспекты структурной организации нуклеиновых кислот. СПб.: АФИ. -2008. -64 с.
- Чесноков Ю.В. Молекулярные маркеры и управление генетическими ресурсами растений//Идентифицированный генофонд растений и селекция. СПб.: ВИР. -2005. -С. 240-250
- Karp A., Kresovich S., Bhat K.V., Ayad W.G., Hodgkin T. Molecular tools in plant genetic resources conservation: a guide to the technologies//IPGRI Technical Bulletin. -1997. -No. 2. -25 p.
- Phillips R.L., Vasil I.K. DNA-Based Markers in Plants. 2nd Edition. Kluwer Academic Publishers. Dordrecht, Boston, London.-2001.
- Spooner D., Van Treuren R., De Vicente M.C. Molecular marker for gene bank management//IPGRI Technical Bulletin. -2005. -No.10. -127 p.
- Чесноков Ю.В. Генетические ресурсы растений и современные методы ДНК-типирования. СПб.: ВИР. -2007а. -80 с.
- Хлесткина Е.К. Молекулярные методы анализа структурно-функциональной организации генов и геномов высших растений//Вавиловский журнал генетики и селекции. -2011. -Т.15, № 4. -С. 757-768
- Botstein D., White R.L., Skolnick M., Davis R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms//Am. J. Hum. Genet. -1980. -Vol. 32. -P. 314-331
- Jeffreys A.J., Wilson V., Thein S.L. Hypervariable 'minisatellite' regions in human DNA//Nature. -1985. -Vol. 314. -P. 67-73
- Tautz D., Renz M. Simple sequences are ubiquitous re-petitive components of eukaryotic genomes//Nucl. Acids Res. -1984. Vol. 12. -P. 4127-4138
- Olson M., Hood L., Cantor C., Dotstein D. A common language for physical mapping of the human genome//Science. -1989. -Vol. 245. -P. 1434-1435
- Paran I., Michelmore R.W. Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce//Theor. Appl. Genet. -1993. -Vol. 85. -P. 985-993
- Orita M., Iwahana H., Kanasawa H., Hayashi K., Sekiya T. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphisms//Proc. Natl. Acad. Sci. USA. -1989. -Vol. 86. -P. 2766-2770
- Konieczny A., Ausubel F.M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers//Plant J. -1993. -Vol. 4. -P. 403-410
- Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A., Tingey S.V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers//Nucl. АоiФб Res. -1990. -Vol. 18. -P. 6531-6535
- Welsh J., Chada K., Dalal S.S., Cheng R., Ralph D., McClelland M. Arbitrarily primed PCR fingerprinting of RNA//Nucl. Acids Res. -1992. -Vol. 20. -P. 4965^970
- Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification//Genomics. -1994. -Vol. 20. -P. 176-183
- Kalendar R., Schulman A.H. IRAP and REMAP for retrotransposon-based genotyping and fingerprinting//Nat. Protoc. -2006. -Vol. 1. -P. 2478-2484
- Vos P., Hogers R., Bleeker M., Reijans M., van de Lee T., Hornes M., Frijters A., Pot J., Peleman J., Kupier M., Zabeau M. AFLP: a new technique for DNA fingerprinting//Nucl. Acids Res. -1995. -Vol. 23. -P. 4407-4414
- Waugh R., McLean K., Flavell A.J., Pearce S.R., Kumar A., T. Thomas B.B., Powell W. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP)//Mol. Gen. Genet. -1997. -Vol. 253. -P. 687-694
- Wang D.G., Fan J.B., Siao C.J., Berno A., Young P., Sapolsky R., Ghandour G., Perkins N., Winchester E., Spencer J., Kruglyak L., Stein L., Hsie L., Topaloglou T., Hubbell E., Robinson E., Mittmann M., Morris M.S., Shen N., Kilburn D., Rioux J., Nusbaum C., Rozen S., Hudson T.J., Lipshutz R., Chee M., Lander E.S. Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome//Science. -1998. -Vol. 280. -P. 1077-1082
- Jaccoud D., Peng K., Feinstein D., Kilian A. Diversity arrays: a solid-state technology for sequence information independent genotyping//Nucl. Acids Res. -2001. -Vol. 29. -P. e25
- Хлесткина Е.К., Салина Е.А. SNP-маркеры: методы анализа, способы разработки и сравнительная характеристика на примере мягкой пшеницы//Генетика. -2006. -T. 42. -C. 725-736
- Чесноков Ю.В. Картирование локусов количественных признаков у растений. ВИР, СПб. -2009. -100 с.
- De Vienne D. Marquerurs moleculares en genetique et biotechnologies vegetales. INRA, Paris. -2002.
- Чесноков Ю.В. Разновидности сцепления генетических маркеров с целевым геном и локусами хромосом//Агрофизика. -2018. (в печати)


