Генетические причины аритмического фенотипа некомпактной кардиомиопатии
Автор: Комиссарова С.М., Чакова Н.Н., Ринейская Н.М., Долматович Т.В., Ниязова С.С.
Журнал: Евразийский кардиологический журнал @eurasian-cardiology-journal
Рубрика: Оригинальные статьи
Статья в выпуске: 2, 2021 года.
Бесплатный доступ
Цель работы. Оценить генотип-фенотип ассоциации у белорусских пациентов с некомпактной кардиомиопатией (НКМ) и клинически значимыми желудочковыми аритмиями.Материалы и методы. В исследование включены 170 неродственных пациентов с НКМ, проспективно наблюдаемых в РНПЦ «Кардиология» в течение 36 мес. [6; 42,0], у которых были данные 24 - часового холтеровского мониторирования ЭКГ в течение 12 месяцев после вступления в исследование. Медиана возраста пациентов, вступивших в исследование, составляла 42 [18;69] года, преобладали мужчины (63,2%). Аритмический фенотип НКМ был диагностирован по наличию необъяснимых синкопальных состояний; неустойчивой желудочковой тахикардии (ЖТ), ≥ 500 желудочковых экстрасистол за сутки. Диагноз НКМ устанавливали на основании эхо кардиографических (ЭхоКГ) критериев Jenni и критериев магнитно-резонансной томографии (МРТ) S. Petersen и А. Jaquier. Поиск мутаций в кодирующих последовательностях 174 генов проведен 30 неродственным пациентам с НКМ методом высокопроизводительного секвенирования (NGS).Результаты. У 76 из 170 (44,7%) пациентов клинически значимые аритмии являлись ведущим проявлением заболевания. Неустойчивая ЖТ была зафиксирована у 54 (71,1%) пациентов, устойчивая ЖТ - у 11 (14,5%) пациентов, ЖЭ более 500 в сутки - у 50 (65,8%). За период наблюдения (медиана наблюдения 36 месяцев) имплантируемые кардиовертеры-дефибрилляторы (ИКД) и ресинхронизирующие устройства с функцией дефибриллятора (CRT-D) были установлены 16 (21,1%) пациентам. В результате проведения NGS у 26 (86,7%) пробандов с аритмическим фенотипом НКМ обнаружено 40 изменения нуклеотидной последовательности (5 патогенных мутаций, 30 вариантов с неопределенной значимостью (VUS), 5 новых замен) в 27 генах. Доля мутаций в генах саркомерных протеинов составила 26,9%, а в генах белков ионных каналов оказалась равной 23,1%. На долю нуклеотидных изменений в генах, кодирующих структурные белки, пришлось 11,5%. В 38,5% случаев выявлена не одна, а две или более редких мутаций, причем в 30,8% аминокислотные изменения затрагивали белки разных функциональных классов.Выводы. В группе пациентов с аритмогенным фенотипом НКМ доля лиц с наличием мутаций в генах, ассоциированных с различными кардиомиопатиями, составила 86,7% и была существенно выше, чем сообщалось у пациентов с НМП в целом (59%). Более высокой оказалась и частота встречаемости множественных мутаций (38,5%) в этой когорте. Генетические особенности пациентов наряду с клиническими характеристиками являются маркерами высокого риска развития жизнеугрожающих аритмий и могут дополнительно использоваться для прогнозирования неблагоприятных событий у пациентов с НКМ, а также для ранней диагностики заболевания у их ближайших родственников.
Некомпактная кардиомиопатия, аритмический фенотип, жизнеугрожающие аритмии, высокопроизводительное секвенирование (ngs)
Короткий адрес: https://sciup.org/143176201
IDR: 143176201 | DOI: 10.38109/2225-1685-2021-2-62-69
Текст научной статьи Генетические причины аритмического фенотипа некомпактной кардиомиопатии
Некомпактная кардиомиопатия (НКМ) – клинически и генетически гетерогенная первичная кардиомиопатия, которая характеризуется интенсивно развитыми желудочковыми трабекулами в сочетании с глубокими выстланными эндокардом межтрабекулярными лакунами, не связанными с коронарным кровотоком и предрасполагающими к образованию тромбов [1]. Результаты популяционных исследований, проведенных в различных странах, свидетельствуют о том, что распространенность НКМ в общей популяции составляет 1:5000 человек, а среди взрослых пациентов с сердечной недостаточностью (СН) – от 3% до 4% [2]. Клинические проявления заболевания разнообразны, НКМ встречается как в изолированной форме, так и в сочетании с другими кардиомиопатиями и врожденными пороками сердца. Одной из основных причин НКМ являются мутации в генах, ассоциируемых с развитием различных кардиомиопатий и кодирующих саркомерные, структурные и регуляторные белки, а также белки, ответственные за функционирование ионных каналов [3]. Мутации наследуются по аутосомно-доминантному типу, реже являются рецессивными или X-сцепленными [4].
Высокая частота жизнеугрожающих желудочковых аритмий с повышенным риском внезапной сердечной смерти (ВСС) у пациентов с НКМ, отмечалась во многих исследованиях [5,6]. В мета-анализе, проведенном Kayvanpour E. с соавт. [7], по данным 35 исследований, включивших 7598 пациентов с НКМ, было показано, что примерно у одной пятой пациентов с НКМ были выявлены жизнеугрожающие аритмии. Однако на сегодняшний день в рекомендациях Европейского общества кардиологов нет каких-либо конкретных критериев выделения пациентов высокого риска развития жизнеугрожающих желудочковых аритмических событий среди пациентов с НКМ, и показания для имплантации кардиовертера-дефибриллятора (ИКД) следуют общему руководству стратегии первичной и вторичной профилактики ВСС c использованием критериев для дилатационной кардиомиопатии (ДКМП) [8]. В 2019 г. американскими экспертами разработан консенсусный документ в отношении НКМ, в котором освещается подход к имплантации КД у этой категории пациентов [9]. Указано, что пациентам с НКМ и желудочковыми тахиаритмиями, ассоциированными с синкопальными состояниями, или ВСС с успешными реанимационными мероприятиями при ожидаемой выживаемости более 1 года, рекомендована терапия КД с целью вторичной профилактики ВСС. Имплантация КД также целесообразна у пациентов с НКМ и доказанной неустойчивой ЖТ при наличии снижения систолической функции миокарда с целью первичной профилактики ВСС [10].
Критерии стратификации риска пациентов с НКМ, разработанные к настоящему времени, были сосредоточены, в основном, на клинико-инструментальных характеристиках, включая семейный анамнез, электрокардиографические(ЭКГ) характеристики, параметры ЭхоКГ-исследования, наличии зон фиброза миокарда по данным МРТ с отсроченным контрастированием без учета генетических факторов. Однако анализ данных многочисленных исследований указывает на существование ассоциаций генотипа и фенотипа заболевания и связи неблагоприятных клинических событий с различными генотипами [11].
Аритмические генотипы, связанные с повышенным риском ВСС, хорошо известны при кардиомиопатиях, таких как ДКМП, аритмогенная кардиомиопатия правого желудочка (АКПЖ), а также гипертрофическая кардиомиопатия (ГКМП) [12]. Генетическая составляющая также играет важную роль в патогенезе НКМ. В ряде крупных исследований изучался спектр генетических причин развития злокачественных желудочковых тахиаритмий при этой патологии. В обзоре J.I. van Waning [13] при изучении результатов 172 исследований, включивших 561 пациентов с НКМ, выделены группы генов, ассоциированные с аритмиями, куда вошли гены, кодирующие белки ионных каналов: ABCC9, ANK2, CACNА2D1, CASQ2, HCN4, KCNE3, KCNH2, KCNQ1, RYR2 и SCN5А, а также генетические варианты в саркомерных и несаркомерных генах, которые часто ассоциируются с желудочковыми тахиаритмиями и имеют риск неблагоприятного исхода: TTN, RBM20, DSP, DTNA, JUP, LMNA, PKP2, PLN, PRDM16 и SGCD.
Цель работы. Оценить генотип-фенотип ассоциации у белорусских пациентов с НКМ и клинически значимыми желудочковыми аритмиями.
МАТЕРИАЛЫ И МЕТОДЫ
В исследование включены 170 неродственных пациентов с НКМ проспективно наблюдаемых в РНПЦ «Кардиология» в течение 36 мес. [6; 42,0], у которых были данные 24 – часового холтеровского мониторирования ЭКГ в течение 12 месяцев после вступления в исследование. Все участники дали добровольное письменное информированное согласие на участие в исследовании и использование соответствующих биоматериалов. Аритмический фенотип НКМ был диагностирован по наличию одного из следующих признаков: 1) необъяснимые синкопальные состояния (вероятно, из-за ЖТ); 2) неустойчивая ЖТ, определяемая как ≥ 3 последовательных желудочковых сокращений продолжительностью <30 сек с частотой ≥ 120 уд/ мин; 3) ≥ 500 желудочковых экстрасистол за сутки и был выявлен у 76 (44,7%) пациентов, медиана возраста которых составляла 42 года (18; 69); женщин – 28 (36,8), мужчин – 48 (63,2%). Клинико-инструментальное обследование помимо стандартных методов обследования (осмотр, сбор индивидуального и семейного анамнеза, ЭКГ, суточное мониторирование ЭКГ по Холтеру, ЭхоКГ) включало МРТ с отсроченным контрастированием, при необходимости коронароангиографическое исследование. Диагноз НКМ устанавливали на основании следующих критериев: 1) ЭхоКГ критериев Jenni и соавт. [14], включающих наличие соотношения некомпактного (NC) и компактного (C) слоев NC/C >2,0 в конце систолы; многочисленных чрезмерно выдающихся трабекул и глубоких межтрабекулярных углублений; наличие более 2-х трабекулярных углублений, снабжаемых внутрижелудочковой кровью по данным цветного допплеровского анализа; 2) МРТ-критериев (Petersen) при конечно-диастолическом соотношении NC/C ≥ 2,3 в одном из сегментов ЛЖ по длинным осям МРТ-изображения [15] и доли некомпактного миокарда >20% согласно критериям А. Jaquier [16].
Протокол МРТ- сканирования включал градиент-эхо последовательности с яркой кровью в кино-режиме для морфологической и функциональной оценки, градиент-эхо последовательности инверсия-восстановление с отсроченным контрстированием через 10 мин после введения контрастного вещества (омнискан) в расчете 0.1 ммоль/кг. Оценка локализации и характера отсроченного распределения контрастного препарата проводилась в 17 сегментах миокарда левого желудочка на срезах по короткой оси ЛЖ на базальном уровне (6 сегментов), среднем уровне (на уровне папиллярных мышц, 6 сегментов), на верхушечном уровне (5 сегментов). Всем пациентам, выполнившим МРТ сердца, визуально проводился анализ участков миокарда с отсроченным накоплением контрастного вещества. Для того, чтобы количественно оценить контрастное накопление препарата на каждом срезе по короткой оси ЛЖ в каждом сегменте вручную обводилась и полуавтоматически рассчитывалась площадь участка с 48 отсроченным контрастированием, затем все полученные результаты суммировались. Объем контрастированного миокарда определялся с помощью умножения суммированной площади с накоплением контраста на толщину среза.
Генетическое исследование. Поиск мутаций в кодирующих последовательностях 174 генов, ассоциируемых с сердечно-сосудистой патологией, проведен 30 неродственным пациентам с НКМ методом высокопроизводительного секвенирования (NGS) на генетическом анализаторе MiSeq (Illumina) с использованием набора «TruSight™ Cardio Sequencing Panel». Обработка и аннотирование результатов секвенирования проводилась с помощью программного обеспечения ANNOVAR rev. 527 [16], позволяющего оценить патогенность выявленного генетического варианта.
Статистическая обработка материала проводилась с использованием пакета прикладных программ «Statistica for Windows 12». Данные в таблицах представлены в виде среднего арифметического значения и стандартного отклонения, а также в виде значений медианы и 25- и 75-го процентилей (интерквартильный размах). Статистически значимыми считали различия при p <0,05.
Результаты и обсуждение
Исходная клиническая характеристика пациентов с клинически значимыми желудочковыми аритмиями представлена в таблице 1.
У 76 из 170 (44,7%) пациентов с самого начала манифестации заболевания клинически значимые желудочковые аритмии выходили на первый план и являлись ведущим проявлением заболевания. Медиана возраста пациентов, вступивших в исследование, составляла 42 [18; 69] года, преобладали мужчины (63,2%). Неустойчивая ЖТ была наиболее частым видом аритмии и зафиксирована у 54 (71,1%) пациентов, устойчивая ЖТ – у 11 (14,5%) пациентов, ЖЭ более 500 в сутки – у 50 (65,8%), хроническая форма ФП с эпизодами неустойчивой ЖТ отмечена у 34 (44,7%) пациентов. Симптомы ХСН имелись у всех пациентов, ХСН III ФК NYHA отмечена у 20 (26,3%) пациентов. У 35 (46%) наблюдалась систолическая дисфункция ЛЖ, медиана ФВ ЛЖ составила 41% [17; 80]. По данным ЭхоКГ-исследования у большинства обследуемых регистрировали дилатацию ЛЖ, так как в данной когорте преобладали пациенты с дилатационным фенотипом НКМ (53,9%). По данным МРТ с отсроченным контрастированием у 37 (48,7%) пациентов обнаружены зоны фиброза миокарда, медиана объема фиброза миокарда составила 14,5%.
За период наблюдения (медиана наблюдения 36 месяцев) имплантируемые устройства (ИКД/СРТ-Д) были установлены 16 (21,1%) пациентам, оправданные шоки отмечались у двух из них; трое пациентов умерли, из них ВСС наступила у одного пациента с CRT-D-терапией, которая оказалась неэффективной в купировании устойчивой ЖТ. Троим пациентам успешно проведена ортотопическая трансплантация сердца (ОТС).
В результате проведения секвенирования методом NGS у 26 из 30 пробандов с аритмогенным фенотипом НКМ 86,7%) об-
Таблица 1. Клиническая характеристика пациентов с клинически значимыми желудочковыми аритмиями Table 1. Clinical characteristics of patients with clinically significant ventricular arrhythmias
|
Характеристика |
Всего, n=76 |
|
Возраст вступления в исследование, лет |
42 [18; 69] |
|
Мужской пол, n (%) |
48/28 (63,2/36,8) |
|
ЧСС, уд/мин |
69 [58,5; 80] |
|
Продолжительность QRS, мс |
110 [99, 133] |
|
Длительность корригированного интервала QT, мс |
437 [409; 480] |
|
ЖЭ более 500 в сутки, n (%) |
50 (65,8) |
|
Наличие ФП, форма постоянная, n (%) |
34 (44,7) |
|
Наличие неустойчивой ЖТ в сутки, n (%) |
54 (71,1) |
|
Наличие устойчивой ЖТ в сутки, n (%) |
11 (14,5) |
|
ФК СН NYHA 0, n (%) I, n (%) II, n (%) III, n (%) |
2 (2,6) 17 (22,4) 37 (48,7) 20 (26,3) |
|
ФВ ЛЖ, % |
41 (17; 80) |
|
Число пациентов ФВ ЛЖ <40%, n (%) |
35 (46) |
|
Характеристика |
Всего, n=76 |
|
Число пациентов с ФВ ЛЖ >40%, n (%) |
41 (54) |
|
ЛП, мм |
41 [28; 70] |
|
КДР ЛЖ, мм |
61 [38; 89] |
|
КСР ЛЖ, мм |
49 [26; 75] |
|
КДО ЛЖ, мл |
177 [40; 470] |
|
КСО ЛЖ, мл |
98 [9; 384] |
|
Число пациентов с фиброзом миокарда, n (%) |
37 (48,7) |
|
% объема фиброза миокарда |
14,35 [0,63; 50] |
|
Фенотип НКМ+ДКМП, n (%) |
41 (53,9) |
|
Фенотип НКМ+ГКМП, n (%) |
8 (10,5) |
|
Изолированный НКМ, n (%) |
24 (31,6) |
|
Фенотип НКМ+ВПС, n (%) |
3 (3,9) |
|
Имплантируемые устройства: |
16 (21,1) |
|
ИКД, n (%) |
7 (9,3) |
|
CRT-D/ИКД, n (%) |
9 (11,8) |
|
ОТС, n (%) |
3 (3,9) |
|
Смерть, n (%) |
3 (3,9) |
Примечания: данные представлены в виде n (%), M±SD, Me [LQ; UQ]; ЖЭ – желудочковая экстрасистолия; ФП – фибрилляция предсердий; ЖТ – желудочковая тахикардия; ФК СН – функциональный класс сердечной недостаточности; ФВ ЛЖ- фракция выброса левого желудочка; ЛП – левое предсердие; КДР ЛЖ – конечно-диастолический размер; КСР ЛЖ – конечно-систолический размер; КДО ЛЖ – конечно-диастолический объём левого желудочка; КСО ЛЖ – конечносистолический объём левого желудочка; НКМ – некомпактный миокард; ДКМП – дилатационная кардиомиопатия; ГКМП – гипертрофическая кадиомиопатия; ВПС – врождённый порок сердца; ОТС – ортотопическая трансплантация сердца. Notes: data are presented as n (%), M±SD, Me [LQ; UQ]; VPB – ventricular premature beats; AF – atrial fibrillation; VT – ventricular tachycardia; FC HF – functional class of heart failure; LV EF - left ventricular ejection fraction; LA – left atrium; LV EDD – left ventricular end-diastolic diameter; LV ESD – left ventricular end-systolic diameter; LV EDV – left ventricular end-diastolic volume; LV ESV – left ventricular end-systolic volume; NCCM – non – compaction cardiomapathy; DCM – dilated cardiomyopathy; HCM – hypertrophic cadiomyopathy; CHD – congenital heart disease; OHT – orthotopic heart transplantation.
наружено 40 изменений нуклеотидной последовательности в 27 генах. На рисунке 1 представлено распределение выявленных мутаций в генах, ответственных за синтез саркомерных, структурных белков и субъединиц ионных каналов. Доля мутаций в генах саркомерных протеинов составила 26,9%, а в генах белков ионных каналов оказалась равной 23,1%. На долю нуклеотидных изменений в генах, кодирующих структурные белки, пришлось 11,5%. Две и более мутаций в различных генах обнаружены в 38,5% случаях, причем в 30,8% аминокислотные изменения затрагивали белки разных функциональных классов.
Доля миссенс-мутаций была наибольшей и равнялась 70%, остальные 12 нуклеотидных вариантов были представлены де-лециями без сдвига рамки считывания (5), изменениями в интроне, нарушающими сплайсинг (2), антисмысловыми мутациями (3) и дупликациями со сдвигом рамки считывания (2).
В таблице 2 представлены выявленные в результате секвенирования мутации в генах, кодирующих саркомерные белки: 3 мутации в гене MYH7 и по одной мутации в генах MYBPC3, TTN и NEXN .
Патогенные мутации составили 28,6% (2 из 7); варианты с неопределенной клинической значимостью (variant of uncertain significance, VUS) – 57,1% (4 из 7) и 1 из мутаций в гене MYH7 была новой (табл. 2). Пациенты с заменами VUS в генах MYH7, MYBPC3, NEXN и LDB3 имели желудочковые аритмии, потребовавшие имплантации ИКД/CRT-D. 2 пациента впоследствии умерло, один (код 4) скончался вследствие развития тромбоэмболических осложнений, а второй пробанд (код 6) с наличием желудочковой аритмии и полной блокады левой ножки пучка Гиса – из-за отсутствия эффективности в купировании устой- чивой ЖТ выполненной CRT-D-терапии. Пациенты (код 1, 2) с мутациями в гене MYH7 и пациент (код 5) с нонсенс-мутацией в гене TTN на данный момент характеризовались отсутствием жизнеугрожающих аритмических событий. В исследованиях последних лет мутации, ведущие к возникновению усеченных вариантов титина, считаются самой частой причиной возникновения НКМ [17]. При этом в нескольких независимых исследованиях на основе семейного скрининга показано, что усеченные варианты TTN часто ассоциируются с аритмогенными событиями и имеют риск неблагоприятного исхода [18,19].
В таблице 3 представлены данные генотипирования пациентов, имеющих аминокислотные изменения в белках ионных каналов.
У 28,8% (8 из 28) пациентов с НКМ были выявлены 10 генетических вариантов в 8 генах, кодирующих белки ионных каналов и ассоциированных с ними протеинов: KCNQ1 (1), KCNH2 (2), KCNE1 (1), SCN2B (1), RYR2 (1), CACNA1C (1), HCN4 (1) и DPP6 (2). Патогенные мутации составили 10,0% (1 из 10), остальные 90,0% классифицировались как VUS. У 2 из 8 пациентов (код 10 и 12) было выявлено сочетание двух редких вариантов в генах ионных каналов. У всех пациентов (код 9 и 10) с мутациями в гене KCNH2 аритмические события потребовали имплантацию ИКД/CRT-D. Интересно отметить, что удлинения интервала QT на сериях ЭКГ ни у одного из пациентов с мутациями в генах, кодирующих белки ионных каналов, зарегистрировано не было.
В исследовании Miszalski-Jamka et al. [20] в когорте из 190 пациентов с НКМ при секвенировании всего экзома (WES) обнаружено значительное количество генетических вариантов, контролирующих ионные каналы: KCNH2, SCN5A, RYR2, из которых
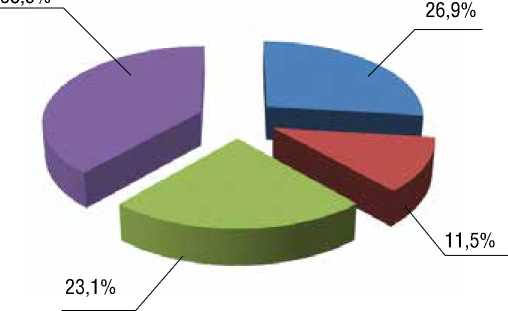
Рисунок 1. Распределение мутаций в генах у пациентов с аритмическим фенотипом НКМП
Figure 1. Mutations distribution in patients’ genes with arrhythmic phenotype of NCCM

Гены саркомерных белков
Гены структурных белков
Гены белков ионных каналов
Две и более мутации
Таблица 2. Выявленные мутации в генах саркомерных белков у пациентов с НКМ
Table 2. Identified mutations in sarcomeric protein genes in patients with NCCM
|
Код пациента |
Ген |
Нуклеотидная замена, Rs |
Аминокислотная замена |
Статус мутации |
События и исходы |
|
1 |
MYH7 |
с.4010G>A, rs368575559 |
p.Arg1337Gln |
VUS |
_ |
|
2 |
MYH7 |
c.2785_2787GAG rs397516172 |
p.Glu931del |
Рathogenic |
_ |
|
3 |
MYH7 |
c. 1169T>G |
p.Leu390Arg |
New |
ИКД |
|
4 |
MYBPC3 |
c.3763G>A, rs727503167 |
p.Ala1255Thr |
VUS |
CRT-D, летальный исход |
|
5 |
TTN |
c.53488G>T, rs759231562 |
p.Gly17830* |
Likely pathogenic |
_ |
|
6 |
NEXN |
c.298+1G>C, rs780476936 |
нарушение сплайсинга |
VUS |
CRT-D, летальный исход |
|
7 |
LDB3 |
c.665G>A, rs139922045 |
p.Ala222Thr |
VUS |
ИКД |
KCNH2 и SCN5A ассоциированы с синдромом удлиненного интервала QT. Несмотря но то, что механизм, с помощью которого мутации в генах ионных каналов могут вызывать НКМ, неясен, данные исследования показывают преобладание редких вариантов в этих генах по сравнению с имеющимися популяционными базами данных. В исследовании Richard et al. [21] в когорте из 95 неродственных пациентов с НКМ сообщалось о высокой доле пациентов с патогенными мутациями в гене HCN4. Среди этих пациентов у 3/5 была выявлена брадикардия (одному пациенту был имплантирован ЭКС). В нашей когорте жизнеугрожающих аритмических событий у пациента (код 12) с вариантами в генах HCN4 и SCN2B зарегистрировано не было. У 2 пробандов (код 13, 14) была выявлена одинаковая делеция в гене DPP6 . В литературе описана мутация в этом гене у пациента с фибрилляцией желудочков (ФЖ), многократно рецидивирующей вопреки многочисленным фармакологическим и абляционным вмешательствам [22]. У 2 наблюдаемых нами пациентов с одинаковой делецией в гене DPP6 в настоящий момент регистрируются злокачественные желудочковые аритмии (индекс эктопии – 21,7%) и проводится медикаментозное лечение соталолом 160 мг/сутки.
Среди 3 пациентов (10,7%) с мутациями в генах, кодирующих структурные белки, имплантация CRT-D и ОТС проведена двум пробандам с нуклеотидными вариантами в генах PKP2 и ACTN2 (см. табл. 4).
В литературе мутации в гене PKP2 представлены, как наиболее частая генетическая причина развития АДПЖ [22]. Имеются также единичные сообщения о связи мутаций в гене PKP2 с развитием НМЛЖ. Так, в одном из исследований описана гомозиготная делеция (hg19, chr12: 32,936,266_33,056,189) размером 120 kb, захватывающей весь ген PKP2, у брата и сестры с диагнозом некомпактного миокарда левого и правого желудочков [23]. Отсутствие гена оказалось летальным: один ребенок умер пренатально, второй – в возрасте 12 дней. Однако у гетерозиготных по данной делеции родителей, состоявших в близкородственном браке, ультразвуковое исследование сердца было без отклонений. У наблюдаемого нами 53-летнего пациента с мутацией c.1892A>C, p.Tyr631Cys в гене РКР2 обнаружена НКМ, подтвержденная МРТ-критериями с дилатацией полости ЛЖ (КДР 74 мм, КСР 65 мм), снижением ФВ ЛЖ до 23% и наличием частой желудочковой экстрасистолии (12800 за сутки) с пробежками неустойчивой ЖТ и полной блокадой левой ножки пучка Гиса. Пациенту было имплантировано CRT-D и назначена стандартная терапия ХСН. В динамике наблюдения клинические проявления СН значительно уменьшились, регистрировались уменьшение размеров ЛЖ (КДД 62 мм, КСД 55 мм) и повышение ФВЛЖ до 30%.
В исследуемой когорте отмечена большая доля пациентов (30,8%) с несколькими нуклеотидными вариантами в двух или более генах (табл. 5), кодирующих белки с различной функциональной направленностью. У 8 пациентов выявлено 20 нуклеотидных вариантов: 2 патогенных мутации, 15 вариантов с неопределенной значимостью и 3 новых замены.
У 5 из 8 пациентов (62,5%) с несколькими мутациями в различных генах наблюдались неблагоприятные события и исходы. Жизнеугрожающие желудочковые аритмические события с необходимостью имплантации ИКД были зарегистрированы у 3 пробандов с различным сочетанием мутаций в саркомерных, структурных генах и генах ионных каналов. Так, у 65-летнего пациента (код 23) с устойчивой ЖТ и синкопальными состояниями, которому был имплантирован ИКД, обнаружены четыре генетический варианта с неустановленной клинической значимостью. У 51-летней пациентки (код 22) с частыми пароксизмами неустойчивой ЖТ и оправданными срабатываниями ИКД при генотипировании обнаружены 3 генетических варианта VUS
Таблица 3. Выявленные мутации в генах ионных каналов и ассоциированных с ними белков у пациентов с НКМ Table 3. Identified mutations in the ion channels genes and associated proteins in patients with NCCM
|
Код пациента |
Ген |
Нуклеотидная замена, Rs |
Аминокислот-ная замена |
Статус мутации |
События и исходы |
|
8 |
KCNQ1 |
c.477+1G>A, rs762814879 |
нарушение сплайсинга |
Pathogenic |
- |
|
9 |
KCNH2 |
c.3107G>A, rs199473022 |
p.Gly1036Asp |
VUS |
ИКД |
|
10 |
KCNH2 |
c.1633C>T, rs143512106 |
p.Arg545Cys |
VUS |
CRT-D |
|
KCNE1 |
c.253G>A, rs1805128 |
p.Asp85Asn |
Risk factor |
||
|
11 |
CACNA1С |
c.2807Т>G |
p.Phe936Cys |
VUS |
_ |
|
12 |
HCN4 |
c.3461G>A, rs145862018 |
p.Arg1154Gln |
VUS |
— |
|
SCN2B |
c.625-626delinsCC, rs1064796044 |
p.Asn209Pro |
VUS |
||
|
13 |
DPP6 |
c.166_167insGCG, rs926747893 |
p.Arg56delins ArgGly |
VUS |
_ |
|
14 |
DPP6 |
c.166_167insGCG, rs926747893 |
p.Arg56delins ArgGly |
VUS |
_ |
|
15 |
RYR2 |
c.12272C>T, rs794728783 |
p.Ala4091Val |
VUS |
_ |
Таблица 4. Выявленные мутации в генах структурных белков у пациентов с НКМ
Table 4. Identified mutations in structural protein genes in patients with NCCM
Наши результаты указывают на то, что в 62,5% случаев наличие мутаций в двух или трех генах привело к жизнеугрожающим желудочковым аритмиям (неустойчивой и устойчивой ЖТ), имплантации КД и неблагоприятным исходам, что позволяет утверждать о кумулятивном эффекте выявленных генетических вариантов, который проявляется злокачественным фенотипом заболевания. С другой стороны, одна из мутаций у этих пациентов идентифицирована в гене саркомерных белков. Этот факт указывает на большую значимость в развитии злокачественных желудочковых аритмий именно мутаций в генах саркомерных белков: половине пациентов даже только с одной мутацией в этих генах установлены имплантируемые устройства (ИКД/ СРТ-Д) (табл. 1), тогда как пациентам с мутациями в других генах имплантация КД требовалась реже (табл. 3-5). Полученные данные являются предварительными и требуют дальнейшего углубленного изучения.
Таблица 5. Множественные мутации у пациентов с НКМ в генах, кодирующих белки с различной функциональной направленностью Table 5. Multiple mutations in patients with NCCM in genes encoding proteins with different functions
|
Код пациента |
Ген |
Нуклеотидная замена, Rs |
Аминокислотная замена |
Статус мутации |
События и исходы |
|
19 |
MYBPC3 |
c.1037G>A, rs397515883 |
p. Arg346His |
VUS |
— |
|
ELN |
c.2032G>A, rs375579231 |
p.Gly678Ser |
VUS |
||
|
MYBPC3 |
c.1037G>A, rs397515883 |
p. Arg346His |
VUS |
_ |
|
|
20 |
DPP6 |
c.166_167insGCG, rs926747893 |
p.Arg56delins ArgGly |
VUS |
|
|
21 |
MYBPC3 |
c.1037G>A, rs397515883 |
p. Arg346His |
VUS |
Летальный исход |
|
TGFB2 |
c.52G>T, rs886045975 |
p.Ala18Ser |
New |
||
|
MYBPC3 |
с.3697C>T, rs397516037 |
p.Gln1233* |
Рathogenic |
ИКД |
|
|
22 |
DTNA |
c.1663A>G, rs779045040 |
p.Asn555Asp |
VUS |
|
|
JPH2 |
c.1225A>G |
p.Asn409Asp |
New |
||
|
NOTCH1 |
c.823G>A, rs371333249 |
p.Gly275Ser |
VUS |
||
|
TTN |
c.12283G>T |
p.Glu4095* |
New |
ИКД |
|
|
23 |
RYR2 |
c.12665_12667delAGA, rs794728838 |
p.4222_4223del |
VUS |
|
|
DSP |
c.2887C>T, rs779931043 |
p.Leu963Phe |
VUS |
||
|
TMPO |
c.629T>A, rs781269460 |
p.Val210Glu |
VUS |
||
|
24 |
TCAP |
c.208C>T, rs775636212 |
p.Arg70Trp |
VUS |
ИКД |
|
KCNQ1 |
c.1450G>A, rs147445322 |
p.Asp484Asn |
VUS |
||
|
JPH2 |
c. 1720A>G, rs773306912 |
p.Thr574Ala |
VUS |
_ |
|
|
25 |
AKAP9 |
c.11714T>C, rs77447750 |
p.Met3905Thr |
Benign (1); Likely benign (1); VUS (3) |
|
|
26 |
PLN |
c.26_29dupCTCG |
p.Thr11fsLeuX10 |
Рathogenic |
ОТС |
ЗАКЛЮЧЕНИЕ
В статье представлено изучение группы пациентов с НКМ с преобладающим аритмическим фенотипом, которую следует рассматривать как группу повышенного риска жизнеугрожающих желудочковых тахиаритмий и ВСС. Результаты генотипирования показали превышение среди этой когорты лиц пациентов с нуклеотидными вариантами (86,7%) по сравнению с общей группой пациентов с НКМ (59%) [20]. У 26 из 30 пациентов выявлено 40 изменений нуклеотидной последовательности в 27 генах, ассоциированных с развитием различных кардиомиопатий. Большинство мутаций зафиксировано в генах, кодирующих саркомерные белки и субъединицы ионных каналов. В 38,5% случаев выявлена не одна, а две или более редких мутаций, причем в 30,8% аминокислотные изменения затрагивали белки разных функциональных классов. Наибольшая частота неблагоприятных событий и исходов наблюдалась у пациентов с несколькими мутациями в генах, кодирующих белки разных функциональных классов (62,5%), при этом одна из мутаций локализовалась в генах саркомерных белков: MYBPC3, TTN, PLN и TCAP . В целом в группе пациентов с имплантируемыми устройствами (ИКД/СРТ-Д) мутации в генах саркомерных белков наблюдались чаще, чем в других генах.
Генетические особенности пациентов наряду с клиническими характеристиками являются маркерами высокого риска развития жизнеугрожающих желудочковых аритмий и могут дополнительно использоваться для прогнозирования неблагоприятных событий у пациентов с НКМ, а также для ранней диагностики заболевания у их ближайших родственников. Определение генетической составляющей открывает новые возможности для персонализации стратификации рисков пациентов с НКМ.
Список литературы Генетические причины аритмического фенотипа некомпактной кардиомиопатии
- Elliott P, Andersson B, Arbustini F, et al. Classification of the cardiomyopathies: a position statement from the European society of cardiology working group on myocardial and pericardial disease. Eur Heart J 2008;29 (2); 270-6. D0l:10.1093/eurheart/ehm342.
- Kovacevic-Preradovic T, Jenni R, Oechslin E et al. Isolated left ventricular noncompaction as a cause for heart failure and heart transplantation: a single center experience. Cardiology. 2009; 112: 158-64. DOI: 10.1159/000147899.
- van Waning J, Caliskan K, Michels M, et al. Cardiac phenotypes, genetics, and risk familian noncompaction cardiomyopathy. J Am Coll Cardiol. 2019; 73:1601-11. DOI: 10.1016/j.jacc.2018.12.085.
- Jefferies J. Barth Syndrome. Am J Med Genet C Semin Med Genet. 2013; 163 С: 198 -205. DOI: 10.1002/ajmg.c.31372.
- Timolo A.Z, Nguyen T, et al. Spectrum of Cardiac Arrhythmias in isolated ventricular n0n-compaction. The J of innovations in cardiac rhythm management.2018:2777-2783. DOI: 10.19102/ icrm. 2017.080701.
- Muser D, Liang J, Witsehey W, et al. Ventricular arrhythmias associated with left ventricular noncompaction: electrophysiological characteristics, mapping, and ablation. Heart Rhythm. 2017; 14 (2): 166 - 75. DOI: 10.1016/j.hrhythm 2016.11.01.14.
- Kayvanpour E, Sedaghat-Hamedani F, Gi W, et al. Clinical and genetic insights into non-compaction: a meta-analysis and systematic review on 7598 individuals. Clin Res Cardiol. 2019. DOI: 10.1007/s00392-019-01465-3.
- Haugaa KH, Dan GA, Iliodromitis K. Management of patients with ventricular arrhythmias and prevention of sudden cardiac death-translating guidelines into practice: results of the European Heart Rhythm Association survey. Europace. 2018;20:f249-f253. DOI: 10.1093/ europace/euy112.
- Towbin JA, McKenna WJ, Abrams DJ. 2019 HRS expert consensus statement on evaluation, risk stratification, and management of arrhythmogenic cardiomyopathy. Heart Rhythm. 2019;16(11):e301-e372. DOI: 10.1016/j.hrthm.2019.05.007.
- Towbin J, Lorts A, Jefferies J. Left ventricular non-compaction cardiomyopathy. The Lancet, 2015; 386: 813-25. DOI: 10.1016/S0140-6736(14)61282-4.
- Towbin J, McKenna W, Abrams D, et al. 2019 HRS expert consensus statement on evaluation, risk stratification, and management of arrhythmogenic cardiomyopathy: Executive summary. Heart Rhythm. 2019;16(11):e373-e407. DOI: 10.1016/j.hrthm.2019.09.019.
- van Waning J, Moesker J, Heijsman D, et al. Systematic Review of Genotype-Phenotype Correlations in Noncompaction Cardiomyopathy. J Am Heart Assoc. 2019;8(23):e012993. DOI: 10.1161/JAHA.119.012993.
- Jenni R, Oechslin E, Schneider J, et al. Echocardiography and pathoanatomical characteristics of isolated left ventricular non-compaction: a step towards classification as a distinct cardiomyopathy. Heart (British Cardiac Society). 2001;86 (6):666-71. DOI: 10.1136/ heart.86.6.666.
- Petersen SE, Selvanayagam JB, Wiesmann F, et al. Left ventricular non-compaction: in- sights from cardiovascular magnetic resonance imaging. Journal of the American College of Cardiology. 2005;46 (1):101-5. DOI: 10.1016/j.jacc.2005.03.045.
- Jacquier A, Thuny F, Jop B, et al. Measurement of trabeculated left ventricular mass using cardiac magnetic resonance imaging in the diagnosis of left ventricular non-compaction. European Heart Journal. 2010;31 (9):1098-104. DOI: 10.1093/eurheartj/ehp595.
- Wang K, et al. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Research - 2010. - Vol. 38. - P. 164. DOI: 10.1093/nar/gkq603.
- Roberts A, Ware J, Herman D, et al, Integrated allelic transcriptional, and phenomic dissection of the cardiac effects of titin truncations in health and disease. Sci Transl Med. 2015; 7:270 - 6. DOI:10,1126 /Sci transmed.3010134.
- Ware J, Cook S. Role of titin in Cardiomyopathy from DNA Variants to patient's stratification. Nat Rev Cardiol 2017; 15 (4): 241 - 52. DOI: 101038/ nrcardio.2017.190.
- Sedaghat-Hamedani F, Haas J, Zhu F, et al. Clinical genetics and outcome of left ventricular non-compaction cardiomyopathy. Eur Heart J. 2017;38(46):3449-3460. DOI: 10.1093/eurheartj/ehx545
- Miszalski-Jamka K, Jefferies JL, Mazur W, et al. Novel Genetic Triggers and Genotype-Phenotype Correlations in Patients with Left Ventricular Noncompaction. Circulation: Cardiovascular Genetics. 2017;10(4):e001763. DOI:10.1161/CIRCGENETICS.117.001763.
- Richard P, Ader F, Roux M, et al. Targeted panel sequencing in adult patients with left ventricular non-compaction reveals a large genetic heterogeneity. Clin Genet. 2019;95:356-367. DOI: 10.1111/cge.13484.
- Trenkwalder T, Deisenhofer J, Hadamitzky M, et al. Novel frome-scift mutations in PKP2 associated with arrhythmogenic right ventricular cardiomyopathy. A case report. BMC Med Genet.2015; 16:117. DOI: 10.1186/s12881-015-0263-1.
- Ramond F, Janin A, Filipo SD, et al. Homozygous PKP2 associated with neonatal left ventricle noncompaction. Clin Genet. 2017;91(1):126-130. DOI: 10.1111/cge.12780.
- Posch M, Perrot A, Geier C, et al. Genetic detection of arginine 14 in causes dilated cardiomyopathy with attenuated electrocardiographic R amplitudes. Heart Rhythm. 2009; 6: 480-6. DOI: 10.1016/j. hrthm.2009.01.016.
- Van Rijsingen J, van der Zwaag P, Groeneweg J, et al. Outome in phospholamban R14del Carries Result of a large multicenter cohort study. Circ Cardiovasc Genet. 2014; 8: 1942-48. DOI 10.1161/ CIRGENETICS.113.000374.
- Haghighi K, Kolokathis F, Pater L, et al. Human phospholamban null results in lethal dilated cardiomyopathy revealing a critical difference between mouse and human. J Clin Invest. 2003;111(6):869-876. DOI:10.1172/ JCI17892.


