Генетическое картирование симбиотических генов у гороха посевного (Pisum sativum L.)
Автор: Жуков В.А., Штарк О.Ю., Неманкин Т.А., Крюков А.А., Борисов А.Ю., Тихонович И.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры, проблемы
Статья в выпуске: 5 т.51, 2016 года.
Бесплатный доступ
Статья обобщает современные данные по методам, результатам и проблемам генетического картирования генов гороха посевного ( Pisum sativum L.), участвующих в развитии и регуляции арбускулярно-микоризного и бобово-ризобиального симбиозов. С помощью мутационного анализа у модельных и сельскохозяйственно ценных бобовых выявлено множество регуляторных симбиотических генов ( Sym -генов), в частности у гороха - более 40 Sym -генов. Некоторые из них клонированы и секвенированы, для различных видов бобовых показано структурное и функциональное сходство ортологичных Sym -генов. Их функции разнообразны и включают контроль рецепции сигнальных молекул микросимбионта, активации сигнального каскада, общего для бобово-ризобиального и арбускулярно-микоризного симбиозов, и последующих транскрипционных изменений в коре корня. Для идентификации последовательностей генов гороха, затронутых мутациями, применяют подход, основанный на сравнительном генетическом картировании и поиске генов-кандидатов в геноме близкородственного бобового растения люцерны слабоусеченной ( Medicago truncatula Gaertn.). На сайте http://www.phytozome.net (D.M. Goodstein с соавт., 2012) представлено текущее состояние работы по секвенированию генома люцерны в формате современного геномного браузера, что облегчает поиск гомологичных генов и анализ последовательностей генов-кандидатов. Высокое сходство геномов гороха и люцерны позволяет создавать геноспецифичные молекулярные маркеры на основе последовательностей генов люцерны, сопоставлять полученные генетические карты гороха с геномом люцерны и (в случае нахождения мутаций со сходным фенотипическим проявлением) клонировать гены гороха. На настоящий момент большинство известных Sym -генов гороха картированы в геноме, что позволило идентифицировать у гороха последовательности 14 симбиотических генов. В частности, авторам настоящего обзора удалось секвенировать гены гороха Sym35 (гомолог NIN лядвенца японского Lotus japonicus (Regel.) K. Larsen) (A.Y. Borisov с соавт., 2003), Sym37 (гомолог NFR1 лядвенца) (V.A. Zhukov с соавт., 2008), Sym33 (гомолог IPD3 люцерны слабоусеченной) (E. Ovchinnikova с соавт., 2011), Cochleata (гомолог NOOT люцерны слабоусеченной) (J.M. Couzigou с соавт., 2012). В последние годы, с развитием современных технологий секвенирования следующего поколения (Next Generation Sequencing - NGS) и массового генотипирования, прослеживается лавинообразное накопление данных по картированию геноспецифичных маркеров у гороха. Насыщение генетической карты маркерами, несомненно, упростит работу по картированию симбиотических генов и выявлению их последовательностей, что поможет расширить понимание того, как функционирует система генов гороха, контролирующих взаимодействие с полезными почвенными микроорганизмами.
Бобовые, бобово-ризобиальный симбиоз, арбуcкулярная микориза, симбиотические гены растений, генетическое картирование, синтения, геноспецифичные молекулярные маркеры
Короткий адрес: https://sciup.org/142213713
IDR: 142213713 | УДК: 635.656:581.557:577.21:575.116 | DOI: 10.15389/agrobiology.2016.5.593rus
Текст обзорной статьи Генетическое картирование симбиотических генов у гороха посевного (Pisum sativum L.)
Статья обобщает современные данные по методам, результатам и проблемам генетического картирования генов гороха посевного (Pisum sativum L.), участвующих в развитии и регуляции арбускулярно-микоризного и бобово-ризобиального симбиозов. С помощью мутационного анализа у модельных и сельскохозяйственно ценных бобовых выявлено множество регуляторных симбиотических генов (Sym-генов), в частности у гороха — более 40 Sym-генов. Некоторые из них клонированы и секвенированы, для различных видов бобовых показано структурное и функциональное сходство ортологичных Sym-генов. Их функции разнообразны и включают контроль рецепции сигнальных молекул микросимбионта, активации сигнального каскада, общего для бо-бово-ризобиального и арбускулярно-микоризного симбиозов, и последующих транскрипционных изменений в коре корня. Для идентификации последовательностей генов гороха, затронутых мутациями, применяют подход, основанный на сравнительном генетическом картировании и поиске генов-кандидатов в геноме близкородственного бобового растения люцерны слабоусеченной (Medicago truncatula Gaertn.). На сайте (D.M. Goodstein с соавт., 2012) представлено текущее состояние работы по секвенированию генома люцерны в формате современного геномного браузера, что облегчает поиск гомологичных генов и анализ последовательностей генов-кандидатов. Высокое сходство геномов гороха и люцерны позволяет создавать геноспецифичные молекулярные маркеры на основе последовательностей генов люцерны, сопоставлять полученные генетические карты гороха с геномом люцерны и (в случае нахождения мутаций со сходным фенотипическим проявлением) клонировать гены гороха. На настоящий момент большинство известных Sym-генов гороха картированы в геноме, что позволило идентифицировать у гороха последовательности 14 симбиотических генов. В частности, авторам настоящего обзора удалось секвенировать гены гороха Sym35 (гомолог NIN лядвенца японского Lotus japonicus (Regel.) K. Larsen) (A.Y. Borisov с соавт., 2003), Sym37 (гомолог NFR1 лядвенца) (V.A. Zhukov с соавт., 2008), Sym33 (гомолог IPD3 люцерны слабоусеченной) (E. Ovchinnikova с соавт., 2011), Cochleata (гомолог NOOT люцерны слабоусеченной) (J.M. Couzigou с соавт., 2012). В последние годы, с развитием современных технологий секвенирования следующего поколения (Next Generation Sequencing — NGS) и массового генотипирования, прослеживается лавинообразное накопление данных по картированию геноспецифичных маркеров у гороха. Насыщение генетической карты маркерами, несомненно, упростит работу по картированию симбиотических генов и выявлению их последовательностей, что поможет расширить понимание того, как функционирует система генов гороха, контролирующих взаимодействие с полезными почвенными микроорганизмами.
Взаимовыгодные (мутуалистические) взаимодействия с микроорганизмами обусловили эволюционно успешную стратегию наземного образа жизни растений (1, 2). Преимущества, приобретаемые растениями в результате вступления в симбиоз, заключаются в облегчении минерального питания и усилении защиты от патогенов и абиотических стрессов (3, 4). Самый распространенный эндосимбиоз, характерный для 80-90 % наземных растений, — арбускулярная микориза (АМ), или симбиоз с грибами отдела Glomeromycota . АМ обеспечивает ассимиляцию питательных (в основном фосфор- и азотсодержащих) веществ из почвы (4). Среди азот-фиксирующих растительно-микробных симбиозов наиболее известен симбиоз между растениями семейства Бобовые ( Fabaceae ) и клубеньковыми бактериями, или ризобиями (альфа-, бета- и гаммапротеобактериями). При формировании бобово-ризобиального симбиоза (БРС) на корнях рас-
Работа финансово поддержана Российским научным фондом (грант ¹ 14-24-00135).
тений развиваются специализированные структуры — клубеньки, заселенные бактериями, фиксирующими атмосферный азот (3). АМ и БРС имеют немало общих черт, указывающих на их эволюционное родство (5, 6). Для обоих типов симбиозов характерна высокая степень генетической и метаболической интеграции партнеров. Развитие АМ и БРС включает ряд четко взаимосвязанных молекулярных и клеточных процессов у партнеров с постоянным обменом сигналами и метаболитами (6, 7).
Для изучения механизмов, лежащих в основе формирования и функционирования мутуалистических растительно-микробных симбиозов, широко применяют экспериментальный мутагенез. В частности, получены обширные коллекции мутантов бобовых, позволившие идентифицировать несколько десятков регуляторных симбиотических генов (8-11), или так называемых Sym -генов (от англ. symbiosis — симбиоз) (12). Многие из них клонированы и секвенированы, и для различных видов бобовых показано структурное и функциональное сходство ортологичных Sym -генов. Функции этих генов разнообразны и включают контроль рецепции сигнальных молекул микросимбионта, активацию общего (для БРС и АМ) сигнального каскада (ОСК) и последующих транскрипционных изменений в коре корня (6, 7, 13).
Горох посевной ( Pisum sativum L.) относится к важнейшим бобовым сельскохозяйственным культурам в России и в мире (14). У гороха получено более 100 независимых мутантов, несущих мутации в 44 Sym -генах, приводящие к нарушению развития БРС (реже АМ) (1, 8, 9, 15-21). Одним из перспективных способов установления последовательности таких генов считается их точная локализация на генетической карте и последующее клонирование на основании поиска генов-кандидатов в геноме модельных бобовых растений люцерны слабоусеченной Medicago truncatula Gaertn. и лядвенца японского Lotus japonicus (Regel.) K. Larsen (8, 22-24).
Цель настоящей статьи — представить обзор современных данных по генетическому картированию Sym -генов гороха, включая результаты, полученные во Всероссийском НИИ сельскохозяйственной микробиологии (ВНИИСХМ).
Методология генетического картирования. Генетическое картирование базируется на методах классической генетики — определении групп сцепления и частот рекомбинации. Единицей измерения расстояний между генами служат проценты рекомбинации, или сантиморганы (сМ) (25). Построение генетических карт проводится на основании статистической обработки массива данных, соответствующих расщеплению по аллельным состояниям маркеров в определенном поколении после проведения скрещивания. Для этого разработано не менее десятка программных средств, например, MAPMAKER 3.0, MapL98, JoinMap 4.1 и многие другие (26-30).
Для генетического картирования генов гороха используются морфологические и молекулярные маркеры (белковые и ДНК-маркеры). Создание масштабных генетических карт с высоким разрешением стало возможным лишь с вовлечением в генетический анализ ДНК-маркеров (31). За последние 25 лет с применением молекулярных маркеров были созданы различные варианты генетических карт генома гороха (22, 32-43).
Для работы по картированию генов гороха, идентифицированных с помощью экспериментального мутагенеза, наиболее подходящими представляются геноспецифичные маркеры, основанные на амплификации фрагментов экспрессирующихся последовательностей (так называемые EST-маркеры — от англ. expressed sequence tag). Известная последовательность 594
EST используется для дизайна праймеров. Если детекция аллельного состояния продукта амплификации осуществляется посредством воздействия эндонуклеазой рестрикции, сайт узнавания которой совпадает с сайтом однонуклеотидного полиморфизма, то маркеры относят к типу CAPS (от англ. cleaved amplified polymorphic sequences). Аллельный полиморфизм таких маркеров проявляется в виде разного числа и размера рестрикционных фрагментов при электрофорезе в агарозном геле (44).
Применение геноспецифичных маркеров для картирования у бобовых растений позволяет эксплуатировать феномен синтении геномов для переноса данных, получаемых для модельных бобовых (лядвенец, люцерна), на экономически значимые сельскохозяйственные культуры, к которым относится и горох посевной (24, 45). Под термином «синтения» понимают одинаковый порядок следования гомологичных генов на хромосомах различных видов. Если этот порядок наблюдается лишь в пределах кластеров из нескольких близко расположенных генов, то имеет место микросин-тения, если же такая закономерность характерна для целых хромосом или их протяженных участков, то употребляют термин «макросинтения».
Исследования, направленные непосредственно на создание новых геноспецифичных маркеров и построение генетических карт с их использованием у бобовых, подтвердили наличие как макро-, так и микросинте-нии их геномов (22, 40). Обнаружение высокой синтении геномов у гороха и люцерны слабоусеченной стало новым стимулом для развития генетики гороха, в особенности после успешного секвенирования генома люцерны. Высокое сходство геномов позволяет использовать знания о генах люцерны для изучения генов гороха, создания геноспецифичных маркеров и клонирования генов в случае нахождения гомологичных мутаций (то есть мутаций со сходным фенотипическим проявлением) (24). На сайте Plant Comparative Genomics portal of the Department of Energy’s Joint Genome Institute (США, (46) представлено текущее состояние работы по секвенированию генома люцерны в формате современного геномного браузера, что облегчает поиск гомологичных генов и анализ генов-кандидатов.
Генетическое картирование как инструмент для идентификации последовательностей симбиотических генов гороха. За последние 10 лет во ВНИИСХМ был создан набор из более чем 100 геноспецифичных молекулярных маркеров для картирования Sym -генов, локализованных во всех группах сцепления у гороха. Для создания молекулярных маркеров использовали EST, позиция которых в геноме известна из данных литературы (так называемые якорные маркеры — от англ. anchor markers).
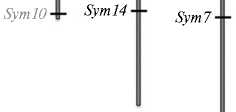
На основании полученных из базы данных National Center for Biotechnology Information (NCBI, нуклеотидных последовательностей выбранных EST были созданы олигонуклеотид-ные праймеры для проведения ПЦР и амплификации целевых фрагментов ДНК у различных генетических линий гороха. С использованием созданного набора маркеров была впервые определена или уточнена локализация десяти симбиотических генов (20, 47, 48). На рисунке представлена обобщенная схема, суммирующая все данные по картированию симбиотических генов гороха, включая полученные во ВНИИСХМ (9, 20, 47-49).
Результаты генетического картирования мутантных генов с использованием геноспецифичных маркеров позволили идентифицировать последовательности 13 Sym -генов гороха. Многие из них были определены одновременно с последовательностями ортологичных генов других бобовых,
II III
SymS5
NodS.
Sym37, Sym2’
Sym5 Syml9/Sym41 SymSS
Sym22

Sym28- ■
Syml6- -
Sym27" "
Coch - -
SymSS" ~
Sym36- ■
(Symi; - -
Sym. - -
Sym 13 - -Symll" "
Sym29 - -
Sym 15- -
Sym40 •*•
Позиции симбиотических ( Sym ) генов гороха ( P. sativum L.) на генетической карте: I-VII — номер группы сцепления гороха . В скобках указана позиция гена Sym17 , требующая дополнительного подтверждения (50). Черным цветом обозначены гены, положение которых определено или подтверждено во Всероссийском НИИ сельскохозяйственной микробиологии (г. Санкт-Петербург), серым — гены, локализованные другими исследователями. Взаимное расположение следующих генов не определено: Coch — Sym38 , Sym16 — Sym27 , Sym14 — Sym22 , Sym11 — Sym13 , Sym15 — Sym29 .
включая модельные виды (20, 48, 51-57). Обобщенная схема определения последовательности гена гороха включала четыре этапы: локализацию гена на генетической карте гороха; поиск известных генов в синтенном регионе генома модельного бобового ( Medicago truncatula или Lotus japonicus ); отбор генов-кандидатов на роль гомологов искомого гена гороха на основании сравнения фенотипа мутантов гороха и модельного бобового; амплификацию и секвенирование последовательности гороха с использованием праймеров, созданных на основе последовательности гена-кандидата модельного бобового (8).
Последовательности семи генов гороха идентифицированы во ВНИИСХМ, в том числе в сотрудничестве с ведущими лабораториями мира. В частности, ген Sym33 был секвенирован на основании гомологии с геном люцерны слабоусеченной IPD3 (56), ген Sym35 — на основе гомологии с геном лядвенца японского NIN (52), ген Sym37 — гомологии с геном NFR1 (54). Эти гены кодируют соответственно белок, взаимодействующий с кальций/кальмодулин-зависимой киназой (центральный компонент ОСК); специфичный для БРС транскрипционный фактор, зависимый от ОСК; рецептор бактериального сигнала (Nod-фактора) (6). Было установлено, что ген гороха Sym40 — гомолог ранее известного гена, кодирующего транскрипционный фактор из группы факторов этиленового ответа EFD у люцерны (48), а мутация, соответствующая гену Sym41 , представляет собой слабое аллельное состояние другого симбиотического гена гороха — Sym19 , кодирующего рецепторную киназу (компонент ОСК) (20). Также недавно показано, что ортологичные гены Cochleata и NOOT , управляющие активностью клубеньковой меристемы, — это гомологи генов BOP1 и BOP2 Arabidopsis thaliana (L.) Heyhn., вовлеченных в морфогенез листьев и цветков (57). Все шесть перечисленных генов гороха были секвенированы на основании результатов генетического картирования. Исключение составляет ген Sym34 с неизвестной локализацией, который секвенирован на ос-596
новании гомологии с геном люцерны слабоусеченной NSP1 (ген общего для БРС и АМ транскрипционного фактора, зависимого от ОСК), предположенной лишь на основании фенотипа мутанта гороха (21).
Таким образом, у гороха на настоящий момент известны нуклеотидные последовательности 14 Sym -генов, что составляет лишь треть от идентифицированных с помощью мутационного анализа, то есть многие гены еще предстоит картировать и секвенировать.
Технологии «следующего поколения» и перспективы. В последние годы с развитием технологий секвенирования следующего поколения и массового генотипирования происходит лавинообразное накопление данных по картированию геноспецифичных маркеров у гороха. Так называемые технологии секвенирования «следующего поколения» (next generation sequencing — NGS) позволяют с высокой скоростью прочитывать огромное число фрагментов, что делает возможным анализ полиморфизма тысяч последовательностей, рассматриваемых в качестве потенциальных маркеров. Массовое генотипирование, осуществляемое, например, с помощью микрочипов GoldenGate («Illumina», США), делает возможным единовременный анализ до нескольких тысяч создаваемых маркеров. С использованием этих подходов в последние два года на генетическую карту гороха были нанесены несколько тысяч геноспецифичных маркеров (43).
Безусловно, работа по генетическому картированию симбиотических генов гороха будет значительно облегчена после секвенирования генома P. sativum . Пока, однако, горох, как и многие другие бобовые культуры, ожидает своего шанса пополнить список видов с секвенированным геномом (58), и секвенирование ограничивается его транскриптомом (59-62).
Итак, использование геноспецифичных маркеров для картирования у бобовых растений позволяет использовать феномен синтении геномов для переноса данных, получаемых для модельных бобовых (лядвенец, люцерна), на экономически значимые сельскохозяйственные культуры, к которым относится и горох посевной. Создание новых, «межвидовых» маркеров (то есть полученных на основе пар ортологичных генов у различных бобовых растений) и насыщение генетической карты гороха, очевидно, способны значительно помочь при осуществлении сборки полного генома гороха на основании результатов секвенирования следующего поколения (NGS). В свою очередь, картирование симбиотических генов и выявление их последовательностей поможет расширить понимание того, как функционирует система генов гороха, контролирующих взаимодействие с полезными почвенными микроорганизмами.
Список литературы Генетическое картирование симбиотических генов у гороха посевного (Pisum sativum L.)
- Gianinazzi-Pearson V. Plant cell responses to arbuscular mycorrhizal fungi: getting to the roots of the symbiosis. The Plant Cell, 1996, 8: 1871-1883 ( ) DOI: 10.1105/tpc.8.10.1871
- Brundrett M.C. Coevolution of roots and mycorrhizas of land plants. New Phytologist, 2002, 154: 275-304 ( ) DOI: 10.1046/j.1469-8137.2002.00397.x
- Dilworth M.J., James E.K., Sprent J.I., Newton W.I. Nitrogen-fixing leguminous symbioses. Springer Science+Business Media B.V., 2008 ( ) DOI: 10.1007/978-1-4020-3548-7
- Smith S.E., Read D.J. Mycorrhizal symbiosis. Academic Press, NY, USA, 2008.
- Parniske M. Arbuscular mycorrhiza: the mother of plant root endosymbioses. Nat. Rev. Microbiol., 2008, 6: 763-775 ( ) DOI: 10.1038/nrmicro1987
- Oldroyd G.E. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nat. Rev. Microbiol., 2013, 11(4): 252-263 ( ) DOI: 10.1038/nrmicro2990
- Gobbato E. Recent developments in arbuscular mycorrhizal signaling. Curr. Opin. Plant Biol., 2015, 26: 1-7 ( ) DOI: 10.1016/j.pbi.2015.05.006
- Борисов А.Ю., Штарк О.Ю., Жуков В.А. и др. Взаимодействие бобовых с полезными почвенными микроорганизмами: от генов растений к сортам. Сельскохозяйственная биология, 2011, 3: 41-47.
- Borisov A.Y., Danilova T.N., Koroleva T.A., Naumkina T.S., Pavlova Z.B., Pinaev A.G., Shtark O.Y., Tsyganov V.E., Voroshilova V.A., Zhernakov A.I., Zhukov V.A., Tikhonovich I.A. Pea (Pisum sativum L.) regulatory genes controlling development of nitrogen-fixing nodule and arbuscular mycorrhiza: fundamentals and application. Biologia, 2004, 59(Suppl 13): 137-144.
- Sandal N., Petersen T.R., Murray J. et al. Genetics of symbiosis in Lotus japonicus: recombinant inbred lines, comparative genetic maps, and map position of 35 symbiotic loci. Mol.-Plant Microbe Interact., 2006, 19: 80-91 ( ) DOI: 10.1094/MPMI-19-0080
- Pislariu C.I., Murray J.D., Wen J., Cosson V., Muni R.R.D., Wang M., Benedito V.A., Andriankaja A., Cheng X., Torres J.I., Mondy S., Zhang S., Taylor M.E., Tadege M., Ratet P., Mysore K.S., Chen R., Udvardi M.K. A Medicago truncatula tobacco retrotransposon insertion mutant collection with defects in nodule development and symbiotic nitrogen fixation. Plant Physiol., 2012, 159(4): 1686-1699 ( ) DOI: 10.1104/pp.112.197061
- Lie T.A. Temperature-dependent root-nodule formation in pea cv. Iran. Plant and Soil, 1971, 34: 751-752.
- Oldroyd G.E., Downie J.A. Coordinating nodule morphogenesis with rhizobial infection in legumes. Annu. Rev. Plant Biol., 2008, 59: 519-546 (doi: 10.1146/annurev.arplant.59.032607.092839).
- Graham P.H., Vance C.P. Legumes: importance and constraints to greater use. Plant Physiol., 2003, 131: 872-877 ( ) DOI: 10.1104/pp.017004
- Guinel F.C., Geil R.D. A model for the development of the rhizobial and arbuscular mycorrhizal symbioses in legumes and its use to understand the roles of ethylene in the establishment of these two symbioses. Can. J. Bot., 2002, 80: 695-720 ( ) DOI: 10.1139/b02-066
- Tsyganov V.E., Voroshilova V.A., Priefer U.B., Borisov A.Y., Tikhonovich I.A. Genetic dissection of the initiation of the infection process and nodule tissue development in the Rhizobium-pea (Pisum sativum L.) symbiosis. Ann. Bot., 2002, 89: 357-366 ( ) DOI: 10.1093/aob/mcf051
- Jacobi L.M., Petrova O.S., Tsyganov V.E., Borisov A.Y., Tikhonovich I.A. Effect of mutations in the pea genes Sym33 and Sym40. I. Arbuscular mycorrhiza formation and function. Mycorrhiza, 2003, 13: 3-7 ( ) DOI: 10.1007/s00572-002-0188-3
- Jacobi L.M., Zubkova L.A., Barmicheva E.M., Tsyganov V.E., Borisov A.Y., Tikhonovich I.A. Effect of mutations in the pea genes Sym33 and Sym40. II. Dynamics of arbuscule development and turnover. Mycorrhiza, 2003, 13: 9-16 ( ) DOI: 10.1007/s00572-002-0189-2
- Voroshilova V.A., Demchenko K.N., Brewin N.J., Borisov A.Y., Tikhonovich I.A. Initiation of a legume nodule with an indeterminate meristem involves proliferating host cells that harbour infection threads. New Phytologist, 2009, 181: 913-923 ( ) DOI: 10.1111/j.1469-8137.2008.02723.x
- Ovchinnikova E. Genetic analysis of symbiosome formation. PhD thesis. Wageningen University, Wageningen, NL, 2012.
- Shtark O.Y., Sulima A.S., Zhernakov A.I., Kliukova M.S., Fedorina J.V., Pinaev A.G., Kryukov A.A., Akhtemova G.A., Tikhonovich I.A., Zhukov V.A. Arbuscular mycorrhiza development in pea (Pisum sativum L.) mutants impaired in five early nodulation genes including putative orthologs of NSP1 and NSP2. Symbiosis, 2016, 68(1): 129-144 ( ) DOI: 10.1007/s13199-016-0382-2
- Aubert G., Morin J., Jacquin F., Loridon K., Quillet M.C., Petit A., Rameau C., Lejeune-Hénaut I., Huguet T., Burstin J. Functional mapping in pea, as an aid to the candidate gene selection and for investigating synteny with the model legume Medicago truncatula. Theor. Appl. Genet., 2006, 112(6): 1024-1041 ( ) DOI: 10.1007/s00122-005-0205-y
- Жуков В.А., Штарк О.Ю., Борисов А.Ю., Тихонович И.А. Молекулярно-генетические механизмы контроля растением ранних стадий развития взаимовыгодных (мутуалистических) симбиозов бобовых. Генетика, 2009, 45(11): 1449-1460.
- Young N.D., Udvardi M. Translating Medicago truncatula genomics to crop legumes. Curr. Opin. Plant Biol., 2009, 12(2): 193-201 ( ) DOI: 10.1016/j.pbi.2008.11.005
- Инге-Вечтомов С.Г. Генетика с основами селекции. СПб, 2010.
- Lander E.S., Green P., Abrahamson J., Barlow A., Daly M.J., Lincoln S.E., Newburg L. MAPMAKER, an interactive computer package for constructing primary genetic linkage maps of experimental and natural populations. Genomics, 1987, 1: 174-181 ( ) DOI: 10.1016/0888-7543(87)90010-3
- Ukai Y. MAPL: a package of computer programs for construction of DNA polymorphism linkage maps and analysis of QTL. Breeding Sci., 1995, 45: 139-142 ( ) DOI: 10.1270/jsbbs1951.45.139
- Van Ooijen J.W. Multipoint maximum likelihood mapping in a full-sib family of an outbreeding species. Genet. Res., 2011, 93(5): 343-349 ( ) DOI: 10.1017/S0016672311000279
- Cheema J., Dicks J. Computational approaches and software tools for genetic linkage map estimation in plants. Briefing in Bioinformatics, 2009, 10(6): 595-608 ( ) DOI: 10.1093/bib/bbp045
- Жернаков А.И. F2Breed. Программа для оценки рекомбинации между генетическими локусами и генетических маркеров в многомерном пространстве при анализе косегрегации признаков в F2-популяции. Свид. о гос. регистр. прогр. для ЭВМ № 2012619482. Зарегистрир. в Реестре программ для ЭВМ 19 октября 2012 г.
- Ежова Т.А., Лебедева О.В., Огаркова О.А., Пенин А.А., Солдатова О.П., Шестаков С.В. Arabidopsis thaliana -модельный объект генетики растений. М., 2003.
- Weeden N.F., Swiecicki W.K., Ambrose M., Timmerman G.M. Linkage groups of pea. Pisum Genetics, 1993, 25: 4.
- Weeden N.F., Swiecicki W.K., Timmerman-Vaughan G.M., Ellis T.H.N., Ambrose M. The current pea linkage map. Pisum Genetics, 1996, 28: 1-4.
- Hall K.J., Parker J.S., Ellis T.H.N., Turner L., Knox M.R., Hofer J.M.I., Lu J., Ferrandiz C., Hunter P.J., Taylor J.D., Baird K. The relationship between genetic and cytogenetic maps of pea. II. Physical maps of linkage mapping populations. Genome, 1997, 40: 755-769 ( ) DOI: 10.1139/g97-798
- Flavell A.J., Knox M.R., Pearce S.R., Ellis T.H. Retrotransposon-based insertion polymorphisms (RBIP) for high throughput marker analysis. Plant Journal, 1998, 16(5): 643-650 ( ) DOI: 10.1046/j.1365-313x.1998.00334.x
- Laucou V., Haurogne K., Ellis N., Rameau C. RAPD-based genetic linkage map of Pisum sativum. Theor. Appl. Genet., 1998, 97: 905-915 ( ) DOI: 10.1007/s001220050971
- Weeden N.F., Ellis T.H.N., Timmerman-Vaughan G.M., Swiecicki W.K., Rozov S.M., Berdnikov V.A. A consensus linkage map for Pisum sativum. Pisum Genetics, 1998, 30: 1-4.
- Irzykowska L., Wolko B., Swiecicki W.K. The genetic linkage map of pea (Pisum sativum L.) based on molecular, biochemical and morphological markers. Pisum Genetics, 2001, 33: 13-18.
- Weeden N.F., Moffet M. Identification of genes affecting root mass and root/shoot ratio in a JI1794 x «Slow» RIL population. Pisum Genetics, 2002, 34: 28-31.
- Bordat A., Savois V., Nicolas M., Salse J., Chauveau A., Bourgeois M., Potier J., Houtin H., Rond C., Murat F., Marget P., Aubert G., Burstin J. Translational genomics in legumes allowed placing in silico 5460 unigenes on the pea functional map and identified candidate genes in Pisum sativum L. G3: Genes, Genomes, Genetics, 2011, 1(2): 93-103 ( ) DOI: 10.1534/g3.111.000349
- Leonforte A., Sudheesh S., Cogan N.O., Salisbury P.A., Nicolas M.E., Materne M., Forster J.W., Kaur S. SNP marker discovery, linkage map construction and identification of QTLs for enhanced salinity tolerance in field pea (Pisum sativum L.). BMC Plant Biol., 2013, 13: 161 ( ) DOI: 10.1186/1471-2229-13-161
- Duarte J., Riviere N., Baranger A., Aubert G., Burstin J., Cornet L., Lavaud C., Lejeune-Henaut I., Martinant J.P., Pichon J.P., Pilet-Nayel M.L., Boutet G. Transcriptome sequencing for high throughput SNP development and genetic mapping in Pea. Genomics, 2014, 15: 126 ( ) DOI: 10.1186/1471-2164-15-126
- Sindhu A., Ramsay L., Sanderson L.A., Stonehouse R., Li R., Condie J., Shunmugam A.S.K., Liu Y., Jha A.B., Diapari M., Burstin J., Aubert G., Tar'an B., Bett K.E., Warkentin T.D., Sharpe A.G. Gene-based SNP discovery and genetic mapping in pea. Theor. Appl. Genet., 2014, 127(10): 2225-2241 ( ) DOI: 10.1007/s00122-014-2375-y
- Konieczny A., Ausubel F.M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers. The Plant Journal, 1993, 4: 403-410 ( ) DOI: 10.1046/j.1365-313X.1993.04020403.x
- Zhukov V.A., Kuznetsova E.V., Ovchinnikova E.S., Rychagova T.S., Titov V.S., Pinaev A.G., Borisov A.Y., Moffet M., Domoney C., Ellis T.H.N., Ratet P., Weeden N.F., Tikhonovich I.A Gene-based markers of pea linkage group V for mapping genes related to symbioses. Pisum Genetics, 2007, 39: 19-25.
- Goodstein D.M., Shu S., Howson R., Neupane R., Hayes R.D., Fazo J., Mitros T., Dirks W., Hellsten U., Putnam N., Rokhsar D.S. Phytozome: a comparative platform for green plant genomics. Nucl. Acids Res., 2012, 40(Database issue): D1178-D1186 ( ) DOI: 10.1093/nar/gkr944
- Жуков В.А., Неманкин Т.А., Овчинникова Е.С., Кузнецова Е.В., Жернаков А.И., Титов В.С., Гришина О.А., Сулима А.С., Борисов Я.Г., Борисов А.Ю., Тихонович И.А. Создание серии ген-специфичных молекулярных маркеров для сравнительного картирования геномов гороха посевного (Pisum sativum L.) и диплоидной люцерны (Medicago truncatula Gaertn.). В сб.: Фактори експериментальної еволюцii органiзмiв. Киев, 2010. Т. 9: 30-34.
- Неманкин Т.А. Анализ генетической системы гороха (Pisum sativum L.), контролирующей развитие арбускулярной микоризы и азотфиксирующего симбиоза. Автореф. канд. дис. СПб, 2011.
- Титов В.С. Генетическое картирование симбиотических генов гороха посевного (Pisum sativum L.). Квалификационная работа магистра. СПб, 2012.
- Kneen B.E., Weeden N.F., LaRue T.A. Non-nodulating mutants of Pisum sativum (L.) cv. Sparkle. J. Hered., 1994, 85(2): 129-133.
- Krusell L., Madsen L.H., Sato S., Aubert G., Genua A., Szczyglowski K., Duc G., Kaneko T., Tabata S., de Bruijn F., Pajuelo E., Sandal N., Stougaard J. Shoot control of root development and nodulation is mediated by a receptor-like kinase. Nature, 2002, 420(6914): 422-426 ( ) DOI: 10.1038/nature01207
- Borisov A.Y., Madsen L.H., Tsyganov V.E., Umehara Y., Voroshilova V.A., Batagov A.O., Sandal N., Mortensen A., Schauser L., Ellis N., Tikhonovich I.A., Stougaard J. The Sym35 gene required for root nodule development in pea is an ortholog of Nin from Lotus japonicus. Plant Physiol., 2003, 131: 1009-1017 ( ) DOI: 10.1104/pp.102.016071
- Lévy J., Bres C., Geurts R., Chalhoub B., Kulikova O., Duc G., Journet E.P., Ané J.M., Lauber E., Bisseling T., Dénarié J., Rosenberg C., Debellé F. A putative Ca2+ and calmodulin-dependent protein kinase required for bacterial and fungal symbioses. Science, 2004, 303(5662): 1361-1364 ( ) DOI: 10.1126/science.1093038
- Zhukov V., Radutoiu S., Madsen L.H., Rychagova T., Ovchinnikova E., Borisov A., Tikhonovich I., Stougaard J. The pea Sym37 receptor kinase gene controls infection-thread initiation and nodule development. Mol. Plant-Microbe Interact., 2008, 21(12): 1600-1608 ( ) DOI: 10.1094/MPMI-21-12-1600
- Krusell L., Sato N., Fukuhara I., Koch B.E., Grossmann C., Okamoto S., Oka-Kira E., Otsubo Y., Aubert G., Nakagawa T., Sato S., Tabata S., Duc G., Parniske M., Wang T.L., Kawaguchi M., Stougaard J. The Clavata2 genes of pea and Lotus japonicus affect autoregulation of nodulation. The Plant Journal, 2011, 65(6): 861-871 ( ) DOI: 10.1111/j.1365-313X.2010.04474.x
- Ovchinnikova E., Journet E.P., Chabaud M., Cosson V., Ratet P., Duc G., Fedorova E., Liu W., Op den Camp R., Zhukov V., Tikhonovich I., Borisov A., Bisseling T., Limpens E. IPD3 controls the formation of nitrogen-fixing symbiosomes in pea and Medicago spp. Mol. Plant-Microbe Interact., 2011, 24(11): 1333-1344 ( ) DOI: 10.1094/MPMI-01-11-0013
- Couzigou J.M., Zhukov V., Mondy S., Abu el Heba G., Cosson V., Ellis T.H., Ambrose M., Wen J., Tadege M., Tikhonovich I., Mysore K.S., Putterill J., Hofer J., Borisov A.Y., Ratet P. NODULE ROOT and COCHLEATA maintain nodule development and are legume orthologs of Arabidopsis BLADE-ON-PETIOLE genes. The Plant Cell, 2012, 24(11): 4498-4510 ( ) DOI: 10.1105/tpc.112.103747
- Varshney R.K., Close T.J., Singh N.K., Hoisington D.A., Cook D.R. Orphan legume crops enter the genomics era! Curr. Opin. Plant Biol., 2009, 12(2): 202-210 ( ) DOI: 10.1016/j.pbi.2008.12.004
- Жуков В.А., Кулаева О.А., Жернаков А.И., Тихонович И.А. «Секвенирование следующего поколения» для изучения транскриптомных профилей тканей и органов гороха посевного (Pisum sativum L.). Сельскохозяйственная биология, 2015, 50(3): 278-287 ( , 10.15389/agrobiology.2015.3.278eng) DOI: 10.15389/agrobiology.2015.3.278rus
- Sudheesh S., Sawbridge T.I., Cogan N.O., Kennedy P., Forster J.W., Kaur S. De novo assembly and characterisation of the field pea transcriptome using RNA-Seq. BMC Genomics, 2015, 16: 611 ( ) DOI: 10.1186/s12864-015-1815-7
- Alves-Carvalho S., Aubert G., Carrère S., Cruaud C., Brochot A.L., Jacquin F., Klein A., Martin C., Boucherot K., Kreplak J., da Silva C., Moreau S., Gamas P., Wincker P., Gouzy J., Burstin J. Full-length de novo assembly of RNA-seq data in pea (Pisum sativum L.) provides a gene expression atlas and gives insights into root nodulation in this species. Plant Journal, 2015, 84(1): 1-19 ( ) DOI: 10.1111/tpj.12967
- Zhukov V.A., Zhernakov A.I., Kulaeva O.A., Ershov N.I., Borisov A.Y., Tikhonovich I.A. De novo assembly of the pea (Pisum sativum L.) nodule transcriptome. International Journal of Genomics, 2015, article 695947 ( ) DOI: 10.1155/2015/695947


