Характеристика генетической структуры аквакультурной и дикой популяций стерляди (Acipenser ruthenus Linnaeus, 1758) на основе анализа микросателлитов
Автор: Харзинова В.Р., Волкова В.В., Никипелов В.И., Доцев А.В., Барминцева А.Е., Бардуков Н.В., Никипелова А.К., Абдельманова А.С., Мюге Н.С., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 4 т.59, 2024 года.
Бесплатный доступ
Стерлядь (Acipenser ruthenus L. 1758) - один из наиболее мелких и короткоцикловых видов среди рыб семейства Acipenseridae с самым широким ареалом. Одно из ключевых условий, обеспечивающих эффективность мероприятий по сохранению и рациональному использованию генетических ресурсов этого в недавнем прошлом важного промыслового вида, находящегося сейчас в депрессивном состоянии, - изучение и оценка генетического разнообразия популяций стерляди. В настоящей работе на основе анализа полиморфизма 12 микросателлитных локусов (STR, short tandem reapets) представлены сведения о генетической изменчивости нескольких как заводских, так и естественных популяций, относящихся к бассейнам Белого, Каспийского, Карского, Азовского и Черного морей. Исследования проводили в 2023-2024 годах, ДНК выделяли из тканей грудных плавников стерляди (n = 191). Выборка состояла из двух групп рыб. Первая группа включала аквакультурные образцы от трех предприятий, на которых стада сформированы из особей стерляди окского (OK, n = 50, Можайский производственно-экспериментальный рыбоводный завод, д. Горетово, Можайский городской округ, Московская обл.), нижневолжского (NV, n = 34, аквакомплекс Астраханского государственного технического университета - АГТУ, г. Астрахань, Астраханская обл.) и сухонского (SX, n = 35, ООО Рыботоварная фирма «Диана», р. Кадуй, Кадуйский р-н, Вологодская обл.) происхождения. Во вторую группу были включены образцы от рыб из пяти природных популяций: куйбышевской (KV, n = 16, р. Волга, Республика Татарстан), саратовской (SV, n = 16, р. Волга, Саратовская обл.), донской (DN, n = 14, р. Дон, Ростовская обл.), вятской (VT, n = 16, р. Вятка, Республика Татарстан) и енисейской (EN, n = 10, р. Енисей, Красноярский край). Полиморфизм 12 STR микросателлитов (Aru13, Spl-163, An20, LS-68, AoxD161, AfuG 41, Afu 68 b, AfuG 51, LS-39, AfuG 63, AfuG 112, Aru18) определяли на генетических анализаторах ABI3130xl («Applied Biosystems», США) и НАНОФОР 05 (НПК «Синтол», Россия). Для оценки генетического разнообразия рассчитывали показатели аллельного разнообразия (AR), скорректированного на размер выборки, наблюдаемой (HO), несмещенной ожидаемой гетерозиготности (UHE) и коэффициента инбридинга (UFIS), основанного на несмещенной ожидаемой гетерозиготности. Для оценки происхождения, дисперсии и родства, характеристики генетической структуры и генетических связей между исследованными выборками стерляди использовали анализ главных компонент (Principal Component Analysis, PCA) и рассчитывали значения матрицы попарных генетических дистанций F ST и Jost’s D с последующим построением дендрограмм «сеть соседей» в программе SplitsTree 4.14.5. По результатам генетического анализа группа аквакультурных образцов из АГТУ (NV) имела наименьшие значения HO (0,566±0,098) и UHE (0,573±0,089). Во всех остальных семи популяциях стерляди величина HO варьировалась от 0,569 у SX до 0,708 у EN, а значения показателя UHE были выше 0,6. В семи исследованных выборках стерляди отмечались невысокие положительные значения коэффициента инбридинга (FIS), область доверительного интервала (CI 95 %) которого перекрывала нулевое значение, указывая на недостоверные отклонения числа гетерозигот от теоретически ожидаемого в указанных выборках. Исключение составила енисейская популяция стерляди, у которой детектировали отрицательные значения коэффициента инбридинга, что свидетельствует об избытке гетерозигот (UFIS(EN) = -0,1). Применение нескольких статистических подходов для оценки генетической структуры выявило, что наиболее обособлена енисейская популяция. При этом между группами рыб, относящимися к Волго-Каспийскому (окского, куйбышевского, саратовского и вятского происхождения) и Азово-Черноморскому (донская популяция) бассейнам было обнаружено сходство по частоте общих аллелей, общность генетической структуры и низкий уровень генетической дифференциации. Полученные в нашей работе данные о состоянии генетического разнообразия, генетическом сходстве и различиях между заводскими стадами стерляди, а также дикими популяциями Каспийского, Карского, Азово-Черноморского бассейнов могут быть востребованы при планировании управления маточными стадами и мер по сохранению уникального генофонда вида A. ruthenus . Для расширения представлений о генетической структуре генофонда российской стерляди эти исследования будут продолжены на выборках большего размера и с привлечением образцов из других стад аквакультуры и природных популяций.
Acipenser ruthenus, стерлядь, аквакультура, природные популяции, генетическое разнообразие, генетическая структура, str
Короткий адрес: https://sciup.org/142243762
IDR: 142243762 | УДК: 639.3:597.4/.5:575.174:575.22 | DOI: 10.15389/agrobiology.2024.4.666rus
Текст научной статьи Характеристика генетической структуры аквакультурной и дикой популяций стерляди (Acipenser ruthenus Linnaeus, 1758) на основе анализа микросателлитов
На территории Российской Федерации обитает 12 видов осетровых, относящихся к семейству Acipenseridae . К самым перспективным видам для товарной аквакультуры можно отнести белугу ( Huso huso Linnaeus, 1758), калугу ( Huso dauricus Georgi, 1775), сибирского ( Acipenser baerii Brandt, 1869) и русского осетра ( Acipenser gueldenstaedtii Brandt, 1833), а также стерлядь ( Acipenser ruthenus Linnaeus, 1758) (1). Стерлядь — один из самых мелких представителей осетровых с самым широким ареалом, встречается в бассейнах Каспийского, Черного, Азовского и Карского морей. Это стайная рыба, обитает в реках, обычно держится на глубине с быстрым течением, изредка встречаются особи, способные заходить в слабосоленые воды. Наиболее многочисленной была в речных бассейнах Дуная, Дона, Волги. По судоходным каналам в XIX веке проникла в бассейн Северной Двины, а из нее в середине XX века была интродуцирована в Онежское озеро и реку Печора (2). В бассейнах Оби и Енисея представлена подвидом сибирской стерлядь Acipenser ruthenus marsiglii , Brandt, 1833.
Несмотря на широкое распространение, численность популяций осетровых видов рыб сокращается из-за разрушительного антропогенного влияния (промысел, ухудшение экологических условий среды обитания и/или разрушение мест нагула, возведение гидроэнергетических сооружений, препятствующих нерестовым миграциям) (3). В настоящее время все виды осетровых включены в Конвенцию о международной торговле видами дикой фауны и флоры (CITES) в связи с угрозой их исчезновения (4), а также в IUCN Red List (5). Если среднегодовой вылов осетровых за 19001917 годы в России составлял 211 т, то в период с 2000 по 2020 годы он сократился до 10,8 т (6), а в настоящее время промышленный и любительский лов осетровых в России запрещен почти повсеместно.
Для решения проблемы снижения природных запасов этих ценных видов рыб приняты программы восстановления популяций. Одно из таких направлений — искусственное воспроизводство в рыбоводных хозяйствах с последующим выпуском подращенной молоди в их естественную среду обитания (7). Так, с 2010 по 2020 год заводской молодью пополнились популяции в Горьковском, Угличском, Иваньковском, Камском, Краснодарском, Волгоградском водохранилищах, а также в реках Сухона, Молога, Днепр, Дон, Кубань, Ока (6).
Активно развивающимся в последние десятилетия направлением по восполнению биологических ресурсов осетровых стало развитие индустриальной аквакультуры на рыбоводных заводах, работающих под эгидой ФГБУ Главное бассейновое управление по рыболовству и сохранению водных биологических ресурсов (ФГБУ Главрыбвод). Особое внимание уделяется искусственному воспроизводство редких и находящихся под угрозой исчезновения видов, таких как осетровые (8). На сегодняшний день в бассейне Азовского моря функционируют четыре осетровых рыбоводных завода (ОРЗ), входящих в структуру ФГБУ Главрыбвод, которые обеспечивают более 95 % выпуска молоди этих ценных видов рыб (Донской ОРЗ, Гривенский ОРЗ, Ачуевский ОРЗ, Темрюкский ОРЗ) (9). В бассейне Каспийского моря искусственное воспроизводство осетровых осуществляется на нескольких рыбоводных заводах (Лебяжий ОРЗ, Александровский ОРЗ, Кизанский ОРЗ, Житнинский ОРЗ, Волгоградский ОРЗ, Сергиевский ОРЗ)
(10), также искусственным воспроизводством занимается НЭБ «БИОС» Каспийского филиала ФГБНУ ВНИРО.
Развитие товарной аквакультуры достигается благодаря применению современных технологий выращивания осетровых в установках замкнутого водоснабжения (УЗВ) и в садковых хозяйствах, что обеспечивает потребительский рынок, снижая при этом нагрузку на природные популяции, осетровых (11). В настоящее время по всей территории России функционирует более 70 подобных предприятий, на которых осуществляется интенсивное товарное выращивание этих уникальных гидробионтов (12). Несмотря на высокий уровень технологии разведения, при репродукции осетровых рыбоводные заводы сталкиваются с серьезными проблемами, такими как дефицит производителей, а также получение нежизнеспособного потомства (6). Одной из возможных причин последнего считается формирование ремонтно-маточного стада из близкородственных особей, что приводит к снижению генетического разнообразия и качества рыбопосадочного материала (13). Поскольку для восполнения природных популяций используют заводские стада, контроль биоразнообразия в них исключительно важен для сохранения аллельного разнообразия в исходных диких популяциях. В свою очередь, уникальные аллели, распространенные в диких популяциях, могут стать важным генетическим ресурсом для селекции в товарной аквакультуре. Этим обусловлена необходимость постоянного контроля за уровнем генетического разнообразия в аквакультурных стадах осетровых, и в частности стерляди (14).
Оценку генетического полиморфизма аквакультурных стад и диких популяций стерляди можно осуществлять с помощью систем ДНК маркеров. Известны молекулярные маркеры, пригодные для изучения большого числа особей в природных и селекционных популяциях (15). За последние десятилетия описано несколько типов молекулярных маркеров для использования в аквакультуре и рыболовстве. Среди маркеров ядерного генома высокую эффективность при получении информации о популяционных процессах и анализе родства у объектов аквакультуры показали микросателлиты (15-17) — простые повторы последовательности ДНК (simple sequence repeat, SSR) (18) и короткие тандемные повторы ДНК (short tandem repeat, STR) (19). Благодаря их преимуществам — высокой степени полиморфизма, кодоминантному типу наследования (20), возможности мультиплексирования различных микросателлитов в ПЦР (21) и частичной автоматизации генотипирования с помощью фрагментного анализа на капиллярных секвенаторах (22) микросателлиты приобрели большое значение в мониторинге генетических процессов в искусственно воспроизводимых популяциях и промышленных стадах осетровых рыб (23). Так, эффективность STR маркеров как инструментов для решения широкого спектра популяционно-генетических задач обеспечила их применение в исследованиях русского осетра ( A. gueldenstaedtii ), сибирского осетра ( A. baerii ), амурского осетра ( A. schren-ckii ), севрюги ( A.stellatus ), калуги ( H. dauricus ) (24), стерляди ( A. ruthenus ) (25, 26), белуги ( H. huso ) (27), китайского осетра ( A. sinensis ) (28).
В настоящей работе с использованием высокополиморфных ДНК маркеров получены данные о генетической изменчивости современных заводских стад и естественных популяций стерляди Каспийского, Карского, Азово-Черноморского бассейнов России, что необходимо для планирования мер по охране этого важного промыслового вида, находящегося под угрозой исчезновения.
Наша цель заключалась в оценке актуального состояния генетического разнообразия и определении генетической структуры диких популяций 668
и аквакультурных стад стерляди в России.
Методика. Работу проводили в 2023-2024 годах. Объектами исследования служили особи заводских и природных популяций стерляди. Для создания карты с точками отбора образцов для анализа использовали R пакет maps (29).
Изученная выборка включала восемь групп рыб. Пять групп были представлены образцами диких популяций: куйбышевской (р. Волга, Республика Татарстан), саратовской (р. Волга, Саратовская обл.), донской (р. Дон, Ростовская обл.), вятской (р. Вятка, Республика Татарстан), енисейской (р. Енисей, Красноярский край). Три группы включали образцы аква-культурной рыбы окской (Можайский производственно-экспериментальный рыбоводный завод, д. Горетово, Можайский городской округ, Московская обл.), нижневолжской (аквакомплекс Астраханского государственного технического университета — АГТУ, г. Астрахань, Астраханская обл.) и сухонской (ООО Рыботоварна фирма «Диана», рп Кадуй, Кадуйский р-н, Вологодская обл.) популяций.
Биологический материал был депонирован в УНУ «Банк генетического материала домашних и диких видов животных и птицы» в составе сетевой биоресурсной коллекции сБРК СХЖ (соглашение с Минобрнауки России ¹ 075-15-2021-1037 от 28 сентября 2021 года), созданной и поддерживаемой в ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста. Образцы ткани стерляди естественных популяций были получены из Российской национальной коллекции эталонных генетических материалов (РНКЭГМ) ВНИРО (свидетельство об официальной регистрации базы данных ¹ 2006620351).
ДНК выделяли из ткани фрагментов грудных плавников. Использовали набор реагентов для выделения геномной ДНК серии ДНК-Экстран-2 (НПК «Синтол», Россия), согласно протоколу изготовителя. Качество и количество полученной ДНК контролировали флуориметрически (Qubit 4.0, «Invitrogen/Life Technologies», США) и спектрофотометрически (NanоDropTM 8000, «Thermo Fisher Scientific, Inc.», США). В результате были отобраны образцы с концентрацией ДНК от 15 нг/мкл и выше и соотношением поглощения при длинах волн 260 нм и 280 нм (OD 260/280 ) от 1,8 и выше.
Полиморфизм 12 микросателлитов изучали в трех мультиплексных ПЦР по четырем STR локусам в каждой панели: панель 1 — локусы Aru13, Spl-163, An20, LS-68, панель 2 — локусы AoxD161, AfuG 41, Afu 68 b, AfuG 51 и панель 3 — локусы LS-39, AfuG 63, AfuG 112, Aru18 (авторская разработка ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста) согласно опубликованным протоколам (30).
Размеры полученных продуктов амплификации определяли по электрофоретической подвижности относительно внутреннего стандарта длин GeneScan-350 ROX («Thermo Fisher Scientific, Inc.», США) и СД-450 (НПК «Синтол», Россия). Капиллярный электрофорез проводили на генетических анализаторах ABI3130xl («Applied Biosystems», США) и НАНОФОР 05 (НПК «Синтол», Россия). Исходные данные о длине каждого аллеля были получены в программном обеспечении GeneMapperTM v.4.0 и GeneMarker® v. 3.0.1 . Матрицу полученных микросателлитных генотипов формировали в программе Microsoft Excel.
Вся исследованная выборка стерляди была протестирована в программе ML-RELATE (31) для выявления родственных особей с их последующим исключением при статистическом анализе.
Внутрипопуляционные показатели, включая рарифицированное аллельное разнообразие (A R ), наблюдаемую (H O ) и несмещенную ожидаемую
( u H e ) гетерозиготность, а также значения коэффициента инбридинга ( U F IS ), основанного на несмещенной ожидаемой гетерозиготности c доверительным интервалом (CI) 95 %, рассчитывали и анализировали с помощью R пакета diveRsity (32).
Для оценки генетической структуры изучаемых популяций рыб применяли анализ главных компонент (Principal Component Analysis, PCA), который выполняли с визуализацией результатов, используя R пакеты adegenet (33) и ggplot2 (34).
На основании матрицы попарных значений индекса фиксации FST (35) и попарных генетических дистанций по Jost’s D (36), рассчитанных в R пакете diveRsity (32), были построены филогенетические деревья по алгоритму Neighbor-Net в программе SplitsTree 4.14.5 (37).
Исходные файлы формировали в формате MS Excel и программной среде R 3.5.0 (R Core Team) (38).
Значения параметров генетического разнообразия представлены в виде средних ( М ) и стандартных ошибок средних (±SEM).
Результаты. Основные характеристики изученной выборки рыб представлены в таблице 1.
1. Образцы, использованные в молекулярно-генетических исследованиях стерляди ( Acipenser ruthenus L.) из заводских и природных популяций (2023-2024 годы)
Популяция Обозначение Выборка, n Места отбора проб от диких/заводских особей Заводские популяции
|
Окская |
OK |
50 |
Можайский производственно-экспериментальный рыбоводный завод, Московская обл. |
|
Сухонская |
SX |
35 |
ООО Рыботоварная фирма «Диана», Вологодская обл. |
|
Нижневолжская |
NV |
34 |
Аквакомплекс Астраханского государственного технического университета, Астраханская обл. |
|
Естест |
венные популяции |
||
|
Куйбышевская |
KV |
16 |
Куйбышевское водохранилище |
|
Саратовская |
SV |
16 |
Саратовское водохранилище |
|
Донская |
DN |
14 |
р. Дон |
|
Вятская |
VT |
16 |
р. Вятка |
|
Енисейская |
EN |
10 |
р. Енисей |
Местоположение точек отбора проб стерляди показано на рисунке 1.
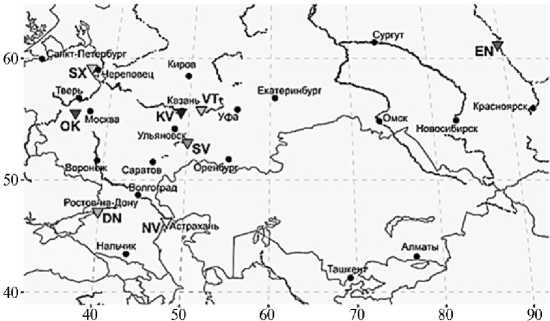
Рис. 1. Географическое распределение особей, отобранных из заводских и природных популяций стерляди ( Acipenser ruthenus L.) для проведения молекулярно-генетических исследований (2023
2024 годы) . Расшифровка аббревиатур представлена в таблице 1.
В результате анализа 12 микросателлитных локусов у исследованных групп стерляди мы суммарно выявили 154 аллеля, что выше среднего значения этого показателя (126 аллелей), детектированного с использованием
12 микросателлитов при изучении популяций стерляди Карпатского бассейна (26). При этом в нашем исследовании наибольшее число аллелей на локус выявили в локусе AfuG 112 (23 аллеля), а наименьшее — в локусе AfuG 63 (4 аллеля). При анализе популяционно-генетической структуры стерляди из р. Молога (Вологодская обл.) микросателлитный локус AfuG 63 также был малоаллельным (25). Однако, по мнению R. Sharma с соавт. (39), при использовании микросателлитного локуса для оценки генетического разнообразия, как это принято при стандартном выборе микросателлитных локусов, желательно наличие четырех аллелей на локус.
На основании полученных микросателлитных генотипов мы проанализировали основные показатели аллельного разнообразия и степени гетерозиготности в исследованных выборках стерляди (табл. 2).
2. Результаты анализа генетического разнообразия в заводских и природных популяциях стерляди ( Acipenser ruthenus L.) на основании полиморфизма 12 мик-росателлитных STR локусов (2023-2024 годы)
|
Популяция |
1 n 1 |
Ar |
1 HO |
1 uHe 1 |
uFis [CI 95 %] |
|
OK |
50 |
6,417±0,956 |
0,608±0,085 |
0,651±0,087 |
0,060[ - 0,007;0,127] |
|
SX |
35 |
5,286±0,700 |
0,569±0,074 |
0,628±0,077 |
0,086[ - 0,012;0,184] |
|
NV |
34 |
4,925±0,731 |
0,566±0,098 |
0,573±0,089 |
0,026[ - 0,1;0,152] |
|
KV |
16 |
6,630±0,974 |
0,594±0,082 |
0,660±0,089 |
0,071[ - 0,028;0,17] |
|
SV |
16 |
6,042±0,873 |
0,589±0,092 |
0,636±0,089 |
0,139[ - 0,026;0,304] |
|
DN |
14 |
6,036±0,815 |
0,631±0,087 |
0,670±0,077 |
0,077[ - 0,039;0,193] |
|
VT |
16 |
6,483±0,949 |
0,609±0,078 |
0,661±0,081 |
0,071[ - 0,022;0,164] |
|
EN |
10 |
5,000±0,718 |
0,708±0,078 |
0,655±0,074 |
- 0,100[ - 0,212;0,012] |
Примечани е. n — число образцов, AR — рарифицированное аллельное разнообразие, HO — наблюда- емая гетерозиготность, UHE — несмещенная ожидаемая гетерозиготность, UFIS — несмещенный коэффициент инбридинга, CI 95 % — доверительный интервал 95 %, в скобках — размах изменчивости UFIS при доверительном интервале 95 %. Выборки аквакультурной стерляди: OK — окская, SX — сухонская, NV — нижневолжская; выборки дикой популяции стерляди: KV — куйбышевская, SV — саратовская, DN — донская, VT — вятская, EN — енисейская (описание см. в табл. 1).
Значения аллельного разнообразия варьировались от 4,925±0,731 в выборке стерляди заводской популяции аквакомплекса АГТУ нижневолжского происхождения (NV) до 6,630±0,974 в выборке природной популяции Куйбышевского водохранилища (KV). Выборка стерляди из аквакомплекса АГТУ также характеризовалась минимальными значениями наблюдаемой (H o ) и ожидаемой ( U H E ) гетерозиготности, что составило соответственно 0,566±0,098 и 0,573±0,089. По распределению значений ожидаемой ( U H E ) гетерозиготности все остальные выборки рыб характеризовались схожими значениями показателя выше 0,6 с варьированием от 0,628±0,077 в заводской популяции рыб сухонского происхождения до 0,670±0,077 в донской популяции. Следует отметить, что во всех исследованных группах значения ожидаемой гетерозиготности были немного выше значений наблюдаемой гетерозиготности, что указывает на дефицит гетерозигот. Исключение составила выборка стерляди из енисейской популяции, у которой значения наблюдаемой гетерозиготности (Н о ) превосходили значения ожидаемой ( u H e ). Кроме того, в указанной популяции стерляди были детектированы отрицательные значения коэффициента инбридинга, что свидетельствует об избытке гетерозигот ( U F IS(EN) = - 0,1). Во всех остальных выборках коэффициент инбридинга имел положительные значения с варьированием от 0,026 у NV до 0,139 у SV. При этом во всех группах рыб область доверительного интервала (CI 95 %) коэффициента инбридинга перекрывала нулевое значение, указывая на недостоверные отклонения числа гетерозигот от теоретически ожидаемого.
Для идентификации и характеристики индивидуумов в исследованных нами заводских стадах и естественных популяциях стерляди, а также для оценки происхождения, дисперсии и родства мы использовали анализ главных компонент (Principal Component Analysis, PCA) (40). Главные компоненты выстраиваются в порядке убывания той дисперсии, которую они объясняют (первой главной компонентой обусловлена большая часть дисперсии, чем второй компонентой, второй — большая, чем третьей, и т.д.) (41). Чем больше главных компонент учитывается, тем большую часть изменчивости можно объяснить. На рисунке 2 представлены результаты ана- лиза по трем главным компонентам.
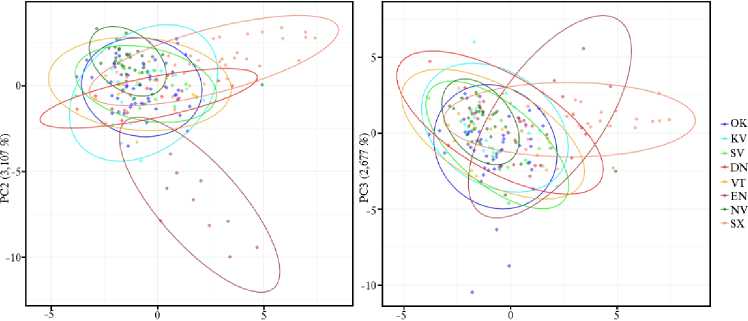
ККЗ.Ш») РС1 (3,131 Я)
Рис. 2. Анализ генетической структуры заводских и природных популяций стерляди ( Acipenser ruthenus L.) методом главных компонент (PCA) на основании полиморфизма 12 микросателлитных STR локусов: выборки аквакультурной стерляди — окская (OK), сухонская (SX), нижневолжская (NV); выборки из природных популяций — куйбышевская (KV), саратовская (SV), донская (DN), вятская (VT), енисейская (EN), описание см. в таблице 1 (2023-2024 годы).
В нашем исследовании первая главная компонента объясняла наибольшее значение дисперсии (3,131 %) и максимально обособила кластер, сформированный особями енисейской популяции. Кроме этого, пара первых двух компонент С1-С2 отделила кластер особей заводской сухонской группы с небольшим перекрытием массива остальных выборок стерляди. Вторая и третья компоненты отражали соответственно 3,107 и 2,677 % генетической изменчивости. При этом все три компоненты показали, что выборки стерляди окского, нижневолжского, куйбышевского, саратовского, донского и вятского происхождения демонстрировали сходство по частоте общих аллелей, образуя единый генетический пул.
-
3. Генетические дистанции между выборками стерляди ( Acipenser ruthenus L.) из заводских и природных популяций, рассчитанные на основании попарных генетических дистанций F ST и показателя аллельной дифференциации Jost’s D (2023-2024 годы)
|
Популяция |
OK |
KV |
SV |
DN |
VT |
EN |
NV |
SX |
|
OK |
0,015 |
0,002 |
0,039 |
0,004 |
0,096 |
0,050 |
0,043 |
|
|
KV |
0,028 |
0,020 |
0,028 |
- 0,001 |
0,075 |
0,045 |
0,043 |
|
|
SV |
0,005 |
0,022 |
0,055 |
0,009 |
0,105 |
0,038 |
0,048 |
|
|
DN |
0,059 |
0,034 |
0,103 |
0,029 |
0,087 |
0,101 |
0,082 |
|
|
VT |
0,008 |
0 |
0,022 |
0,012 |
0,101 |
0,055 |
0,041 |
|
|
EN |
0,192 |
0,138 |
0,178 |
0,172 |
0,175 |
0,159 |
0,124 |
|
|
NV |
0,099 |
0,090 |
0,060 |
0,182 |
0,106 |
0,286 |
0,070 |
|
|
SX |
0,092 |
0,072 |
0,076 |
0,162 |
0,091 |
0,179 |
0,114 |
Примечани е. Значения F ST указаны над диагональю, значения D — под диагональю при парном сравнении. Выборки аквакультурной стерляди — окская (OK), сухонская (SX), нижневолжская (NV); выборки из природных популяций — куйбышевская (KV), саратовская (SV), донская (DN), вятская (VT), енисейская (EN), описание см. в таблице 1.
Для оценки степени генетической близости изученных выборок стерляди мы рассчитали значения и индекса фиксации FST (35), и показателя D, предложенного L. Jost (36) (табл. 3). Далее полученные значения показателей были визуализированы с помощью алгоритма «сети соседей» (Neighbor Net) и представлены в виде филогенетической дендрограммы (рис. 3).
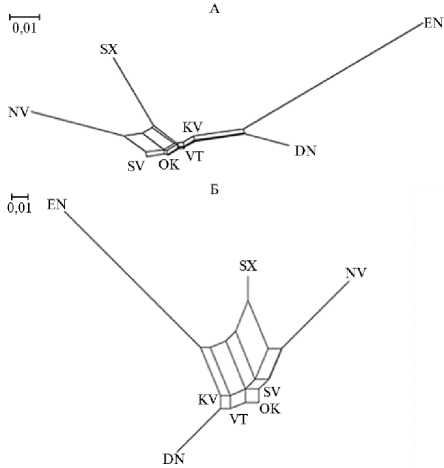
Рис. 3. Визуализация степени генетической близости изученных выборок стерляди ( Acipenser ruthenus L.) из заводских и природных популяций на основе матрицы попарных генетических дистанций F ST (А) и Jost’s D (Б) по алгоритму «сети соседей» (NeighborNet): выборки аквакуль-турной стерляди — окская (OK), сухонская (SX), нижневолжская (NV); выборки из природных популяций — куйбышевская (KV), саратовская (SV), донская (DN), вятская (VT), енисейская (EN), описание см. в таблице 1 (2023-2024 годы).
Анализ значений матрицы попарных генетических дистанций F ST и Jost’s D, а также структуры генетических сетей показал отсутствие генетической дифференциации куйбышевской и вятской популяций, между которыми значения обоих показателей равнялись 0, указывая на полный обмен генетическим материалом между ними. Кроме этого была выявлена наибольшая генетическая близость окской группы с вятской и саратовской популяциями: F ST OK/SV = 0,002, F ST OK/VT = 0,004, D OK/SV = 0,005, D OK/VT = 0,008. Более высокую степень генетической дифференциации детектировали между енисейской и нижневолжской группами стерляди: F ST EN/NV = 0,159, D en/nv = 0,286. Следует отметить, что на топологии дендрограмм (см. рис. 3) мы обнаружили кластер, образованный окской, нижневолжской, куйбышевской, саратовской и вятской выборками. При этом две естественные популяции р. Дон и р. Енисей, так же как две заводские группы р. Волги и р. Сухоны (приток Северной Двины) сформировали отдельные ветви.
Планирование и сохранение генетических ресурсов объектов аквакультуры все больше зависят от генетических данных, которые используются для оценки генетической структуры популяции, репродуктивной динамики и их адаптации (17, 42). Применение микросателлитных маркеров играет в этом важную роль, и во многих исследованиях показано, что именно такой тип ДНК-маркеров востребован как для управления маточным стадом, так и для оценки структуры популяций рыбы (43, 44). Вместе с тем в настоящее время информации об актуальном состоянии генетического разнообразия и генетических взаимоотношениях между популяциями стерляди из бассейнов Белого, Каспийского, Карского, Азовского и Черного морей нашей страны недостаточно.
Согласно Н.В. Козловой с соавт. (23), генетическое разнообразие или генетический полиморфизм (один из компонентов генетической характеристики популяции или вида) имеет большое значение для экологической пластичности популяции, позволяет ей адаптироваться к изменяющимся условиям среды обитания. В нашей работе анализ внутрипопуляционных параметров, рассчитанных на основе полиморфизма 12 микросателлитов, показал, что наименьший уровень полиморфности имела выборка заводской стерляди нижневолжского происхождения: A R = 4,925, H O = 0,566 и u H e = 0,573. Учитывая сниженный по сравнению с другими выборками (в том числе с дикими популяциями из р. Волги) уровень полиморфизма, а также выделение этой группы в самостоятельную ветвь на филогенетических дендрограммах (см. рис. 3), можно предположить, что на формирование генетической структуры нижневолжского стада стерляди оказал влияние эффект основателя. При этом максимальные значения аллельного разнообразия были детектированы в трех популяциях Волго-Каспийского бассейна (окская, куйбышевская, вятская), что может свидетельствовать о крупном размере природной популяции и генетическом обмене между популяциями стерляди из различных участков течения р. Волга как важном факторе поддержания генетического разнообразия и структуры популяций рыб (45). Следует отметить, что выявленное нами сходство по частоте общих аллелей (см. рис. 2) и низкий уровень генетической дифференциации (см. табл. 3) между окской, куйбышевской, саратовской, донской и вятской популяциями также предполагает наличие более высокого межпопуляционного потока генов и, следовательно, большую способность к миграции представителей указанных популяций. Она влияет на географическую структуру генетической изменчивости внутри вида и, возможно, является результатом неконтролируемого зарыбления р. Дон молодью стерляди волжского происхождения.
Высокий уровень генетической дифференциации популяций пресноводных рыб (в сравнении с морскими) обусловлен большим числом миграционных барьеров и уменьшением потока генов (46, 47). Анализ генетической структуры исследованных нами популяций стерляди, проведенный с использованием нескольких статистических подходов, показал, что наиболее обособленной структурой характеризуется енисейская популяция. Наше наблюдение согласуется с опубликованными ранее выводами о значительном отличии енисейской популяций стерляди, постулируемом на основе анализа как митохондриальных (48), так и ядерных (49) ДНК-маркеров. На популяции осетровых, населяющих сибирские реки, могли повлиять плейстоценовые оледенения, когда северный сток рек был перекрыт ледниками и большинство рек, изменив свое русло и образовав систему озер, стали впадать в Каспий, а затем и в Черное море (48, 50). Анадромные виды пострадали от таких радикальных изменений в большей степени, чем неанадромные и пресноводные. Таким образом, стерлядь и сибирский осетр, возможно, обладали выгодными адаптационными свойствами, которые позволили им выжить в периоды оледенения за счет ограничения путей миграции в пределах пресноводных бассейнов.
Результаты наших исследований также показали, что еще одной группой, продемонстрировавшей более высокую степень генетической обособленности при наличии нескольких гибридных особей, является выборка заводской стерляди сухонского происхождения из ООО РТФ «Диана». Известно, что с 2001 года на этом предприятии велась работа по сохранению уникальной локальной сухонской популяции стерляди, обитающей в р. Сухона (51), откуда были выловлены экземпляры дикой стерляди, которые 674
использовались для получения молоди и ее последующего выпуска с целью восстановления исходной популяции. Вероятно, избыток молоди стерляди, полученной на предприятии, был использован для формирования ремонтноматочного стада, аллелофонд которого в настоящий момент содержит высокую долю редких генотипов, характерных для дикой популяции р. Сухона.
Итак, проведенные нами исследования не только подчеркнули потенциал микросателлитных маркеров для проведения базовых генетических исследований недостаточно изученных видов рыб, требующих сохранения, но и предоставили уникальную информацию о состоянии генетического разнообразия, генетическом сходстве и различиях между дикими популяциями Acipenser ruthenus в реках бассейнов Каспийского, Карского, Азовского и Черного морей, а также заводскими стадами стерляди. Так, установлено, что среди трех исследованных групп аквакультурной стерляди показатели аллельного и генетического разнообразия были выше в окской популяции: A r = 6,417±0,956, H o = 0,608±0,085, u H e = 0,651±0,087. В изученных нами естественных популяциях наибольшие значения по аллельному разнообразию выявлены в выборке стерляди из р. Вятки (A R = 6,483±0,949), по наблюдаемой гетерозиготности — в выборке из р. Енисей (H O = 0,708±0,078), по ожидаемой гетерозиготности — в выборке из р. Дон ( U H E = 0,670±0,077). При этом, выборка аквакультурной стерляди из Астраханской области характеризовалась наименьшим как аллельным (A RNV = 4,925±0,731), так и генетическим разнообразием (H ONV = 0,566±0,098, U H ENV = 0,573±0,089). Полученные результаты могут быть использованы при генетическом мониторинге популяций стерляди для сохранения их генетической структуры и изменчивости, при управлении маточным стадом и резервировании уникального генофонда вида A. ruthenus . Для получения более детальной информации о генетической вариабельности и генетической структуре генофонда российской стерляди мы продолжим исследования на выборках большего размера и с привлечением образцов из других стад аквакультурной стерляди и ее природных популяций.
Выражаем благодарность ООО РТФ «Диана» и Союзу осетроводов за предоставление образцов биоматериала для исследований.
Список литературы Характеристика генетической структуры аквакультурной и дикой популяций стерляди (Acipenser ruthenus Linnaeus, 1758) на основе анализа микросателлитов
- Макеева А.П., Павлов Д.С., Павлов Д.А. Атлас молоди пресноводных рыб России. М., 2011.
- Волосников Г.И. Обзор данных по биологии стерляди Acipenserruthenus (Linnaeus, 1758). Вестник Астраханского государственного технического университета, 2017, 2(64): 67-72.
- Рубан Г.И., Ходоревская Р.П., Кошелев В.Н. О состоянии осетровых в России. Астраханский вестник экологического образования, 2015, 1(31): 42-50.
- Niu X., Wang X., Zhang Y., Zhao Z., Fan E. Integration and application of sturgeon identification methods. Biodiversity Science, 2022, 30(6): 22034 (doi: 10.17520/biods.2022034).
- IUCN Biodiversity Assessment & Knowledge Team: Red List Unit. The IUCN Red List of Threatened Species. Режим доступа: https://www.iucnredlist.org/search?query=Sturgeon&searchType=species. Дата обращения: 01.05.2024.
- Быков А.Д., Бражник С.Ю. Современное состояние запасов и искусственного воспроизводства стерляди в России. Вопросы рыболовства, 2022, 23(3): 5-30 (doi: 10.36038/0234-2774-2022-23-3-5-30).
- Чебанов М.С., Галич Е.В. Руководство по искусственному воспроизводству осетровых рыб. Технические доклады ФАО по рыбному хозяйству и аквакультуре. № 558. Анкара, 2013.
- ФГБУ «Главрыбвод». Деятельность. Режим доступа: https://glavrybvod.ru/. Дата обращения: 01.05.2024.
- Бугаев Л.А., Небесихина Н.А., Алимова А.Ш., Гайдамаченко В.Н. Генетическое разнообразие производителей русского осетра из ремонтно-маточного стада Донского осетрового завода ФГБУ «Главрыбвод» в 2018-2023 гг. Рациональная эксплуатация водных биологических ресурсов. Материалы Международной научно-технической конференции, Владивосток, 26-27 октября 2023 года. Владивосток, 2023: 140-146.
- Щербакова В.Д., Барминцева А.Е., Сафронов А.С. Оценка генетического разнообразия стад русского осетра (Acipenser gueldenstaedtii Brandt, 1833) каспийской популяции семи осетровых рыбоводных заводов ФГБУ «Главрыбвод». Современные проблемы и перспективы развития рыбохозяйственного комплекса. Материалы XI Международной научно-практической конференции молодых ученых и специалистов, СПб, 25-26 сентября 2023 года. М., 2023: 243-245.
- Судакова Н.В., Эльхетави А., Васильева Л.М., Анохина А.З. Оценка экономической эффективности выращивания русского осетра товарной массы в бассейнах УЗВ, садках и прудах. Технологии пищевой и перерабатывающей промышленности АПК — продукты здорового питания, 2019, 2(28): 40-45.
- ГК «Агриконсалт». Рынок осетровых: состояние и перспективы. Режим доступа: https://agri-cons.ru/publikatsii/nashi-publikatsii/rynok-osetrovykh-sostoyanie-i-perspektivy/ . Дата обращения: 02.05.2024.
- Павлюк А.А., Горбенко Е.В. Современные проблемы искусственного воспроизводства осетровых видов рыб в азовском бассейне. Сборник тезисов докладов участников пула научно-практических конференций, Сочи, 23-27 января 2020 года. Керчь, 2020: 220-222.
- Васильева Л.М. Проблемы и перспективы развития аквакультуры осетровых рыб в современных условиях. Сборник статей Международной научно-практической конференции «Аквакультура осетровых рыб: проблемы и перспективы», Астрахань, 10-12 октября 2017 года. Астрахань, 2017: 7-10.
- Wenne R. Microsatellites as molecular markers with applications in exploitation and conservation of aquatic animal populations. Genes (Basel), 2023, 14(4): 808 (doi: 10.3390/genes14040808).
- Hauser L., Baird M., Hilborn R., Seeb L.W., Seeb J.E. An empirical comparison of SNPs and microsatellites for parentage and kinship assignment in a wild sockeye salmon (Oncorhynchus nerka) population. Mol. Ecol. Resour., 2011, 11: 150-161 (doi: 10.1111/j.1755-0998.2010.02961.x).
- Wilson A.B., Ashe J., Padron M., Hamilton H. Comprehensive genus-wide screening of seahorse microsatellite loci identifies priority species for conservation assessment. Conserv. Genet. Resour., 2021, 13: 221-230. (doi: 10.1007/s12686-021-01198-4).
- Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Research, 1989, 17: 6463-6471 (doi: 10.1093/nar/17.16.6463).
- Edwards A., Civitello A., Hammond H.A., Caskey C.T. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats. The American Journal of Human Genetics, 1991, 49(4): 746-775.
- Baron E.E., Mário L.M., Verneque R.S., Coutinho L.L. Parentage testing and effect of misidentification on the estimation of breeding value in Gir cattle. Genet. Mol Biol., 2002, 25(4): 389-394 (doi: 10.1590/S1415-47572002000400006).
- Kuleung C., Baenziger P.S., Dweikat I. Transferability of SSR markers among wheat, rye and triticale. Theor. Appl .Genet., 2004, 108(6) :1147-1150 (doi: 10.1007/s00122-003-1532-5).
- Yang W., Kang X., Yang Q., Lin Y., Fang M. Review on the development of genotyping methods for assessing farm animal diversity. Anim. Sci. Biotechnol., 2013, 4(1): 2 (doi: 10.1186/2049-1891-4-2).
- Козлова Н., Базелюк Н., Файзулина Д., Стоногина Е. Применение молекулярно-генетических исследований в аквакультуре осетровых рыб. Вестник Астраханского государственного технического университета. Серия: Рыбное хозяйство, 2013, 3: 113-117.
- Барминцева А.Е., Мюге Н.С. Использование микросателлитных локусов для установления видовой принадлежности осетровых (Acipenseridae) и выявления особей гибридного происхождения. Генетика, 2013, 49(9): 1093 (doi: 10.7868/S0016675813090038).
- Игнашев А.А., Щербакова В.Д., Борисов М.Я., Барминцева А.Е., Думнич Н.В., Мюге Н.С. Популяционно-генетическая структура стерляди Acipenser ruthenus L., 1758 реки Молога (Вологодская область) в условиях ее реакклиматизации. Трансформация экосистем, 2023, 6(4): 79-95 (doi: 10.23859/estr-230810).
- Fazekas G., Kovács G., Sándor Z.J., Bogár K., Géczi A., Kovács B. Genetic characterization of endangered Sterlet (Acipenser ruthenus, Linnaeus 1758) Gene Bank Broodstock, natural and cultured populations in Hungary. Fishes, 2024, 9: 201 (doi: 10.3390/fishes9060201).
- Алимова А.Ш., Небесихина Н.А., Абросимова Е.Б. Исследование генетического разнообразия белуги (Huso huso Linnaeus, 1758) в ремонтно-маточном стаде Донского осетрового завода на основе микросателлитных локусов. Материалы Всероссийской (национальной) научно-практической конференции «Актуальные проблемы науки и техники», Ростов-на-Дону, 16-18 марта 2022 года. Ростов-на-Дону, 2022: 133-134.
- Hu Y., Liu X., Yang J., Xiao K., Wang B., Du H. Development and characterization of a duplex PCR assay in Chinese sturgeon (Acipenser sinensis) for genetic analysis. Sci. Rep., 2020, 10(1): 3451 (doi: 10.1038/s41598-020-60401-y).
- Becker R.A., Wilks A.R., Brownrigg R., Minka T.P., Deckmyn A. Draw geographical maps. R package version 3.3.0. 2018. Режим доступа: https://CRAN.R-project.org/package=maps. Без даты.
- Никипелов В.И., Харзинова В.Р., Волкова В.В., Никипелова А.К., Бардуков Н.В., Грозеску Ю.Н., Зиновьева Н.А. Разработка мультилокусной STR-панели и оценка достоверности происхождения аквакультурной стерляди (Acipenser ruthenus). Известия НВ АУК, 2024. 3(75): 273-282 (doi: 10.32786/2071-9485-2024-03).
- Kalinowski S.T., Taper M.L. Maximum likelihood estimation of the frequency of null alleles at microsatellite loci. Conservation Genetics, 2006, 7(6): 991-995 (doi: 10.1007/s10592-006-9134-9).
- Keenan J.D., Hotez P.J., Amza A., Stoller N.E., Gaynor B.D., Porco T.C., Lietman T.M. Elimination and eradication of neglected tropical diseases with mass drug administrations: a survey of experts. PLoS Neglected Tropical Diseases, 2013, 7(12): 2562 (doi: 10.1371/journal.pntd.0002562).
- Jombart T., Ahmed I. Adegenet 1.3-1: new tools for the analysis of genome-wide SNP data. Bioinformatics, 2011, 27(21): 3070-3071 (doi: 10.1093/bioinformatics/btr521).
- Wickham H. Ggplot2: elegant graphics for data analysis (use R!). Springer, New York, 2009.
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 (doi: 10.2307/2408641).
- Jost L. G(ST) and its relatives do not measure differentiation. Molecular Ecology, 2008, 17: 4015-4026 (doi: 10.1111/j.1365-294x.2008.03887.x).
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 (doi: 10.1093/molbev/msj030).
- R Core Team. R: A language and environment for statistical computing. R Foundation for statistical computing. Vienna, Austria, 2012. Режим доступа: https://www.semanticscholar.org/paper/R%3A-A-language-and-environment-for-statistical-Team/659408b243cec55de8d0a3bc51b81173007aa89b. Без даты.
- Sharma R., Kishore A., Mukesh M., Ahlawat S., Maitra A., Pandey A.K., Tantia M.S. Genetic diversity and relationship of Indian cattle inferred from microsatellite and mitochondrial DNA markers. BMC Genet., 2015, 16: 73 (doi: 10.1186/s12863-015-0221-0).
- Elhaik E. Principal Component Analyses (PCA)-based findings in population genetic studies are highly biased and must be reevaluated. Sci. Rep., 2022, 12: 14683 (doi: 10.1038/s41598-022-14395-4).
- Борякова Е.Е., Кочетков И.Б. Применение метода главных компонент для анализа зоологических данных. Актуальные вопросы современной науки, 2009, 7-1: 6-11.
- Garner B.A., Hoban S., Luikart G. IUCN Red List and the value of integrating genetics. Conservation Genetics, 2020, 21: 795-801 (doi: 10.1007/s10592-020-01301-6).
- Moghim M., Javanmard A., Pourkazemi M., Tan S., Panandam J., Kor D., Laloei F. Application of microsatellite markers for genetic conservation and management of Persian sturgeon (Acipenser persicus, Borodin, 1897) in the Caspian Sea. Journal of Applied Ichthyology, 2013, 29: 696-703 (doi: 10.1111/JAI.12195).
- Boscari E., Pujolar J.M., Dupanloup I., Corradin R., Congiu L. Captive breeding programs based on family groups in polyploid sturgeons. PLoS ONE, 2014, 9: e110951 (doi: 10.1371/journal.pone.0110951).
- Song N., Nwafili S.A., Gao T.X. Genetic diversity and population structure of Chrysichthys nigrodigitatus from Niger Delta based on AFLP analysis. Biochemical Systematics and Ecology, 2011, 39(4-6): 320-327 (doi: 10.1016/j.bse.2011.04.002).
- Abbas K., Zhou X., Li Y., Gao Z., Wang W. Microsatellite diversity and population genetic structure of yellowcheek, Elopichthys bambusa (Cyprinidae) in the Yangtze River. Biochemical Systematics and Ecology, 2010, 38(4): 806-812 (doi: 10.1016/j.bse.2010.08.003).
- Cvijanović G., Adnadević T., Jarić I., Lenhardt M, Marić S. Genetic analysis of sterlet (Acipenser ruthenus L.) populations in the Middle and Lower Danube sections. North-Western Journal of Zoology, 2017, 13(1): 34-43.
- Pobedintseva M.A., Makunin A.I., Kichigin I.G., Kulemzina A.I., Serdyukova N.A., Romanenko S.A., Vorobieva N.V., Interesova E.A., Korentovich M.A., Zaytsev V.F., Mischenko A.V., Zadelenov V.A., Yurchenko A.A., Sherbakov D.Y., Graphodatsky A.S., Trifonov V.A. Population genetic structure and phylogeography of sterlet (Acipenser Ruthenus, Acipenseridae) in the Ob and Yenisei river basins. Mitochondrial DNA. Part A, DNA Mapping, Sequencing, and Analysis. 2019, 30(1): 156-164 (doi: 10.1080/24701394.2018.1467409).
- Fopp-Bayat D., Kuzniar P., Kolman R., Liszewski T., Kucinski M. Genetic analysis of six sterlet (Acipenser ruthenus) populations —recommendations for the plan of restitution in the Dniester River. Iranian Journal of Fisheries Science, 2015, 14: 634-645.
- Барминцева А.Е., Мюге Н.С. Генетический полиморфизм сибирского осетра Acipenser baerii Brandt, 1869 в аквакультуре. Генетика, 2018, 54(2): 216-223 (doi: 10.7868/S0016675818020030).
- Слынько Е.Е., Слынько Ю.В. Генетические последствия реинтродукции стерляди в бассейне реки Сухона. Сборник научных трудов по материалам Национальной научно-практической конференции с международным участием «Научно-прикладные аспекты производства, переработки и ветеринарно-санитарного контроля сельскохозяйственной продукции», Ярославль, 27-28 марта 2019 года. Ярославль, 2019: 150-154.


