Характеристика железосодержащих нитрилгидратаз почвенных бактерий рода Rhodococcus
Автор: Максимова А.В., Кузнецова М.В.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 1, 2016 года.
Бесплатный доступ
Проведена детекция генов, кодирующих α- и β-субъединицы железосодержащих нитрилгидратаз, среди альдоксим- и нитрилутилизирующих микроорганизмов, выделенных из образцов природных и антропогенно-загрязненных почв. Встречаемость генов фермента не зависела от субстрата, на котором были выделены бактерии (р = 0.73). Показана высокая гомология nha и nhb с известными последовательностями нитрилгидратаз из базы данных GenBank (от 84 до 99%). Секвенирование генов не выявило мутаций в областях, кодирующих активные или регуляторные центры ферментов. Отмечено преобладание замен среди штаммов, выделенных из загрязненных почвенных образцов (66.7% для α-субъединицы и 70.0% для β-субъединицы). Полученные аминокислотные последовательности ферментов почвенных изолятов содержали как характерные для рода Rhodococcus, так и уникальные замены шинокислотнык остатков. У штамма R. erythropolis Б4-4, выделенного из почвы, загрязненной акриламидом, обнаружена радикальная аминокислотная замена тирозина на серин в 85 сайте а-субъединицы.
Почвенные бактерии рода rhodococcus, нитрилгидратаза, аминокислотные замены
Короткий адрес: https://sciup.org/147204753
IDR: 147204753 | УДК: 579.25
Текст научной статьи Характеристика железосодержащих нитрилгидратаз почвенных бактерий рода Rhodococcus
За последние 20 лет опубликовано большое количество работ, посвященных исследованию бактериальных культур, способных утилизировать нитрилы карбоновых кислот. Это связано с тем, что нитрилгидролизующие бактерии имеют большой потенциал для промышленного производства акриламида и никотинамида, а также для биоремедиации загрязненных ширильными соединениями территорий [Yamada, Kobayashi, 1996; Holtze et al., 2008].
Главными источниками для выделения нитрил конвертирующих организмов являются почва, прибрежные морские осадки и глубоководные отложения. Известно, что в почвах с химической нагрузкой происходит активное размножение и накопление бактерий, способных усваивать загрязняющие вещества. Так, Verma и Sangave выделили Bacillus thuringiensis из почвы, отобранной с предприятия по производству бензонитрила, способный утилизировать ароматические нитрилы [Verma, Sangave, 2014]. Штамм Rhodococcus sp. МТВ5,
изолированный из загрязненной нитрильными гербицидами сельскохозяйственной почвы, полностью конвертировал 30 мМ бензонитрил и был способен расти в среде с добавлением 60 мМ субстрата. Также этот штамм оказался активен в отношении алифатических нитрилов [Mukram et al., 2015]. Большинство трансформирующих нитрилы микроорганизмов являются представителями ак-тиномицетов, среди которых преобладают родо-кокки. В работе [Brandao et al., 2002] авторы установили, что Rhodococcus erithropolis доминирует среди культивируемых нитрилутилизирующих бактерий в морских и почвенных образцах.
Конверсия нитрильных соединений может происходить несколькими способами, в том числе одностадийным гидролизом, осуществляемым нит-рилазой, или двустадийным, в котором участвуют два фермента - нитрилгидратаза и амидаза. Нитрилгидратаза (ЕС4.2.1.84) - это водорастворимый металлофермент, который катализирует гидратацию нитрильных соединений в соответствующие амиды. Впервые нитрилгидратаза была обнаружена в клетках бактерий Rhodococcus rhodochrous J-l (ранее Arthrobacter sp. J-l) в 1980 г. [Asano et al., 1980]. К настоящему времени изучены десятки ферментов из различных видов бактерий, но и сегодня родококки и другие актиномицеты являются наиболее распространенными источниками новых нитрилгидратаз [Prasad, Bhalla, 2010]. Нитрилгид-ратазы состоят из двух субъединиц (а и |3), структурные гены (nha и nhb) которых существенно отличаются в пределах различных таксонов. Несмотря на то, что у представителей рода Rhodococcus описана высокая гомология этих ферментов, субстратная специфичность их может существенно варьировать [Brandao et al., 2003].
Цель данной работы - изучение разнообразия нитрилгидратаз у почвенных бактерий рода Rhodococcus.
Объекты и методы исследования
Объектами исследования являлись бактериальные культуры, утилизирующие альдоксимы (и = 47) и нитрилы карбоновых кислот (и = 52), полученные ранее (2002-2004 гг. и 2006-2008 гг.) в результате селекции на ацето- и/или изобутиронитриле из образцов почв и вод естественной среды и почвы промышленных предприятий (ФГУП «ПЗ им. С.М. Кирова» и ОАО «Бератон»),
Препараты хромосомной ДНК бактерий получали фенольным методом, модифицированным для выделения ДНК из актиномицетов [Гловер и др., 1988].
Для ПЦР-детекции генов, кодирующих а-субъединицу нитрилгидратазы, использовали праймеры АМ5 (5’- CATATGTCAGTAACGA-TCGAC-3’) и АМ8 (5’- ATGCATCAGACGGT-GGGAACCTG-3’), [3-субъединицу - АМ4 (5’-
CATATGGATGGAGTACACGAT-3’) и АМ6 (5’-ATGCATCAGGCCGCAGGCTCGAG-3 ’), сконструированные на основе последовательности штамма Rhodococcus sp. N-774 (Х54074.1). Режим амплификации для всех праймеров включал начальный цикл денатурации - 1 мин. при 94°С, 93°С - 40 с.; 48°С - 60 с.; 72°С - 150 с. (5 циклов); 93°С - 50 с.; 54°С - 70 с.; 72°С - 180 с. (30 циклов) и завершающий цикл 3 мин. при 72°С. Электрофоретическое разделение ПЦР-продуктов проводили в 1.2%-ном агарозном геле с последующим окрашиванием бромидом этидия.
Секвенирование генов nha и nhb проводили с праймерами, используемыми в ПЦР, с применением набора реактивов Big Dye Terminator Cycle Sequencing Kit на автоматическом секвенаторе 3500XL Genetic Analyzer (Applied Biosystems, США), согласно производителю. Полученные последовательности идентифицировали с помощью программы BLAST и базы данных GenBank . Перевод нуклеотидных последовательностей в аминокислотные осуществляли в программе BLASTX. Выравнивание последовательностей производили при помощи программы YACWGUI 1.2. Построение дендрограмм производили с помощью программы Vector NTI 10 (Invitrogen, США).
Характер аминокислотных замен определяли по коэффициенту П. Снита (ср) [Sneath, 1966], показателю М. Волькенштейна (АН) [Волькенштейн, 1978], показателю А. Бачинского (ФБА) [Бачинский, 1976], физико-химической дистанции Р. Грэнтсема (GD) [Grantham, 1974] и ее модифицированным значениям (GDm), универсальному эволюционному индексу X. Танга (U) [Tang et al., 2004].
Статистическую обработку полученных данных проводили с использованием программы STATISTICA 5.0, используя тест Фишера (F-тест).
Результаты и их обсуждение
Проведена детекция генов железосодержащих нитрилгидратаз среди альдоксим- и нитрилутилизирующих микроорганизмов. Встречаемость кодирующих последовательностей фермента не зависела от субстрата, на котором были выделены бактерии (р = 0.73).
Обнаруженные гены nha (н = 28) и nhb (н = 26) у штаммов Rhodococcus, изолированных из географически различных почвенных сред, были сек-венированы. Размер прочитанных фрагментов а-субъединиц варьировал от 302 и. и. у штамма Rhodococcus sp. 84 до 630 п.н. у штамма Rhodococcus sp. ПО28, тогда как длина целого гена а-субъединицы составляет 624 п.н.. Все фрагменты имели высокое сходство с последовательностями нитрилгидратаз из базы данных GenBank (от 87 до 99%). Чаще всего такое сходство обна- руживалось с генами штаммов R. erythropolis ССМ2595 (JQO23O3O.1) и DCM13002 (AY223836). Также полученные прочтения оказались гомологичными генам железосодержащей нитрилгидратазы референтного штамма Rhodococcus sp. N-774 (от 84 до 96% гомологии). Длина [3-субъединицы у представителей рода Rhodococcus составляет 639 и. и. Секвенированные фрагменты этого гена находились в пределах от 389 п.н. у штамма R. erythropolis Б20-4 до 641 п.н. у Rhodococcus sp. 3213. Максимально идентичными (от 94 до 99%) по этой субъединице оказались штаммы R. erythropolis ССМ2595 (JQO23O3O.1), ARG-AN025 (AY223831.1) и ENG-AN033 (AY223832.1). Гомология с последовательностью штамма N-774 составила от 86 до 97%.
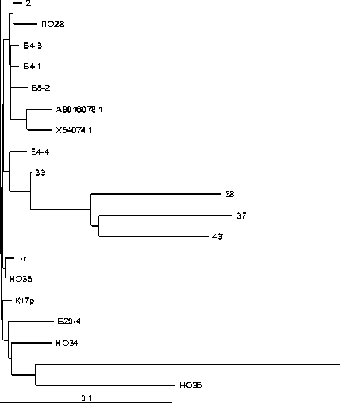
При переводе последовательности нуклеотидов в последовательность аминокислот оказалось, что и на этом уровне гомология исследуемых ферментов с известными ранее была очень высокой: 8999% для а-субъединицы и 87-99% - для 13-субъединицы. Более высокая консервативность последовательности а-субъединицы выявляется также при анализе дендрограмм, построенных для 21 изучаемой и 6 референтных последовательностей нитрилгидратазы (табл. 1). Здесь располагается CSLCSC-мотив, кодирующий активный центр фермента. Данный мотив идентичен у всех извест
Таблица 1
Аминокислотные замены в первичной структуре а- (А) и р-субъединиц (Б) нитрилгидратазы
|
А Штамм |
Размер фрагмента (а.о.) |
Аминокислотные замены (по отношению к Rhodococcus sp. N-774) |
|
2 |
182 |
I156V, I201V |
|
9-33 |
121 |
I156V, I201V |
|
9-37 |
137 |
I156V |
|
9-38 |
176 |
E69D, I156V, I190V, I201V |
|
9-43’ |
141 |
E69D, 120IV |
|
9-60 |
118 |
E69D, I156D, I201V |
|
3213 |
206 |
F48L, E69D, I156V, I201V |
|
1-г |
205 |
E69D, I156V, I201V |
|
П-г |
205 |
E69D, I156V, I190V, I201V |
|
Б4-1 |
207 |
I156V, I201V |
|
Б4-3 |
207 |
I156V, I201V |
|
Б4-4 |
203 |
E69D, Y85S, I156V, I201V |
|
Б5-2 |
207 |
Р125Н, I156V, I201V |
|
Б20-4 |
195 |
E69D, I156V, Q184R |
|
К17р |
205 |
E69D, I156V, I201V |
|
НО34 |
168 |
E69D, I156V, I201V |
|
НО35 |
167 |
E69D, I156V, I201V |
|
П1 |
207 |
E69D, I156V, I201V |
|
ПО27 |
206 |
E69D, I156V, I201V |
|
ПО28 |
206 |
E49Q, I156V, I201V |
|
ПО29 |
204 |
E69D, I156V, I201V |
JQD23DM.1
ных на сегодня нитрилгидратаз [Marron et al., 2012].
На сайте GenBank были взяты аминокислотные последовательности нитрилгидратазы разных штаммов родококков и проведено их выравнивание с помощью программы YACWGUI 1.2. относительно друг друга. Полученные аминокислотные последовательности почвенных изолятов содержали как характерные для рода Rhodococcus, так и уникальные замены аминокислотных остатков. Последовательность Nha содержит 3 типичных для родококков вариабельных участка: Glu 69A/Asp 69А, Vai 156А/11е 156А и Vai 201A/Ile 201А. Обнаружены уникальные замены, которые не представлены в базе данных GenBank: Туг 85 Ser у штамма Б4-4, Phe 48 Leu - 3213, Glu 49 Gin - ПО28, Pro 125 His- Б5-2, lie 190 Vai у штаммов П-г и 38.
Nhb характеризуется наличием 9 вариабельных участков: Thr 82В/А1а 82в, Glu 93B/Asp 93в, Pro 117B/Arg 117в, Glu 119B/Asp 119B, Vai 125вЛ1е 125в, Ser 154В/А1а 154в, Ser 158B/Thr 158в, Lys 183B/Thr 183в и Ala 185B/Asp 185в. Уникальные замены: Не 78 Vai у штамма Б5-2, Met 84 Lys и Vai 85 Phe у штаммов Б20-4 и НО35, Leu 98 Pro - ПО27, Arg 128 Lys -ПО28, His 155 Arg - Б4-1, lie 167 Vai - Б4-3, lie 167 Phe - ПО28, Ser 198 Gly - Б4-3.
Окончание табл. 1
|
Б Штамм |
Размер фрагмента (а.о.) |
Аминокислотные замены (по отношению к Rhodococcus sp. N-774) |
||||
|
2 |
202 |
D93E, V125I, T158S |
J |
0023030.1 213 ---33 —28 |
||
|
9-33 |
212 |
Т82А, D93E, P117R, E119D, S154A, К183Т |
||||
|
9-37 |
206 |
Т82А, D93E, P117R, E119D, S154A, К183Т |
||||
|
9-38 |
193 |
Т82А, D93E, P117R, E119D, S154A |
||||
|
9-43’ |
206 |
Т82А, D93E, P117R, E119D, S154A, К183Т, |
||||
|
9-60 |
175 |
Т82А, D93E, P117R, E119D, S154A, К183Т |
- |
|||
|
3213 |
212 |
Т82А, D93E, P117R, E119D, S154A, К183Т |
||||
|
1-г |
211 |
Т82А, D93E, P117R, VI181, E119D, S154A, К183Т |
---------------------ПО27 |
|||
|
— |
||||||
|
П-г |
197 |
Т82А, D93E, P117R, VI181, E119D, S154A, К183Т |
||||
|
Б4-1 |
173 |
V125I, H155R |
-------------------------------------ПЭ2Й — --------------------Б4-1 -----------------------------------------------------Б4-3 |
|||
|
Б4-3 |
173 |
V125I, S154P, H155G, R156K, T157G, I167V, S198G |
||||
|
Б4-4 |
209 |
D93E |
||||
|
Б5-2 |
154 |
I78V, K87L, G88T, D93E, V125I, T158S |
_ |
---------------XE4074.1 |
||
|
Б20-4 |
129 |
М84К, V85F, D93E, P117R, E119D, S154A |
||||
|
3-4 |
||||||
|
К17р |
173 |
D93E, V125I |
||||
|
НО34 |
173 |
Т82А, D93E, P117R, VI181, E119D, R156P, К183Т |
— |
-----------------------------------------HO 34 ---------------------HO35 |
||
|
НО35 |
153 |
М84К, V85F, D93E, P117R, VI181, E119D, R156P, К183Т |
||||
|
П1 |
211 |
Т82А, D93E, P117R, E119D, S154A, К183Т |
----------------------------Il-r -43 |
|||
|
ПО27 |
175 |
D93E, L98P, V125I, T158S |
1--------эд |
|||
|
ПО28 |
171 |
D93E, V125I, R128K, H155S, I167F |
||||
|
ПО29 |
175 |
D93E, V125I, R156P, T158S |
V. 1 |
|||
Примечание. Реконструированные последовательности сопоставлены с базой GenBank при помощи программы
BLAST. Полужирным начертанием выделены уникальные замены. Дендрограммы построены на основе сравнения аминокислотных последовательностей а- и [3- субъединиц нитрилгидратазы. Масштаб -0.1 замена на сайт.
Аминокислотные замены могут быть локализованы не в активном центре, а в соседних регионах. Так, Nakasako et al. показали, что штамм Rhodococcus sp. ACV2 содержит единичную замену (Vai 40 Met), расположенную на участке 13-субъединицы, формирующем входной канал к активному центру фермента. Активность такой мутантной нитрилгидратазы отличалась от активности фермента дикого типа по оптимуму pH и скорости гидролиза циановалерамида и циановалериановой кислоты в 30 и 15 раз соответственно
[Nakasako et al., 1999]. Piersma et al. [2000] путем сайт-направленного мутагенеза получили штамм Rhodococcus sp. N-771 (Arg56B), фермент которого показал ограниченную стабильность и более низкую активность по сравнению со штаммом дикого типа. Данная аминокислота является консервативной для большинства известных нитрилгидратаз. Она необходима для каталитической активности, поскольку вовлечена не только в связывание субстрата, но также взаимодействует с железом в активном центре фермента [Piersma et al., 2000].
При сравнении встречаемости аминокислотных замен среди штаммов, выделенных из природных и антропогенно-измененных почв, статистически-значимой разницы не выявлено (р=0.717 по F-тесту). Однако чаще замены встречались среди штаммов, выделенных из загрязненных почвенных образцов (66.7% для а-субъединицы и 70.0% - для (3-субъединицы).
Учитывая, что при молекулярном скрининге а- и (3-субъединиц нитрилгидратазы были обнаружены уникальные аминокислотные замены, представлялось интересным установить степень их консервативности и радикальности. Консервативной заменой аминокислоты называется мутация, не приводящая к значительным изменениям структуры и функции белка. В процессе эволюции консервативные замены аминокислот происходят чаще, чем радикальные. Эти замены преимущественно встречаются в функционально важных участках белковой молекулы (например, сайтах связывания лигандов). Радикальные замены аминокислот, напротив, существенно меняют структуру и функции белка [Бутвиловский, Черноус, 2008].
Для определения характера уникальных аминокислотных замен в 1966 г. предложен метод Снита. В его основе лежит анализ всех доступных для того времени физико-химических свойств аминокислот: наличие/отсутствие -ОН групп, растворимость L-изомеров, наличие бензольного кольца, свободные электроны и т.д. Поскольку достаточно сложно установить степень влияния каждого эффекта на активность белка, априори был принят равный вклад каждого эффекта в изменение биологической активности. Одним из свойств, определяющих конформацию белка, является гидрофобность составляющих его аминокислот. В 1970-е гг. Волькенштейн показал, что наиболее многочисленны и предпочтительны замены с минимальными изменениями гидрофобности аминокислотного остатка. Для оценки степени взаимоза меняемости аминокислот в 1976 г. А. Бачинский предложил показатель «функциональной близости аминокислот» (ФБА). Под ФБА подразумевается способность аминокислот заменять друг друга в белках с полным или частичным сохранением их активности. Величины ФБА были получены на основе анализа числа замен аминокислот в 28 семействах изофунк-циональных белков. Согласно представлениям Грэн-тсема, частота аминокислотных замен зависит от физико-химических свойств их боковых радикалов. Формула расчета физико-химической дистанции учитывает три параметра аминокислотных остатков: полярность, объем и состав. Для удобства анализа М. Джонсон предложил использовать относительные (модифицированные) значения физико-химической дистанции. Аминокислотная замена считается консервативной при ф □ 0.416, ДН< 1.28, ФБА 012.4, GD < 100 и GDM □ 57.9.
В 2004 г. X. Танг предложил новый метод определения характера аминокислотной замены -вычисление универсального эволюционного индекса (U). Он был разработан на основании анализа белок-кодирующих участков 4 383 генов, т.е. универсальный эволюционный индекс является эмпирической системой классификации аминокислотных замен по степени их эволюционной изменчивости. Чем больше значение U, тем чаще происходят взаимные замены аминокислот данной пары (индекс варьирует в пределах от 0.241 до 2.49) [Бутвиловский, Черноус, 2008].
При анализе уникальных аминокислотных замен, обнаруженных среди штаммов рода Rhodococcus, оказалось, что большинство из них являются консервативными. Замены Pro 125 His, Met 84 Lys, Vai 85 Phe, Leu 98 Pro, His 155 Arg определяются как радикальные по двум показателям из пяти, а замена Не 167 Phe - только по показателю функциональной близости аминокислот Бачинского (табл. 2).
Таблица 2
Определение характера уникальных аминокислотных замен в белок-кодирующей последовательности нитрилгидратазы
|
F48L |
Phe 48 Leu |
0.570 |
0.23 |
16 |
22 |
86 |
0.732 |
|
E49Q |
Glu 49 Gin |
0.685 |
0.45 |
25 |
29 |
86 |
1.634 |
|
¥85 S |
Туг 85 Ser |
0.354 |
2.83 |
7 |
144 |
59 |
0.503 |
|
Р 125 Н |
Pro 125 His |
0.172 |
1.20 |
3 |
70 |
64 |
0.784 |
|
I 190 V |
lie 190 Vai |
0.843 |
1.28 |
35 |
29 |
86 |
2.415 |
(3-субъединица
|
178 V |
lie 78 Vai |
0.843 |
1.28 |
35 |
29 |
86 |
2.415 |
|
M84K |
Met 84 Lys |
0.482 |
0.20 |
5 |
95 |
54 |
0.559 |
|
V85F |
Vai 85 Phe |
0.380 |
0.96 |
8 |
50 |
77 |
0.548 |
|
L98P |
Leu 98 Pro |
0.432 |
0.18 |
4 |
98 |
54 |
0.388 |
|
R 128 К |
Arg 128 Lys |
0.733 |
0.77 |
25 |
26 |
86 |
1.583 |
|
H 155 R |
His 155 Arg |
0.396 |
0.67 |
7 |
29 |
86 |
0.784 |
Окончание табл. 2
|
Аминокислотные замены |
Показатели |
||||||
|
Ф |
AH |
ФБА |
GD |
GDM |
U |
||
|
R 156 К |
Arg 156 Pro |
0.733 |
0.77 |
25 |
26 |
86 |
1.583 |
|
I 167 V |
lie 167 Vai |
0.843 |
1.28 |
35 |
29 |
86 |
2.415 |
|
I 167 F |
lie 167 Phe |
0.487 |
0.32 |
10 |
21 |
86 |
0.545 |
|
S 198G |
Ser 198 Gly |
0.323 |
0.04 |
25 |
56 |
73 |
1.360 |
Примечание. <р - коэффициент Спита, АН - показатель Волькенштейна, ФБА - показатель Бачинского, GD -физико-химическая дистанция Грэнтсема, GDm - относительная физико-химическая дистанция Грэнтсема, U - универсальный эволюционный индекс Танга. Полужирным начертанием выделены радикальные замены аминокислот.
Обнаруженная у штамма R. erythropolis Б4-4 мутация в 254 положении а-субъединицы нитрилгидратазы является трансверсией (С—>А) и наблюдается во втором положении кодона (ТАС—>ТСС) (рисунок). Данная замена несинонимична, так как приводит к изменению кодируемой аминокислоты с тирозина на серин в 85 сайте, и определяется как радикальная по всем используемым показателям, за исключением GDM. Универсальный эволюционный индекс Танга U=0.503 отражает редкость такой замены.
-
(1) 61 CTTCGAGGAGGACTTCAGTCCAAGGCGCGGAGCGGAATTGGTCGCGCGGGCTTGGACCGA 120
-
(2) 141 CTTCGAGGAGGACTTCAGTCCAAGGCGCGGAGCGGAATTGGTCGCGCGGGCTTGGACCGA 200
-
(1) 121 ccccgatttccggcaactgcttctcaccgacggtaccgccgcggttgcccagtCcggata 180
11 I I I I I I I I I 1111111 I I I I I I I I I I 111 Н 11 I I I I I I I I I 1111111 I I HI I I I
-
(2) 201 ccccgatttccggcaactgcttctcaccgacggtaccgccgcggttgcccagtAcggata 260
-
(1) 181 tctgggcccccagggcgaatacatcgtggcagtcgaagacaccccgaccctcaagaacgt 240
I III I I I I I I I I I I I I I! I I I I I I I I I I I I IHI I I I I I I I I I I I I I I HI I I HI I I 1 I
-
(2) 261 TCTGGGCCCCCAGGGCGAATACATCGTGGCAGTCGAAGACACCCCGACCCTCAAGAACGT 320
Участок выровненной секвенированной последовательности гена, кодирующего а-субъединицу нитрилгидратазы:
1 - полученный сиквенс nha штамма R. erythropolis Б4-4, 2 - участок nha штамма R. erythropolis ARG-AN025, AY223831.1
Заключение
Нитрилгидратазы родококков характеризуются высокой гомологией первичной структуры, однако их активность и субстратная специфичность могут существенно отличаться даже при наличии точечных мутаций. Анализ секвенированных генов железосодержащих нитрилгидратаз почвенных штаммов рода Rhodococcus не выявил различий в областях, кодирующих активные или регуляторные центры ферментов. Однако в первичной структуре белков обнаружено множество как характерных, так и уникальных для данного рода аминокислотных замен, что также может повлиять на активность ферментов. Несмотря на то, что встречаемость аминокислотных замен в субъединицах нитрилгидратаз не различалась у штаммов, выделенных из природных и антропогенно-измененных почв, тем не менее, у последних выявлена большая гетерогенность фермента. Единственная замена, определенная нами как радикальная, обнаружена в последовательности а-субъединицы нитрилгидра тазы штамма R. erythropolis Б4-4, изолированного из почвы предприятия по производству акриламида.
Список литературы Характеристика железосодержащих нитрилгидратаз почвенных бактерий рода Rhodococcus
- Бачинский А.Г. Структура и помехоустойчивость генетического кода//Общая биология. 1976. Т. 37. С. 163-173
- Бутвиловский А.В., Черноус Е.А. Методы определения характера аминокислотных замен: учеб.метод. пособие. Минск: БГМУ, 2008. 28 с
- Волькенштейн М. Общая биофизика. М.: Наука, 1978. 590 с
- Гловер Д. Клонирование ДНК. Методы. М.: Мир, 1988. 538 с
- Asano Y., Tani Y., Yamada H. A new enzyme «nitrile hydratase» which degrade acetonitrile in combination with amidase//Agricultural and biological chemistry. 1980. Vol. 44. P. 22512252
- Brandao P.F.B., Clapp J.P., Bull A.T. Discrimination and taxonomy of geographically diverse strains of nitrile-metabolising actinomycetes using chemometric and molecular sequencing techniques//Environmental Microbiology. 2002. Vol. 4. P. 262-276
- Brandao P.F.B., Clapp J.P., Bull A.T. Diversity of nitrile hydratase and amidase enzyme genes in Rhodococcus erythropolis recovered from geographically distinct habitats//Applied and Environmental Microbiology. 2003. Vol. 69, № 10. P. 5754-5766
- Grantham R. Amino acid difference formula to help explain protein evolution//Science. 1974. Vol. 185. P. 862-864
- Holtze M.S. et al. Microbial degradation of the ben-zonitrile herbicides dichlobenil, bromoxynil and ioxynil in soil and subsurface environments-insights into degradation pathways, persistent metabolites and involved degrader organism//Environmental Pollution. 2008. Vol. 154, № 2. P. 155-158
- Marron A.O., Akam M., Walk G. Nitrile hydratase genes are present in multiple eukaryotic supergroups//PLoS ONE. 2012. Vol. 7. P. 1-10
- Mukram I. et al. Isolation and identification of a ni-trile hydrolyzing bacterium and simultaneous utilization of aromatic and aliphatic nitriles//International Biodeterioration and Biodegradation. 2015. Vol. 100. P. 165-171
- Nakasako M. et al. Tertiary and quaternary structures of photoreactive Fe-type nitrile hydratase from Rhodococcus sp. N-771: roles of hydration water molecules in stabilizing the structures and the structural origin of the substrate specificity of the enzyme//Biochemistry. 1999. Vol. 38. P. 9887-9898
- Piersma S.R. et al. Arginine 56 mutation in the beta subunit of nitrile hydratase: importance of hydrogen bonding to the non-heme iron center//Journal of Inorganic Biochemistry. 2000. Vol. 80. P. 283-288
- Prasad S., Bhalla T.C. Nitrile hydratases (NHases): At the interface of academia and industry//Biotechnology Advances. 2010. Vol. 28. P. 725-741
- Sneath P.H.A. Relations between chemical structure and biological activity in peptides//Journal of Theoretical Biology. 1966. Vol. 12. P. 157-195
- Tang H. et al. Universal evolutionary index for amino acid changes//Molecular Biology and Evolution. 2004. Vol. 21. P. 1548-1556
- Verma V., Sangave P. Screening, isolation and identification of nitrilase producing bacteria from soil//International Journal of Pharmaceutical Sciences and Research. 2015. Vol. 6. P. 19501957
- Yamada H., Kobayashi M. Nitrile hydratase and its application to industrial production of acrylamide//Bioscience Biotechnology and Biochemistry. 1996. Vol. 60. Р. 1391-1400


