Идентификация SNPS для показателей роста и развития коз (Capra hircus Linnaeus, 1758) из ресурсной популяции в возрастной динамике
Автор: Сермягин А.А., Денискова Т.Е., Гусев И.В., Петров С.Н., Родионов А.Н., Доцев А.В., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 4 т.59, 2024 года.
Бесплатный доступ
Поиск генетических вариантов, влияющих на рост и развитие коз, актуален для создания специализированной мясной породы и повышения продуктивности животных. В настоящей работе впервые идентифицированы 34 потенциальных QTL, сопряженных с генами, ассоциированными с параметрами роста и развития, мясной продуктивностью и воспроизводительными качествами, адаптационными способностями, развитием волосяных фолликулов, молочной продуктивностью и доместикацией у коз из группы возвратных кроссов ресурсной популяции, полученных при скрещивании местных карачаевских коз, гибридных по дикому туру, c производителями калахарской породы. Цель исследований - идентифицировать гены-кандидаты, ассоциированные с хозяйственно полезными признаками в специально созданной ресурсной популяции коз (Capra hircus ). Исследования проводили в 2021-2023 годах в ФГБНУ Федеральный исследовательский центр животноводства - ВИЖ им. академика Л.К. Эрнста. Объектом были кроссы животных из специально созданной ресурсной популяции коз (n = 237), в том числе 109 козликов и 128 козочек. Кроссы были получены при скрещивании местных карачаевских коз, генотип которых несет до 25 % генов дикого тура (3/4 карачаевская коза × 1/4 дикий тур) и калахарских козлов-производителей красной масти (порода Kalahari Red). При создании базы фенотипов учитывали параметры роста и развития молодняка коз, которые соответствовали среднему возрасту в 10, 110, 190 и 390 сут, что соизмеримо с учетом величин при рождении, в 3-4 мес, 6 и 12 мес. Фиксировали следующие параметры роста и развития: живая масса, высота в холке, высота в спине, высота в крестце, высота в маклоках, высота в седалищных буграх, косая длина туловища, обхват груди, обхват пясти, ширина груди в плечах, глубина груди, ширина в маклоках, ширина в седалищных буграх, длина головы. Генотипирование экспериментальных животных проводили на основе SNP-панелей средней плотности ДНК-чипа Caprine SNP 50K BeadChip (59727 точечных мутаций) («Illumina, Inc.», США). GWAS анализ выполняли по стандартной линейной регрессионной модели, реализованной в программе Plink 1.9, с учетом популяционной структуры изучаемой выборки коз. Аннотацию генов осуществляли с использованием международной базы данных NCBI по сборке генома ARS1.2 Capra hircus (goat). Всего было идентифицировано 1036 генов с входящими в них или находящимися вблизи ±0,2 Mb SNPs, сопряженными с изменчивостью фенотипических оценок по показателям экстерьера и живой массы молодняка коз из ресурсной популяции. Из них 341 полиморфизм был определен непосредственно в 326 генах на 1-29-й хромосомах. Для детального анализа выявленных в результате GWAS генов, в части их биологической функции, посредством генной онтологии проводили дальнейшее исследование с привлечением международной базы данных генетической информации DAVID. Наибольшее число выявленных ассоциаций (пределы значимости от p function show_abstract() { $('#abstract1').hide(); $('#abstract2').show(); $('#abstract_expand').hide(); }
Capra hircus, козы, gwas, snp, qtl, гены-кандидаты, ресурсная популяция
Короткий адрес: https://sciup.org/142243759
IDR: 142243759 | УДК: 636.39:575.162 | DOI: 10.15389/agrobiology.2024.4.633rus
Текст научной статьи Идентификация SNPS для показателей роста и развития коз (Capra hircus Linnaeus, 1758) из ресурсной популяции в возрастной динамике
При выполнении исследований было использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста. Работа выполнена при поддержке Российского научного фонда, проект ¹ 21-66-00007.
Важные условия успешного развития отечественного животноводства — это, во-первых, достаточное генетическое разнообразие в популяциях сельскохозяйственных животных, во-вторых, наличие выдающихся генотипов и адаптированных к конкретным условиям разведения особей. К сожалению, стремительное сужение и обеднение генофонда практически всех видов культурных животных стало следствием глобализации, которая коснулась и селекционно-племенной работы. В этой связи в российском животноводстве стоит задача надлежащим образом использовать имеющийся генофонд для увеличения производства, повышения его эффективности и расширения ассортимента продукции.
В России созданы уникальные культурные породы коз пухового и шерстного направлений. Иностранные породы прочно заняли нишу молочного направления. Однако нет специализированной мясной породы коз, а в качестве источника козлятины используются местные грубошерстные популяции, не имеющие племенной базы (1). В связи с этим поиск генетических вариантов, вовлеченных в регуляцию процессов роста и развития молодняка коз, весьма актуален для увеличения производства качественной козлятины как источника дешевых полноценных диетических продуктов питания, и для создания специализированной мясной породы на основе поголовья местных коз локальной популяции, происходящих из горной местности Карачаево-Черкесской Республики.
В козоводстве успешно реализуется поиск полногеномных ассоциаций с признаками, имеющими экономическую значимость. Так, L. Zhang с соавт. (2) применили GWAS анализ (genome-wide association study) для выявления генов, связанных с живой массой при рождении, отъеме и в возрасте 1 года у кашемировых коз, которых разводят на территории Внутренней Монголии. Установлен 21 статистически значимый однонуклеотидный полиморфизм (single nucleotide polymorphism, SNP), расположенный в 10 генах, в том числе MAPK3 , LDB2 и LRP1B (2). M. Selionova с соавт. (3) на основе GWAS, проведенного для карачаевских козлят в возрасте 4 и 8 мес, выявили 58 общих статистически значимых SNPs: 11 SNPs — для массы тела, 4 SNPs — для группы признаков высота в холке, высота крупа, длина тела, 2 SNPs — для высоты в холке и высоты крупа, 1 SNP — для длины тела и глубины груди. Эти SNPs пересекались с генами, регулирующими транскрипцию, клеточную пролиферацию, ангиогенез, рост тела, метаболизм жирных кислот и липидов, развитие нервной системы и сперматогенез (3). Тем же авторским коллективом впервые проведен поиск геномных ассоциаций с составом молока местных карачаевских коз, в результате которого выявлены 8 генов, влияющих на содержание сухого вещества и жирных кислот ( METTL , SLC1A8 , PHACTR1 , FMO2 , ECI1 , PGP , ABCA3 , AMDHD2 ) (4). Выявлены SNPs, связанные с числом козлят на одно козление (snp54094-scaffold824-899720 на CHI11) и влияющие на цвет шерстного покрова (snp11508-scaffold142-1990450 в гене SORCS3 на CHI26 и snp55048-scaffold842-324525 в гене LOC10218777 на CHI12) в китайской юго-западной популяции коз породы Youzhou (5).
В наших более ранних работах показана эффективность использования массива животных, происходящих из специально созданных ресурсных популяций, в качестве объектов для проведения GWAS. Так, идентифицированы SNPs и гены-кандидаты, достоверно ассоциированные с живой массой (6) и промерами туловища (7) у возвратных кроссов овец из ресурсной популяции, созданной посредством серии скрещиваний баранов породы ка-тадин и овцематок романовской породы.
В настоящей работе впервые идентифицированы 34 потенциальных
QTL, сопряженных с генами, ассоциированными с параметрами роста и развития, мясной продуктивностью и воспроизводительными качествами, адаптационными способностями, развитием волосяных фолликулов, молочной продуктивностью и доместикацией у коз из группы возвратных кроссов в ресурсной популяции. Ресурсная популяция была получена при скрещивании местных карачаевских коз, гибридных по дикому туру, с производителями калахарской породы.
Цель исследований — идентифицировать гены-кандидаты, ассоциированные с хозяйственно полезными признаками в ресурсной популяции особей Capra hircus , которая была получена при скрещивании карачаевских коз, созданных методами народной селекции, со специализированной породной группой калахарских козлов-производителей красной масти мясного высокоинтенсивного направления продуктивности.
Методика. Исследования проводили на кроссах животных из специально созданной ресурсной популяции коз ( n = 237, 109 козликов и 128 козочек; 2021-2023 годы). Кроссы были получены при скрещивании местных карачаевских коз, генотип которых несет до 25 % генов дикого тура (3/ 4 карачаевская коза ½ 1/ 4 дикий тур) и калахарских козлов-производителей (порода Kalahari Red). Ресурсная популяция коз создана и поддерживается на базе филиала ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста — племенного завода «Ладожский» (Усть-Лабинский р-н, Краснодарский край).
При создании базы фенотипов учитывали параметры роста и развития молодняка коз, которые соответствовали среднему возрасту в 10, 110, 190 и 390 сут, что соизмеримо с учетом величин при рождении, в 3-4 мес, 6 и 12 мес (возрастные периоды выбраны в связи с продолжительным коз-лением маток с марта по май 2022 года). Фиксировали следующие параметры роста и развития: живая масса (ЖМ), высота в холке (ВХ), высота в спине (ВС), высота в крестце (ВК), высота в маклоках (ВМ), высота в седалищных буграх (ВСБ), косая длина туловища (КДТ), обхват груди (ОГ), обхват пясти (ОП), ширина груди в плечах (ШГ), глубина груди (ГГ), ширина в маклоках (ШМ), ширина в седалищных буграх (ШCБ), длина головы (ДГ).
Генотипирование экспериментальных животных проводили с использованием SNP-панелей средней плотности ДНК-чипа Caprine SNP 50K BeadChip (59727 точечных мутаций) («Illumina, Inc.», США). Качество генотипирования (call rate) составило от 97,9 до 98,3 %, или 100 % результативности генотипирования всех образцов с порогом выше 95,0 %. После контроля качества для последующего анализа осталось 51167 SNPs при условии наличия не менее 90 % результативности генотипирования по выборке для каждого из индивидуумов, частоте минорного аллеля 5 % и соответствия критерию по Харди-Вайнбергу 1½10 - 6.
GWAS анализ проводили по стандартной линейной регрессионной модели, реализованной в программе Plink 1.9 (8), с учетом популяционной структуры изучаемой выборки коз (учитывали ассоциации не ниже порога значимости p < 0,0001-0,0002). Аннотацию генов осуществляли с привлечением международной базы данных NCBI по сборке генома ARS1.2 Capra hircus (goat) .
Для определения биологической функции выявленных в результате GWAS генов на основе генной онтологии использовали международную базу данных генетической информации DAVID (9), анализировали только те полиморфизмы, которые находились внутри генов. Для описания выбирали только гены, которые несли максимальное число биологических функций — 38.
Статистическую обработку осуществляли стандартными биомет- рическими методами, рассчитывали средние значения (M), стандартные ошибки средних (±SEM) и фенотипические коэффициенты вариации (Cv, %) с использованием встроенного пакета Анализ данных (Microsoft Excel 2013).
Результаты. Между козликами и козочками отмечались различия по фенотипическим показателям роста и развития (табл. 1). Первые характеризовались большей живой массой при рождении и в динамике по возрастам, а также большей высотой, шириной и глубиной тела. Наибольшая изменчивость показателей наблюдалась в 10-суточном возрасте (6,0-12,9 % для козликов и 7,0-14,4 % для козочек) при плавном ее снижении к годовалому возрасту (соответственно 4,1-14,5 и 4,1-12,6 %). Живая масса козликов (валушков — кастрированных самцов) и козочек на 390-е сут достигала соответственно 26,2 и 25,8 кг. Все параметры оценки экстерьера имели нормальное распределение и были использованы при поиске полногеномных ассоциаций показателей роста и развития у молодняка коз.
-
1. Фенотипические параметры роста и развития коз ( Capra hircus L.) в группе возвратных кроссов из ресурсной популяции, полученной при скрещивании местных гибридных карачаевских коз с производителями калахарской породы (ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста — филиал племенной завод «Ладожский», Усть-Лабинский р-н, Краснодарский край, 2021-2023 годы)
-
2. Результаты аннотации выявленных при GWAS анализе генов с наибольшим числом биологических функций, сопряженных с ростом и развитием молодняка коз ( Capra hircus L.) в группе возвратных кроссов из ресурсной популяции, полученной при скрещивании местных гибридных карачаевских коз с производителями калахарской породы ( n = 237, ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста — филиал племенной завод «Ладожский», Усть-Ла-бинский р-н, Краснодарский край, 2021-2023 годы)
Ген
CHI
Число и описание основных биологических функций по генной онтологии (GO)
Признак
p
ADAM10
10
19 GO, в том числе: GO:0001701 — внутриутробное эмбриональное развитие, GO:0008284 — положительная регуляция пролиферации клеток, GO:1901342 — регуляция развития сосудистой сети
КДТ 10
ОГ 10
ГГ 10
ШСБ 10
ЖМ 10
7,05E - 05
1,34E - 05
2,47E - 05
3,49E - 05
2,62E-05
ADAMTS12
20
16 GO, в том числе: GO:0002062 — дифференцировка хондроцитов, GO:0030199 — организация коллагеновых фибрилл, GO:0043931 — оссификация, участвующая в созревании костей
ГГ 110
1,31E - 04
ARL8B
22
14 GO, в том числе: GO:0007059 — сегрегация хромосом, GO:0002747 — процессинг и презентация антигена после фагоцитоза, GO:0015031 — транспорт белка
КДТ 110
ОГ 110
ЖМ 110
9,40E - 06
1,79E - 05
9,15E - 05
AKT3
16
11 GO, в том числе: GO:0000002 — поддержание митохондриального генома, GO:0045793 — положительная регуляция размера клеток, GO:0045766 — положительная регуляция ангиогенеза
ОГ 390
1,23E - 04
ABCA1
8
24 GO, в том числе: GO:0002790 — секреция пептидов, GO:0006497 — липидизация белков, GO:0008203 — метаболический процесс по холестерину
ВХ 10
ВС 10
ВК 10
ВМ 10
ВСБ 10
КДТ 10
ОГ 10
ШГП 10
ГГ 10
ШМ 10
ШСБ 10
ЖМ 10
6,49E - 07
3,99E - 07
4,31E - 07
6,48E - 07
2,56E - 06
1,61E - 07
2,91E - 06
2,50E - 06
5,76E - 06
8,91E - 07
1,48E - 05
6,98E - 08
BANK1
6
8 GO, в том числе: GO:0009617 — ответ на бактериальную инфекцию, GO:0050869 — негативная регуляция активации B-клеток, GO:0051898 — негативная регуляция передачи сигналов протеинкиназы B
ШГП 390
2,38E - 04
CDK5RAP2
8
12 GO, в том числе: GO:0000132 — установление ориентации митотического веретена, GO:0000226 — организация цитоскелета микротрубочек, GO:0007420 — развитие мозга
ЖМ 10
3,22E - 05
FBXW7
17
21 GO, в том числе: GO:0045741 — положительная регуляция активности рецептора, активируемого эпидермальным фактором роста, GO:0016567 — убиквитинирование белка, GO:0050821 — стабилизация белка
ОГ 10
ШГП 10
ЖМ 10
1,79E - 05
6,58E - 05
2,64E - 05
LRP2
2
25 GO, в том числе: GO:0001822 — развитие почек, GO:0008283 — пролиферация клеток, GO:0015031 — транспорт белка
ВХ 110
ВС 110
ВК 110
ВМ 110
6,84E - 05
1,01Е - 04
9,38E - 05
9,75E - 05
Продолжение таблицы 2
MYCBP2
12 11 GO, в том числе: GO:0016567 — убиквити- ДГ 390 1,38E - 04
нирование белка, GO:0019222 — регуляция ме
таболического процесса, GO:0050905 — нейро
мышечный процесс
RBFOX2
5 9 GO, в том числе: GO:0010724 — регуляция ОГ 390 1,15E - 04
окончательной дифференцировки эритроци
тов, GO:0030520 — сигнальный путь внутриклеточного рецептора эстрогена, GO:0043484 — регуляция сплайсинга РНК
UVRAG
15 15 GO, в том числе: GO:0006468 — фосфори- (SNP в позиции 27634379 п.н.)
лирование белка, GO:0032465 — регуляция ци- КДТ 10 2,07E - 05
токинеза, GO:0071900 — регуляция активности ОГ 10 1,73E - 05
протеинсерин/треонин киназы ШГП 10 8,04E - 06
ГГ 10 2,84E - 06
ЖМ 10 1,97E - 05
(SNP в позиции 27670405 п.н.)
ВС 10 1,02E - 04
КДТ 10 9,76E - 05
ОГ10 4,98E - 05
ЖМ10 1,21E - 05
WNT3A
7 54 GO, в том числе: GO:0001701 — внутри- ОП 390 8,82E - 05
утробное эмбриональное развитие,
GO:0035556 — передача внутриклеточного сиг
нала, GO:0010468 — регуляция экспрессии ге
нов, GO:0030879 — развитие молочной железы
YAP1
15 49 GO, в том числе: GO:0000902 — клеточный ШМ 390 1,63E - 04
морфогенез, GO:0008283 — пролиферация
клеток, GO:0030307 — положительная регуля
ция роста клеток
ACACB
17 12 GO, в том числе: GO:0010884 — положи- ШГП 10 6,66E - 05
тельная регуляция накопления липидов, ГГ 10 2,96E - 06
GO:0010906 — регуляция метаболического ЖМ 10 9,81E - 05
процесса глюкозы, GO:0031667 — реакция на уровень питательных веществ
ANO6
5 16 GO, в том числе: GO:0007596 — свертыва- КДТ 10 3,78E - 05
ние крови, GO:0030501 — положительная ре- ШГП 10 4,71E - 05
гуляция минерализации костей, GO:0035725 — ШМ 10 3,70E - 05
трансмембранный транспорт ионов натрия ЖМ 10 1,11E - 05
AGO2
14 21 GO, в том числе: GO:0045766 — положи- ДГ 390 2,58E - 04
тельная регуляция ангиогенеза, GO:1901165 — положительная регуляция миграции клеток трофобласта, GO:1900153 — положительная регуляция катаболического процесса ядерно-транскрибируемой мРНК, зависимый от деаденилирования распад
CDH5
18 23 GO, в том числе: GO:0001932 — регуляция ШМ 10 7,55E - 05
фосфорилирования белков, GO:0006874 — клеточный гомеостаз ионов кальция, GO:0045766 — положительная регуляция ангиогенеза
CACNA1C
5 10 GO, в том числе: GO:0002520 — развитие ШГП 110 7,85E - 05
иммунной системы, GO:0007507 — развитие ШМ 110 3,38E - 05
сердца, GO:0035115 — эмбриональный морфо- ВХ 190 8,70E - 06
генез передних конечностей ВС 190 7,80E - 06
ВК 190 1,60E - 05
ВМ 190 1,60E - 05
ГГ 190 9,97E - 05
ДГ 190 1,49E - 05
CARMIL1
23 12 GO, в том числе: GO:0016477 — миграция ОГ 10 4,16E - 05
клеток, GO:0030335 — положительная регуля- ГГ 10 3,83E - 05
ция миграции клеток, GO:0051639 — формирование сети актиновых нитей
DNM1L
5 24 GO, в том числе: GO:0006816 — транспорт ШГП 10 5,26E - 05
ионов кальция, GO:0010468 — регуляция экс
прессии генов, GO:0050714 — положительная
регуляция секреции белка
EDNRA
17 66 GO, в том числе: GO:0001701 — внутри- (SNP в позиции 60223612 п.н.)
утробное эмбриональное развитие, КДТ 10 1,15E - 05
GO:0006468 — фосфорилирование белков, ШМ 10 6,05E - 05
GO:0006874 — клеточный гомеостаз ионов ЖМ 10 2,08E - 06
кальция (SNP в позиции 60280444 п.н.)
КДТ 10 1,15E - 05
|
Показатель |
Возраст, сут |
|||||||
|
10 |
110 |
190 |
390 |
|||||
|
M ±SEM \ |
Cv |
M ±SEM \ |
Cv |
M ±SEM \ |
Cv |
M ±SEM 1 |
Cv |
|
|
ЖМ |
5,5±0,2 |
Козлики (вал 19,7 14,0±0,6 |
ушки) ( n = 109) 25,8 15,7±0,5 |
19,3 |
26,2±0,7 |
17,8 |
||
|
ВХ |
39,0±0,4 |
6,3 |
50,9±0,6 |
7,5 |
53,4±0,5 |
5,9 |
61,9±0,4 |
4,1 |
|
ВС |
39,1±0,4 |
6,0 |
50,9±0,6 |
7,5 |
53,4±0,5 |
5,9 |
61,7±0,4 |
4,2 |
|
ВК |
39,3±0,4 |
6,1 |
50,9±0,6 |
7,5 |
53,4±0,5 |
5,9 |
61,9±0,4 |
4,2 |
|
ВМ |
39,1±0,4 |
6,3 |
50,9±0,6 |
7,5 |
53,4±0,5 |
5,9 |
62,0±0,4 |
4,3 |
|
ВСБ |
34,0±0,4 |
7,0 |
43,1±0,5 |
7,3 |
45,7±0,4 |
6,0 |
52,9±0,4 |
5,4 |
|
КДТ |
37,6±0,4 |
6,0 |
53,7±0,8 |
9,3 |
56,3±0,6 |
7,5 |
67,2±0,8 |
7,3 |
|
ОГ |
40,0±0,5 |
8,0 |
54,3±0,8 |
8,8 |
56,7±0,6 |
6,5 |
70,8±0,7 |
6,4 |
|
ОП |
6,3±0,1 |
6,2 |
6,7±0,1 |
8,2 |
6,4±0,1 |
8,8 |
7,7±0,1 |
7,9 |
|
ШГ |
9,2±0,1 |
9,0 |
13,5±0,3 |
14,9 |
13,5±0,2 |
10,7 |
15,3±0,3 |
14,5 |
|
ГГ |
13,8±0,2 |
7,8 |
20,2±0,3 |
10,4 |
21,0±0,2 |
6,8 |
26,4±0,2 |
5,1 |
|
ШМ |
6,8±0,1 |
10,2 |
9,0±0,2 |
10,7 |
9,6±0,1 |
9,3 |
11,2±0,3 |
16,7 |
|
ШCБ |
5,3±0,1 |
12,9 |
6,2±0,1 |
10,7 |
6,6±0,1 |
7,5 |
8,0±0,1 |
9,6 |
|
ДГ |
17,0±0,2 |
8,1 |
19,8±0,3 |
8,7 |
22,4±0,2 |
6,4 |
28,5±0,4 |
9,0 |
|
ЖМ |
4,9±0,1 |
20,0 |
Козочки 13,4±0,6 |
( n = 128) 28,3 13,5±0,5 |
21,7 |
25,8±0,6 |
18,1 |
|
|
ВХ |
36,7±0,4 |
8,0 |
49,5±0,8 |
9,3 |
50,3±0,6 |
6,6 |
60,9±0,3 |
4,1 |
|
ВС |
36,8±0,4 |
7,3 |
49,5±0,8 |
9,5 |
50,4±0,6 |
6,7 |
60,8±0,3 |
4,1 |
|
ВК |
37,1±0,4 |
7,0 |
49,6±0,7 |
9,1 |
50,4±0,6 |
6,7 |
60,9±0,4 |
4,3 |
|
ВМ |
36,6±0,4 |
7,6 |
49,5±0,7 |
9,0 |
50,4±0,6 |
6,7 |
60,9±0,4 |
4,4 |
|
ВСБ |
32,2±0,4 |
8,3 |
41,9±0,6 |
9,4 |
44,1±0,6 |
7,9 |
51,9±0,4 |
5,8 |
|
КДТ |
37,2±0,5 |
8,8 |
52,6±1,1 |
12,8 |
53,1±0,9 |
10,4 |
67,9±0,6 |
7,0 |
|
ОГ |
38,9±0,4 |
7,6 |
54,8±1,2 |
13,8 |
54,6±0,6 |
7,0 |
69,2±0,6 |
6,5 |
|
ОП |
6,0±0,1 |
7,7 |
6,4±0,1 |
10,6 |
6,0±0,1 |
7,3 |
7,4±0,1 |
6,0 |
|
ШГ |
9,3±0,2 |
16,3 |
13,0±0,2 |
11,6 |
12,3±0,3 |
15,1 |
15,3±0,2 |
10,5 |
|
ГГ |
14,7±0,3 |
14,4 |
20,0±0,3 |
10,3 |
19,6±0,3 |
9,7 |
25,4±0,2 |
6,0 |
|
ШМ |
6,5±0,1 |
9,8 |
8,5±0,2 |
11,7 |
9,1±0,1 |
8,9 |
12,3±0,2 |
10,2 |
|
ШCБ |
5,1±0,1 |
11,0 |
6,3±0,1 |
12,9 |
6,7±0,1 |
10,8 |
8,4±0,1 |
12,6 |
|
ДГ |
16,5±0,2 |
9,8 |
19,9±0,4 |
13,7 |
21,0±0,3 |
8,1 |
27,7±0,3 |
7,4 |
Примечани е. ЖМ — живая масса, ВХ — высота в холке, ВС — высота в спине, ВК — высота в крестце, ВМ — высота в маклоках, ВСБ — высота в седалищных буграх, КДТ — косая длина туловища, ОГ — обхват груди, ОП — обхват пясти, ШГ — ширина груди в плечах, ГГ — глубина груди, ШМ —ширина в маклоках, ШCБ — ширина в седалищных буграх, ДГ — длина головы. ЖМ указана в кг, промеры туловища представлены в см.
Всего было идентифицировано 1036 генов с находящимися в них или вблизи (±0,2 Mb) SNPs, сопряженными с изменчивостью фенотипических оценок показателей экстерьера и живой массы у молодняка коз из ресурсной популяции. Из них 341 полиморфизм был определен непосред- ственно в 326 генах на 1-29-й хромосомах.
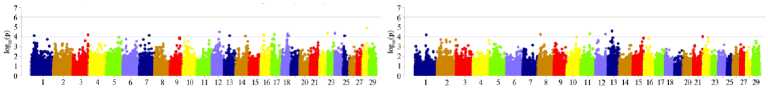
Установлены общие закономерности в распределении высокозначимых SNPs коз (p < 0,0001-0,00000001) для каждого из возрастных периодов. В возрасте 10 сут наблюдались общие мутации на 1-й, 8-й, 14-й, 16-й, 21-й и 24-й хромосомах (рис. 1, А), оказывающие влияние на изменчивость высотных промеров. Для КДТ, ОГ и ОП, ШГ и ГГ, ШМ отмечали SNPs, локализованные в QTL на 1-й, 2-й, 8-й, 12-й, 14-й, 15-й, 21-й и 22-й хромосомах. Для ДГ обнаружены суггестивные мутации (нижняя линия, p < 1,00E - 4) на 1-й, 5-й, 12-й, 14-й и 21-й хромосомах, однако они не преодолевали порог значимости по Бонферрони (верхняя линия, p < 9,77E - 7, 0,05/51167 SNP). По ЖМ на момент определения промеров экстерьера четко выделялись QTL на 1-й, 12-й, 14-й и 15-й хромосомах.
А
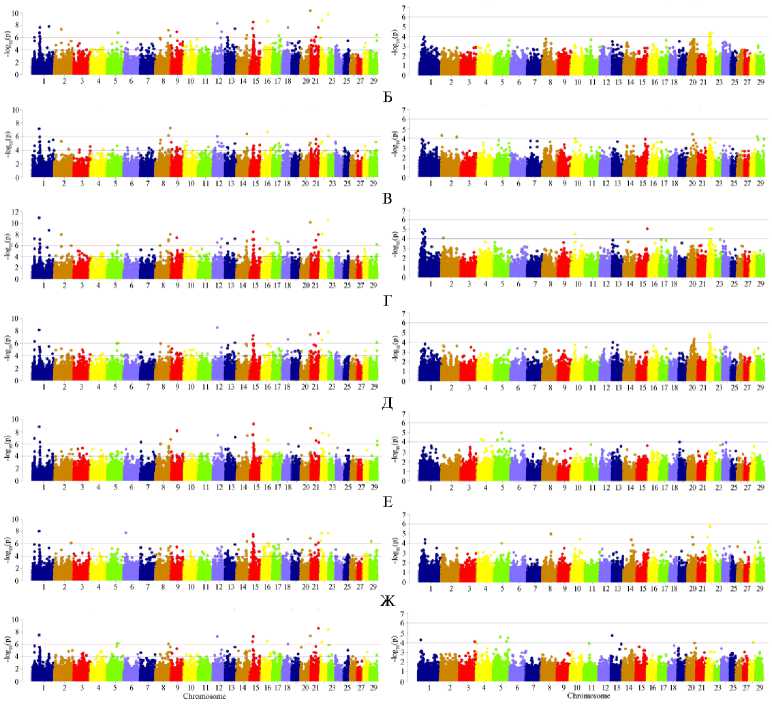
Рис. 1. Результаты GWAS анализа по показателям роста и развития молодняка коз ( Capra hir-cus L.) в группе возвратных кроссов из ресурсной популяции, полученной при скрещивании местных гибридных карачаевских коз с производителями калахарской породы, в разные возрастные периоды: слева — 10 сут, справа — 110 сут; А — живая масса, Б — высота в холке, В — косая длина туловища, Г — обхват груди, Д — ширина груди в плечах, Е — глубина груди, Ж — ширина в маклоках. Верхняя горизонтальная линия — порог по Бонферрони, нижняя горизонтальная линия — суггестивный уровень значимости ( n = 237, ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста — филиал племенной завод «Ладожский», Усть-Лабинский р-н, Краснодарский край, 2021-2023 годы).
В возрасте 110 сут наблюдали снижение достоверности детектируемых SNP-мутаций, однако общие из них, группируемые по промерам высоты, широты и глубины тела, сохранялись (см. рис. 1, Б). Так, для ВХ, ВС, ВК, ВМ и ВСБ были показаны значимые SNPs на 1-й, 2-й, 8-й, 14-й, 15-й,
20-й, 22-й хромосомах. По КДТ установлены QTL на 1-й, 10-й и 22-й хромосомах. Для ОГ и ОП отмечали схожие по своей локализации QTL (CHI1, CHI22). Широтные промеры экстерьера ввиду их коррелированности с ЖМ также показали значимые SNPs на 1-й, 8-й, 11-й, 14-й и 22-й хромосомах.
В 190-суточном возрасте результаты были схожи с наблюдаемыми в 110-суточном возрасте, что может указывать на наличие общих компонент изменчивости на хромосомах в активный период роста. Наиболее сильные ассоциации были установлены для ОГ (CHI16), ШГ (CHI17) и ЖМ (CHI7 и CHI16) (рис. 2, А).
А
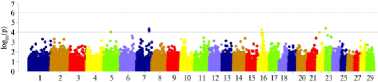
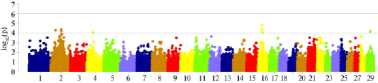
Б
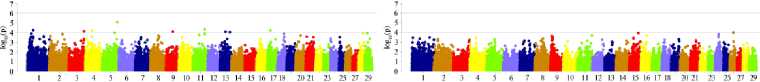
В
Г
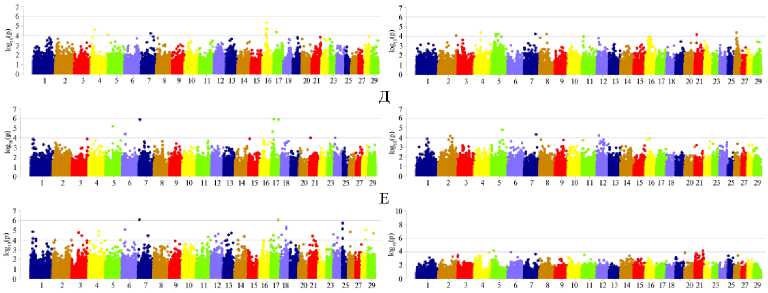
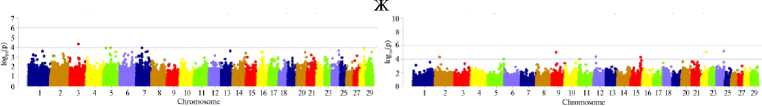
Рис. 2. Результаты GWAS анализа по показателям роста и развития молодняка коз ( Capra hir-cus L.) в группе возвратных кроссов из ресурсной популяции, полученной при скрещивании местных гибридных карачаевских коз с производителями калахарской породы, в разные возрастные периоды: слева — 190 сут, справа — 390 сут; А — живая масса, Б — высота в холке, В — косая длина туловища, Г — обхват груди, Д — ширина груди в плечах, Е — глубина груди, Ж — ширина в маклоках. Верхняя горизонтальная линия — порог по Бонферрони, нижняя горизонтальная линия — суггестивный уровень значимости ( n = 237, ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста — филиал племенной завод «Ладожский», Усть-Лабинский р-н, Краснодарский край, 2021-2023 годы).
Для животных в возрасте более 1 года ассоциации показали по большей части суггестивные мутации (до порога значимости по Бонферрони), что, по нашему мнению, может быть связано как с изменчивостью учтенных показателей экстерьера, так и с объемом выборки, снижением относительной скорости роста молодняка (с учетом интенсивности формирования интерьерных показателей и развития внутренних органов). Отмечены общие и значимые SNPs, входящие в QTL, на 1-й, 2-й, 5-й, 9-й, 12-й, 13-й, 15-й, 16-й, 21-й, 24-й и 26-й хромосомах (см. рис. 2, Б).
Максимальное число ассоциаций было выявлено в возрасте 10 сут по ВХ (35 SNPs), КДТ (52 SNPs), ОГ (19 SNPs), ОП (20 SNPs), ШГ (18 SNPs), ГГ (19 SNPs), ЖМ (12 SNPs). Полагаем, что эти параметры обладали наибольшей генетической вариабельностью, поскольку именно по ним можно точно оценить интенсивность развития особей. Для возрастных периодов 110 и 190 сут число значимых полиморфизмов, выявленных по результатам GWAS анализа, и их распределение было меньшим и колебалось от 1 до 9. В возрасте около 1 года у животных проявились значимые ассоциации для ШМ и ШCБ (6 SNPs), ШГ (8 SNPs), ВСБ и ОГ (10 SNPs), ДГ (12 SNPs). По нашему мнению, меньшее число значимых SNPs было связано со стадиями формирования костяка, внутренних органов и достижением животными оптимального соотношения высотных и широтных промеров, которые при снижении относительных приростов в дальнейшем мало изменяются. Однако в возрасте 1 года при окончательной структурной перестройке физиологических систем организма, когда начинается половое созревание, в условиях доращивания молодняка значительное влияние оказывали внешние факторы. В период раннего постнатального онтогенеза наблюдалось влияние материнского фактора (эффекта) при потреблении молока козлятами и активном развитии всех биологических систем и тканей.
Наиболее значимые ассоциации при уровне достоверности, близком к порогу по Бонферрони (p = 1,34E - 06) или выше него, были обнаружены на 1-й, 2-й, 5-й, 7-й, 8-й, 10-й, 12-й, 13-й, 14-й, 15-й, 16-й, 17-й, 19-й, 22-й и 25-й хромосомах. Ассоциации с наибольшим порогом значимости показали ВХ (10 сут), ОП (10 сут), ГГ (10 и 190 сут), КДТ (10 и 110 сут), ШГ (10 и 190 сут), ДГ (190 сут).
При анализе 326 генов, в которых мы выявили значимые ассоциации 341 однонуклеотидного полиморфизма с изученными нами фенотипическими признаками, с использованием базы данных DAVID были установлены следующие функциональные группы, по генной онтологии наиболее близко сопряженные с развитием организма животных и формированием фенотипических особенностей: GO:0001701 — внутриутробное эмбриональное развитие ( MYO1E , EDNRA , ADCY9 , SH3PXD2A , WNT3A , PTCH1 , ADAM10 , ZFPM2 , CMIP , DSC3 ), GO:0035556 — передача внутриклеточного сигнала ( SOCS2 , TGFBR3 , AKAP13 , UNC13C , ADCY9 , PLCL1 , AKT3 , ADCY8 , PLCB1 , CDC42BPA , DCLK1 ), GO:0016567 — убиквитинирование белка ( SOCS2 , ZNRF3 , FBXW7 , SIAH2 , MYCBP2 , TULP4 , TRIP12 ), GO:0045766 — положительная регуляция ангиогенеза ( CDH5 , FLT1 , AKT3 , AGO2 , ITGB8 ).
Для следующих полиморфизмов в генах, идентифицированных при GWAS анализе, было обнаружено множественное влияние на изменчивость показателей экстерьера кроссбредного потомства коз в разные возрастные периоды: CACNA1C (CHI5, 107498021 п.н.) для ШГ (110 сут), ГГ и ДГ (190 сут) (табл. 2); CTNNA2 (CHI11, 55401545 п.н., 54913498 п.н. и 55479696 п.н.) для ЖМ и ШГ (10 сут), ОП (390 сут); CDC42BPA (CHI16, 28553652 п.н.) для ЖМ (при рождении), ГГ и ШГ (10 сут); NPAS3 (CHI21, 43152627 п.н.) для ЖМ (10 сут) и ШМ (390 сут); RBMS3 (CHI22, 3929321 п.н., 4004416 п.н. и 4128373 п.н.) для ВХ, ГГ и ШСБ (10 сут), ГГ (110 сут); DLG2 (CHI29, 10526202 п.н. и 10555217 п.н.) для ГГ и ШГ (10 сут), ГГ и ВК (110 сут).
При детальном анализе генной онтологии мы вывили 38 генов с максимальным числом биологических функций (см. табл. 2) и провели поиск их сопряженности с QTL хозяйственных и функциональных особенностей по источникам литературы. Колебания числа функций для каждого из генов составляло от 8 (BANK1) до 66 (EDNRA). Наиболее часто встречающимися функциями генной онтологии были внутриутробное эмбриональное развитие (GO:0001701), регуляция пролиферации клеток (GO:0008283) и метаболических процессов, положительная регуляция ангиогенеза (GO:0045766), фосфорилирование белков (GO:0006468), клеточный гомеостаз ионов кальция (GO:0006874), регуляция экспрессии генов (GO:0010468), транспорт белка (GO:0015031), положительная регуляция роста клеток (GO:0030307), развитие молочной железы (GO:0030879), морфогенез задних конечностей (GO:0035137), нейромышечный процесс (GO:0050905) и ряд других.
ШМ 10 6,05E - 05
ЖМ 10 2,08E - 06
|
EFNA5 |
7 |
16 GO, в том числе: GO:0022407 — регуляция |
|
межклеточной адгезии, GO: 0061178 — регуляция секреции инсулина, участвующая в клеточном ответе на стимул глюкозы, GO:0071372 — клеточный ответ на стимул фолликулостимулирующего гормона |
||
|
FLT1 |
12 |
11 GO, в том числе: GO:0001525 — ангиогенез, GO:0030335 — положительная регуляция миграции клеток, GO:0045766 — положительная регуляция ангиогенеза |
|
FMN1 |
10 |
9 GO, в том числе: GO:0035136 — морфогенез передних конечностей, GO:0035137 — морфогенез задних конечностей, GO:0048705 — морфогенез скелетной системы |
|
GRIN2A |
25 |
19 GO, в том числе: GO:0022008 — нейрогенез, GO:0030431 — сон, GO:0042417 — метаболический процесс дофамина |
|
ITGB8 |
4 |
14 GO, в том числе: GO:0045766 — положительная регуляция ангиогенеза, GO:0051216 — развитие хряща, GO:1901388 — регуляция активации трансформирующего фактора роста бета |
|
MAP3K7 |
9 |
10 GO, в том числе: GO:0006915 — апоптоз-ный процесс, GO:0032147 — активация активности протеинкиназы, GO:0032743 — положительная регуляция продукции интерлейкина-2 |
|
MYO1E |
10 |
9 GO, в том числе: GO:0001701 — внутриутробное эмбриональное развитие, GO:0006807 — метаболический процесс соединений азота, GO:0006897 — эндоцитоз |
|
OXSR1 |
22 |
9 GO, в том числе: GO:0006979 — ответ на окислительный стресс, GO:0007231 — осмо-сенсорный сигнальный путь, GO:0010820 — положительная регуляция хемотаксиса Т-клеток |
Продолжение таблицы 2
КДТ 10 6,32E - 06
ГГ 10 4,59E - 06
ЖМ 10 1,23E - 05
ШГП 10 8,92E - 05
ЖМ 10 8,28E - 05
КДТ 390 1,14E - 04
КДТ 190 8,23E - 05
ОП 190 7,33E - 05
ЖМ 10 6,25E - 05
|
КДТ 10 |
1,97E - 05 |
|
КДТ 10 |
7,98E - 05 |
PTCH1 8
RRM2B 14
PDE4B 3
STAT1 2
SOCS2 5
TGFBR3 3
TRIM71 22
39 GO, в том числе: GO:0001701 — внутриутробное эмбриональное развитие, GO:0030326 — морфогенез эмбриональных конечностей, GO:0035137 — морфогенез задних конечностей
12 GO, в том числе: GO:0001822 — развитие почек, GO:0003014 — процесс почечной системы, GO:0009263 — процесс биосинтеза дезоксирибонуклеотидов
13 GO, в том числе: GO:0001780 — гомеостаз нейтрофилов, GO:0006198 — катаболический процесс цАМФ, GO:0086004 — регуляция сердечной мышцы
19 GO, в том числе: GO:0032727 — положительная регуляция продукции интерферона-альфа, GO:0035458 — клеточный ответ на ин-терферон-бета, GO:0051607 — защитный ответ на вирусную инфекцию
10 GO, в том числе: GO:0016567 — убиквити-нирование белка, GO:0035556 — передача внутриклеточного сигнала, GO:0060396 — сигнальный путь рецептора гормона роста
10 GO, в том числе: GO:0035556 — передача внутриклеточного сигнала GO:0006955 — иммунный ответ, GO:0007179 — сигнальный путь бета-рецептора трансформирующего фактора роста
11 GO, в том числе: GO:0008543 — сигнальный путь рецептора фактора роста фибробластов, GO:0010586 — метаболический процесс микроРНК, GO:0072089 — пролиферация стволовых клеток
|
ВХ 10 |
5,31E - 06 |
|
ВС 10 |
4,10E - 06 |
|
ВК 10 |
5,18E - 06 |
|
ВМ 10 |
4,12E - 06 |
|
КДТ 10 |
1,01E - 08 |
|
ОГ 10 |
2,80E - 07 |
|
ОП 10 |
1,21E - 05 |
|
ШГП 10 |
1,53E - 08 |
|
ГГ 10 |
2,15E - 08 |
|
ШМ 10 |
4,21E - 07 |
|
ШСБ 10 |
6,08E - 05 |
|
ЖМ 10 |
1,70E - 09 |
|
КДТ 10 |
1,07E - 05 |
|
ШМ 10 |
3,68E - 05 |
|
ЖМ 10 |
2,67E - 05 |
|
ДГ 390 |
5,49E - 06 |
|
КДТ 10 |
4,75E - 05 |
|
ОГ 10 |
5,76E - 05 |
|
ШГП 10 |
7,77E - 05 |
|
КДТ 10 |
9,89E - 05 |
ШГП 110 6,07E - 05
|
ОГ 10 |
2,72E - 05 |
|
ШМ 10 |
2,79E - 05 |
|
ДГ 10 |
9,46E - 05 |
|
ЖМ 10 |
4,40E - 05 |
Продолжение таблицы 2
ZFPM2 14 18 GO, в том числе: GO:0001701 — внутри- ГГ 190 1,04E - 04
утробное эмбриональное развитие, GO:0009653 — морфогенез анатомической структуры, GO:0048513 — развитие органов животных
П р и м е ч ани е. CHI — аутосома коз, p — значимость результатов GWAS анализа; ВХ — высота в холке, ВС — высота в спине, ВК — высота в крестце, ВМ — высота в маклоках, ВСБ — высота в седалищных буграх, КДТ — косая длина туловища, ОГ — обхват груди, ОП — обхват пясти, ШГП — ширина груди в плечах, ГГ — глубина груди, ШМ — ширина в маклоках, ШСБ — ширина в седалищных буграх, ДГ — длина головы. В нижних индексах указан возраст животных — 10, 110, 190 и 390 сут. В таблице указаны по 3-4 основные GO для каждого гена.
Среди идентифицированных были выявлены известные гены-кандидаты, влияющие на формирование экономически значимых признаков у коз. Обнаружены гены, связанные с процессами роста и развития. Так, ген AKT3 влияет на дифференцировку остеокластов (10). Ген ABCA1 потенциально связан с морфогенезом эмбриональной скелетной системы (11). Анализируя результаты GWAS, B. Gu с соавт. (12) установили, что ген CDK5RAP2 ассоциирован с окружностью пясти у коз породы черная дазу. Ген MYCBP2 влияет на мясную продуктивность у пакистанских коз (13). Ген RBFOX2 , необходимый для поддержания массы скелетных мышц и белкового гомеостаза, контролирует слияние миобластов в миогенезе посредством координации альтернативного сплайсинга Mef2d и Rock2 (14). Ген EFNA5 опосредует связь между островковыми клетками поджелудочной железы, регулируя стимулируемую глюкозой секрецию инсулина, который необходим для правильного роста, обмена веществ и восстановления тканей. Этот ген предложен в качестве кандидата, связанного с ростом и развитием домашних коз (15).
Идентифицирована большая группа генов, влияющих на шерстную продуктивность. Ген CDH5 , связанный с клеточной адгезией, вовлечен в регуляцию фаз роста волосяных фолликулов у кашемировых коз (16). В ряде исследований показано, что lncRNA-599,554 способствует индуктивному свойству клеток дермального сосочка у кашемировых коз, которое может быть реализовано посредством экспрессии WNT3A (17). Ген YAP1 — это позитивно закрепленный отбором ген, вовлеченный в развитие волосяных фолликулов, рост волос и, вероятно, в формирование адаптаций к выживанию в холодной среде Тибетского плато у тибетских кашемировых коз (18). Ген ITGB8 — важный функциональный кандидат, влияющий на формирование и развитие кашемирового пуха (19). Анализ транскриптома показал, что MAP3K7 участвует в циклировании волосяных фолликулов у кашемировой козы (20). Кроме того, выявлены гены-кандидаты, связанные с молочной продуктивностью: PDE4B (21) и AGO2 (22) .
Ген PTCH1 — один из генов-мишеней, регулирующих репродуктивные функции (23). Ген ACACB кодирует фермент, ограничивающий окисление митохондриальных жирных кислот при низких запасах глюкозы и гликогена. Отмечена связь этого гена с формированием воспроизводительных качеств у животных (24). Ген FLT1 влияет на развитие трофобласта на ранних стадиях суягности коз (25).
Предполагается, что мутация в гене FMN1 , играющем роль в росте и развитии конечностей млекопитающих, может быть ответственна за развитие полипоподобных придатков кожи у коз (сережек — придатков на горле) (26). Число копий гена EDNRA коррелирует со степенью белой пятнистости у коз (27).
Ген STAT1 связан с иммунными функциями (28). Ген TGFBR3 влияет на развитие системы кровообращения и ряда заболеваний, включая гипоплазию trabecula carnea. Согласно биоинформационной фенотипической базе млекопитающих MGI-MP, основанной на модели генома мыши, также существует значимая связь (p < 0,05) этого гена с аномальным развитием скелета у тибетских кашмирских коз (29).
-
H. Signer-Hasler с соавт. (30) представили описание 15 островков ROH, общих для 217 коз швейцарских пород и 9 безоаровых козлов. В одном из регионов был аннотирован ген ZFPM2 , вовлеченный в процесс доместикации и формирование хозяйственно полезных признаков у современных коз (30). У диких коз ген CACNA1C , участвующий в высвобождении серотонина, находился под давлением естественного отбора, что может быть связано с более сложными условиями обитания (31). Известно, что активация гена RRM2B повышалась у коз при экстенсивной технологии разведения (32). Ген SOCS2 связан с адаптацией коз к условиям гипоксии (33).
Кроме того, нами аннотированы гены, которые ранее не были описаны у домашних коз, но имеют известные функции у других видов сельскохозяйственных животных.
Ген GRIN2A может играть роль в регуляции связанных с половым созреванием молекулярных механизмов, в оси гипоталамус–гипофиз–яич-ники у крупного рогатого скота (34). Ген CARMIL1 связан с концентрацией нейротрансмиттеров, влияющих на здоровье и поведенческие особенности животных (35). ANO6 закреплен позитивным отбором у жвачных в числе генов адаптационных признаков (36). Ген LRP2 у местных и коммерческих пород крупного рогатого скота связан с ростом и мясной продуктивностью (37). Подтверждена связь гена BANK1 с характеристиками эффективности использования корма у китайских овец породы Ху (Hu), что дает возможности по оптимизации стратегий кормления (38). Ген ADAM10 связан с дифференцировкой Т-клеток — группы лейкоцитов, ответственных за защиту организма от антигенов (39). Высказано предположение, что ген DNM1L участвует во взаимодействии между белками, клетками и возбудителем лентивирусной инфекции (40). Ген ADAMTS12 — это член семейства дезинтегринов и металлопротеиназ с тромбоспондиновыми мотивами, которые играют важную роль в обмене белков внеклеточного матрикса в различных тканях (41). По данным H. Zhang с соавт. (42), этот ген может быть связан с репродуктивными способностями и концентрацией тестостерона, что играет роль в регуляции развития рогов. Ген ARL8B влияет на развитие костей и вызывает различия в ростовых параметрах во время онтогенеза (42). Кроме того, обнаружено, что этот ген наряду с AMD1 и DTNBP1 обусловливает более легкое течение родового процесса (43). Ген TRIM71 входит в геномный регион, связанный с потерей слуха, и демонстрирует признаки древнего положительного адаптивного отбора у людей (44). Ген UVRAG , маркированный CNV (copy number variation), обнаружен у трех пород коз C. nubiana и связан с признаками адаптивной эволюции диких африканских коз (45). Ген OXSR1 влияет на формирование ответа на окислительный стресс (46).
Таким образом, в геноме коз из группы возвратных кроссов ресурсной популяции, полученных при скрещивании местных гибридных карачаевских коз с производителями калахарской породы, были определены гены, связанные с параметрами роста и развития, фертильностью и воспроизводительными качествами, адаптационными способностями, шерстной продуктивностью животных. Были определены 34 потенциальных QTL, сопряженных с генами, которые связаны с защитной реакцией организма животных ( ADAM10 ), репродуктивными особенностями ( ADAMTS12 , ACACB , PTCH1 ), параметрами роста (остеогенеза и миогенеза) и развития ( ARL8B , 644
AKT3 , CDK5RAP2 , RBFOX2 , CDH5 , EFNA5 , FMN1 , TGFBR3 ), эмбриональным развитием ( ABCA1 ), эффективностью использования корма ( BANK1 ), мясной продуктивностью и воспроизводительными качествами ( LRP2 , MYCBP2 , OXSR1 ), адаптационными способностями ( UVRAG , ANO6 , CACNA1C , FLT1 , RRM2B , STAT1 , SOCS2 , TRIM71 ), развитием волосяных фолликулов, пигментацией и шерстной/пуховой продуктивностью ( WNT3A , YAP1 , EDNRA , ITGB8 , MAP3K7 ), молочной продуктивностью ( AGO2, PDE4B ), устойчивостью к заболеваниям ( DNM1L ) и доместикацией ( ZFPM2 ). В сочетании идентифицированные гены в наибольшей степени влияли на глубину и обхват груди, ширину груди в плечах, ширину в маклоках, косую длину туловища, живую массу и высоту спины.
Список литературы Идентификация SNPS для показателей роста и развития коз (Capra hircus Linnaeus, 1758) из ресурсной популяции в возрастной динамике
- Новопашина С.И., Санников М.Ю., Хататаев С.А., Кузьмина Т.Н., Хмелевская Г.Н., Степанова Н.Г., Тихомиров А.И., Маринченко Т.Е. Состояние и перспективные направления улучшения генетического потенциала мелкого рогатого скота. Научный аналитическийобзор. М., 2019.
- Zhang L., Wang F., Gao G., Yan X., Liu H., Liu Z., Wang Z., He L., Lv Q., Wang Z., Wang R., Zhang Y., Li J., Su R. Genome-wide association study of body weight traits in Inner Mongolia cashmere goats. Frontiers in Veterinary Science, 2021, 8: 752746 (doi: 10.3389/fvets.2021.752746).
- Selionova M., Aibazov M., Sermyagin A., Belous A., Deniskova T., Mamontova T., Zharkova E., Zinovieva N. Genome-wide association and pathway analysis of carcass and meat quality traits in Karachai young goats. Animals, 2023,13(20):3237 (doi: 10.3390/ani13203237).
- Selionova M., Trukhachev V., Aibazov M., Sermyagin A., Belous A., Gladkikh M., Zinovieva N. Genome-wide association study of milk composition in Karachai goats. Animals, 2024, 14(2): 327 (doi: 10.3390/ani14020327).
- Sun X., Niu Q., Jiang J., Wang G., Zhou P., Li J., Chen C., Liu L., Xu L., Ren H. Identifying candidate genes for litter size and three morphological traits in Youzhou Dark goats based on genome-wide SNP markers. Genes, 2023, 14(6): 1183 (doi: 10.3390/genes14061183).
- Денискова Т.Е., Петров С.Н., Сермягин А.А., Доцев А.В., Форнара М.С., Reyer H., Wimmers К., Багиров В.А., Brem G., Зиновьева Н.А. Поиск геномных вариантов, ассоциированных с живой массой у овец, на основе анализа высокоплотных SNP генотипов. Сельскохозяйственная биология, 2021, 56(2): 279-291 (doi: 10.15389/agrobiology.2021.2.279rus).
- Денискова Т.Е., Кошкина О.А., Петров С.Н., Сермягин А.А., Зиновьева Н.А. Идентификация генов-кандидатов, связанных с ростом и развитием овец из кроссбредной популяции, с использованием полногеномного поиска ассоциаций. АграрнаянаукаЕвро-Северо-Востока, 2024, 25(2): 236-250 (doi: 10.30766/2072-9081.2024.25.2.236-250).
- Sherman B.T., Hao M., Qiu J., Jiao X., Baseler M.W., Lane H.C., Imamichi T., Chang W. DAVID: a web server for functional enrichment analysis and functional annotation of gene lists (2021 update). Nucleic Acids Research, 2022, 50(W1): W216-W221 (doi: 10.1093/nar/gkac194).
- Chang C.C., Chow C.C., Tellier L.C., Vattikuti S., Purcell S.M., Lee J.J. Second-generation PLINK: rising to the challenge of larger and richer datasets. GigaScience, 2015, 4: s13742-015-0047-8 (doi: 10.1186/s13742-015-0047-8).
- Tao L., He X., Jiang Y., Liu Y., Ouyang Y., Shen Y., Hong Q., Chu M. Genome-wide analyses reveal genetic convergence of prolificacy between goats and sheep. Genes, 2021, 12(4): 480 (doi: 10.3390/genes12040480).
- Guan D., Martínez A., Castelló A., Landi V., Luigi-Sierra M.G., Fernández-Álvarez J., Cabrera B., Delgado J.V., Such X., Jordana J., Amills M. A genome-wide analysis of copy number variation in Murciano-Granadina goats. Genetics, Selection, Evolution: GSE, 2020, 52(1): 44 (doi: 10.1186/s12711-020-00564-4).
- Gu B., Sun R., Fang X., Zhang J., Zhao Z., Huang D., Zhao Y., Zhao Y. Genome-wide association study of body conformation traits by whole genome sequencing in Dazu Black goats. Animals, 2022, 12(5): 548 (doi: 10.3390/ani12050548).
- Saif R., Mahmood T., Zia S., Henkel J., Ejaz A. Genomic selection pressure discovery using site-frequency spectrum and reduced local variability statistics in Pakistani Dera-Din-Panah goat. Tropical Animal Health and Production, 2023, 55(5): 331 (doi: 10.1007/s11250-023-03758-2).
- Yuan Y., Zhang W., Liu C., He Y., Zhang H., Xu L., Yang B., Zhao Y., Ma Y., Chu M., Zhao Z., Huang Y., Han Y., Zeng Y., Ren H., Wang G., E G. Genome-wide selective analysis of Boer goat to investigate the dynamic heredity evolution under different stages. Animals, 2022, 12(11): 1356 (doi: 10.3390/ani12111356).
- Moaeen-Ud-Din M., Danish Muner R., Khan M.S. Genome wide association study identifies novel candidate genes for growth and body conformation traits in goats. Scientific Reports, 2022, 12(1): 9891 (doi: 10.1038/s41598-022-14018-y).
- Bhat B., Yaseen M., Singh A., Ahmad S.M., Ganai N.A. Identification of potential key genes and pathways associated with the Pashmina fiber initiation using RNA-Seq and integrated bioinformatics analysis. Scientific Reports, 2021, 11(1): 1766 (doi: 10.1038/s41598-021-81471-6).
- Li C., Feng C., Ma G., Fu S., Chen M., Zhang W., Li J. Time-course RNA-seq analysis reveals stage-specific and melatonin-triggered gene expression patterns during the hair follicle growth cycle in Capra hircus. BMC Genomics, 2022, 23(1): 140 (doi: 10.1186/s12864-022-08331-z).
- Wu C., Ma S., Zhao B., Qin C., Wu Y., Di J., Suo L., Fu X. Drivers of plateau adaptability in cashmere goats revealed by genomic and transcriptomic analyses. BMC Genomics, 2023, 24(1): 428 (doi: 10.1186/s12864-023-09333-1).
- Wang W., Li Z., Xie G., Li X., Wu Z., Li M., Liu A., Xiong Y., Wang Y. Convergent genomic signatures of cashmere traits: evidence for natural and artificial selection. International Journal of Molecular Sciences, 2023, 24(2): 1165 (doi: 10.3390/ijms24021165).
- Geng R., Yuan C., Chen Y. Exploring differentially expressed genes by RNA-Seq in cashmere goat (Capra hircus) skin during hair follicle development and cycling. PloS ONE, 2013, 8(4): e62704 (doi: 10.1371/journal.pone.0062704).
- Waineina R.W., Okeno T.O., Ilatsia E.D., Ngeno K. Selection signature analyses revealed genes associated with adaptation, production, and reproduction in selected goat breeds in Kenya. Frontiers in Genetics, 2022, 13: 858923 (doi: 10.3389/fgene.2022.858923).
- Massender E., Oliveira H.R., Brito L.F., Maignel L., Jafarikia M., Baes C.F., Sullivan B., Schenkel F.S. Genome-wide association study for milk production and conformation traits in Canadian Alpine and Saanen dairy goats. Journal of Dairy Science, 2023, 106(2): 1168-1189 (doi: 10.3168/jds.2022-22223).
- Sun Z., Zhang Z., Liu Y., Ren C., He X., Jiang Y., Ouyang Y., Hong Q., Chu M. Integrated analysis of mRNAs and long non-coding RNAs expression of oviduct that provides novel insights into the prolificacy mechanism of goat (Capra hircus). Genes, 2022, 13(6): 1031 (doi: 10.3390/genes13061031).
- Ren C., Chen Y., Tang J., Wang P., Zhang Y., Li C., Zhang Z., Cheng X. TMT-based comparative proteomic analysis of the spermatozoa of buck (Capra hircus) and ram (Ovis aries). Genes, 2023, 14(5): 973 (doi: 10.3390/genes14050973).
- Luo N., Cheng W., Zhou Y., Gu B., Zhao Z., Zhao Y. Screening candidate genes regulating placental development from trophoblast transcriptome at early pregnancy in Dazu Black goats (Capra hircus). Animals, 2021, 11(7): 2132 (doi: 10.3390/ani11072132).
- Reber I., Keller I., Becker D., Flury C., Welle M., Drögemüller C. Wattles in goats are associated with the FMN1/GREM1 region on chromosome 10. Animal Genetics, 2015, 46(3): 316-320 (doi: 10.1111/age.12279).
- Menzi F., Keller I., Reber I., Beck J., Brenig B., Schütz E., Leeb T., Drögemüller C. Genomic amplification of the caprine EDNRA locus might lead to a dose dependent loss of pigmentation. Scientific Reports, 2016, 6: 28438 (doi: 10.1038/srep28438).
- Wani S.A., Sahu A.R., Khan R.I.N., Pandey A., Saxena S., Hosamani N., Malla W.A., Chaudhary D., Kanchan S., Sah V., Rajak K.K., Muthuchelvan D., Mishra B., Tiwari A.K., Sahoo A.P., Sajjanar B., Singh Y.P., Gandham R.K., Mishra B.P., Singh R.K. Contrasting gene expression profiles of monocytes and lymphocytes from peste-des-petits-ruminants virus infected goats. Frontiers in Immunology, 2019, 10: 1463 (doi: 10.3389/fimmu.2019.01463).
- Guo J., Zhong J., Li L., Zhong T., Wang L., Song T., Zhang H. Comparative genome analyses reveal the unique genetic composition and selection signals underlying the phenotypic characteristics of three Chinese domestic goat breeds. Genetics, Selection, Evolution: GSE, 2019 51(1): 70 (doi: 10.1186/s12711-019-0512-4).
- Signer-Hasler H., Henkel J., Bangerter E., Bulut Z., VarGoats Consortium, Drögemüller C., Leeb T., Flury C. Runs of homozygosity in Swiss goats reveal genetic changes associated with domestication and modern selection. Genetics, Selection, Evolution: GSE, 2022, 54(1): 6 (doi: 10.1186/s12711-022-00695-w).
- Dong Y., Zhang X., Xie M., Arefnezhad B., Wang Z., Wang W., Feng S., Huang G., Guan R., Shen W., Bunch R., McCulloch R., Li Q., Li B., Zhang G., Xu X., Kijas J. W., Salekdeh G.H., Wang W., Jiang Y. Reference genome of wild goat (Capra aegagrus) and sequencing of goat breeds provide insight into genic basis of goat domestication. BMC Genomics, 2015, 16(1): 431 (doi: 10.1186/s12864-015-1606-1).
- Ncube K.T., Dzomba E.F., Rosen B.D., Schroeder S.G., Van Tassell C.P., Muchadeyi F.C. Differential gene expression and identification of growth-related genes in the pituitary gland of South African goats. Frontiers in Genetics, 2022, 13: 811193 (doi: 10.3389/fgene.2022.811193).
- Wang X., Liu J., Zhou G., Guo J., Yan H., Niu Y., Li Y., Yuan C., Geng R., Lan X., An X., Tian X., Zhou H., Song J., Jiang Y., Chen Y. Whole-genome sequencing of eight goat populations for the detection of selection signatures underlying production and adaptive traits. Scientific Reports, 2016, 6: 38932 (doi: 10.1038/srep38932).
- Tahir M.S., Porto-Neto L.R., Gondro C., Shittu O.B., Wockner K., Tan A.W.L., Smith H.R., Gouveia G.C., Kour J., Fortes M.R.S. Meta-analysis of heifer traits identified reproductive pathways in Bos indicus cattle. Genes, 2021, 12(5): 768 (doi: 10.3390/genes12050768).
- Chen Q., Qu K., Ma, Z., Zhan J., Zhang F., Shen J., Ning Q., Jia P., Zhang J., Chen N., Chen H., Huang B., Lei C. Genome-wide association study identifies genomic loci associated with neurotransmitter concentration in cattle. Frontiers in Genetics, 2020, 11: 139 (doi: 10.3389/fgene.2020.00139).
- Wang Y., Zhang C., Wang N., Li Z., Heller R., Liu R., Zhao Y., Han J., Pan X., Zheng Z., Dai X., Chen C., Dou M., Peng S., Chen X., Liu J., Li M., Wang K., Liu C., Lin Z., Chen L., Hao F., Zhu W., Song C., Zhao C., Zheng C., Wang J., Hu S., Li C., Yang H., Jiang L., Li G., Liu M., Sonstegard T.S., Zhang G., Jiang Y., Wang W., Qiu Q. Genetic basis of ruminant headgear and rapid antler regeneration. Science, 2019, 364(6446): eaav6335 (doi: 10.1126/science.aav6335).
- Stronen A.V., Pertoldi C., Iacolina L., Kadarmideen H.N., Kristensen T.N. Genomic analyses suggest adaptive differentiation of northern European native cattle breeds. Evolutionary Applications, 2019, 12(6): 1096-1113 (doi: 10.1111/eva.12783).
- Zeng X., Wang W., Zhang D., Li X., Zhang Y., Zhao Y., Zhao L., Wang J., Xu D., Cheng J., Li W., Zhou B., Lin C., Yang X., Zhai R., Ma Z., Liu J., Cui P., Weng X., Wu W., Zhang X., Zheng W. Expression of the ovine gene and the relationship between its polymorphism and feed efficiency traits. DNA and Cell Biology, 2023, 42(4): 194-202 (doi: 10.1089/dna.2022.0529).
- Berton M.P., de Oliveira Silva R.M., Peripolli E., Stafuzza N.B., Martin J.F., Álvarez M.S., Gavinã B.V., Toro M.A., Banchero G., Oliveira P.S., Eler J.P., Baldi F., Ferraz J.B.S. Genomic regions and pathways associated with gastrointestinal parasites resistance in Santa Inês breed adapted to tropical climate. Journal of Animal Science and Biotechnology, 2017, 8: 73 (doi: 10.1186/s40104-017-0190-4).
- Pławińska-Czarnak J., Majewska A., Zarzyńska J., Bogdan J., Kaba J., Anusz K., Bagnicka E. Gene expression profile in peripheral blood nuclear cells of small ruminant lentivirus-seropositive and seronegative dairy goats in their first lactation. Animals, 2021, 11(4): 940 (doi: 10.3390/ani11040940).
- Namous H., Peñagaricano F., Del Corvo M., Capra E., Thomas D. L., Stella A., Williams J.L., Marsan P.A., Khatib H. Integrative analysis of methylomic and transcriptomic data in fetal sheep muscle tissues in response to maternal diet during pregnancy. BMC Genomics, 2018, 19(1): 123 (doi: 10.1186/s12864-018-4509-0).
- Zhang H., Yang P., Liu C., Ma Y., Han Y., Zeng Y., Huang Y., Zhao Y., Zhao Z., He X., E G. Novel heredity basis of the four-horn phenotype in sheep using genome-wide sequence data. Animals, 2023, 13(20): 3166 (doi: 10.3390/ani13203166).
- Zhang X., Zhang S., Tang Q., Jiang E., Wang K., Lan X., Pan C. Goat sperm associated antigen 17 protein gene (SPAG17): small and large fragment genetic variation detection, association analysis, and mRNA expression in gonads. Genomics, 2020, 112(6): 5115-5121 (doi: 10.1016/j.ygeno.2020.09.029).
- Trigila A.P., Pisciottano F., Franchini L.F. Hearing loss genes reveal patterns of adaptive evolution at the coding and non-coding levels in mammals. BMC Biology, 2021, 19(1): 244 (doi: 10.1186/s12915-021-01170-6).
- Chebii V. Genome scan for signatures of adaptive evolution in wild African goat (Capra nubiana). PhD Theses and Dissertations. Arusha, Tanzania, 2021
- Naldurtiker A., Batchu P., Kouakou B., Terrill T.H., Shaik A., Kannan G. RNA-Seq exploration of the influence of stress on meat quality in Spanish goats. Scientific Reports, 2022, 12(1): 20573 (doi: 10.1038/s41598-022-23269-8).


