Идентификация сортов, линий и мутантов гороха посевного с помощью RAPD-маркеров
Автор: Дрибноходова О.П., Кокаева З.Г., Гостимский С.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: ДНК-технологии в растениеводстве
Статья в выпуске: 5 т.40, 2005 года.
Бесплатный доступ
Проводили анализ ДНК-полиморфизма 19 форм гороха посевного (Pisum sativum L.) по 134 RAPD-маркерам. Определяли генетическое сходство, отражающее степень различий RAPD-спектров исследованных образцов. Рассчитывали генетические расстояния между линиями, сортами и мутантами гороха посевного. Оценивали эффективность RAPD-метода для паспортизации и идентификации линий и сортов растений.
Короткий адрес: https://sciup.org/142133119
IDR: 142133119 | УДК: 633.358:577.2
Текст научной статьи Идентификация сортов, линий и мутантов гороха посевного с помощью RAPD-маркеров
В последние годы проблема идентификации видов и сортов растений становится все более актуальной в связи с ускорением селекционного процесса и появлением большого числа новых форм. Для создания нового сорта необходимы предварительная регистрация существующих генотипов, контроль за уровнем полиморфизма полученных при скрещивании партий семян и чистотой селектируемой линии. Экспресс-анализы сортопринадлежности образцов семян могут быть использованы для решения спорных вопросов в семеноводстве и для защиты прав оригинатора сорта (1, 2). Очевидно, что современная практика семеноводства нуждается в разработке быстрых и точных методов идентификации каждого сорта. Морфологические признаки не пригодны для быстрой проверки семян на сортовую принадлежность, так как требуют больших затрат времени и труда, особенно в случае необходимости разделения сортов, имеющих сходные фенотипы (3). Кроме того, при таком подходе трудно отличить генетические изменения от модификаций, вызванных влиянием окружающей среды (4).
В настоящее время для генотипирования все чаще используют ДНК-маркеры, причем наиболее часто — RAPD- и SSR-маркеры (2, 5). Эти методы обладают высокой производительностью, получаемые с их помощью результаты не зависят от условий выращивания и стадии развития растений, продолжительности и условий хранения семян. С помощью RAPD-метода можно анализировать изучаемые формы одновременно по многим локусам, случайно распределенным по всему геному, что позволяет различать даже очень близкородственные сорта (6). Хотя многие авторы отмечают недостаточную надежность RAPD-метода, оптимизация условий и отбор стабильно амплифицирующихся ДНК-маркеров позволяют добиться воспроизведения данных, полученных в разных лабораториях, и поставить вопрос о разработке дешевых, быстрых и эффективных технологий паспортизации разных линий, сортов и гибридов гороха на основе RAPD-метода. За последние годы были проведены работы по изучению генетического полиморфизма различных культур: ячменя, кукурузы, подсолнечника, картофеля, хлопчатника, пшеницы и др. (1, 3, 4, 6-9).
В задачу нашей работы входила идентификация и паспортизация сортов, линий и мутантов гороха посевного с помощью RAPD-маркеров.
технология», Москва) в следующем режиме: предварительная денатурация 2,5 мин при 94 о С; денатурация 30 с при 94 о С; отжиг 30 с при 40 о С; синтез 1,3 мин при 70,5 о С (всего 5 циклов); денатурация 20 с при 93 о С; отжиг 30 с при 37 о С; синтез 1 мин при 72 о С (всего 35 циклов); конечная элонгация 5 мин при 72 о С. Реакционная смесь объемом 25 мкл содержала 2 е.а. Taq-полимеразы («Силекс М», Москва), 2,5 мкл стандартного 10-кратного буфера для ПЦР («Силекс М», Москва), 25 пкмоль праймера, 2,5 мМ MgCl 2 , 0,5 или 0,25 мМ дезоксинуклеозидтрифосфата. На смесь наслаивали две капли минерального масла.
Продукты амплификации разделяли электрофорезом в 1,7 % агарозном геле с использованием буфера ТВЕ и окрашивали бромистым этидием. Для определения длины фрагментов использовали маркеры молекулярной массы 100 bp + 1,5 Kb DNA Ladder и 1 Kb DNA Ladder («Сибэнзим», Новосибирск). После электрофореза гели анализировали в УФ-свете и фотографировали с использованием цифровой фотокамеры.
Для проверки воспроизводимости полученных результатов эксперименты проводили в нескольких повторностях с использованием двух концентраций MgCl 2 (0,5 и 0,25 мМ) и разных партий полимеразы. В дальнейшей работе использовали только те фрагменты, о наличии или отсутствии которых в спектрах всех сортов можно было судить с достаточной степенью уверенности. Каждому полученному маркеру было дано название, состоящее из двух частей: первая часть соответствует праймеру, при амплификации с которым был обнаружен фрагмент, вторая — отражает приблизительный размер фрагмента (п.н.).
Для количественной оценки полиморфизма и определения генетического расстояния между исследованными формами данные RAPD-анализа были представлены в виде матрицы, в которой наличие или отсутствие в RAPD-спектрах фрагментов одинаковой длины соответствовало значениям 1 или 0. На основе этой матрицы были составлены две матрицы различий, генетические расстояния (ГР) в которых рассчитывали по формуле
Ч исло полиморфных маркеров
ГР =-----------—----- X 100%.
Общее число маркеров
Генетическое расстояние между сравниваемыми линиями определяли по формуле Nei с соавт. с помощью компьютерной программы Treecon (11, 12).
На основании полученной невзвешенным парно-групповым методом (Unweighted Pair-Group Method — UPGMA) матрицы мы составили генеалогическую дендрограмму, отражающую степень различий между RAPD-спектрами исследуемых форм гороха (12). Дендрограмму строили по компьютерной программе Treecon с применением 100 реплик бутстрепа (12, 13). Матрица различий, полученная по формуле Nei
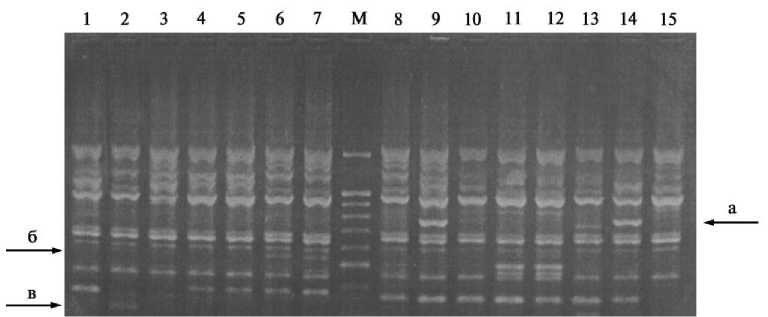
Рис. 1. RAPD-спектры линий, мутантов и сортов гороха посевного, полученные при амплификации ДНК с праймером B474: 1 — Флагман; 2 — Филби; 3 — Труженик; 4 — Виола; 5 — Виола (В); 6 — Хл-1; 7 — Хл-2; 8 — Хл-15; 9 — Хл-18; 10 — Хл-42; 11 — L-1238; 12 — L-1132; 13 — L-851; 14 — L-111; 15 — L-108; М — маркер молекулярной массы 100 bp + 1,5 Kb DNA Ladder. Стрелками отмечены полиморфные фрагменты: а, б и в — соответственно B474#730, B474#530 и B474#300.
с соавт., лучше отвечает известным данным о происхождении сортов и линий, что соответствует данным литературы (9).
Результаты. Межлинейный и межсортовой полиморфизм оценивали у семи сортов, шести линий и шести мутантов гороха посевного с использованием десяти высокоэффективных RAPD-праймеров (рис. 1). При амплификации ДНК разных образцов гороха посевного отдельные праймеры выявляли от 9 до 19 фрагментов размером 230-2900 п.н., 7-14 из которых были полиморфными; всего получено 134 RAPD-маркера, 102 из которых были полиморфными среди изученных форм гороха
(табл. 1).
Для количественной оценки полиморфизма и определения степени дивер-
0,2
0,1
76 % । -------------Немчиновский
^-----------R 64
--------------------Хл-42
-----------------Флагман
-----------------Труженик
--------------------------------------L-111
I-------------------------Филби
1-------------------------- Хл-18
Хл-115 Капитал Хл-1 Хл-2
|
1 |
|
70 %
77 %
100 % । Виола Виола (В) Демон L-108 L-851
L-1238
L-1132
генции между изученными формами данные, полученные при оценке RAPD-полиморфизма, были представлены в виде матрицы по 134 бинарным признакам, которую использовали для расчета генетических расстояний и построения дендрограммы (рис. 2). Степень различий между мутантами, полученными от одного сорта, составляла 1,514,9 %; от исходных форм — Виола и Хл-42 — они отличались соответственно на 0 и 20,9 %. Средний уровень полиморфизма между изученными формами составлял 25,6 %, что согласуется с ранее полученными данными (14).
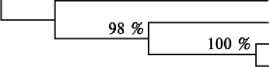
Рис. 2. Генеалогическая дендрограмма, отражающая степень различий между RAPD-спектрами линий, сортов и мутантов гороха посевного (UPGMA-метод) (12): 0,1 и 0,2 — генетическое расстояние, по Nei с соавт. (11) .
-
"^ 1. Оценка полиморфизма ДНК у различных линий, сортов и мутантов гороха посевного ( Pisum sativum L.) с помощью RAPD-маркеров
Сорт, мутант, линия
Длина ДНК-фрагментов, полученных при амплификации с праймером QR2, п.н.
1600
1550
1500
1400
1300
1250
1200
1100
1000
910
810
790
740
620
535
410
350
320
Флагман
+
+
–
+
–
+
+
–
+
+
–
+
+
+
–
+
–
–
Филби
+
–
–
–
–
+
+
–
+
+
–
–
+
+
–
–
–
–
Труженик
+
+
–
+
+
+
+
–
+
+
–
–
+
+
–
–
+
–
Виола
+
+
–
+
–
+
+
–
+
+
–
+
+
+
–
–
–
–
Виола (В)
+
+
–
+
–
+
+
–
+
+
–
+
+
+
–
–
–
–
Капитал
+
+
–
–
+
+
+
–
+
+
–
–
+
+
–
–
–
+
Немчиновский
+
–
–
–
–
+
+
–
+
+
–
–
+
+
–
–
+
–
Демон
–
–
–
+
+
+
+
–
+
+
+
–
+
+
–
–
–
+
Хл-1
+
–
+
–
+
+
+
–
+
+
–
–
+
+
–
–
–
–
Хл-2
+
–
+
–
+
+
+
–
+
+
–
–
+
+
–
–
–
–
Хл-115
+
–
–
–
–
+
+
–
+
+
–
+
+
+
–
–
+
+
Хл-18
–
–
+
–
+
+
+
–
+
+
+
–
+
+
–
+
–
+
Хл-42
+
–
–
–
–
+
+
–
+
+
–
–
+
+
–
–
+
+
L-1238
+
–
–
+
–
+
+
+
+
+
–
–
–
+
+
–
–
–
L-1132
+
–
–
+
–
+
+
+
+
+
–
–
+
+
+
+
–
+
L-851
+
–
–
+
–
+
+
+
+
–
–
–
+
+
+
+
+
–
L-111
–
+
–
–
–
+
+
–
+
+
–
+
+
+
+
–
+
+
L-108
–
+
–
+
+
+
+
+
+
+
–
–
+
+
–
–
+
–
R-64
+
–
–
–
–
+
+
–
+
+
–
–
+
+
–
–
+
+
-
2. Матрица генотипов для тестирования линий, сортов и мутантов гороха посевного ( Pisum sativum L.) с помощью RAPD-маркеров (фрагмент общей матрицы)
3. Специфические маркеры для тестирования различных образцов гороха посевного
П р и м е ч а н и е: (+) и (–) означает соответственно наличие и отсутствие маркера.
|
Сорт, мутант, линия (исходный сорт) |
Длина ДНК-фрагментов, полученных при амплификации с праймером QR2, п.н. |
||||||||||||
|
1600 |
1550 |
1500 |
1400 |
1300 |
1100 |
810 |
790 |
740 |
535 |
410 |
350 |
320 |
|
|
Флагман |
+ |
+ |
– |
+ |
– |
– |
– |
+ |
+ |
– |
+ |
– |
– |
|
Филби |
+ |
– |
– |
– |
– |
– |
– |
– |
+ |
– |
– |
– |
– |
|
Труженик |
+ |
+ |
– |
+ |
+ |
– |
– |
– |
+ |
– |
– |
+ |
– |
|
Виола |
+ |
+ |
– |
+ |
– |
– |
– |
+ |
+ |
– |
– |
– |
– |
|
Капитал |
+ |
+ |
– |
– |
+ |
– |
– |
– |
+ |
– |
– |
– |
+ |
|
Немчиновский |
+ |
– |
– |
– |
– |
– |
– |
– |
+ |
– |
– |
+ |
– |
|
Демон |
– |
– |
– |
+ |
+ |
– |
+ |
– |
+ |
– |
– |
– |
+ |
|
Хл-115 (Торсдаг) |
+ |
– |
– |
– |
– |
– |
– |
+ |
+ |
– |
– |
+ |
+ |
|
Хл-18 (Ранний зеленый) |
– |
– |
+ |
– |
+ |
– |
+ |
– |
+ |
– |
+ |
– |
+ |
|
L-1238 |
+ |
– |
– |
+ |
– |
+ |
– |
– |
– |
+ |
– |
– |
– |
|
L-111 |
– |
+ |
– |
– |
– |
– |
– |
+ |
+ |
+ |
– |
+ |
+ |
|
L-108 |
– |
+ |
– |
+ |
+ |
+ |
– |
– |
+ |
– |
– |
+ |
– |
П р и м е ч а н и е. То же, что в таблице 1.
|
Маркер |
1 Сорт, мутант, линия |
|
B474#530 |
Капитал |
|
B474#300 |
Филби |
|
QR2#1500 |
Хл-18 |
|
K10#570 |
Виола |
|
V#630 |
Виола (В) |
|
V#480 |
Флагман |
|
V#430 |
Хл-18 |
|
AD04#650 |
L-1238 |
|
Q20#650 |
Капитал |
|
Pr10#410 |
Флагман |
На основе матрицы различий всех исследованных образцов гороха посевного UPGMA-методом была построена дендрограмма, отражающая степень различий между RAPD-спект-рами исследованных форм; значимость генеалогических реконструкций определяли методом бутстрепа (13) (см. рис. 2).
Топология генеалогического дерева в участках со значениями больше 50 % может считаться на- дежно установленной. Полученная схема соответствует известной информации о происхождении изучаемых форм.
Для оценки возможности применения RAPD-метода с целью идентификации линий и сортов гороха посевного из исследованных форм было отобрано семь сортов, три маркерные линии и два мутанта. На основе RAPD-спектров изученных форм (см. табл. 1) была разработана матрица генотипов, в которую включены 95 полиморфных маркеров (табл. 2).
Для однозначной идентификации генотипов выбранных сортов, линий и мутантов гороха посевного достаточно анализа 10-15 полиморфных маркеров, то есть возможно определение сорта на основе анализа его RAPD-спектра по нескольким праймерам. В нашей работе каждый из трех RAPD-праймеров (QR2, K10, R03) позволял получать такой набор фрагментов анонимной ДНК, который точно соответствовал одному из RAPD-спектров исследованных 12 сортов, линий и мутантов гороха посевного. Следовательно, определение сортового соответствия выбранных нами образцов гороха посевного (ПЦР, электрофорез и анализ полученных данных) занимает 8 ч (один рабочий день). Среди полиморфных маркеров было выявлено 10 маркеров, амплифицирующихся только в одном генотипе (табл. 3).
Итак, полученные нами данные свидетельствуют о том, что RAPD-метод позволяет получать специфические геномные маркеры, которые можно использовать для паспортизации и идентификации видов и сортов гороха. RAPD-маркеры, специфические для определенных линий или сортов, можно применять для быстрой идентификации этих генотипов, проведения генетического анализа, изучения родственных связей между различными формами гороха посевного, а также в работе по картированию хромосом.
Л И Т Е Р А Т У Р А
-
1. С и в о л а п Ю.М., Т о п ч и е в а Е.А., Ч е б о т а р ь С.В. Идентификация и паспортизация сортов мягкой пшеницы методами RAPD- и SSR-анализа. Генетика, 2000, 36, 1: 44-51.
-
2. Х а в к и н Э.Е. Молекулярная селекция растений: ДНК-технология создания новых сортов сельскохозяйственных культур. С.-х. биол., 2003, 3: 26-41.
-
3. П о м о р ц е в А.А., Л я л и н а Е.В. Идентификация и оценка сортовой чистоты ячменя методом электрофореза запасных белков семян. М., 2003.
-
4. К о ч и е в а З.Г., О г а н и с я н А.С., Р ы с к о в А.П. RAPD-маркеры генома картофеля: клонирование и использование для определения межвидовых и межсортовых различий. Мол. биол., 1999, 33, 5: 893-895.
-
5. Г о с т и м с к и й С.А., К о к а е в а З.Г., Б о б р о в а В.К. Использование молекулярных маркеров для анализа генома растений. Генетика, 1999, 35, 11: 1538-1549.
-
6. К о ч и е в а Е.З., Р ы ж о в а Н.Н., Х р а п а л о в а И.А. и др. Использование метода RAPD-анализа в определении генетического полиморфизма и филогенетических связей у представителей рода Lico-persicon (Tourn.) Mill. Генетика, 2002, 38: 1298-1303.
-
7. К о ж у х о в а Н.Э., С и в о л а п Ю.М. Идентификация и регистрация генотипов кукурузы при помощи молекулярных маркеров. Генетика, 2004, 40, 1: 59-66.
-
8. С и в о л а п Ю.М., С о л о д е н к о Е.А., Б у р л о в В.В. RAPD-анализ молекулярно-генетического полиморфизма подсолнечника ( Heliantus annuus ). Генетика, 1998, 34, 2: 266-271.
-
9. К у д р я в ц е в А.М., М а р т ы н о в С.П., Б р о д ж и о М. и др. Оценка правомерности использования RAPD-анализа для выявления филогенетических связей между сортами яровой твердой пшеницы ( T. durum Defs.). Генетика, 2003, 39, 9: 1237-1246.
-
10. T o r r e s A.M., W e e d e n N.F., M a r t i n A. Linkage among sozyme, RFLP and RAPD markers in Vicia faba . Theor. Appl. Genet., 1993, 5: 937-945.
-
11. N e i M., L i W.-H. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. of the Nat. Acad. of Sci. (USA), 1979, 76: 5269-5273.
-
12. V a n d e P e e r Y. Treecon for Windows: a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment. Computer Application in the Biosciences, 1994, 10, 5: 569-570.
-
13. F e l s e n s t e i n J. Confidence limits in phylogenies an approach using bootstrap. Evolution, 1985, 39, 4: 783-791.
-
14. К о в е з а О.В., К о к а е в а З.Г., К о н о в а л о в Ф.А. и др. Использование RAPD-метода для исследования генома гороха ( Pisum sativum L.). Генетика, 2005, 41, 4: 1-8.


