Индукция PTI (pattern-triggered immunity) и транскрипционное репрограммирование при персистентной аллексивирусной инфекции
Автор: Архипов А.В., Вишниченко В.К.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Иммунитет и болезни растений
Статья в выпуске: 5 т.53, 2018 года.
Бесплатный доступ
Растения обладают сложной антивирусной иммунной системой, в функционировании которой ключевую роль играет РНК-сайленсинг - индуцибельный молекулярный механизм, контролирующий уровень репликации вируса и степень выраженности симптомов. В ряде случаев сайленсинг подавляется при действии вирусных белков-супрессоров, предотвращающих фрагментацию или блокирование трансляции вирусных РНК. Активность белков-супрессоров считают непременным условием успешной репродукции фитовирусов. В то же время РНК-сайленсинг нельзя рассматривать исключительно как беспощадный антивирусный механизм, действие которого необходимо полностью подавить, в действительности, по-видимому, происходит тонкая регуляция этого процесса. Известно, что многие фитовирусы кодируют супрессоры, обладающие очень слабой или преходящей активностью (transiently active suppressors). В частности Х вирус шалота (ХВШ), прототип рода Allexivirus, успешно репродуцируется и формирует персистентную инфекцию в отсутствие активного белка-супрессора и, следовательно, преодолевает иммунный барьер сайленсинга с помощью иного механизма...
Аллексивирусы, х вирус шалота, персистентная инфекция, рнк-сайленсинг, врожденная иммунная система растений, транскрипционное репрограммирование
Короткий адрес: https://sciup.org/142216601
IDR: 142216601 | УДК: 632.3:635.263:578.85/.86:578.2:577.217 | DOI: 10.15389/agrobiology.2018.5.947rus
Текст научной статьи Индукция PTI (pattern-triggered immunity) и транскрипционное репрограммирование при персистентной аллексивирусной инфекции
Растения обладают сложной защитной антивирусной системой, в
Работа выполнена в рамках научно-исследовательского проекта «Исследование молекулярных механизмов супрессии РНК-сайленсинга вирусами растений (на модели вирусного комплекса шалота и вируса шарки сливы)» (Госзадание ¹ 0574-2015-0003 на 2017 год).
процессе функционирования которой ключевую роль играет РНК-сайленсинг — индуцируемый вирусными двуцепочечными РНК молекулярный механизм, контролирующий уровень репликации вируса и степень выраженности симптомов (1, 2). При этом активность вирусных белков-супрессоров, предотвращающих фрагментацию или блокирование трансляции вирусных РНК в процессе сайленсинга, считают непременным условием успешной репродукции фитовирусов (3, 4). Известно, однако, что многие фитовирусы кодируют супрессоры, обладающие очень слабой или преходящей активностью (transiently active suppressors) (5). В частности, как мы показали ранее (6), Х вирус шалота (ХВШ) — прототип рода Allexivirus — вызывает персистентную инфекцию и успешно репродуцируется в отсутствие активного белка-супрессора, следовательно, преодолевает иммунный барьер сайленсинга с помощью иного механизма. Мы предположили, что таким механизмом может быть специфическое транскрипционное репрограммирование (ТРП) (7, 8), обусловленное PTI (Pattern-triggered immunity) — первой «линией обороны» врожденной иммунной системы растений (9); в результате ТРП может избирательно изменяться уровень экспрессии целого ряда генов-мишеней, участвующих в репродукции фитовирусов, и, в частности, подавляться до критического уровня экспрессия ключевых факторов РНК-сайленсинга: DCL-белков, белков-аргонавтов (Ago) и клеточных РНК-зависимых РНК-полимераз (RDR). Полученные нами ранее результаты свидетельствовали в пользу этого предположения: инфекция ХВШ оказалась сопряженной с подавлением транскрипции генов DCL-белков и RDR в корнях и листьях инфицированных растений (10).
В последние 2-3 года в нескольких зарубежных лабораториях были получены экспериментальные данные, свидетельствующие о том, что фитовирусы подобно бактериальным патогенам индуцируют процесс, аналогичный классическому PTI (11-13); при этом индукторами (вирусными PAMPs) служат двуцепочечные репликативные формы вирусных РНК (12, 13). Таким образом, в растительной клетке функционируют по меньшей мере два антивирусных механизма, запускаемых при участии двуцепочечных РНК, — РНК-сайленсинг и PTI.
В классическом варианте PTI инициируется в результате специфического узнавания консервативных молекулярных структур (паттернов) патогена (PAMPs, Pathogen-associated molecular patterns), например флагеллина бактерий, клеточными рецепторами (PRRs, Pattern recognition receptors), представляющими собой RLKs (receptor-like kinases) или LRR-RKs (leucine-rich repeat receptor kinases) и локализованными на плазматической мембране растительной клетки (14-18). При взаимодействии молекулярных паттернов с PRRs наблюдается немедленная и интенсивная индукция транскрипционного репрограммирования, что выражается, в частности, в дифференциальной экспрессии ряда белков, служащих маркерами PTI (19). Специфические антивирусные PRR растительной клетки до настоящего времени не идентифицированы, хотя косвенно их существование достаточно убедительно подтверждается недавним исследованием роли полифункционального ко-шаперона Hop/Sti1 в симптоматике инфекции вируса Y картофеля (20).
В России это направление изучения молекулярных механизмов антивирусного фитоиммунитета пока не получило должного развития. В настоящей работе впервые в мировой литературе представлены экспериментальные данные, свидетельствующие об индукции PTI в растениях в условиях персистентной вирусной инфекции и об участии этого механизма в транскрипционном репрограммировании; показано, что в результате ТРП в инфицированных растениях шалота наблюдается подавление экс948
прессии всех факторов сайленсинга и некоторых R-генов (например, гомологов гена Tm22 ).
В представленной работе нами впервые получены экспериментальные данные, свидетельствующие об индукции PTI в растениях шалота в условиях персистентной вирусной инфекции и об участии этого механизма в транскрипционном репрограммировании.
Целью настоящего исследования была экспериментальная проверка нашей гипотезы (10) о ключевой роли транскрипционного репрограммирования как механизма супрессии РНК-сайленсинга, а также предположения, согласно которому транскрипционное репрограммирование сопряжено с PTI и в результате этого процесса могут избирательно изменяться уровни экспрессии ряда генов, участвующих в репродукции фитовирусов (например, TCTP-комплекс) (21).
Методика. Растения шалота ( Allium cepa L. var. aggregatum L.G. Don) размножали луковицами в условиях персистентной инфекции ХВШ. В каждом эксперименте высаживали 5-6 луковиц одного кластера побегов, объединенные образцы корней, проростков и листьев отбирали через 3 сут и через 2 нед после высадки луковиц.
Получение препаратов суммарной РНК и детекцию вируса методом ПЦР в классическом варианте осуществляли, как описано нами ранее (10).
Поиск нуклеотидных последовательностей, кодирующих гомологи выбранных генов-мишеней, в транскриптоме наиболее близкого к шалоту вида A. cepa L. проводили с помощью программ tblastn, tblastx и базы данных TSA (Transcriptome Shotgun Assembly, . При конструировании праймеров, специфичных в отношении шалотных гомологов исследуемых генов-мишеней, был применен подход, использованный нами ранее (10, 23); набор праймеров генерировали с помощью программы Primer3 v.4.1.0 .
Представленности транскриптов, кодирующих белки-мишени в исследуемых образцах, определяли методом ПЦР в реальном времени в формате Comparative CT Experiment: алгоритм delta-delta CT; калибратор — безвирусные сеянцы шалота; нормалайзер — 18S РНК, что в условиях наблюдаемого динамического процесса транскрипционного репрограммирования представляется оптимальным выбором (22). Амплификатор — 7500/7500 Fast Real-Time PCR Systems или QuantStudio («Applied Biosystems», США), набор реагентов SYBR® Green Reagents (ОАО «Синтол», Москва).
Результаты. В таблице 1 приведены маркеры PTI, в таблице 2 — соответствующие праймеры. В первой серии экспериментов исследовали представленность транскриптов 10 классических маркеров PTI (рис. 1).
-
1. Гены-мишени, уровни экспрессии которых исследовали в настоящей работе в условиях персистентной инфекции шалота ( Allium cepa L. var. aggregatum ) Х вирусом шалота
-
2. Нуклеотидные последовательности праймеров, использованных при определении уровней экспрессии маркеров Pattern-Triggered Immunity (PTI) при персистентной инфекции шалота ( Allium cepa L. var. aggregatum ) Х вирусом шалота (ПЦР в реальном времени)
Праймер
Последовательность
ID транскрипта Allium cepa L. в базе данных TSA (NCBI)
ARM 530-L
5´- ATGATGCGGGCCTAGTAGAC -3´
АТ7ТТ7Л1 Л7 1 ^Л/1 1
ARM 766-R
5´- CTCCCTCGATCAGTCCACTC -3´
GETF01031504.1
RBOHD 2641-L
5´- GTTTGATCCTAGACGACGCG -3´
ат?т>кгл 1 ллл/^со 1
RBOHD 2868-R
5´- TCAACATACCCGACCCGAAA -3´
GBRN01002659.1
EDS5 303-L
5´- TCGCTTGGTCTTGGCTTCTA -3´
EDS5 549-R
5´- CGTCTGAGAATCCAACGACG -3´
GBRQ01023449.1
LOX3 1753-L
5´- ATGCCACTCGTACGCTTTTC -3´
LOX3 1943-R
5´- GACGCCTGCATCATTAGAGC -3´
GBRQ01024907.1
BRI1 1541-L
5´- TGTTCCCGCTCAGCTGATTA -3´
АТ?Т? АЛ 1 1 1
BRI1 1749-R
5´- TACTTTCGGTGGCAATGGGA -3´
SOBIR 812-L
5´- CAAGTCATGCAAGCTTCCGT -3´
SOBIR 1041-R
5´- CTGGAAAGATGATCGCGGTG -3´
GBRQ01012958.1
AtCRK4 1496-L
5´- CTTTCTTGACCTTGGCCTCG -3´
AtCRK4 1731-R
5´- TCCCCAGCTAAGCACATCAA -3´
GBGJ01061169.1
SERK1 1291-L
5´- CTTCTTCAGCGGGAACATCG -3´
SERK1 1478-R
5´- TCCACCTCCTCCATTTGTCC -3´
GBGJ01064935.1
PR1 443-L
5´- GTCAAGATCGGTTGCGCTAG -3´
PR1 660-R
5´- CCAAGCAAACTCTCATCGCA -3´
GBJZ01171295.1
PR5 616-L
5´- ACTGTCTACGGGCCCAAAAT -3´
ат?АТЛ1 Л700А4 1
GBGJ01079964.1
PR5 803-R
5´- ATATGCTGCCTCCGGAACTC -3´
18s rRNA-L
5´- CATCAGCTCGCGTTGACTAC -3´
18s rRNA-R
5´- GATCCTTCCGCAGGTTCAC -3´
(22)
-
3. Нуклеотидные последовательности праймеров, использованных при определении уровней экспрессии факторов РНК-сайленсинга, NB-LRR рецепторов и генов, принимающих участие в репродукции фитовирусов, при персистентной инфекции шалота ( Allium cepa L. var. aggregatum ) Х вирусом шалота (ПЦР в реальном времени)
Праймер
Последовательность
ID транскрипта Allium cepa в базе данных TSA (NCBI)
Ago-L-672
Ago-R-851
5´-AACTCCCAAGAAGCTTTGCG-3´
5´-CCCTCCTTGAGCAGTTCTGA-3´
GBGJ01050630.1
Tm22-L-3013
Tm22-R-3257
5´- TCGTGGGCTCTTTCACTGAT-3´
5´- CACCCGCTTCATTGGTGTAG-3´
GBRN01023560
FRK1-L-421
FRK1-R-635
5´-AGTCACGCTCAATGGCAATG-3´
5´-CTGCCGCAACATCATAGCAT-3´
GAAO01012059.1
NHL10-L-327
NHL10-R-572
5´-TGCTCCTCACATCGTTCACA-3´
5´-AGCTCACCACCTTCACTCTC-3´
GBRQ01023138.1
ACRE31-L-473
ACRE31-R-699
5´-GCAGTTCTTCGAAAGCAGGA-3´
5´-ATTGAGCACATCCTCCCCTT-3´
GBRO01073928.1
ACRE132-L-272
ACRE132-R-451
5´-GCCATGCCTCAACCTGATTT-3´
5´-CCTTCTTGATCGGGAAAGCG-3´
GBRQ01011764.1
PR6-L-100
PR6-R-274
5´-ATGAGGGGTACATGGCAGAC-3´
5´-AAGCATCGGAAGCGAAGAAG-3´
GBRQ01165078.1
LTP pcr-59- L
LTP pcr-274-R
GCA-GTC-CGT-ATG-CAA-AT
TAG-GGT-TTC-GTC-TCA-GAC-CG
AF004946.1
WRKY-L-1367
WRKY-R-1550
5´-ACGTGGAAAGGGCATCAAAC-3´
5´-GCGACCGGTCTTTGAACATT-3´
GBRO01047677.1
TCTP-L-172
TCTP-R-382
5´-AGGGCAAGTGGGTAGTTCAA-3´
5´-TCCAATTTCGAAGGCAGAAGT-3´
JR844934.1
PIRL-L-84
PIRL-R-377
DBP-L-392
DBP-R-623
5´-ATCATGGATCCAAGCCCCAA-3´
5´-CTTTGCGAGGTCAACAGCTT-3´
5´-AGGGTCGTTGTGCTCTTGTA-3´
5´-CAACGGTCAGCTCAACGTAG-3´
GAAN01019083
GBRO01024689.1
СBP-L-429
СBP-R-673
5´- GAAGCAGAGGGAAAGCAACC-3´
5´- AAGCCAACACCATCATGCAG-3´
GBRO01059419.1
GRF6-L-607
GRF6-R-829
5´-TCCAGTCTTGAATTCGGCCA-3´
5´-TTCGATCGAGCAGAAGGAGG-3´
GAAN01023832.1
18S rRNA-L
18S rRNA-R
CATCAGCTCGCGTTGACTAC GATCCTTCCGCAGGTTCAC
(22)
Символ гена ] Название белка — продукта экспрессии I Вид растения, ID ортолога
Гены-маркеры PTI
ARM Armadillo repeat family proteins Arabidopsis thaliana AT3G02840
RBOHD Respiratory Burst Oxidase Homologue D Arabidopsis thaliana AT5G47910
EDS5 Mate Efflux Family Protein (Enhanced Disease Susceptibility 5) Arabidopsis thaliana AT4G39030
|
LOX3 |
Lipoxygenase 3 |
Arabidopsis thaliana |
AT1G17420 |
|
BRI1 |
Bri1-like 3 (Brassinosteroid insensitive) |
Arabidopsis thaliana |
AT3G13380 |
|
SOBIR |
Leucine-Rich Repeat Protein Kinase Family Proteins (Suppressor Of Bir1-1/Evershed) |
Arabidopsis thaliana |
AT2G31880 |
|
CRK4 |
Calcium-dependent protein kinase (CDPK) family proteins (Cysteine-rich receptor-like kinase 4) |
Arabidopsis thaliana |
AT5G24430 |
|
SERK1 |
Somatic embryogenesis receptor-like kinase 1 |
Arabidopsis thaliana |
AT1G71830 |
|
PR1 |
Pathogenesis-related proteins group 1 |
Arabidopsis thaliana |
AT2G14610 |
|
PR5 |
Pathogenesis-related proteins group 5 |
Arabidopsis thaliana |
AT1G75040 |
|
NHL10 |
Late embryogenesis abundant (lea) hydroxyproline-rich glycoprotein family |
Arabidopsis thaliana |
NP_181142.1 |
|
ACRE31 |
Avr9/cf-9 rapidly elicited protein 31 |
Nicotiana tabacum |
AAG43547.1 |
|
ACRE132 |
Avr9/cf-9 rapidly elicited protein 132 |
Nicotiana tabacum |
AF211532.1 |
|
Другие гены-мишени |
|||
|
DCL |
Dicer-Like proteins |
Allium sativum EPP005KGAA12S003959 |
|
|
RDR 6 |
RNA-dependent RNA polymerase 6 |
Arabidopsis thaliana NP_001327617.1 |
|
|
AGO |
Argonaute family proteins |
Triticum aestivum |
AGB34311.1 |
|
Tm22 |
ToMV resistance protein |
Solanum lycopersicum AAQ10736.1 |
|
|
PR6 |
Pathogenesis-related proteins group 6 |
Arabidopsis thaliana |
NP_199170.2 |
|
LTP |
Lipid transfer proteins (Pathogenesis-related proteins group 14), (Antimicrobial protein Ace-amp1 precursor mRNA) |
Allium cepa |
AF004946.1 |
|
WRKY |
WRKY transcription factors family proteins |
Arabidopsis thaliana |
AEC09374.1 |
|
TCTP |
Translationally controlled tumor proteins |
Jatropha curcas |
EF091818.1 |
|
PIRL |
Plant intracellular Ras group-related LRR proteins |
Arabidopsis thaliana |
NP_196204.1 |
|
DBP1 |
DNA-binding protein phosphatase 1 |
Arabidopsis thaliana NP_001324148.1 |
|
|
СBP60g |
СBP60G (Calmodulin-binding protein 60 G) |
Arabidopsis thaliana |
OAO89604.1 |
|
GRF6 |
G-box regulating factor 6 |
Arabidopsis thaliana |
NP_001190276 |
|
FRK1 |
FRK (Fertilization-related kinase 1) |
Arabidopsis thaliana |
OAP09570.1 |
|
Примеч |
а ни е. Указаны ортологи, с помощью которых были получены оптимальные |
результаты при |
|
|
конструировании соответствующих праймеров (см. табл. 2, 3). |
|||
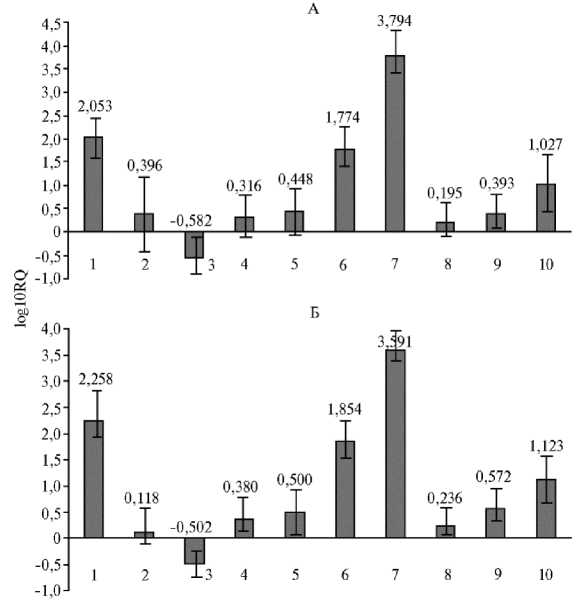
Рис. 1. Представленность транскриптов генов-маркеров Pattern-Triggered Immunity (PTI) в листьях (А) и в корнях (Б) растений шалота ( Allium cepa L. var. aggregatum ) при персистентной инфекции Х вирусом шалота через 2 нед после высадки луковиц: RQ — Relative Quantification (изменения в экспрессии мРНК относительно величины для РНК внутреннего контроля); 1 — ARM , 2 — BRI1 , 3 — CRK4 , 4 — EDS5 , 5 — LOX3 , 6 — PR1 , 7 — PR5 , 8 — RBOHD , 9 — SERK1 , 10 — SOBIR (обозначения генов-маркеров PTI см. таблицу 1).
Как следует из полученных результатов (см. рис. 1), в той или иной мере вирусная инфекция влияет на экспрессию всех исследованных генов, однако при этом выявляется группа генов с очень высокой экспрессией и в листьях, и в корнях инфицированных растений: SOBIR: log10RQ ∼ 1.0; ARM: log10RQ ∼ 2.0; PR1: log10RQ ∼ 2.0; PR5: — log10RQ ∼ 4.0 (!). Следовательно, можно сделать вывод, что ХВШ в период инициирования инфекции взаимодействует с компонентами механизма PTI и в результате индукции PTI запускается процесс ТРП.
На начальной стадии инфекционного процесса в проростках (рис. 2, А) активировалась экспрессии всех генов-мишеней (табл. 3) (за исключением AGO и PIRL ), в том числе четырех дополнительных маркеров PTI и nsLTP (= PR14, Pathogenesis-Related Proteins Group 14). Через 2 нед инфекции ситуация в целом изменялась на противоположную (см. рис. 2, Б): в листьях подавлялась экспрессия большинства генов-мишеней, в том числе всех факторов РНК-сайленсинга (более других — белков-аргонавтов), трех маркеров PTI, NB-LRR рецепторов и белков CBP60g, контролирующих синтез салициловой кислоты. Однако в те же сроки в листьях экспрессия ACRE 132 и LTP (маркеры PTI) демонстрировала заметную тенденцию к росту, а экспрессия TCTP оставалась достаточно высокой и стабильной.
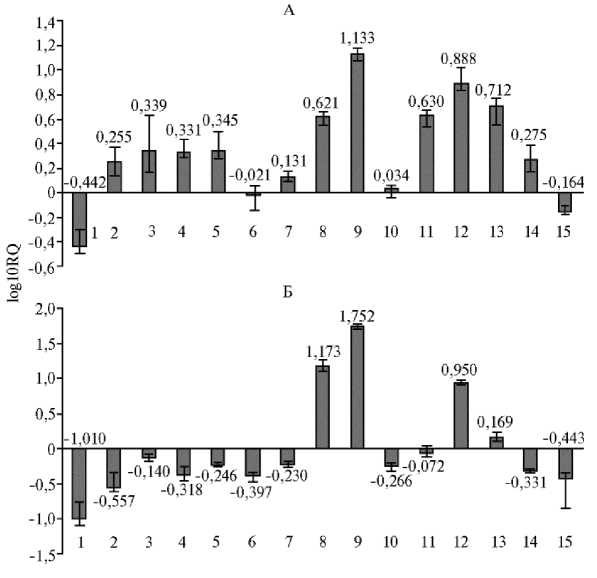
Рис. 2. Представленность транскриптов генов-мишений в надземных органах растений шалота ( Allium cepa L. var. aggregatum ) на разных стадиях инфекции Х вирусом шалота: А — проростки (3 сут после высадки луковиц), Б — листья ( 2 нед после высадки луковиц); RQ — Relative Quantification (изменения в экспрессии мРНК относительно величины для РНК внутреннего контроля); 1 — AGO , 2 — DCL , 3 — RDR , 4 — Tm22 , 5 — FRK1 , 6 — NHL10 , 7 — ACRE31 , 8 — ACRE32 , 9 — LTP , 10 — WRKY , 11 — CBP60g , 12 — TCTP , 13 — GRF6 , 14 — DBP1 , 15 — PIRL (обозначения генов-маркеров PTI см. таблицу 1).
В корнях (рис. 3, А, Б) ингибирование экспрессии генов-мишеней отмечали уже на начальной стадии инфекционного процесса, а через 2 нед подавленной оказывается уже большая часть генов-мишеней, но экспрес952
сия LTP снижалась незначительно.
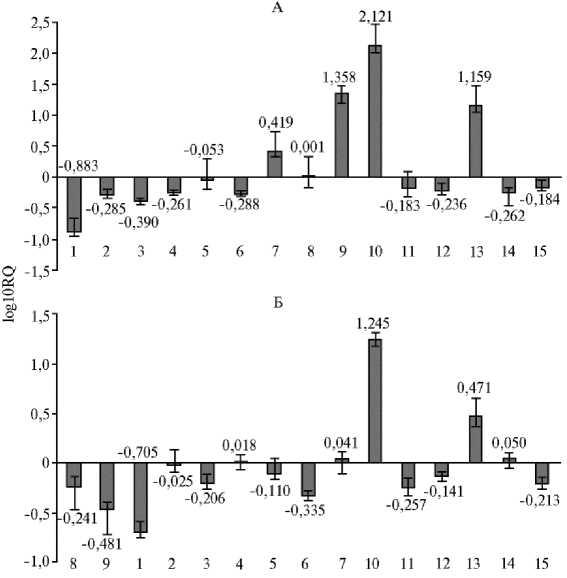
Рис. 3. Представленность транскриптов генов-мишений в корнях растений шалота ( Allium cepa L. var. aggregatum ) на разных стадиях инфекции Х вирусом шалота: А — 3 сут после высадки луковиц, Б — 2 нед после высадки луковиц; RQ — Relative Quantification (изменения в экспрессии мРНК относительно величины для РНК внутреннего контроля); 1 — AGO , 2 — FRK1 , 3 — NHL10 , 4 — CBP60g , 5 — DBP1 , 6 — DCL , 7 — GRF6 , 8 — ACRE31 , 9 — ACRE132 , 10 — LTP , 11 — PIRL , 12 — RDR , 13 — TCTP , 14 — Tm22 , 15 — WRKY (обозначения генов-маркеров PTI см. таблицу 1).
Из представленных результатов следует, что в условиях персистентной инфекции ХВШ индуцирует в корнях и листьях растений динамический процесс транскрипционного репрограммирования (ТРП), избирательное изменение экспрессии ряда генов-мишеней, включая гены, кодирующие белки-маркеры PTI, факторы РНК-сайленсинга, NB-LRR рецепторы, липид-трансферные белки, а также белки, принимающие участие в репликации вируса (TCTP-комплекс). В конечном итоге в исследуемой патосистеме подавляется экспрессии всех факторов сайленсинга и некоторых NB-LRR рецепторов, что подтверждает нашу первоначальную гипотезу. Следует отметить высокую степень индукции фактора транскрипции WRKY (Group III) и генов PR-белков, в частности PR1, PR5 и PR14 (nsLTP), как характерную особенность репрограммирования.
Мы полагаем, что индукция PTI, ETI (Effector triggered immunity) (9) и обусловленное этим транскрипционное репрограммирование — механизмы, общие для всех РНК-содержащих вирусов. В результате реализации рецепторных и сигнальных функций, присущих факторам врожденной иммунной системы, в растениях активируются многочисленные и разнообразные иммунные реакции, в то числе РНК-сайленсинг. Их задача — контроль репродукции вируса, но не полная его элиминация. Со своей стороны, вирусы выработали механизмы противодействия иммунным реакциям растения-хозяина, в частности через белки-супрессоры. Многие фитовирусы кодируют супрессоры с очень слабой или преходящей активностью ли- бо неактивные вовсе. Таким образом, с точки зрения вируса РНК-сайленсинг — далеко не абсолютное оружие клетки: его можно проигнорировать или ослабить с помощью белков-супрессоров либо транскрипционного репрограммирования. Выбор вирусом способа подавления системы антивирусного иммунитета зависит от того, как растение-хозяин решает дилемму «рост (развитие) или защита» (concept of growth-defence tradeoffs) (24).
Описанный в настоящем сообщении процесс ТРП обладает органной специфичностью и выраженным двухфазным характером: на ранних стадиях развития инфекции экспрессия большей части генов-мишеней активируется, на более поздних — подавляется. Анализ представленных фактов позволяет также сделать вывод о том, что в проростках, листьях и корнях инфицированных растений индуктором транскрипционного репрограммирования, вероятно, служит PTI, но не сайленсинг с участием не идентифицированного в настоящей работе паралога семейства Ago-белков, экспрессия которого, как следует из представленных данных (см. рис. 2, 3), интенсивно подавляется на всех стадиях инфекционного процесса. Можно, однако, предположить, что на той или иной стадии инфекции транскрипционное репрограммирование может быть обусловлено также активностью специфического комплекса факторов сайленсинга (например, Ago2 + DCL4 + RDR1), экспрессия которых в условиях настоящего эксперимента, вероятно, не подавлялась. В результате этой активности генерируются эндогенные малые интерферирующие РНК (принадлежат к обширному классу активируемых вирусной инфекцией клеточных siRNA — vasiRNAs, virus-activated siRNAs) (25), мишенями которых служат различные гены растения-хозяина. Не исключено также, что при персистентной инфекции ХВШ индукция транскрипционного репрограммирования может быть обусловлена РНК-сайленсингом с участием малых интерферирующих РНК, источником которых служит вирусный геном (26). Кроме того, в исследуемой патосистеме в результате действия неизвестного пока механизма может изменяться уровень экспрессии определенных видов микроРНК, контролирующих процесс ТРП (27).
Мы считаем, что ключевое направление дальнейших исследований механизма индукции и роли ТРП в формировании антивирусного фитоиммунитета — идентификация участвующих в PTI антивирусных PRR растительной клетки и выяснение молекулярных механизмов взаимодействия этих рецепторов с репликативными (двуцепочечными) формами вирусных РНК. Фундаментальные особенности индуцируемого вирусными PAMPs процесса PTI дают основание полагать, что клонирование и межвидовой перенос генов антивирусных PRR растительной клетки может оказаться перспективной технологией создания новых форм сельскохозяйственных растений, обладающих высокой длительной резистентностью к широкому кругу патогенных вирусов (28).
Итак, впервые в мире экспериментально показано, что персистентная инфекция Х вируса шалота индуцирует в корнях и листьях растений шалота динамический процесс транскрипционного репрограммирования, в результате чего наблюдается изменение экспрессии широкого спектра генов-мишеней, включая гены, кодирующие белки-маркеры PTI, факторы РНК-сайленсинга, NB-LRR рецепторы, PR-белки, а также белки, принимающие участие в репликации вируса.
Список литературы Индукция PTI (pattern-triggered immunity) и транскрипционное репрограммирование при персистентной аллексивирусной инфекции
- Calil I.P., Fontes E.P.B. Plant immunity against viruses: antiviral immune receptors in focus. Annals of Botany, 2016, 119(5): 711-723 () DOI: 10.1093/aob/mcw200
- Ghoshal B., Sanfaçon H. Symptom recovery in virus-infected plants: revisiting the role of RNA silencing mechanisms. Virology, 2015, 479-480: 167-179 ( ) DOI: 10.1016/j.virol.2015.01.008
- Burgyan J., Havelda Z. Viral suppressors of RNA silencing. Trends Plant Sci., 2011, 16(5): 265-72 ( ) DOI: 10.1016/j.tplants.2011.02.010
- Csorba T., Kontra L., Burgyan J. Viral silencing suppressors: tools forged to fine-tune host-pathogen coexistence. Virology, 2015, 479-480: 85-103 ( ) DOI: 10.1016/j.virol.2015.02.028
- Sanfaçon H. Grand challenge in plant virology: understanding the impact of plant viruses in model plants, in agricultural crops, and in complex ecosystems. Front. Microbiol., 2017, 8: 860 ( ) DOI: 10.3389/fmicb.2017.00860
- Архипов А.В., Соловьев А.Г., Вишниченко В.К. Репродукция Х вируса шалота в отсутствие собственного активного белка-супрессора. Доклады РАСХН, 2013, 2: 4-7.
- Li B., Meng X., Shan L., He P. Transcriptional regulation of pattern-triggered immunity in plants. Cell Host & Microbe, 2016, 19(5): 641-650 ( ) DOI: 10.1016/j.chom.2016.04.011
- Hillmer R.A., Tsuda K., Rallapalli G., Asai S., Truman W., Papke M.D., Sakakibara H., Jones J.D.G., Myers C.L., Katagiri F. The highly buffered Arabidopsis immune signaling network conceals the functions of its components. PloS Genet., 2017, 13(5): e1006639 ( ) DOI: 10.1371/journal.pgen.1006639
- Jones J.D.G., Dangl J.L. The plant immune system. Nature, 2006, 444: 323-329 ( ) DOI: 10.1038/nature05286
- Архипов А.В., Соловьев А.Г., Вишниченко В.К. Персистентная инфекция Х-вируса шалота сопряжена с подавлением транскрипции генов клеточной РНК-зависимой РНК-полимеразы и DCL-белков в корнях инфицированных растений. Молекулярная биология, 2017, 51(1): 126-130 ( ) DOI: 10.7868/S0026898417010037
- Korner C.J., Klauser D., Niehl A., Dominguez-Ferreras A., Chinchilla D., Boller T., Heinlein M., Hann1 D.R. The immunity regulator BAK1 contributes to resistance against diverse RNA viruses. Mol. Plant Microbe Interact., 2013, 26: 1271-1280 ( ) DOI: 10.1094/MPMI-06-13-0179-R
- Niehl A., Wyrsch I., Boller T., Heinlein M. Double-stranded RNAs induce a pattern-triggered immune signaling pathway in plants. New Phytol., 2016, 211(3): 1008-1019 ( ) DOI: 10.1111/nph.13944
- Mitter N., Worrall E.A., Robinson K.E., Ping X.Z., Carroll B.J. Induction of virus resistance by exogenous application of double-stranded RNA. Curr. Opin. Virol., 2017 26: 49-55 ( ) DOI: 10.1016/j.coviro.2017.07.009
- Zipfel C. Pattern-recognition receptors in plant innate immunity. Curr. Opin. Immunol., 2008, 20(1): 10-16 ( ) DOI: 10.1016/j.coi.2007.11.003
- Zipfel C. Plant pattern-recognition receptors. Trends Immunol., 2014, 35: 345-351 ( ) DOI: 10.1016/j.it.2014.05.004
- Macho A.P., Zipfel C. Plant PRRs and the activation of innate immune signaling. Mol. Cell, 2014, 54(2): 263-272 ( ) DOI: 10.1016/j.molcel.2014.03.028
- Bartels S., Boller T. Quo vadis, Pep? Plant elicitor peptides at the crossroads of immunity, stress, and development. J. Exp. Bot., 2015, 66(17): 5183-5193 ( ) DOI: 10.1093/jxb/erv180
- Boutrot F., Zipfel C. Function, discovery, and exploitation of plant pattern recognition receptors for broad-spectrum disease resistance. Annu. Rev. Phytopathol., 2017, 55: 257-286 ( ) DOI: 10.1146/annurev-phyto-080614-120106
- Hruz T., Laule O., Szabo G., Wessendorp F., Bleuler S., Oertle L., Widmayer P., Gruissem W., Zimmermann P. Genevestigator v3: a reference expression database for the meta-analysis of transcriptomes. Advances in Bioinformatics, 2008, 2008: Article ID 420747 ( ) DOI: 10.1155/2008/420747
- Lamm C.E., Kraner M.E., Hofmann J., Börnke F., Mock H.-P., Sonnewald U. Hop/Sti1 -a two-faced cochaperone involved in pattern recognition receptor maturation and viral infection. Front. Plant Sci., 2017, 8: 1754 ( DOI: 10.3389/fpls.2017.01754
- Bruckner F.P., Xavier A.D., Cascardo R.S., Otoni W.C., Zerbini F.M., Alfenas-Zerbini P. Translationally controlled tumour protein (TCTP) from tomato and Nicotiana benthamiana is necessary for successful infection by a potyvirus. Mol. Plant Pathol., 2017, 18(5): 672-683 ( ) DOI: 10.1111/mpp.12426
- Meng X., Chen Q., Fan H., Song T., Cui N, Zhao J., Jia S., Meng K. Molecular characterization, expression analysis and heterologous expression of two translationally controlled tumour protein genes from Cucumis sativus. PLoS ONE, 2017, 12(9): e0184872 ( ) DOI: 10.1371/journal.pone.0184872
- Архипов А.В., Вишниченко В.К. Обнаружение нуклеотидных последовательностей, кодирующих DCL-белки в растениях шалота A. cepa L. var. aggregatum L.G. Don. Сельскохозяйственная биология, 2011, 5: 51-55.
- Huot B., Yao J., Montgomery B.L., He S.Y. Growth-defense tradeoffs in plants: a balancing act to optimize fitness. Mol. Plant, 2014, 7(8): 1267-1287 ( ) DOI: 10.1093/mp/ssu049
- Cao M., Du P., Wang X., Yu Y.Q., Qiu Y.H., Li W., Gal-On A., Zhou C., Li Y., Ding S.W. Virus infection triggers widespread silencing of host genes by a distinct class of endogenous siRNAs in Arabidopsis. PNAS USA, 2014, 111(40): 14613-14618 ( ) DOI: 10.1073/pnas.1407131111
- Xu D., Zhou G. Characteristics of siRNAs derived from Southern rice black-streaked dwarf virus in infected rice and their potential role in host gene regulation. Virol. J., 2017, 14(1): 27 ( ) DOI: 10.1186/s12985-017-0699-3
- Bao D., Ganbaatar O., Cui X., Yu R., Bao W., Falk B.W., Wuriyanghan H. Down-regulation of genes coding for core RNAi components and disease resistance proteins via corresponding microRNAs might be correlated with successful Soybean mosaic virus infection in soybean. Mol. Plant Pathol., 2018, 19(4): 948-960 ( ) DOI: 10.1111/mpp.12581
- Nicaise V. Boosting innate immunity to sustainably control diseases in crops. Curr. Opin. Virol., 2017, 26: 112-119 ( ) DOI: 10.1016/j.coviro.2017.07.030


