Инфекционная опасность носителей провируса вируса бычьего лейкоза и ее оценка в связи с лейкоцитозом
Автор: Косовский Г.Ю., Глазко В.И., Андрейченко И.А., Ковальчук С.Н., Глазко Т.Т.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Вирусные и бактериальные инфекции - молекулярная идентификация возбудителя
Статья в выпуске: 4 т.51, 2016 года.
Бесплатный доступ
Распространение вируса бычьего лейкоза (Bovine leukemia virus, BLV) наносит существенный экономический ущерб молочному и мясному скотоводству. Одна из причин заключается в том, что до сих пор не удается разработать оптимальные способы предупреждения заболевания. Ситуация усугубляется отсроченным проявлением лимфолейкоза, который примерно у 5-7 % животных наблюдается через 5-10 лет после инфицирования. Подразделенность инфицированных BLV В-клеток на продуцентов зрелых вирусных частиц и предшественников формирования лимфом приводит к необходимости рассматривать отдельно инфекционную опасность животных и прогноз развития у них лимфолейкоза. Тестирование антител к вирусным антигенам или встройки провирусной ДНК в геном хозяина не позволяет охарактеризовать животных с точки зрения роли в распространении инфекции, что наиболее существенно для ее контроля. Поэтому особую актуальность приобретают методы, позволяющие оценить инфекционную опасность инфицированных особей, связанную с большим числом В-лимфоцитов, способных продуцировать зрелые вирусные частицы. В настоящей работе мы сопоставили содержание вирусной РНК в образцах крови и пролиферативную активность лейкоцитов, чтобы изучить возможность использовать эти показатели для индивидуального тестирования животных по обеим характеристикам. С использованием разработанных нами праймеров к генам BLV gag и pol (Г.Ю. Косовский с соавт., 2013) у черно-пестрых голштинизированных коров ( n = 57) из промышленного стада определили наличие или отсутствие провирусной ДНК BLV, интегрированной в геном, и выявили группу инфицированных особей. С помощью RT-PCR (reverse transcription polymerase chain reaction) и праймеров к консервативному фрагменту гена pol BLV оценили относительное количество экспрессирующегося провируса у индивидуальных инфицированных животных. Дополнительно у всех коров, включенных в анализ, подсчитали число лейкоцитов в образцах крови. Инфицированные животные по этому показателю разделились на две подгруппы: у первой значения не превышали 17х109 кл/л, у второй - оказались существенно выше, достигая у некоторых особей 28х109 кл/л. У коров, свободных от инфекции (за исключением одной особи), число лейкоцитов было ниже 12х109 кл/л. Оказалось, что достаточное для выявления в RT-PCR количество РНК вируса BLV обнаруживается только у второй подгруппы инфицированных коров. Повышение количества лейкоцитов (> 20х109 кл/л) в сочетании с накоплением вирусной РНК в образцах крови инфицированных животных свидетельствует о том, что именно они представляют наибольшую инфекционную опасность. Это позволяет предполагать, что совместное использование двух показателей (число лейкоцитов и количество вирусной РНК BLV) может быть достаточно надежным подходом при первоочередных мероприятиях по оздоровлению стад крупного рогатого скота.
Вирус бычьего лейкоза, провирусная днк, ген pol, лейкоцитоз, инфекционная опасность
Короткий адрес: https://sciup.org/142213954
IDR: 142213954 | УДК: 636.2:575.174.015.3:578.2:577.2.08:51-76 | DOI: 10.15389/agrobiology.2016.4.475rus
Текст научной статьи Инфекционная опасность носителей провируса вируса бычьего лейкоза и ее оценка в связи с лейкоцитозом
Вирус бычьего лейкоза (Bovine leukemia virus — BLV) принадлежит к дельтавирусам, подсемейству орторетровирусов семейства Retroviradae и филогенетически связан c вирусом Т-клеточного лейкоза человека 1-го типа (HTLV-1) (1). HTLV-1 инфицирует CD4+ T-клетки человека, у крупного рогатого скота BLV инфицирует B-клетки, оба вируса интегрируют геномные копии в геном хозяина как провирусную ДНК (2, 3). У большинства инфицированных BLV животных не проявляются какие-либо клинические симптомы, однако у некоторых, как правило, через 5-10 лет после инфицирования отмечается повышенная пролиферация В-клеток, и в конечном итоге у них формируются В-клеточные лимфомы. Распространение BLV приводит к существенному экономическому ущербу в молочном и мясном скотоводстве (4) еще и потому, что отсутствуют эффективные методы иммунизации животных. Предполагается, что трудности вакцинации против
BLV могут быть обусловлены сложным влиянием, которое продукты генов провирусной ДНК оказывают на иммунную систему хозяина (5). Кроме того, дискуссионной остается сама стратегия оздоровления стад, поскольку до настоящего времени основным приемом считается удаление всех животных, несущих антитела к BLV или провирусную ДНК в геноме, без выделения собственно инфекционно опасных.
У крупного рогатого скота, инфицированного BLV, в периферической крови выявляется менее 1 % клеток, несущих в геноме провирусную ДНК (6). После первых стадий инфицирования провирусная ДНК массово встраивается в активно транскрибируемые участки генома хозяина, однако в последующем только единичные клеточные клоны сохраняют интегрированные в геном копии провирусной ДНК, и из них только единичные в дальнейшем малигнизируются (7). Инфекционный процесс сопровождается массовой гибелью клеток, несущих интегрированный провирусный геном (6, 7). Такая гибель обусловлена как реакцией иммунной системы хозяина на антигены вируса, так и тем, что интеграция провирусной ДНК в активно транскрибирующиеся районы генома хозяина может приводить к снижению жизнеспособности клеток, особенно при встраивании прови-русной ДНК в регуляторные или кодирующие последовательности жизненно важных структурных генов (8).
То есть в общем случае естественный отбор направлен против клеточных популяций, в которых активно экспрессируется провирусная ДНК. Более того, оказалось, что инфицированные BLV В-клетки подразделяются на две субпопуляции — в одной активно экспрессируется IgM и прови-русная ДНК BLV, а в другой отсутствует экспрессия как IgM, так и про-вирусной РНК. Предполагается, что именно вторая клеточная популяция служит предшественником формирования лимфом, поскольку в опухолевых клетках провирусная ДНК не экспрессируется (8). Накапливаются данные, свидетельствующие о том, что ключевым регуляторным фактором, способствующим инициации неопластической трансформации В-клеток, служит экспрессия микроРНК провирусной ДНК BLV, транскрипция которой обнаруживается в не экспрессирующих полноразмерный геном BLV В-лимфоцитах (9-11).
Подразделенность инфицированных BLV В-клеток на продуцентов зрелых вирусных частиц и предшественников формирования лимфом приводит к необходимости рассматривать отдельно инфекционную опасность животных и прогноз развития у них лимфолейкоза (12). Важно подчеркнуть, что современные методы, основанные на тестировании антител к вирусным антигенам или встройки провирусной ДНК в геном хозяина, не позволяют оценить инфекционную опасность животных, что наиболее существенно для контроля распространения инфекции. Одним из подходов могло бы быть тестирование количества полноразмерной вирусной РНК в крови животных, однако лабильность пула РНК не позволяет считать такой контроль надежным. В поисках дополнительной характеристики инфекционной опасности животных в настоящей работе выполнен сравнительный анализ экспрессии провирусной ДНК на основании количественной оценки фрагмента транскрипта гена pol BLV (методом reverse transcription polymerase chain reaction — RT-PCR, ОТ-ПЦР) и числа лейкоцитов (с использованием автоматического гематологического анализатора) у животных, инфицированность которых BLV установлена по выявлению в их геноме интегрированной провирусной ДНК с использованием разработанных нами ранее праймеров к генам BLV gag и pol (13).
Методика. Кровь для исследования отбирали из яремной вены у черно-пестрых голштинизированных коров в возрасте 2-5 лет (57 гол.) из хозяйства ЗАО «Можайское» (Московская обл.).
ДНК получали из цельной крови, используя набор реагентов для обработки клинических образцов М-Сорб («Синтол», Россия) согласно протоколу, описанному производителем. Тотальную РНК выделяли из цельной крови коров с помощью набора ExtractRNA («Евроген», Россия) в соответствии с прилагаемой инструкцией. Синтез первой цепи кДНК выполняли с набором MMLV RT kit («Евроген», Россия), как рекомендовано производителем. Качество полученной кДНК оценивали по амплификации гена рибосомального белка RPLPO. Реакцию проводили в 20 мкл смеси для ПЦР, содержащей 1 х буфер для амплификации, 3 мМ MgCl2, по 0,2 мМ каждого из нуклеозидтрифосфатов (dNTPs), 1 ед. HS Таq ДНК-полимеразы («Евроген», Россия), прямой (0,2 мкМ) и обратный (0,2 мкМ) праймеры. Использовали следующие пары праймеров: для Pol — 5´-GCA-GGCCGATATAACCCAT-3´ и 5´-TGCTGGCAAAACCTGACAAAG-3´, для RPLPO — 5 ′ -CA-ACCCTGAAGTGCTTGACAT-3 ′ и 5 ′ -CAGATGGATCAG-
CCAAGAAG-3 ’ . Режим амплификации: денатурация при 95 ° С в течение 15 с, отжиг праймеров при 60 ° С в течение 15 с, элонгация при 72 ° С в течение 15 с (40 циклов). Результаты ПЦР оценивали методом электрофореза в 1,2 % агарозном геле.
Сравнительный анализ экспрессии гена pol проводили методом количественной полимеразной цепной реакции в реальном времени (real time PCR, ПЦР-РВ) на приборе LightCycler 480 («Roche», Швейцария), используя интеркалирующий краситель SYBR Green и набор реагентов qPCRmix-HS SYBR («Евроген», Россия). Для более точной характеристики экспрессии генов контролировали присутствие примеси геномной ДНК в кДНК. Внутренним контролем служил фрагмент гена рибосомального белка RPLPO.
Общее число лейкоцитов в крови исследуемых коров посчитывали на автоматическом гематологическом анализаторе Abacus junior Vet5 («Dia-tron», Австрия; принцип работы основан на методе Культера). Для подсчета лейкоцитов в образцах использовали 100 мкл свежей цельной крови (индивидуально от каждого животного), стабилизированной EDTA.
Результаты . С использованием ранее разработанных праймеров (13) среди черно-пестрого голштинизированного скота были выявлены 35 коров, свободных от инфекции, и 22 особи, несущие в геноме интегрированную провирусную ДНК BLV (рис. 1, А).
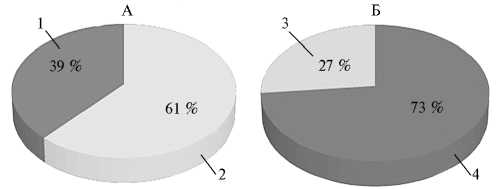
Рис. 1. Инфицированность Bovine leukemia virus (BLV) (А) и транскрипция вирусной ДНК у инфицированных особей (Б) в выборке ( n = 57) черно-пестрых голшти-низированных коров: 1 и 2 — инфицированные и неинфициро-ванные; 3 и 4 — с активной транскрипцией провируса и без его экспрессии (промышленное стадо, Московская обл.).
Для оценки транскрипции провирусной ДНК BLV выполнили нормирование кДНК, полученной на матрице тотальной РНК из клеток крови, по гену рибосомального белка RPLPO. Экспрессия гена pol не выявлялась ни у одного животного, свободного от инфекции, но была обнаружена у 27 % коров, несущих в геноме встроенную провирусную ДНК (см. рис. 1, Б).
Подсчет лейкоцитов в образцах крови (рис. 2) показал, что у сво- бодных от инфекции коров, выявленных с использованием подобранных праймеров (13), этот показатель варьировал от 3x109 до 18x109 кл/л (рис. 2, А). При этом группа инфицированных животных разделилась на две подгруппы: в одной содержание лейкоцитов составило 7x109-17x109 кл/л (значение верхней границы у большинства в подгруппе), во второй — находилось в пределах от > 20x109 кл/л до 28x109 кл/л.
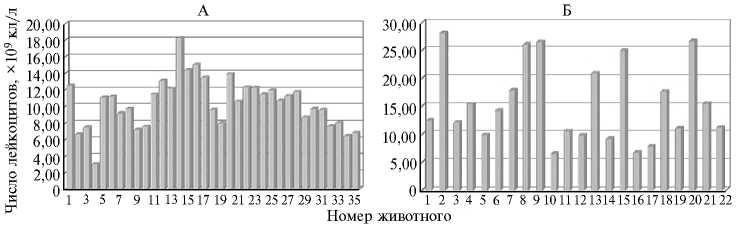
Рис. 2. Число лейкоцитов в крови черно-пестрых голштинизированных коров , свободных от инфицирования Bovine leukemia virus (BLV) (А, n = 35) и инфицированных вирусом BLV (Б, n = 22) (исследуемая выборка — n = 57, промышленное стадо, Московская обл.).
Во второй подгруппе было шесть коров, при этом у каждой без исключения наблюдалась высокая экспрессия гена pol BLV. В первой подгруппе (несмотря на наличие в геномах животных интегрированной про-вирусной ДНК BLV) экспрессию pol BLV выявить не удалось, что согласуется с относительно пониженным числом лейкоцитов в образцах крови по сравнению с соответствующими значениями у коров из второй подгруппы. Представленные результаты RT-PCR (рис. 3), свидетельствуют о довольно высокой экспрессии провирусной ДНК у всех животных, у которых отмечали лейкоцитоз.
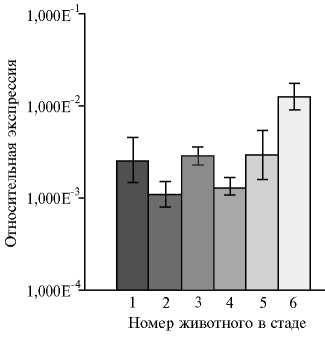
Рис. 3. Результаты RT-PCR анализа экспрессии гена pol Bovine leukemia virus (BLV) в подгруппе чернопестрых голштинизированных коров с лейкоцитозом, инфицированных BLV ( n = 6, указаны номера коров) : 1 — 7412, 2 — 7575, 3 — 7652, 4 — 7243, 5 — 7722, 6 — 7345 (промышленное стадо, Московская обл.). Экспрессию гена pol выражали как кратность превышения количества его продукта относительно образующегося при амплификации референтного гена рибосомального белка RPLPO для суммарной РНК в пробе.
Следует отметить, что в настоящее время приняты достаточно жесткие правила оздоровления стад крупного рогатого скота, основанные главным обра- зом на серологических исследованиях с использованием реакции иммунодиффузии (РИД) или вариантов имму-ноферментного анализа (ИФА, ELISA), то есть на выявлении антител, продуцируемых у животного в ответ на контакт с патогеном (как правило, с продуктами гена env BLV). Гликопротеин Env gp51 играет важную роль в жизненном цикле BLV, он необходим для проникновения вируса в клетку и служит основной мишенью для нейтрализующих антител (14).
Однако диагностика BLV на основе выявления антител у животных, инфицированных вирусом, достаточно проблематична. Так, у крупного рогатого скота успешное выявление BLV-специфических антигенов возможно не ранее, чем у полугодовалых особей, к тому же антителогенез зависит от количества клеточных клонов, продуцирующих зрелые вирусные частицы, и, соответственно, представленности антигенов гликопротеинов оболочки, а также от структуры самих антигенов. Например, единичная мутация в сайте гликозилирования на N-конце поверхностного гликопротеина может приводить к резкому повышению патогенности BLV. Последнее рассматривается как свидетельство коэволюции хозяина и патогена, препятствующей росту патогенности выше определенного предела (15). Показано отсутствие корреляций между кинетикой размножения вируса и иммунореактивностью животных, а также способность BLV инфицировать не только B-лимфоциты, но и другие клеточные популяции (16). В некоторых случаях у инфицированных BLV животных обнаруживается отсутствие синтеза соответствующих антител, несмотря на наличие интегрированной провирусной ДНК в клетках хозяина (17). Описано ингибирующее влияние инфицированности животных BLV на регуляторную функцию субпопуляции Т-лимфоцитов (CD4+CD25highFoxp3+), приводящее к подавлению антивирусной активности, в частности у естественных киллеров — NK-клеток (18).
В последние годы с целью выявления носителей ретровирусных инфекций широко используют методы полимеразной цепной реакции (ПЦР) для обнаружения провирусной ДНК, интегрированной в геном хозяина. Критические факторы при такой диагностике — консервативность фрагментов провирусной ДНК, подобранной в качестве праймеров для ПЦР, и отсутствие участков гомологии к ним в геноме хозяина. В то же время известно, что скорость мутирования у ретровирусов достаточно высока: в среднем частота мутаций составляет 10-3-10-5 на нуклеотид за один транскрипционный цикл (19). Таким образом, имеющиеся методы выявления инфицированных животных, основанные на диагностике интеграции про-вирусной ДНК ретровирусных патогенов в геном хозяина, осложняются высокой изменчивостью провирусных генов и перегруженностью генома млекопитающих эндогенными ретровирусами и их обломками, поскольку при этом осуществляется подбор праймеров к участкам провирусной ДНК с недостаточно изученной спонтанной мутабильностью и потенциальной предрасположенностью к перекрестной гибридизации с диспергированными повторами в геноме хозяина. Кроме того, показано, что существенное влияние на инфекционную опасность инфицированных BLV животных, а также на развитие у них лейкоза оказывает генотип носителей провируса BLV по генам II класса главного комплекса гистосовместимости. Так, содержание инфицированных BLV животных, несущих аллель BoLA-DRB3*0902 , с неинфицированными коровами в течение достаточно длительного времени не приводит к заражению последних. При этом инфицированные особи были позитивны по антителам к BLV (хотя и в низких титрах) и несли провирусную ДНК в геномах (20).
Таким образом, современные способы выявления инфицированных животных, основанные на обнаружении либо антител к гликопротеинам — продуктам гена env BLV, либо провирусной ДНК BLV в геноме хозяина, отражают только факт контакта животного с патогеном, но, в общем, не позволяют прогнозировать ни инфекционную опасность особи, ни вероятность развития у нее лейкоза.
Дополнительным функциональным показателем предрасположенности инфицированных BLV животных к развитию лейкоза служит персистирующий лейкоцитоз, который связывают с усиленной пролиферацией В-лимфоцитов, индуцируемой BLV. Содержание лейкоцитов у половозрелых животных выше 10000 x 10 9 кл/л принято считать преднеопластической 479
характеристикой (21). Однако собственно лейкоцитоз как показатель достаточно неспецифичен, поскольку развивается при ряде неинфекционных и других инфекционных заболеваний, часто сопутствующих основному, в том числе у животных, инфицированных BLV (22, 23).
Следует отметить, что в проведенном нами исследовании превышение этого значения отмечали у 19 из 35 коров, свободных, по нашим данным, от инфицированности BLV (см. рис. 2, А). В то же время среди инфицированных BLV животных у 6 особей из 22 число лейкоцитов не превышало порогового значения 10000 x 10 9 кл/л (см. рис. 2, Б). То есть сам по себе лейкоцитоз не может быть диагностическим признаком инфициро-ванности животных именно BLV. Известно, что у животных, несущих аллельные варианты по BoLA DRB3.2 , ассоциированные с относительно повышенной устойчивостью к инфицированию BLV, этот факт не препятствует заражению другими патогенами, что, в свою очередь, может приводить к повышению содержания лейкоцитов (24).
Следовательно, разрозненные данные о количестве антител к белкам BLV, интеграции провирусной ДНК BLV в геном хозяина или лейкоцитозу у животных не позволяют выделять наиболее инфекционно опасных особей. Очевидно, что этим затрудняется оздоровление стад, но в то же время не исключается выбраковка тех животных из ценного племенного поголовья, у которых носительство BLV не представляет выраженной угрозы распространения инфекции. Мы полагаем, что объединение таких функциональных показателей, как относительное количество вирусной РНК и уровень лейкоцитов в периферической крови, будет наиболее близко соответствовать задаче выявления и обоснованного удаления из стада инфекционно опасных особей. Размер клеточной популяции, активно продуцирующей полноразмерные вирусные частицы, можно оценить на основании титра вирусной РНК BLV в периферической крови животных, который определятся в количественной ПЦР (ПЦР-РВ).
Итак, в исследованных нами группах животных высокую экспрессию провирусной ДНК Bovine leukemia virus ( BLV) отмечали только у коров с выраженным лейкоцитозом: для таких животных (в отличие от остальных) характерно самое большое количество РНК BLV и наиболее высокая активность пролиферации лейкоцитов. Можно ожидать, что сочетание именно этих характеристик инфекционного процесса, индуцируемого BLV, позволит выявлять наиболее опасных с инфекционной точки зрения животных, удаление которых — первоочередная задача при оздоровлении стад.
Список литературы Инфекционная опасность носителей провируса вируса бычьего лейкоза и ее оценка в связи с лейкоцитозом
- Sagata N., Yasunaga T., Tsuzuku-Kawamura J., Ohishi K., Ogawa Y., Ikawa Y. Complete nucleotide sequence of the genome of bovine leukemia virus: its evolutionary relationship to other retroviruses. PNAS USA, 1985, 82: 677-681 ( ) DOI: 10.1073/pnas.82.3.677
- Mirsky M.L., Olmstead C.A., Da Y., Lewin H.A. The prevalence of proviral bovine leukemia virus in peripheral blood mononuclear cells at two subclinical stages of infection. J. Virol., 1996, 70: 2178-2183.
- Schwartz I., Bensaid A., Polack B., Perrin B., Berthelemy M., Levy D. In vivo leukocyte tropism of bovine leukemia virus in sheep and cattle. J. Virol., 1994, 68: 4589-4596.
- Gillet N., Florins A., Boxus M., Burteau C., Nigro A., Vandermeers F., Balon H., Bouzar A.B., Defoiche J. Mechanisms of leukemogenesis induced by bovine leukemia virus: prospects for novel anti-retroviral therapies in human. Retrovirology, 2007, 4: 18 ( ) DOI: 10.1186/1742-4690-4-18
- Gutiérrez G., Rodríguez S.M., de Brogniez A., Gillet N., Golime R., Burny A., Jaworski J.P., Alvarez I., Vagnoni L., Trono K., Willems L. Vaccination against d-retroViruses: the bovine leukemia virus paradigm. Viruses, 2014, 6(6): 2416-2427 ( ) DOI: 10.3390/v6062416
- Barez P.Y., de Brogniez A., Carpentier A., Gazon H., Gillet N., Gutiérrez G., Hamaidia M., Jacques J.R., Perike S., Neelature Sriramareddy S., Renotte N., Staumont B., Reichert M., Trono K., Willems L. Recent advances in BLV research. Viruses, 2015, 7(11): 6080-6088 ( ) DOI: 10.3390/v7112929
- Gillet N.A., Gutiérrez G., Rodriguez S.M., de Brogniez A., Renotte N., Alvarez I., Trono K., Willems L. Massive depletion of Bovine leukemia virus proviral clones located in genomic transcriptionally active sites during primary infection. PLoS Pathog., 2013, 9(10): e1003687 ( ) DOI: 10.1371/journal.ppat.1003687
- Ikebuchi R., Konnai S., Okagawa T., Nishimori A., Nakahara A., Murata S., Ohashi K. Differences in cellular function and viral protein expression between IgMhigh and IgMlow B-cells in bovine leukemia virus-infected cattle. J. Gen. Virol., 2014, 95: 1832-1842 ( ) DOI: 10.1099/vir.0.065011-0
- Gillet N.A., Hamaidia M., de Brogniez A., Gutiérrez G., Renotte N., Reichert M., Trono K., Willems L. The bovine leukemia virus microRNAs permit escape from innate immune response and contribute to viral replication in the natural host. Retrovirology, 2015, 12(Suppl 1): O9 ( ) DOI: 10.1186/1742-4690-12-S1-O9
- Gillet N.A., Hamaidia M., de Brogniez A., Gutiérrez G., Renotte N., Reichert M., Trono K., Willems L. Bovine leukemia virus small noncoding RNAs are functional elements that regulate replication and contribute to oncogenesis in vivo. PLoS Pathog., 2016, 12(4): e1005588 ( ) DOI: 10.1371/journal.ppat.1005588
- Rosewick N., Momont M., Durkin K., Takeda H., Caiment F., Cleuter Y., Vernin C., Mortreux F., Wattel E., Burny A., Georges M., Van den Broeke A. Deep sequencing reveals abundant noncanonical retroviral microRNAs in B-cell leukemia/lymphoma. PNAS USA, 2013, 110(6): 2306-2311 ( ) DOI: 10.1073/pnas.1213842110
- Глазко В.И., Косовский Г.Ю., Глазко Т.Т. Взаимодействия вируса бычьего лейкоза с организмом хозяина. Farm animals, 2016, 2(12): 42-46.
- Косовский Г.Ю., Сотникова Е.А., Мудрик Н.Н., Cuong V.C., Toan T.X., Hoan T.X., Глазко В.И. Диагностика лейкоза КРС с помощью праймеров к генам gag и pol. Ветеринария, 2013, 8: 58-61.
- Polat M., Takeshima S.N., Hosomichi K., Kim J., Miyasaka T., Yamada K., Arainga M., Murakami T., Matsumoto Y., de la Barra Diaz V., Panei C.J., González E.T., Kanemaki M., Onuma M., Giovambattista G., Aida Y. A new genotype of bovine leukemia virus in South America identified by NGS based whole genome sequencing and molecular evolutionary genetic analysis. Retrovirology, 2016, 13(1): 4 ( ) DOI: 10.1186/s12977-016-0239-z
- de Brogniez A., Bouzar A.B., Jacques J.-R., Cosse J.-P., Gillet N., Callebaut I., Reichert M., Willems L. Mutation of a single envelope N-linked glycosylation site enhances the pathogenicity of bovine leukemia virus. J. Virol., 2015, 89: 8945-8956 ( ) DOI: 10.1128/JVI.00261-15
- Panei C.J., Takeshima S., Omori T., Nunoya T., Davis W.C., Ishizaki H., Matoba K., Aida Y. Estimation of bovine leukemia virus (BLV) proviral load harbored by lymphocyte subpopulations in BLV infected cattle at the subclinical stage of enzootic bovine leucosis using BLV-CoCoMo-284 qPCR. BMC Vet. Res., 2013, 9: 95. Режим доступа: http://www.biomedcentral.com/1746-6148/9/95. Без даты.
- Jimba M., Takeshima S.N., Murakami H., Kohara J., Kobayashi N., Matsuhashi T., Ohmori T., Nunoya T., Aida Y. BLV-CoCoMo-qPCR: a useful tool for evaluating bovine leukemia virus infection status. BMC Vet. Res., 2012, 8(1): 167 ( ) DOI: 10.1186/1746-6148-8-167
- Ohira R., Nakahara A., Konnai S., Okagawa T., Nishimori A., Maekawa N., Ikebuchi R., Kohara J., Murata S., Ohashi K. Bovine leukemia virus reduces anti-viral cytokine activities and NK cytotoxicity by inducing TGF-b secretion from regulatory T cells. Immunity, Inflammation and Disease, 2016, 4(1): 52-63 ( ) DOI: 10.1002/iid3.93
- Duffy S., Shackelton L.A., Holmes E.C. Rates of evolutionary change in viruses: patterns and determinants. Nat. Rev. Genet., 2008, 9: 267-276 ( ) DOI: 10.1038/nrg2323
- Juliarena M.A., Barrios C.N., Ceriani M.C., Esteban E.N. Hot topic: Bovine leukemia virus (BLV)-infected cows with low proviral load are not a source of infection for BLV-free cattle. J. Dairy Sci., 2016, 99(6): 4586-4589 ( ) DOI: 10.3168/jds.2015-10480
- Trueblood E.S., Brown W.C., Palmer G.H., Davis W.C., Stone D.M., McElwain T.F. B-lymphocyte proliferation during bovine leukemia virus-induced persistent lymphocytosis is enhanced by T-lymphocyte-derived interleukin-2. J. Virol., 1998, 72(4): 3169-3177.
- Suzuki S., Konnai S., Okagawa T., Ikebuchi R., Shirai T., Sunden Y., Mingala C.N., Murata S., Ohashi K. Expression analysis of Foxp3 in T cells from bovine leukemia virus infected cattle. Microbiol. Immunol., 2013, 57: 600-604 ( ) DOI: 10.1111/1348-0421.12073
- Okagawa T., Konnai S., Ikebuchi R., Suzuki S., Shirai T., Sunden Y., Onuma M., Murata S., Ohashi K. Increased bovine tim-3 and its ligand expressions during bovine leukemia virus infection. Vet. Res., 2012, 43: 45-48 ( ) DOI: 10.1186/1297-9716-43-45
- Juliarena M.A., Poli M., Ceriani C., Sala L., Rodríguez E., Gutierrez S., Dolcini G., Odeon A., Esteban E.N. Antibody response against three widespread bovine viruses is not impaired in Holstein cattle carrying bovine leukocyte antigen DRB3.2 alleles associated with bovine leukemia virus resistance. J. Dairy Sci., 2009, 92(1): 375-381 ( ) DOI: 10.3168/jds.2008-1143


