Использование молекулярных маркеров, связанных с устойчивостью к биотическим и абиотическим факторам среды при создании селекционного материала томата и перца в Беларуси
Автор: Бабак О.Г., Дрозд Е.В., Некрашевич Н.А., Анисимова Н.В., Яцевич К.К., Шестерень П.В., Баева И.Е., Невестенко Н.А., Пугачва И.Г., Добродькин М.М., Кильчевский А.В.
Журнал: Овощи России @vegetables
Рубрика: Селекция, семеноводство и биотехнология растений
Статья в выпуске: 1 (81), 2025 года.
Бесплатный доступ
Актуальность. Разработка системы молекулярных маркеров, позволяющих выявлять генетические детерминанты устойчивости к возбудителям болезней, а также типировать аллели, участвующие в регуляции накопления антоцианов, является важнейшим условием повышения эффективности селекционного процесса, направленного на повышение устойчивости возделываемых культур к биотическим и абиотическим стрессам. Материал и методика. В работе использовались молекулярно-генетические методы выделения ДНК, ПЦР-анализ, рестрикция, оценка продуктов амплификации и рестрикции в агарозном или полиакриламидном геле. В качестве материала использовались широкие коллекции Solanum lycopersicum и Capsicum annuum, а также образцы близкородственных диких видов.
S. lycopersicum, c. annuum, днк-маркеры, устойчивость к болезням, гены, регулирующие биосинтез антоцианов
Короткий адрес: https://sciup.org/140308655
IDR: 140308655 | УДК: (635.64+635.649):631.52.85(476) | DOI: 10.18619/2072-9146-2025-1-5-13
Текст научной статьи Использование молекулярных маркеров, связанных с устойчивостью к биотическим и абиотическим факторам среды при создании селекционного материала томата и перца в Беларуси
Оригинальная статья / Original article
У стойчивость к воздействию неблагоприятных факторов – необходимое условие успешного возделывания культурных растений. Способность растительного организма к эффективной защите от негативных воздействий среды является важнейшим резервом повышения урожайности, поскольку обеспечивает возможность максимальной реализации потенциала продуктивности. В этой связи повышение устойчивости сельскохозяйственных растений к неблагоприятным факторам среды биотической и абиотической природы неизменно остается важной и актуальной задачей генетико-селекционных исследований [1].
Серьезный экономический ущерб возделыванию овощных культур наносят патогены, которые вызывают многочисленные болезни растений. Применение химических методов борьбы с инфекциями не всегда дает желаемые результаты и снижает качество продукции, нанося вред окружающей среде и здоровью человека.Перспективным способом защиты растений является создание и выращивание устойчивых форм,которые невосприимчивы к воздействию патогенов.Поиск генетических детерминант устойчивости и ДНК-маркеров к ним позволяет с применением инструментов маркер-ассоции-рованной селекции (МАС) создавать генотипы, способные противостоять разнообразным негативным воздействиям. В литературе опубликованы сведения о идентификации генов,связанных с сопротивляемостью биотическим стрессам у томата и перца, и о разработанных к ним молекулярных маркерах. Выявлены гены, контролирующие устойчивость к ряду экономически значимых заболеваний, вызываемых возбудителями грибной, бактериальной, вирусной природы и нематодой [2-12].
В Институте генетики и цитологии НАН Беларуси проводится работа с ДНК-маркерами генов устойчивости к болезням и вредителям пасленовых,которая направлена на апробацию, адаптацию, верификацию представленных в литературе маркеров. С применением методов ДНК-маркирования ведется скрининг коллекционного и селекционного материала для выявления источников целевых аллелей и создания новых устойчивых генотипов [13-15].
Не менее важна для сельскохозяйственных культур, особенно выращиваемых в зонах рискованного земледелия, устойчивость к негативным абиотическим факторам. К генетическим детерминантам, ассоциированным с неспецифической устойчивостью к стрессам, относят гены, связанные с регуляцией синтеза антоциановых пигментов.Имеются све- дения, что растительные организмы с повышенным содержанием флавоноидов в тканях более устойчивы к неблагоприятным воздействиям среды [16-18].
В литературе и генетических базах данных представлены сведения по идентификации генов, кодирующих Myb транскрипционные факторы (ТФ), участвующие в регуляции биосинтеза антоцианов у томата: Anthocyanin1, доминантный аллель которого Ant1 из S. chilense увеличивает количество флавоноидов в плодах, An2 и An2-like, которые тесно сцеплены с Ant1. [16, 19]. Ген Atroviolacеa (ATV), рецессивный аллель которого (atv) из S. cheesmaniae связан с активным накоплением антоциана как в плодах (наряду с Ant1, An2), так и в вегетативных органах [20]. Для идентификации указанных генов предложены ДНК-маркеры, позволяющие дифференцировать аллели Ant1/ant1, Atv/atv. [19-20].
В Институте генетики и цитологии НАН Беларуси разработаны новый SCAR Ant1.1 (FAM) маркер для выявления аллелей Ant1 и ant1 , SCAR маркеры An2-AFT(ОM) и An2-4F/R для генотипирования аллелей гена An2: An2-Aft, An2-Ins57 и Myb75, SCAR маркер Atv2 для дифференциации аллей гена Аtv ( Atv/atv ). Выполнены исследования по изучению накопления антоцианов в различных частях растений томата в зависимости от комбинации аллелей [21].
У Capsicum annuum выявлены наиболее схожие по нуклеотидному составу и функциям с геном Ant1 S . lycopersicum последовательности генов Мyb113-like 1 и Мyb113-like 2 , установлены генетические полиморфизмы этих генов,приводящие к изменению структуры кодируемого белка и нарушению процесса биосинтеза антоцианов в плодах и вегетативных органах, разработаны молекулярные маркеры для типирования выявленных аллелей ( Myb113-like1-delT, Myb113-like1-promIns148, Myb113-like1-prоmIns2, Myb113-like2-(C/A )) [22-23].
В данной работе дана характеристика используемых молекулярных маркеров, представлены результаты ДНК-типирования с их применением на коллекциях образцов томата и перца для использования в генетических исследованиях, а также для сопровождения селекционного процесса, направленного на повышение устойчивости к неблагоприятным биотическим и абиотическим факторам.
Материалы и методы
Материалом для исследований на различных этапах были формы томата и перца для защищенного и открытого грунта коллекций Института генетики и цитологии НАН Беларуси, Белорусской государственной сельскохозяйственной академии, а также коллекции, используемые при выполнении совместных проектов с Федеральным научным центром овощеводства, Всероссийским институтом генетических ресурсов растений имени Н.И. Вавилова, Научноисследовательским институтом плодоовощевод-ства., Научно-исследовательского института плодо-овощеводства им. Л. Димитровой (Республика Куба). В работе использовались молекулярно-генетические методы выделения ДНК, ПЦР-анализ, рестрикция, оценка продуктов амплификации и рестрикции в агарозном или полиакриламидном гелях [13].
Результаты и обсуждение
На основании литературных данных проведены исследования по апробации,адаптации и верификации имеющихся маркеров, связанных c устойчивостью к болезням и вредителям у томата и перца.В таблицах 1 и 2 представлены данные о используемых маркерах по литературным источникам и выполненной верификации.При апробации указанных в таблице 1 маркеров устойчивости к болезням томата были использованы коллекционные образцы, а также близкородственные дикие виды.
Таблица 1. Наименование и характеристика маркеров, используемых для ДНК-типирования образцов S. lycopersicum Table 1. Name and characteristics of markers used for the DNA typing of S. lycopersicum samples
|
Ген |
Тип и название маркера |
Праймеры для ПЦР |
Т отжига, ºС |
Размер ПЦР продукта, п.н. |
Литературный источник |
|
Mi-1 |
SCAR, Mi-1.2 |
Mi23F: TGGAAAAATGTTGAATTTCTTTTG Mi23R: GCATACTATATGGCTTGTTTACCC |
55 |
R - 380; S - 430 |
Seah S. et.al., 2007 |
|
Mi-9 |
SCAR, C8B-Mi9 |
F: ACCCACGCCCCATCAATG R: TGCAAGAGGGTGAATATTGAGTGC |
59 |
R - 400; S - 360 |
El-Sappah A. H., 2019 |
|
Ve |
CAPS, Ve ( XbaI ) |
F: CGAACTTGAC TACATTGACC CTG R: CAGTCTTGAAAGGTTGCTCAGCC |
59 |
R - 410, 332; S - 410, 310, 22 |
Je Min Lee et.al., 2015 |
|
I-2 |
SCAR, I-2 |
I-2/5F: CAAGGAACTGCGTCTGTCTG I-2/5R: ATGAGCAATTTGTGGCCAGT |
55 |
R ( I-2 ) - 633; R ( I-2C ) - 566; S - 693, 760 |
Davis R.M. et al., 2010 |
|
I-3 |
SCAR, P7-43D |
F: GGTAAAGAGATGCGATGATTATGTGGAG R: GTCTTTACCACAGGAACTTTATCACC |
60 |
R - 875; S - 650 |
Je Min Lee. et al., 2015 |
|
I-7 |
СAPS, I-7 ( AgeI ) |
F: AAGAAGTTCCCTTCTTCCCTTA R: GGAATAACCAAGGGGGTGTT |
56 |
R - 608, 200; S - 808 |
Je Min Lee. et al., 2015 |
|
Cf2, Cf5 |
SCAR, 2-5Cf |
F: GCTATCTTTGGGTATCAACTT R: AGATGACATCGACAAAATGTG |
58 |
R ( Cf-2 ) - 1600; R ( Cf-5 ) - 1163, 880; S - нет продукта |
Kanwar J.S. et al., 1980 |
|
Cf5 |
SCAR, CF5 |
F: GTAATATCAGTGACCTTCACA R: ATTTTCCAAACTGAAAAG |
55 |
R - 1600; S - нет продукта |
Dixon M.S. et al.,1998 |
|
Cf4 |
SCAR, Cf4/4A |
F: TTACGACAGAAGAACTCTTTCTTGG R: AGTCCGCTTACGTTGGATGG |
55 |
R ( Cf-4 ) - 816; R ( Cf-4A ) - 965; S - нет продукта |
Parniske M. et al., 1997 Takken F.L. et al., 1998 |
|
Сf9 |
SCAR Cf9/9DC |
CS5: TTTCCAACTTACAATCCCTTC DS1: GAGAGCTCAACCTTTACGAA CS1: GCCGTTCAAGTTGGGTGTT |
55 |
R ( Cf-9 ) - 378; R ( 9DC ) - 507; S - нет продукта |
Van der Hoorn R.A. et al., 2001 |
|
Ph-2 |
CAPS, dTG63 ( HinfI ) |
F: CTACTCTTTCTATGCAATTTGAATTG R: AATAATTTTCAACCATAGAATGATT |
55 |
R - 246; S - 220 |
Panthee D.R. et al., 2012 |
|
Ph-3 |
SCAR, NC-LB-9-6678 |
F: CCTTAATGCAATAGGCAAAT R: ATTTGAATGTTCTGGATTGG |
52 |
R - 600; S - 900 |
Panthee D.R. et al., 2012 |
|
Pto |
СAPS, Pto_RsaI |
F: ATCTACCCACAATGAGCATGAGCTC R: GTGCATACTCCAGTTTCCAC |
56 |
R - 552; S - 113, 439 |
Je Min Lee. et al., 2015 Yang W. et al., 2005 |
|
Rx4 |
SCAR, pcc12 InDel |
F: TCCACATCAAATGCGTTTCT R:TTCCAATCCTTTCCATTTCG |
60 |
R - 113; S - 119 |
Yang W. et al., 2005 |
|
Tm1 |
SCAR, Tm1:8рог |
F: CCACTGTATGATTTCTGCTAGTGAA R1: AGCTTTAACAAATATAAGAATAAAGAC R2: GCAAGCTAAGGTTTACATATATGCC |
56 |
R - 131; S - 212 |
Je Min Lee. et al., 2015 |
|
Тm-2 |
CAPS, Tm2RS-f3/r3_ HpaI |
F: TGGAGGGGAATATTTGTGGA R: ACTTCAGACAACCCATTCGG |
61 |
R ( Tm2 ) - 458, 245; S - 703 |
Shi A. et al., 2011 |
|
Тm-22 |
CAPS, Tm2RS-f3/ r3_BsiHKAI |
F: TGGAGGGGAATATTTGTGGA R: ACTTCAGACAACCCATTCGG |
61 |
R ( Tm22 ) - 358, 353; R (Tm2) - 703 |
Shi A. et al., 2011 |
|
Sw-5 |
SCAR, Sw5-2 |
F: CGGAACCTGTAACTTGACTG R:GAGCTCTCATCCATTTTCCG |
60 |
R ( Sw-5b ) - 541; S - нет продукта |
Shi A. et al., 2011 |
|
Ty2 |
SCAR, Ty-2 |
F: TGGCTCATCCTGAAGCTGATAGCGC R: AGTGTACATCCTTGCCATTGACT |
50 |
R - 900; S - 790 |
Je Min Lee. et al., 2015 |
|
Ty3 |
SCAR, Ty-3 |
F: GGTAGTGGAAATGATGCTGCTC R: GCTCTGCCTATTGTCCCATATATAACC |
50 |
R ( Ty-3 ) - 450; R ( Ty-3a ) - 630; S - 320 |
Je Min Lee. et al., 2015 |
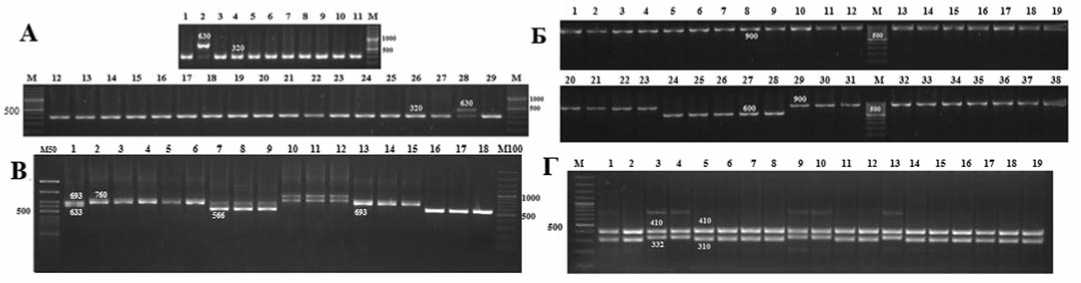
Рис.1.ДНК-типирование аллелей устойчивости к болезням у форм томата:гена Ty-3 (А), гена Ph-3 (Б)гена I-2 (В), гена Ve (Г
По большинству апробированных маркеров были подтверждены размеры ожидаемых фрагментов, подобраны оптимальные температуры отжига, выполнена адаптация под используемые наборы реагентов. На рис. 1 показаны результаты электрофоретической разгонки продуктов ПЦР при оценке материала по аллелям устойчивости к вирусу желтой курчавости листьев (А), фитофторе (Б), фузарио-зу (В) и вертициллезу (Г).
Анализ коллекций томата на устойчивость к мелойдогинозу, позволил выявить образцы с аллелями устойчивости гена Мi-1 у различных форм для защищенного и открытого грунта. При этом устойчивые образцы c R-аллелем гена Mi-9 чаще были характерны для детерминантных форм, используемых для возделывания в открытом грунте.
Фитофтороз ( late blight ), вызываемый грибами класса Oomycetes рода Phytophtora , является наиболее опасной болезнью для томата в условиях Беларуси. В связи с этим выполнен анализ широких коллекций томата на наличие источников устойчивости по генам Ph-2 и Ph-3 , дана оценка эффективности представленных в литературе маркеров [15], рис.1 Б.
Устойчивость к фузариозному увяданию томата, вызываемому Fusarium oxysporum f. sp. Lycopersici, оценивалась с помощью маркеров I-2, P7-43D, I-7_AgeI (табл.1), связанных с резистентностью к определенным расам возбудителя.Анализ образцов белорусской и российской селекции с помощью маркера I-2 в наших исследованиях позволил идентифицировать два R-аллеля I-2 (633 п.н.) и I-2C (566 п.н.), причем аллель I-2C , как правило, был характерен для индетерминантных форм томата черри.
Исследования образцов кубинской коллекции выявили наличие отличного,не описанного авторами фрагмента около 900 п.н. (рис.1 В, номера образцов 10-12, Lyto).
Бурая пятнистость листьев томата, или кладоспо-риоз, вызываемая грибом Cladosporium fulvum Cke., является одним из наиболее вредоносных заболеваний томата защищенного грунта. Устойчивость к кладоспориозу является непременным требованием при создании современных сортов и гибридов. В наших исследованиях широко использованы доминантные маркеры к генам Cf2, Cf4, Cf5, Cf9. Кроме того, нами апробирован кодоминантный маркер Cf8/CR12 [3] к аллелям гена Сf-9. При сопоставлении результатов, полученных с помощью доминантных маркеров и кодоминантного,выявилось несоответствие размеров ПЦР продуктов, заявляемых авторами по кодоминантному маркеру. Согласно авторам, размеры фрагментов у устойчивых / восприимчивых образцов составляют 1500/1600 п.н. [3], согласно нашим полученным данным – 1675/1909 п.н. соответственно. На рис. 2 представлены результаты ДНК-типирования форм томата с применением SC AR маркеров C f9/9DC (А) и Cf8/CR12 (Б). Использование широких коллекций форм позволило показать, что по большинству образцов результаты типирования обоими маркерами совпадают, однако ряд образцов показали различные результаты. Так у образца кубинской коллекции №24 (L33/1) наряду с фрагментом, характеризующим наличие восприимчивого аллеля, маркер Cf8 /CR12 позволил выявить фрагмент размером 1500 п.н., нехарактерный для других образцов.
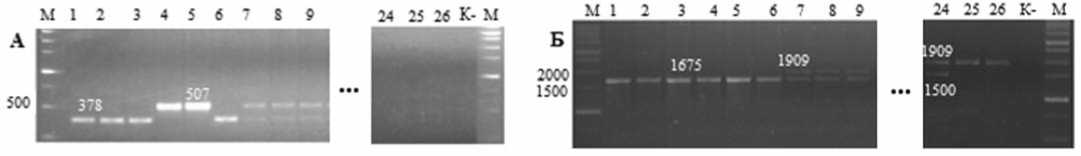
Рис.2.ДНК-типирование аллелей гена CF9у форм томата белорусской и кубинской селекции маркерами Cf9/9DC (А)и Cf8/CR12 (Б)
Таблица 2. Наименование и характеристика маркеров, используемых для ДНК-типирования образцов рода Capsicum Table 2. Name and characteristics of markers used for the DNA typing of Capsicum genus specimens
|
Ген |
Тип и название маркера |
Праймеры для ПЦР |
Т отжига, ºС |
Размер ПЦР продукта, п.н. |
Литературный источник |
|
Me1 |
CAPS, C2At2g06530-F/R_ HpyCH4IV |
F: TTGGTGCTGTAAGGGACTAAA R: TCTTAATCAATCATTCACACAGCA |
55 |
R - 119; S - 55, 59 |
Uncu A.T. et al., 2015 |
|
Phyto.9 QTL |
CAPS, F/R_MvaI |
F:CCAACCCTATTGAACGTCTT R:CTGATTCTTGATGCCTCTTG |
55 |
R - 700, 150; S - 850 |
Kim H. et al., 2008 |
|
Phyto.5.2 QTL |
SCAR, OpD04.717-F/R |
F: CCATAAGGGTTGGTAAATTTACAAAG R: TCGAGAGATAATTCAGATAGTATAATC |
55 |
R - 717; S - нет продукта |
Quirin E.A. et al., 2005 |
|
Хр.5, QTL |
STS, InDel_F/R |
F: GGTATCTTATTTCATAGGGACCAGGCA R: TTTGCGGTAGTGACAACAACTTTACAGCCA |
55 |
R - 100; S - 90 |
ChunYing S. et al., 2015 |
|
pvr1 |
CAPS, PVRL-F/R_ Bse NI |
F: TGAGGCAGATGATGAAGTTGA R: CAACCATAAATATACCCCGAG |
60 |
R - 710; S - 580, 130 |
Paran I. et al., 2009 |
|
Cvr1 |
CAPS, CVMV3-F/R_EcoRI |
F: GAACCTCAATCATCTTAGCA R: ACATCAAATTGTTGCATTATAC |
55 |
R - 311, 466; S - 762 |
Lee H.R. et al., 2013 |
|
Cmr1 |
CAPS, CaTm-int1-F/R_HinfI |
F: TCAGCAAAGAAAGATTCACGAAC R: ACGTACACTTGATGATGCCTTGT |
58 |
R - 336, 160; S - 496 |
Kang W. Et al., 2010 |
|
Tsw |
CAPS, SCAC568-F/R_XbaI |
F: GTGCCAGAGGAGGATTTAT R: GCGAGGTGGACACTGATACT |
55 |
R - 300, 268; S - 568 |
Özkaynak E. et al., 2014 |
|
Bs3 |
PR-Bs3-F/R |
F: GCACACCCTGGTTAAACAATGAACACG R: GATGATAACTTGAAGTTGTGAGGATGG |
58 |
R - 97; S - 110 |
Römer P. et al., 2010 |
В таблице 2 представлены данные по 9 маркерам, которые были успешно апробированы, а также по которым найдены источники устойчивости среди образцов изучаемых коллекций: к корневым нематодам ( Meloidogyne spp.: M. incognita, M. javanica, M. arenaria ) – Me1 ; к корневым и плодовым гнилям, вызываемым Phytophthora capsici – Phyto.9, Phyto.5.2, QTLs; к антракнозу, вызываемому Colletotrichum acutatum – хромосома 5, QTL; к Y вирусу картофеля – pvr1; к вирусу огуречной мозаики – Cmr1 ; к вирусу пятнистого увядания TSWV – Tsw ; к вирусу крапчатости сосудов чили – Cvr1 ; к бактериальной пятнистости – Bs3.
По результатам оценки материала выявлены образцы c маркерами устойчивости к корневым нематодам (к мелойдогинозу): C. annuum Л-97, Златозар, Шоколадная красавица, Л-160-10, Фиолетовый красавец и др., к фитофторе плодов: C.annuum Златозар, C. chinense Хабанеро, C. baccatum
Маленький принц; к корневым гнилям, вызываемым Phytophthora capsici – C.annuum МТПЕ053, МТПЕ137, МТПЕ154, МТПЕ204, МТПЕ209, C.chinense Огненная дева, Хабанеро; к антракнозу: C.chinense Огненная дева, Хабанеро; к Y вирусу картофеля: C.annuum ZongKao, F 2 Премьер, F 2 Валентина, F 2 Корнелия, к вирусу огуречной мозаики: C.chinense Огненная дева; к вирусу пятнистого увядания TSWV : C. chinense Хабанеро; к вирусу крапчатости сосудов чили: C. annuum Златозар, ZongKao, Требиа F 2 , Джемини F 2 , МТПЕ 006, МТПЕ 196, C. baccatum Маленький принц, C. chinense Огненная дева, Хабанеро; к бактериальной пятнистости: образцы европейской селекции EVA_Ca_№20, 26, 111, 128 и др. На рис. 3 представлены примеры ДНК-типирования образцов по генам, связанным с устойчивостью к Y вирусу картофеля (А), вирусу крапчатости сосудов чили (Б), мелойдогинозу (В), вирусу огуречной мозаики (Г).
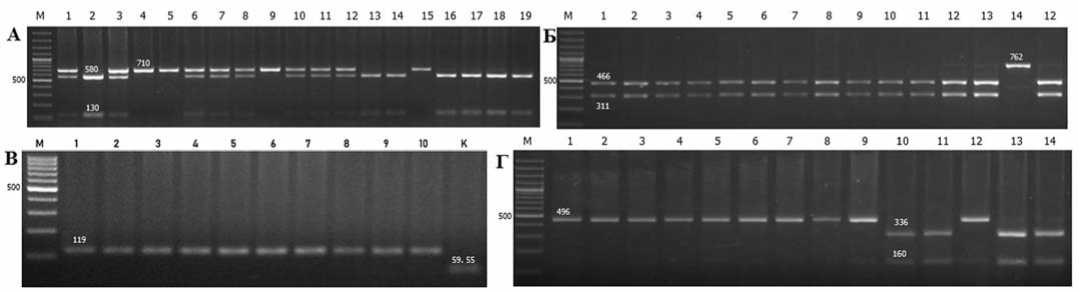
Рис.3.ДНК-типирование аллелей устойчивости к болезням у форм перца:гена pvr1 (А), гена Cvr1 (Б), гена Me1 (В), гена Cmr1 (Г
Второй группой маркеров, связанной с общей устойчивостью к биотическим и абиотическим факторам, являются маркеры аллелей, участвующих в регуляции накопления антоцианов в растениях.В таблице 3 представлены апробированные и разработанные маркеры, позволяющие выделять формы с накоплением / отсутствием халкон-нарингенина (SC AR MYB12-603del) и антоцианов (CAPS и SCAR маркеры к генам Ant1, An2, ATV, An2-like ) в плодах.
В представленных ранее наших исследованиях на основе данных молекулярного маркирования аллелей генов Ant1, An2 , atv в популяциях F 2 -F 3 изучаемых гибридов и оценки фенотипического проявления признаков на различных этапах онтогенеза,связанных с накоплением антоцианов в растениях, показаны высокая эффективность разработанных молекулярных маркеров, тесное сцепление аллелей генов Ant1 и An2 ( Ant1 и An2-Aft, ant1 и Myb75 ). Подтверждено повышение накопления антоцианов в плодах и вегетативных органах при усилении потока солнечного света, а также при понижении температуры в конце вегетационного периода. Выявлены особенности накопления антоцианов в растениях томата в зависимости от аллелей генов Ant1, An2, Atv. Показано максимальное накопление антоцианов в вегетативных органах и плодах у образцов с аллелями Ant1/An2-Aft/atv в генотипе [21].
Наряду с описанными ранее, в таблице 3 представлены маркеры к аллелям гена An2-like , разработанные по результатам секвенирования данного гена у форм с различным накоплением антоцианов в кожице плодов, которые позволяют разделить образцы с высоким и низким накоплением антоцианов. Данный ген входит в комплекс генов R2R3Myb транскрипционных факторов и тесно сцеплен с геном Ant1 . На рис. 4 показаны результаты ДНК-типирования форм томата с высоким (Октябрята, Бурштын, Дзiвосны: №1-6), и низким накоплением антоцианов (ИСИ63, Дачный: №7-10) в плодах с помощью SCAR маркера SlAn2-like и dCAPS SlAn2-like-AvaI маркера. SCAR маркер подобран к области гена,охватывающей делеции 3и 6 п.н.в четвертом экзоне, приводящие к выпадению трех аминокислот в белке. Маркер dCAPS SlAn2-like-AvaI подобран к SNP-замене C/А в третьем экзоне, приводящей к аминокислотной замене лейцина у диких форм (без накопления антоциана)на метионин у форм с фенотипом Aft. Данная замена расположена в области второго ДНК-связывающего домена синтезируемого белка. Считаем, что данная замена, а также выпадение трех аминокислот вносят вклад в функциональность синтезируемого протеина и, как следствие, в регуляцию накопления антоцианов в плодах.
Таблица 3. Наименование и характеристика маркеров, связанных с накоплением антоцианов, используемых для ДНК-типирования образцов S. lycopersicum
Table 3. Name and characteristics of markers associated with anthocyanin accumulation, used for the DNA typing of S. lycopersicum samples
|
Ген |
Тип и название маркера |
Праймеры для ПЦР |
Т отжига, ºС |
Размер ПЦР продукта, п.н. |
Литературный источник |
|
SlMyb12 |
SCAR, MYB12-603del -aF1/aR6 и MYB12-603del -aF1/aR5 |
F1: GTGACGAACAACCGAACCTAGAATAA R5: ATTCTAGCGTTATCAGTCGGCATACA R6: GCGGACAAAGTTAATTGGTCACTCA |
60 |
Y - 950, 614; y - 347 |
Veerappan K. et.al., 2016 |
|
Ant1 |
CAPS, Ant1-NcoI |
F: GGAAGGACAGCTAACGATGTG; R: GTTGCATGGGTGGTAAATTAAG |
55 |
Ant1 - 478; ant1 - 271, 207 |
Sapir M. et.al., 2008 |
|
Ant1 |
SCAR, Ant1.1(FAM |
F: TTCATTGGGAGTGAGAAAAGGTT R(FAM): AACTTGCATGCCTGTTGCCTA |
58 |
Ant 1 - 340; ant1 - 344 |
Babak O.G. et al., 2024 |
|
An2-Aft |
SCAR, An2-AFT |
F: ATTACAAGTGTCATTTTGTGGAAAG R (FAM): AAACTTTTGAATGAAATAATTGC |
51 |
An-Aft - 661; Myb 75 - 647; Ant-Ins - 659 |
Бабак. О.Г. и др., 2024 |
|
An2-Aft |
SCAR, An2-4 |
F: ACTTCACAAACTCTTAGGCAATAG R: AGTCTACCAGCAATAAGTGACCAC |
61 |
An-Aft - 186; Myb 75 - 580; Ant-Ins - 647 |
Бабак. О.Г. и др., 2024 |
|
An2-Like |
SCAR, SlAn2-like |
F: ACACACACCTACACAAGAAGTT R: GTTGTTCGTCATCTTTGTCTAAT |
58 |
Aft - 204 wt - 213 |
|
|
dCAPS, SlAn2-like-AvaI |
F: CGTTCAACCCACAAATCCCC R: AAGTCTCAAAATGAGATCTATTTCATCCC |
59 |
Aft - 161 wt -131 |
||
|
ATV |
SCAR, ATV-In |
F(FAM): GAGGTTTCTTCGTTGGTAGTC R: CTAAATAAAAGTTATTGAGTTCACG |
53 |
Atv - 81; atv - 85. |
Cao X. et al., 2017 |
|
SCAR, Atv2 |
F: GTTGGATAAGTAAGAATGTTGTAGA R: CTTCTGAAAGTACATAAACCACA |
55 |
Atv - 460; atv - 852 |
Бабак. О.Г. и др., 2024 |
|
|
M 1 2 3 4 5 < |
5 7 8 9 10 М М 1 |
2 3 |
4 5 6 7 |
8 9 10 |
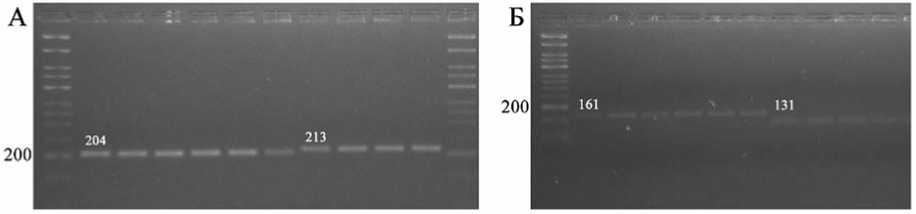
Таблица 4. Наименование и характеристика маркеров, используемых для ДНК-типирования образцов C. annuum Table 4. Name and characteristics of markers used for the DNA typing of C. annuum samples
|
Ген, полиморфизм |
Тип и название маркера |
Праймеры для ПЦР |
Т отжига, ºС |
Размер ПЦР продукта, п.н. |
Литературный источник |
|
Myb113-like1, delT |
CAPS, Myb113-AccI F / R |
F: ATGCAGATGGTCACTTATGGCT R: ATGAATTTTTGAACCCCTACAGCAA |
60 |
Aft - 589, 25; delT - 393, 195, 25 |
Бабак О.Г. и др., 2019 |
|
Myb113-like1, prom Ins148 |
SCAR, Myb113-like1-promIns148 F / R |
F: TCCGCCCCTCGTTAATTT R: AGCAGGAACAAGATGCCACT |
55 |
Aft - 700; Ins 148 - 848 |
Бабак О.Г. и др., 2022 |
|
Myb113-like1, prоm Ins2 |
SCAR, PromIns2-F(FAM)/R, |
F: TTTTAATATTACGTTAATTTGGGAACG FAM R: AATTAGGCGGGTTAGCCTCA |
59 |
prоmIns 2 - 241; без вставки - 239 |
Бабак О.Г. и др., 2022 |
|
Myb113-like2, SNP C/A |
dCAPS, Myb113-AciI F / R |
F(FAM): AAAGTACAATACTGCCCTCAAGATCACCG R: GACGACGTTTCACTTGTGAGC |
60 |
Aft (C) - 170, 28 ; wt (A) - 198 |
Бабак О.Г. и др., 2019 |
|
MYB-like ETC3-2, del C и SNP С/G |
CAPS, OGF_PagI |
F: AGCTATGCTTCACTAGCTCACC R: TCACTGCCTCTTTCGCATCT |
60 |
Aft - 284 и 61; wt - 345 |
В таблице 4 представлены маркеры, разработанные к генам Myb113-like1 и Myb113-like2 перца, являющимся аналогами томатному гену Ant1 . Ранее представлено описание полиморфизма данных генов,выявленно-го по результатам секвенирования, и его связи с фенотипом Aft (накопление антоцианов в плодах на стадии технической спелости, а также в вегетативных органах, чаще всего в междоузлиях стебля и жилках листьев) [22, 23]. Как правило, фенотип Aft характерен для форм со следующими размерами маркируемых областей у гена Myb113-like1 : 589 п.н. и 25 п.н. (CAPS маркер Myb113-AccI), а также 700 п.н. (SCAR, Myb113-like1-promIns148); у гена Myb113-like2 – 170 п.н. и 28 п.н. (dCAPS маркер Myb113-AciI) [22-24], рис.5 А, Б, В.
В настоящее время основное количество возделываемых сортов перца не накапливают антоцианы в плодах, при этом у большинства из них наблюдается накопление антоцианов в междоузлиях стебля. В нашей изучаемой коллекции накопление антоцианов в междоузлиях стебля не зависело от описанных выше полиморфизмов генов Myb113-like1 и Myb113-like2 . При этом в последовательности гена Myb113-like1 сорта
Златозар, фенотип которого характеризовался полным отсутствием антоцианов в плодах и вегетативных органах, обнаружена наряду с описанными выше маркируемыми полиморфизмами, нарушающими синтез антоцианов, инсерция размером 2 п.н. в промоторной области [24]. Для типирования данной инсерции разработан ДНК-маркер MYB113-like1-prоmIns2, для чего были подобраны праймеры PromIns2-F(FAM)/R (табл.4). Отличным от других образцов в изучаемой коллекции был сорт Блондин, который по разработанным маркерам характеризовался наличием аллелей генов Myb113-like1 и Myb113-like2 без нарушения синтеза антоцианов и, предположительно, мог проявлять фенотип Aft, при этом он характеризовался полным отсутствием синтеза антоцианов во всех частях растения. Данный сорт также характеризовался бледно-зеленой окраской вегетативных органов и светлой оранжевокрасной окраской плодов. Такие фенотипические признаки могут говорить о возможных нарушениях в генотипе, связанных с синтезом пигментов в целом.
Наряду с поиском генов-гомологов Ant1 томата, у перца был осуществлен поиск гена-гомолога ATV
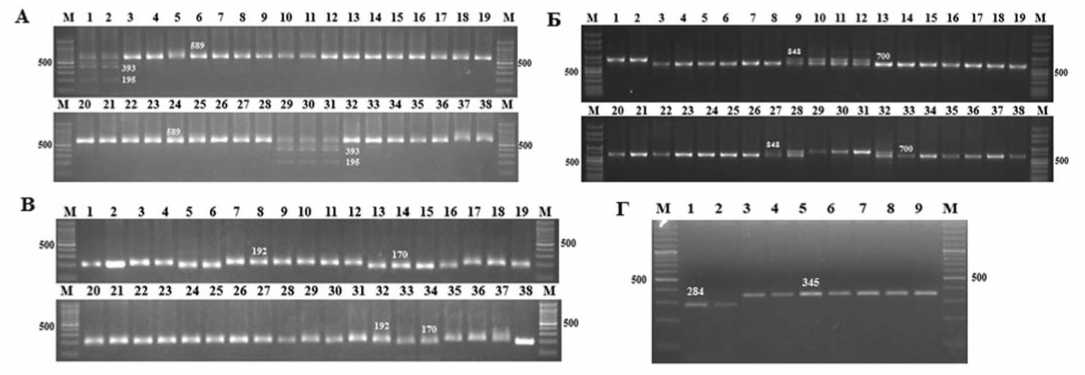
Рис.5.ДНК-типирование аллелей генов, связанных с накоплением антоцианов у перца: Myb113-like1-delT/TT(A),Myb113-like1-promIns148(Б),Myb113-like2-(C/A)(В), ETC3-2 (Г)
Myb113-like1-delT/TT (A),Myb113-like1-promIns148 (B),Myb113-like2-(C/A)(С),ETC3 -2 (D)
( Atroviolaceae ). Одним из выделенных генов - кандидатов является ген ETC3-2 MYB-like ТФ ETC3-2 (GenBank -XM_016723933). По результатам секвенирования последовательностей данного гена у представителей рода Capsicum у формы с максимальным накоплением антоцианов в вегетативных органах, венчике цветка и плодах (OGF) выявлены отличные от других образцов изменения в структуре гена ETC3-2 : однонуклеотидная делеция ( del C ) и SNP-замена С/G перед старт кодоном в первом экзоне гена (м-РНК XM_016723933), приводящие к образованию более раннего старт-кодона, а также однонуклеотидная делеция в первом экзоне ( del T ), приводящая к сдвигу рамки считывания и образованию раннего стоп-кодона во втором экзоне. Данные изменения приводят к формированию новой короткой белковой последовательности и нарушению функций гена, в результате чего возможно активное накопление антоцианов во всех частях растения. Для выявления полиморфизма, связанного с высоким накоплением антоцианов в вегетативных и генеративных органах к del C и SNP С/G разработан CAPS маркер OGF_PagI (табл. 4). На рис. 5 Г показаны размеры маркируемых фрагментов: у образца OGF – 284 п.н., у форм с ненарушенной последовательностью гена – 345 п.н.
Заключение
По результатам апробации и верификации выделен комплекс наиболее эффективных представленных в литературе ДНК-маркеров, связанных с устойчивостью к возбудителям грибных,бактериальных, вирусных инфекций и нематодам у томата и перца. Отобраны образцы,являющиеся донорами аллелей резистентности к возбудителям болезней.
На основании оценки полиморфизма генов MYB транскрипционных факторов, связанных с регуляцией накопления флавоноидов (халкон-нарингени-на, антоцианов), разработаны молекулярные маркеры, позволяющие дифференцировать образцы томата и перца по накоплению антоцианов в плодах и вегетативных органах. Представлены результаты разработки новых молекулярных маркеров,связан-ных с накоплением антоцианов у томата к аллелям гена An2-like и у перца к аллелям гена ETC3-2 . С применением системы разработанных ДНК-маркеров выделены образцы для использования в селекционном процессе, направленном на повышение устойчивости к биотическим и абиотическим факторам окружающей среды.
16. Liu Y., Tikunov Y., Schouten R.E., Marcelis L., Visser R., Bovy A. Anthocyanin biosynthesis and degradation mechanisms in Solanaceous Vegetables: a review. Frontiers in Chemistry. 2018;6(52):1–17.
(In Russ.)
(In Russ.)
Об авторах:
Ольга Геннадьевна Бабак ‒ кандидат биологических наук, доцент, ведущий научный сотрудник лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси, SPIN-код: 8702-4355, ,
Елизавета Валерьевна Дрозд – младший научный сотрудник лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси, ,
Наталья Александровна Некрашевич – научный сотрудник лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси, SPIN-код: 8288-8067, ,
Наталья Владимировна Анисимова – кандидат биологических наук, старший научный сотрудник лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси, SPIN-код: 3000-1523, ,
Констанция Константиновна Яцевич – научный сотрудник лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси,
,
Павел Владимирович Шестерень — студент магистратуры Университета Национальной академии наук Беларуси, ,
Ирина Евгеньевна Баева ‒ кандидат с.-х. наук, зав. учебно-научно-исследовательской генетической лабораторией Белорусской государственной сельскохозяйственной академии, SPIN-код: 8736-7550, ,
SPIN-код: 1917-9851,
,
Ирина Геннадьевна Пугачёва ‒ кандидат с.-х. наук, доцент кафедры сельскохозяйственной биотехнологии, экологии и радиологии Белорусской государственной сельскохозяйственной академии, SPIN-код: 3875-8999, ,
Михаил Михайлович Добродькин – кандидат с.-х. наук, доцент кафедры сельскохозяйственной биотехнологии, экологии и радиологии Белорусской государственной сельскохозяйственной академии, SPIN-код: 2238-2173, ,
Александр Владимирович Кильчевский ‒ академик НАН Беларуси, доктор биологических наук, профессор, научной руководитель лаборатории экологической генетики и биотехнологии Института генетики и цитологии Национальной академии наук Беларуси, SPIN-код: 2496-4294, ,
Aboutthe Authors:
Olga G.Babak – Cand. Sci. (Biology), Assistant Professor, Leading Researcher of the Laboratory of Ecological Genetics and Biotechnology,
Institute of Genetics and Cytology of National Academy of Science of Belarus,
SPIN-code: 8702-4355, ,
Elizaveta V. Drozd – Junior Researcher of the Laboratory of Ecological Genetics and Biotechnology, Institute of Genetics and Cytology of National Academy of Science of Belarus, ,
Natalya A. Nekrashevich – Researcher of the Laboratory of Ecological Genetics and Biotechnology, Institute of Genetics and Cytology of National Academy of Science of Belarus, SPIN-code: 8288-8067,
,
Natalya V.Anisimova – Cand. Sci. (Biology), Senior Researcher of the Laboratory of Ecological Genetics and Biotechnology, Institute of Genetics and Cytology of National Academy of Science of Belarus,
SPIN-code: 3000-1523,
Kаnstantsyia K. Yatsevich – Researcher of the Laboratory of Ecological Genetics and Biotechnology, Institute of Genetics and Cytology of National Academy of Science of Belarus, ,
Pavel V. Shesteren – Master’s degree student, University of the National Academy of Sciences of Belarus,
,
Iryna Е. Bayeva – Cand. Sci. (Agriculturе), Chief of the Academic-scientific-investigation Genetics Laboratory, Belarusian State Agricultural Academy,
SPIN-code: 8736-7550,
,
Natalya A. Nevestenko – Cand. Sci. (Agriculturе), Senior Lecturer of the Department of the Agricultural Biotechnology, Ecology and Radiology, Belarusian State Agricultural Academy, SPIN-code: 1917-9851,
,
Iryna G.Puhachova – Cand. Sci. (Agriculturе), Associate Professor of the Department of the Agricultural Biotechnology, Ecology and Radiology, Belarusian State Agricultural Academy, SPIN-code: 3875-8999,
,
Mihail M. Dobrodkin – Cand. Sci. (Agriculturе), Associate Professor, Belarusian State Agricultural Academy,
SPIN-code: 2238-2173,
,
Alexander V. Kilchevsky – Academician of the National Academy of Sciences of Belarus, Dr. Sci. (Biology), Professor, Scientific Director of the Laboratory of Environmental Genetics and Biotechnology, Institute of Genetics and Cytology of National Academy of Science of Belarus, SPIN-code: 2496-4294,
,
ISSN 2618-7132 (Online) Овощи России №1 2025 [ 13 ] Vegetable crops of Russia №1 2025 ISSN 2072-9146 (Print)
Список литературы Использование молекулярных маркеров, связанных с устойчивостью к биотическим и абиотическим факторам среды при создании селекционного материала томата и перца в Беларуси
- Biotechnology and plant disease management / ed. by Z.K.Punja, S.H.De Boer, H.Sanfaçon / Biddles Ltd, King’s Lynn, UK. 2007. 574 p
- Lee J.M., Oh C., Yeam I. Molecular Markers for Selecting Diverse Disease Resistances in Tomato Breeding Programs. Plant Breeding and Biotechnology. 2015;(3):308-322. https://doi.org/10.9787/PBB.2015.3.4.308
- Truong H.T.H., Choi H., Cho M.C., Lee H.E., Kim J.H. Use of Cf-9 genebased markers in marker-assisted selection to screen tomato cultivars with resistance to Cladosporium fulv um. Hortic. Environ. Biotechnol. 2011;(52):204–210. https://doi.org/10.1007/s13580-011-0164-y
- Kang W., Hoang N., Yang H., Kwon J., Jo S., Seo J., Kim K., Choi D., Kang B. Molecular mapping and characterization of a single dominant gene controlling CMV resistance in peppers (Capsicum annuum L.). Theor. Appl. Genet. 2010;(120):1587–1596. https://doi.org/10.1007/s00122-010-1278-9
- Uncu A.T., Celik I., Devran Z., Ozkaynak E., Frary A., Frary A., Doganla S. Development of SNP-based CAPS assay for the Me1 gene conferring resistance to root knot nematode in pepper. Euphytica. 2015;(206):393–399. https://doi.org/10.1007/s10681-015-1489-x
- Kim H., Nahm S., Lee H., Yoon G., Kim K., Kang B., Choi D., Kweon O.Y., Cho M., Kwon J., Han J., Kim J., Park M., Ahn J.H., Choi S.H., Her N.H., Sung J., Kim B. BAC-derived markers converted from RFLP linked to Phytophthora capsici resistance in pepper (Capsicum annuum L. Theor. Appl. Genet. 2008;(118):15–27.
- ChunYing S., Li M., Hai Z., Alainb P., Haoa W., Xi Z. Resistances to anthracnose (Colletotrichum acutatum) of Capsicum mature green and ripe fruit are controlled by a major dominant cluster of QTLs on chromosome P5. Scientia Horticulturae. 2015;(181):81-88. https://doi.org/10.1016/j.scienta.2014.10.033
- Paran I., Benarous S., Ashkenazi V. Disease resistant pepper plants. Hazera Genetics Ltd, The Agricultural Research Organization. Volcani Center, 2009. Patent WO2009098685 A2
- Lee H.R., An H.J., You Y.G., Lee J., Kim H.J., Kang B.C., Harn C.H. Development of a novel codominant molecular marker for Chili veinal mottle virus resistance in Capsicum annuum L. Euphytica. 2013;(193):197–205.
- Quirin E.A., Ogundiwin E.A., Prince J.P., Mazourek M., Briggs M.O., Chlanda T.S., Kim K., Falise M., Kang B., Jahn M.M. Development of sequence characterized amplified region (SCAR) primers for the detection of Phyto.5.2, a major QTL for resistance to Phytophthora capsici Leon. in pepper. Theor. Appl. Genet. 2005;(110):605–612. https://doi.org/10.1007/s00122-004-1874-7
- Özkaynak E., Devran Z., Kahveci E., Doğanlar S., Başköylü B., Doğan F., İşleyen M., Yüksel A., Yüksel M. Pyramiding Multiple Genes for Resistance to PVY, TSWV and PMMoV in Pepper Using Molecular Markers. Europ. J. Hort. Sci. 2014;79(4):233–239.
- Rӧmer P., Jordan T., Lahaye T. Identification and application of a DNAbased marker that is diagnostic for the pepper (Capsicum annuum) bacterial spot resistance gene Bs3. Plant Breed. 2010;(129):737–740. https://doi.org/10.1111/j.1439-0523.2009.01750.x
- Бабак О.Г., Некрашевич Н.А., Анисимова Н.В., Дрозд Е.В., Яцевич К.К., Кильчевский А.В. Технология маркер-сопутствующего отбора форм томата с высокими биохимическими и технологическими свойствами плодов: методические рекомендации. Минск: Министерство сельского хозяйства и продовольствия Республики Беларусь; Национальная академия наук Беларуси; Институт генетики и цитологии Национальной академии наук Беларуси Право и экономика, 2023. 74 c.
- Игнатова С.И., Бабак О.Г., Багирова С.Ф. Создание высоколикопиновых гибридов томата для теплиц с использованием традиционных методов селекции и молекулярных маркеров. Овощи России. 2020;(5):22-28. https://doi.org/10.18619/2072-9146-2020-5-22-28 https://elibrary.ru/avqfuj
- Бабак О.Г., Дрозд Е.В., Некрашевич Н.А., Анисимова Н.В., Яцевич К.К., Баева И.Е., Пугачева И.Г., Французенок А.В., Добродькин М.М., Кильчевский А.В. Оценка и применение молекулярных маркеров в селекции на устойчивость томата (Solanum lycopersicum L.) к фитофторе (Phytophthora Infestans). Молекулярная и прикладная генетика. 2021;(31):22-30. https://doi.org/10.47612/1999-9127-2021-31-22-30 https://elibrary.ru/kgdhdp
- Liu Y., Tikunov Y., Schouten R.E., Marcelis L., Visser R., Bovy A. Anthocyanin biosynthesis and degradation mechanisms in Solanaceous Vegetables: a review. Frontiers in Chemistry. 2018;6(52):1–17. https://doi.org/10.3389/fchem.2018.00052
- Bassolino L., Zhang Y., Schoonbeek H.J., Kiferle C., Perata P., Martin C. Accumulation of anthocyanins in tomato skin extends shelf life. New Phytol. 2013;200(3):650-655. https://doi.org/10.1111/nph.12524
- Zhang Y., Butelli E., De Stefano R., Schoonbeek H.J., Magusin A., Pagliarani C., Wellner N., Hill L., Orzaez D., Granell A., Jones J.D., Martin C. Anthocyanins double the shelf life of tomatoes by delaying overripening and reducing susceptibility to gray mold. Curr. Biol. 2013;23(12):1094-100. https://doi.org/10.1016/j.cub.2013.04.072
- Sapir M., Oren-Shamir M., Ovadia R., Reuveni M., Evenor D., Tadmor Y., Nahon S., Shlomo H., Chen L., Meir A., Levin I. Molecular Aspects of Anthocyanin fruit Tomato in Relation to high pigment-1. Journal of Heredity. 2008;99(3):292–303. https://doi.org/10.1093/jhered/esm128
- Cao X., Qiu Z., Wang X., Van Giang T., Liu X., Wang J., Wang X., Gao J., Guo Y., Du Y., Wang G., Huang Z. A putative R3 MYB repressor is the candidate gene underlying atroviolacium, a locus for anthocyanin pigmentation in tomato fruit. J. Exp. Bot. 2017;68(21-22):5745-5758. https://doi.org/10.1093/jxb/erx382
- Бабак О.Г., Дрозд Е.В., Некрашевич Н.А., Анисимова Н.В., Яцевич К.К., Кильчевский А.В. Разработка молекулярных маркеров накопления антоцианов в плодах и изучение особенностей взаимодействия генов Ant1, An2 и Atv у Solanum lycopersicum. Молекулярная и прикладная генетика. 2024;(36):7-23. https://elibrary.ru/gdbfhm
- Бабак О.Г., Некрашевич Н.А., Никитинская Т.В., Яцевич К.К., Кильчевский А.В. Изучение полиморфизма генов Myb-факторов на основе сравнительной геномики овощных пасленовых культур (томат, перец, баклажан) для поиска ДНК-маркеров, дифференцирующих образцы по накоплению антоцианов. Доклады Национальной академии наук Беларуси. 2019;63(6):721-729. – https://doi.org/10.29235/1561-8323-2019-63-6-721-729 https://elibrary.ru/bmbsqh
- Babak O., Nikitinskaya T., Nekrashevich N., Yatsevich K., Kilchevsky A. Identification of DNA Markers of Anthocyanin Biosynthesis Disorders Based on the Polymorphism of Anthocyanin 1 Tomato Ortholog Genes in Pepper and Eggplant. Crop Breed Genet Genom. 2020;2(3):e200011. https://doi.org/10.20900/cbgg20200011
- Бабак О.Г., Никитинская Т.В., Некрашевич Н.А., Яцевич К.К., Дрозд Е.В., Фатеев Д.А.., Беренсен Ф.А., Артемьева А.М., Кильчевский А.В. Изучение полиморфизма генов R2R3MYB транскрипционных факторов культур семейства Solanaceae и гена Myb114 рода Brassica в связи с регуляцией биосинтеза антоцианов. Доклады Национальной академии наук Беларуси. 2022;66(4):414-424. https://doi.org/10.29235/1561-8323-2022-66-4-414-424 https://elibrary.ru/fuwcwf


