Изменение профиля метилирования ДНК в тамоксифен-резистентных сублиниях клеток MCF-7
Автор: Андреева Ольга Евгеньевна, Сигин Владимир Олегович, Стрельников Владимир Викторович, Танас Александр Сергеевич, Щербаков Александр Михайлович, Красильников Михаил Александрович
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 5 т.18, 2019 года.
Бесплатный доступ
Введение. Ранее мы показали возможность горизонтального распространения гормональной резистентности от клетки к клетке при совместном культивировании чувствительных и резистентных клеток и/или через экзосомы, секретируемые резистентными клетками. Каков механизм подобного распространения резистентности, и в какой мере клетки со вторичной резистентностью воспроизводят характеристики донорских резистентных клеток - для ответа на эти вопросы был проведен анализ общего уровня метилирования ДНК в клетках эстрогензависимого рака молочной железы MCF-7 и эстрогеннезависимых сублиниях. Цель исследования - изучение профиля метилирования ДНК при развитии гормональной резистентности и его значения в закреплении резистентного фенотипа клеток рака молочной железы. Методы. Метилирование ДНК исследовали методом RRBS (Reduced Representation Bisulfite Sequencing) в клетках рака молочной железы MCF-7 и резистентных сублиниях. Результаты. Выявлено 19 динуклеотидов CpG, дифференциально и в целом однонаправленно метилированных в клетках c первичной и вторичной резистентностью к тамоксифену. Дифференциальное изменение метилирования было обнаружено для участков ДНК, регулирующих экспрессию шести белок-кодирующих генов: PRKCZ, TRAPPC9, ASIC2, C2CD4a, ZNF787, CRTAC1. Проведенный биоинформатический анализ показал, что два из этих шести генов, PRKCZ (protein kinase С Zeta) и TRAPPC9 (Trafficking Protein Particle Complex Subunit 9), напрямую вовлечены в регуляцию активности NF-kB. Заключение. Полученные данные свидетельствуют о существовании общих паттернов ДНК, метилирование которых изменяется в одном направлении в клетках с первичной и вторичной резистентностью. Участие двух из идентифицированных генов в регуляции NF-kB может свидетельствовать о включении последнего в формирование резистентного фенотипа опухолевых клеток, в том числе в условиях горизонтального переноса резистентности.
Рак молочной железы, тамоксифен, экзосомы, гормональная резистентность, метилирование
Короткий адрес: https://sciup.org/140254440
IDR: 140254440 | УДК: 618.19:576.385:577.2 | DOI: 10.21294/1814-4861-2019-18-5-45-53
Текст научной статьи Изменение профиля метилирования ДНК в тамоксифен-резистентных сублиниях клеток MCF-7
Рак молочной железы (РМЖ) является наиболее часто диагностируемым злокачественным новообразованием у женщин и основной причиной смерти женщин от онкологических заболеваний. По уровню заболеваемости за РМЖ следуют колоректальный рак и рак легких (по показателю смертности у этих двух нозологий порядок обратный) [1]. Рак молочной железы – крайне гетерогенное заболевание [2–4]. В современной литературе растет количество доказательств поликлональности канцерогенеза, описанной для большинства опухолей, за исключением миелоидных (для последних характерно моноклональное происхождение) [5, 6]. Гетерогенность опухолевых клеток частично обусловлена эпигенетическими вариациями; эпигенетическая пластичность вместе с генетическими нарушениями стимулируют опухолевую прогрессию [7].
Особую роль в развитии РМЖ имеет рецептор эстрогенов. Рецептор эстрогенов (ER) является транскрипционным фактором, который после связывания лиганда-эстрадиола, димеризации и попадания в ядро активирует транскрипцию своих генов-мишеней через эстроген-респонсивные участки в их промоторах; это классическое геномное действие сигнального каскада эстрогенового рецептора [8]. Неклассические геномные эффекты включают образование комплексов с транскрипционными факторами, ER/Sp1 и ER/AP-1, в ответ на связывание рецептора эстрогенов с лигандом [9, 10].
Антигормональные соединения, такие как антиэстроген тамоксифен, блокируют эстрогенза-висимый клеточный рост. Важную роль в развитии резистентности к тамоксифену играет активация сигнальных путей в обход рецептора, в том числе активация негативных регуляторов ERα, таких как транскрипционный фактор NF-kB, SNAIL1 и ряд других [11–13]. Особую роль как в канцерогенезе РМЖ, так и в развитии резистентности к антиэ-строгену тамоксифену играет эпигенетический контроль [14, 15], однако вопрос, каким образом эпигенетическая регуляция связана с развитием и передачей резистентности к антиэстрогенам, до сих пор во многом остается без ответа.
Целью исследования явилось изучение эпигенетических изменений и выявление паттернов метилирования при развитии гормональной резистентности клеток РМЖ.
Материал и методы
Культивирование клеток
Клетки эстрогензависимого РМЖ человека линии MCF-7 культивировали в стандартной среде DMEM, содержавшей 7 % эмбриональную сыворотку телят (FBS HyClone, США) и гентамицин (50 ед/мл) («ПанЭко», Россия), при 37 °С и 5 % СО2. При анализе скорости роста и чувствительности к тамоксифену количество клеток определяли либо при подсчете в камере Горяева, либо с использованием МТТ-теста, основанного на утилизации живыми клетками реагента МТТ (3-[4,5-диметилтиазол-2]-2,5-дифенилте-тразол бромида). Тамоксифен-резистентная субпопуляция MCF-7/T была получена путем длительной (60 сут) селекции клеток MCF-7 в присутствии антиэстро-гена тамоксифена. После окончания селекции полученную сублинию культивировали в стандартных условиях (не более 6 мес). Субпопуляция MCF-7/R была получена при ко-культивировании клеток MCF-7 с меченными GFP клетками MCF-7/Т в течение 14 сут с последующим клонированием клеток MCF-7; контрольная сублиния MCF-7/С была получена при клонировании родительских клеток MCF-7. Сублинии MCF-7/exoТ и MCF-7/exoС были получены путем культивирования в течение 10 сут клеток MCF-7 с экзосомами клеток MCF-7/Т и MCF-7 соответственно. Сравнительный анализ чувствительности к тамоксифену в парах MCF-7/R – MCF-7/С и MCF-7/exoТ – MCF-7/exoС выявил частичную резистентность к тамоксифену клеток MCF-7/R и MCF-7/exoТ [16], что позволило в дальнейшем использовать эти линии в качестве моделей вторичной гормональной резистентности.
Выделение ДНК
При выделении геномной ДНК использовали следующий протокол:
-
1. Клетки помещали в пробирку и добавляли 0,5 мл экстракционного буфера (10mM Tris-HCL, 2mM ЭДТА, 4mM NaCl, pH=8,0).
-
2. Добавляли протеиназу К до концентрации 50 мкг/мл.
-
3. Инкубировали 10–16 ч при 56 °C.
-
4. Последовательно проводили экстракцию ДНК равными объемами фенола, смеси фенолхлороформ и хлороформом.
-
5. К образцу добавляли 1/10 объема 5 М ацетата натрия (рН 5.3) перемешивали и осаждали ДНК 2,5 объемами холодного 96 % этанола, выдерживали образец 30 мин при температуре -70 °C.
-
6. Пробу центрифугировали 15 мин с ускорением 12 000 g. Высушивали осадок ДНК на воздухе и растворяли в 0,2 мл ТЕ, рН8.0.
Широкогеномное бисульфитное секвенирование XmaI-RRBS
RRBS (Reduced representation bisulfite sequencing) увеличивает относительную информационную ценность анализа метилирования ДНК методом NGS в сравнении с полногеномным бисульфитным секвенированием, так как каждая прочитанная последовательность RRBS включает, по меньшей мере, одну информативную последовательность CpG. В отличие от полногеномного секвенирования, библиотеки RRBS изготавливаются с использованием специфической эндонуклеазы рестрикции, которая образует пул фрагментов, богатых CpG-участками. Это значительно снижает долю генома, подвергающегося секвенированию, и обогащает его наиболее релевантными регионами [17].
Подготовка библиотек XmaI-RRBS проводилась согласно разработанному Tanas et. al. протоколу [18]. Геномная ДНК, обработанная эндонуклеазой рестрикции XmaI (Сибэнзим, Россия), была частично затуплена 5-метил-цитозинами с помощью фрагмента Кленова (3’-5’ – exo-). Частично затупленные фрагменты ДНК лигировали с синтезированными 5- метилцитозин-содержащими адаптерами. После лигирования неметилирован-ные цепи адапетера ник-транслировали путём добавления dATP, dTTP, dGTP, 5-метил-dCTP и Taq ДНК-полимеразы. Полученные фрагменты библиотеки отбирали по длине с помощью препаративного электрофореза Pippin Prep (Sage Science) в соответствии с инструкциями производителя для получения фракции 181–311 п.н. фрагментов (186–316 п.н. в случае адаптеров со штрих-кодом). Бисульфитное конвертирование элюированной фракции проводили с помощью набора Bisulfite EpiTect (Qiagen Cat # 59104). Для предотвращения неспецифического праймирования полимеразной реакции 3’-концами деградированных фрагментов ДНК фрагменты обрабатывали терминирующими нуклеотидами (ddNTP) с помощью набора SNaPshot Multiplex (Thermo Fisher Scientific, MA, USA, Cat # 4323163). Терминированные фрагменты ДНК обрабатывали РНКазой A (Sigma-Aldrich, MO, USA, Cat # 9001-99-4) и щелочной фосфатазой (SibEnzyme Cat # E328), что позволяет удалить РНК-носитель, использованный в протоколе EpiTect Bisulfite, и дефосфорилировать остаточные ddNTP соответственно. Аликвоту продукта использовали для проведения аналитической количественной ПЦР с целью определения количества циклов последующей препаративной ПЦР. Конечные библиотеки были получены с помощью ПЦР с количеством циклов, определенным для образца с помощью аналитической количественной ПЦР (обычно в диапазоне от 12 до 18 циклов). Полученные библиотеки очищали, измеряли их концентрации на флуорометре Qubit 2.0 (Thermo Fisher Scientific) и разводили до рабочей концентрации 100 пМ.
Эмульсионную ПЦР с объединенными библиотеками проводили с использованием прибора Ion OneTouch и набора Ion OneTouch 200 Template Kit (Thermo Fisher Scientific, Cat # 4480974) в соответствии с инструкциями производителя. Полученные ISP были обогащены при 37 °C с использованием системы Ion OneTouch ES (Thermo Fisher Scientific). Библиотеки XmaI-RRBS секвенировали на Ion Personal Genome Machine (PGM) с использованием набора для секвенирования Ion PGM 200 и чипа Ion 318 (Thermo Fisher Scientific).
Обработка результатов секвенирования
Результаты секвенирования обрабатывали с использованием программного обеспечения Ion Torrent Suite™ Software. Полученные последовательности выравнивали на полный референсный геном человека (build GRCh37/hg19) с помощью программы Bismark [19], последовательности конвертировали в варианты С>T и G>A (соответственно конверсии С>T на обратной нити), затем выравнивали на эквивалентно конвертированные варианты референсного генома и определяли лучший результат среди полученных в итоге четырех параллельных процессов выравнивания. Используемое в процессе подготовки библиотек XmaI-RRBS частичное тупление концов фрагментов ДНК, подразумевающее их достройку остатками 5-метилцитозина, создаёт артефактное метилирование 3′-концевых CpG-динуклеотидов фрагментов. Такие искусственно метилированные цитозины исключали из анализа с помощью скрипта на языке программирования Python (код опубликован нами ранее [17]). Также с помощью программы Bismark определяли уровень метилирования CpG-динуклеотидов. Дифференциально метилированными считали CpG-динуклеотиды при изменении уровня метилирования |Δ(b-value)| >0.9.
Чтобы отобрать дифференциально метилированные CpG-пары, в целом однонаправленно изменяющие уровень метилирования при развитии резистентности к тамоксифену, во всех трех парах клеточных линий, рассчитывали значение
D = (bMCF-7/Т – bMCF-7) + (bMCF-7/R – bMCF-7) – – (bMCF-7/C – bMCF-7) – (bMCF-7/exoC – bMCF-7), где b – b-value – уровень метилирования CpG-пары в клеточной линии. Отбирали CpG-пары, удовлетворяющие |D|>0.9.
Результаты
Эксперименты проводились на клетках эстро-гензависимого рака молочной железы MCF-7 и тамоксифен-резистентной сублинии MCF-7/Т, полученной в результате длительного культивирования клеток MCF-7 с тамоксифеном. Ранее мы показали возможность передачи гормональной резистентности горизонтальным путем, от резистент- ных MCF-7/Т к чувствительным клеткам MCF-7, при их совместном культивировании в условиях in vitro. Клетки с приобретенной таким образом вторичной резистентностью были клонированы и получили название MCF-7/R, клонированные контрольные клетки – MCF-7/С. При исследовании роли экзосом в подобной передаче резистентного фенотипа обнаружено, что регулярное добавление (в течение 10 сут) к родительским клеткам MCF-7 экзосом, полученным от резистентных клеток MCF-7/Т, приводит к развитию частичной резистентности клеток MCF-7 к тамоксифену (сублиния MCF-7/exoТ). В то же время экзосомы, полученные от родительских клеток MCF-7, не обладают такой активностью, и их добавление не приводит к изменению гормональной чувствительности клеток (сублиния MCF-7/exoС) [16].
Основной задачей настоящего исследования явилось изучение возможного механизма горизонтального пути распространения резистентности и дальнейшего закрепления резистентного фенотипа опухолевых клеток. В какой мере характеристики клеток с вторичной резистентностью, развившейся в результате совместного культивирования чувствительных клеток с резистентными, или при действии экзосом резистентных клеток, воспроизводят характеристики донорских резистентных клеток – для ответа на этот вопрос был проведен широкогеномный анализ метилирования ДНК в чувствительных и резистентных клетках MCF-7.
Анализ метилирования методом RRBS выявил 95 динуклеотидов CpG с дифференциальным метилированием в первой паре сублиний MCF-7 и MCF-7/T, во второй паре (MCF-7/C и MCF-7/R) обнаружен 91 дифференциально метилированный динуклеотид, а в третьей паре сублиний MCF-7/exoС и MCF-7/exoT обнаружено 23 таких динуклеотида. Сопоставление выборок выявило 19 динуклеотидов CpG, дифференциально и в целом однонаправленно метилированных при развитии резистентности к тамоксифену во всех трех парах клеточных линий. Схематическое изображение выборок представлено на рис. 1.
Для дальнейшего анализа были отобраны 19 динуклеотидов CpG с дифференциальным метилированием, обнаруженные во всех трех парах сублиний. На рис. 2 представлены значения параметра B-value, характеризующие уровень метилирования цитозинов по шкале от 1 (метилирование 100 %) до 0 (отсутствие метилирования) по каждой сублинии. Для большей части CpG (15 из 19) было отмечено повышение метилирования в резистентных сублиниях по сравнению с чувствительными. Гиперметилирование во всех резистентных сублиниях по сравнению с контрольными было отмечено в:
-
1) межгенных участках: chr2:130344496-130344497, chr4:1040977-1040978, chr2:52799788-52799789, chr7:101361449-101361450, chr11:1858802-1858803;
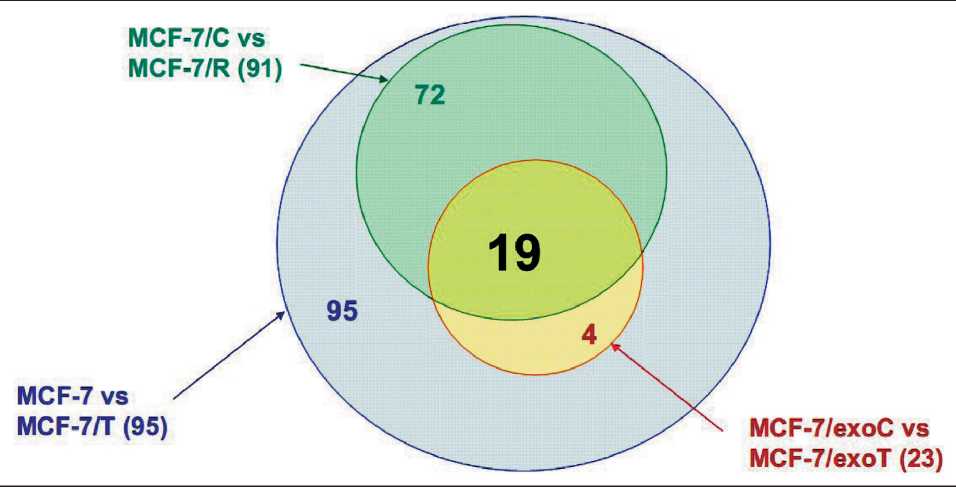
Рис. 1. Диаграмма Эйлера – Венна. Цифрами указано количество дифференциально метилированных CpG-пар для каждой пары клеточных линий
метилирование (B-value)
g
^
g
X
координаты GRCh3 7/hg 19
6
S
U
S
U
S
U
2
U
S
>
S
1
chrl:2036833-2036834
0,67
0,04
0,37
0,22
0,88
0,23
2
chrlO:99734998-99734999
0,37
0,05
0,48
0,11
1
1
3
chrl2:132107115-132107116
0,51
0,31
0,7
0,32
1
0
4
chrl9:2462305-2462306
0,3
0,27
0,66
0,37
1
0,18
5
chrlO:99734891-99734892
0,75
1
0,28
1
0,52
1
6
chrll:1858802-1858803
0,89
0,91
0,16
0,38
0,08
0,86
7
chrl4:102026722-102026723
0,68
0,57
0,24
0,58
0
0,62
8
chrl5:62360801-62360802
0,54
0,72
0,2
0,4
0
0
9
chrl7:31620119-31620120
0,56
0,54
0
0,61
0,21
0,47
10
chrl9:56609978-56609979
0,76
0,87
0,56
0,89
0,25
1
11
chr2:130344496-130344497
0,29
1
0,15
0,69
0,15
0,38
12
chr2:16154295-16154296
0,35
0,96
0,43
0,48
0
1
13
chr2:52799788-52799789
0
0,58
0,13
0,63
0
0,96
14
chr4:1040977-1040978
0,39
0,9
0,53
1
0,25
0,42
15
chr7:101361449-101361450
0
0,46
0
0,87
0,28
0,79
16
chr8:141109628-141109629
0,42
0,75
0,27
0,66
0,23
0,91
17
chr8:141109651-141109652
0,42
0,81
0,27
0,71
0,28
1
18
chr8:141109655-141109656
0,42
0,89
0,24
0,7
0,31
1
19
chr8:141109686-141109687
0,42
0,92
0,28
0,67
0,31
1
Рис. 2. Уровни метилирования динуклеотидов CpG в трех парах сублиний по результатам RRBS. Приведены значения B-value для каждой координаты дифференциально метилированных CpG по 6 клеточным линиям
-
2) внутригенных участках РНК-генов AC010145.4 (chr2:16154295-16154296), DIO3OS (chr14:102026722-102026723);
-
3) внутригенных участках генов, кодирующих белки (по большей части в экзонах и внутригенных энхансерах, подробнее см. ниже).
Гиперметилирование отмечено в CpG генов CRTAC1 (chr10:99734891-99734892), TRAPPC9 (chr8:141109628-141109629, chr8:141109651-141109652, chr8:141109686-141109687, chr8:141109655-141109656), ASIC2 (chr17:31620119-31620120), C2CD4A (chr15:62360801-62360802), ZNF787 (chr19:56609978-56609979).
Гипометилирование во всех резистентных сублиниях по сравнению с контрольными было отмечено для 4 CpG, среди которых:
-
1) 2 CpG относятся к последовательностям белок-кодирующих генов: PRKCZ (chr1:2036833-2036834), CRTAC1 (chr10:99734998-99734999);
-
2) РНК-ген RP11-495K9.9 (chr12:132107115-132107116);
-
3) CpG из межгенного пространства (chr19:2462305-2462306).
На рис. 3 приведены координаты дифференциально метилированных CpG, названия генов (при наличии), к которым относятся CpG, а также направление изменения их метилирования у резистентных сублиний по сравнению с контрольными.
Обсуждение
Ранее мы показали, что ко-культивирование клеток эстрогензависимого рака молочной железы MCF-7 с тамоксифен-резистентными клет-
|
ген |
координаты GRCh37/hgl9 |
|
|
PRKCZ |
chrl: 2036833-2036834 |
метилирование снизилось |
|
CRT AC 1 |
chrl 0:997349 98-99734999 |
|
|
RP1M95K9.9 |
chrl 2:132107115-132107116 |
|
|
chrl 9:2462305-2462306 |
||
|
CRTAC1 |
chrl 0:997348 91-99734892 |
метилирование повысилось |
|
chrl 1:1858802-1858803 |
||
|
chrl4:102026722-102026723 |
||
|
C2CD4A |
chrl5:62360801-62360802 |
|
|
ASIC2 |
chr!7:31620119-31620120 |
|
|
ZNF787 |
chrl 9:56609978-56609979 |
|
|
chr2:130344496-130344497 |
||
|
AC010145.4 |
chr2:16154295-16154296 |
|
|
chr2:52799788-52799789 |
||
|
chr4:1040977-1040 978 |
||
|
chr7:101361449-101361450 |
||
|
TRAPPC9 |
chr8:141109628-141109629 |
|
|
TRAPPC9 |
chr8:141109651-141109652 |
|
|
TRAPPC9 |
chr8:141109655-141109656 |
|
|
TRAPPC9 |
chrS: 141109686-141109687 |
|
Рис. 3. Координаты дифференциально метилированных CpG в резистентных линиях и изменение их метилирования
ками MCF-7/T приводит к развитию частичной резистентности клеток MCF-7 [20]. В дальнейших исследованиях было продемонстрировано непосредственное участие экзосом в подобном горизонтальном пути распространения резистентности [16]. Для ответа на вопрос, в какой мере горизонтальный путь развития резистентности воспроизводит основные характеристики первично-резистентных клеток, был проведен анализ общего уровня метилирования ДНК в чувствительных и резистентных клетках MCF-7.
Дифференциальное изменение метилирования было обнаружено для участков ДНК, регулирующих экспрессию шести белок-кодирующих генов: PRKCZ, TRAPPC9, ASIC2, C2CD4a, ZNF787, CRTAC1. Ген CRTAC1 даже дважды попал в этот список, поскольку направление изменения метилирования у двух соседних динуклеотидов (на расстоянии 100 bp) различалось (гипо- и гиперметилирование). Общие паттерны дифференциального метилирования динуклеотидов CpG во всех шести сублиниях свидетельствуют о необходимости дальнейшего исследования потенциальной значимости этого набора генов при развитии резистентности к тамоксифену, в том числе с помощью экзосом.
Для трех генов из приведенного списка, C2CD4a, ZNF787, CRTAC1, не удалось обнаружить какой-либо возможной связи с РМЖ или с развитием каких-либо опухолей в целом. Стоит отметить, что про функцию этих генов известно довольно мало. Однако для трех других генов существуют определенные доказательства участия в регуляции опухолевой прогрессии.
Так, ген ASIC2 (acid sensing ion channel subunit 2) кодирует кислотно-чувствительный ионный канал, принадлежащий к суперсемейству дегенеринов/ эпителиальных натриевых каналов (DEG / ENaC) [21, 22]. В работе [23] была показана связь ASIC2
с развитием и метастазированием колоректального рака (CRC) за счет активации сигнального пути calcineurin/NFAT1 в условиях ацидоза [23]. По данным Protein Atlas (https://www.proteinatlas. org/ENSG00000108684-ASIC2/pathology), мРНК и белок ASIC2 определяются в линиях клеток РМЖ. За тканеспецифичную экспрессию гена ASIC2 отвечают, по данным GeneHancer, 92 энхансер-ных элемента. Координаты дифференциально метилированного динуклеотида CpG: 17:3162011931620120 относятся к дистальной части последовательности внутригенного энхансера, GH17J033292 (chr17:31619018-31620430 (GRCh37/hg19). Помимо ASIC2 данный энхансер регулирует экспрессию GC17P033138 и GC17M033432, и сам регулируется следующими транскрипционными факторами: GLIS2, ZIC2, ZBTB8A, PRDM10, CTCF, PATZ1, KLF9, ZBTB48, ZBTB26, ZFHX2, SIN3A, ZNF660, ZNF335, EGR2.
Ген PRKCZ (протеинкиназа С Zeta) кодирует атипичную потеинкиназу семейства серин/треони-новых киназ PKC, которые участвуют в различных клеточных процессах, таких как пролиферация, дифференцировка и секреция. В отличие от классических изоферментов РКС, PRKCZ имеет единственный домен типа цинковых пальцев. Функциональная активность PRKCZ включает реорганизацию микротрубочек цитоскелета, позитивную регуляцию адгезии к внеклеточному матриксу, фосфорилирование белков, участие в воспалительном ответе, в том числе NF-κB – одного из негативных регуляторов ER. CpG с дифференциальным метилированием в резистентных к тамоксифену сублиниях был обнаружен в 4-м интроне из 17 (1:2036833-2036834) и относится к последовательности внутригенного энхансера GH01J002104, контролирующего экспрессию 7 генов: помимо PRKCZ, это SKI (протоонкоген SKI), ANKRD65 (домен повторения Ankyrin 65), FAAP20
(белок 20, связанный с комплексом FA), РНК-гены ENSG00000271806 и lncRNAs GC01M002070 и GC01M002102.
Еще один дифференциально метилированный ген TRAPPC9 (Trafficking Protein Particle Complex Subunit 9) кодирует субъединицу транспортного комплекса TRAPP, являющегося одним из позитивных регуляторов NF-κB [24, 25]. Интересно, что подавление экспрессии TRAPPC9 ослабляет пролиферацию, инвазию, миграцию, образование колоний и рост ксенографтов для клеточной линии РМЖ MDA-MB-231 [25]. Участие TRAPPC9 в регуляции сигналлинга NF-κB и в онкогенезе было в дальнейшем подтверждено еще в нескольких работах [25–27].
Следует отметить, что полученные нами данные расширяют и дополняют спектр генов, метилирование которых коррелирует с развитием гормональной резистентности. Так, судя по известным литературным данным, изменения метилирования ДНК, сопровождающие развитие гормональной резистентности, представляют собой достаточно вариабельный процесс и могут охватывать широкий спектр генов в каждом отдельном случае [28, 29]. Какие из этих изменений являются ведущими, необходимыми и достаточными для развития резистентности – именно для ответа на этот вопрос было выполнено настоящее исследование на трех различных моделях резистентности, результатом которого явилась идентификация общих паттернов метилирования ДНК, ассоциированных с формированием резистентного фенотипа клеток.
Анализируя вопрос об эпигенетических факторах, участвующих в формировании гормональной резистентности, нельзя не отметить микроРНК как класс биологически активных соединений, чью роль в развитии и поддержании резистентности трудно переоценить. Среди них: mir-27, mir-181, mir-221/222, mir-21, mir-101 и ряд других, обладающих широким спектром действия, в первую
Список литературы Изменение профиля метилирования ДНК в тамоксифен-резистентных сублиниях клеток MCF-7
- Ferlay J., Colombet M., Soerjomataram I., Mathers C., Parkin D.M., Pineros M., Znaor A., Bray F. Estimating the global cancer incidence and mortality in 2018: GLOBOCAN sources and methods. Int J Cancer. 2019 Apr 15; 144(8): 1941-1953. DOI: 10.1002/ijc.31937
- Meric-Bernstam F. Heterogenic loss of BRCA in breast cancer: the "two-hit" hypothesis takes a hit. Ann Surg Oncol. 2007. 14(9): 2428-2429. DOI: 10.1245/s10434-007-9379-7
- Di Cosimo S., Baselga J. Management of breast cancer with targeted agents: importance of heterogeneity. [corrected]. Nat Rev Clin Oncol. 2010 Mar; 7(3): 139-47. DOI: 10.1038/nrclinonc.2009.234
- Sachs N., de Ligt J., Kopper O., Gogola E., Bounova G., Weeber F., Balgobind A.V., Wind K., Gracanin A., Begthel H., Korving J., van Boxtel R., Duarte A.A., Lelieveld D., van Hoeck A., Ernst R.F., Blokzijl F., Nijman I.J., Hoogstraat M., van de Ven M., Egan D.A., Zinzalla V., Moll J., Boj S.F., Voest E.E., Wessels L., van Diest P.J., Rottenberg S., Vries R.G.J., Cuppen E., Clevers H. A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity. Cell. 2018 Jan 11; 172(1-2): 373-386.e10. DOI: 10.1016/j.cell.2017.11.010
- Keller R.R., Gunther E.J. Evolution of Relapse-Proficient Subclones Constrained by Collateral Sensitivity to Oncogene Overdose in Wnt-Driven Mammary Cancer. Cell Rep. 2019 Jan 22; 26(4): 893-905. e4. DOI: 10.1016/j.celrep.2018.12.096


