Изоферментный анализ эстераз в зрелых семенах гексаплоидной мягкой пшеницы (Triticum aestivum L.)
Автор: Рудакова А.С., Рудаков С.В., Давыдова Н.В., Мирская Г.В., Журавлева Екатерина Васильевна, Чесноков Юрий Валентинович
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетические основы устойчивости, цитогенетические и биохимические маркеры
Статья в выпуске: 3 т.51, 2016 года.
Бесплатный доступ
Эстеразы - большая группа ферментов, катализирующих расщепление сложноэфирных связей. Обычно их разделяют на четыре типа: холинэстеразы (наиболее часто идентифицируются при традиционном электрофоретическом анализе), ацетилэстеразы, арилэстеразы и карбоксилэстеразы. Эстеразы растений катализируют превращение карбоксильных эфиров в биоактивные кислоты и спирты, благодаря чему играют ключевую роль во многих биологических процессах. Отсутствие эпистатических взаимодействий и кодоминантный характер наследования изоформ эстераз делают их значимыми для быстрого и доступного изучения процессов биохимической адаптации к изменяющимся условиям окружающей среды. Этот тип маркеров удобен и для решения практических задач селекции в качестве средства, способного ускорить и упростить процесс отбора селекционно значимого материала. Целью настоящей работы была оценка изоферментного профиля эстераз, выделенных из зрелых семян, и установление полиморфизма между образцами перспективного селекционного материала у гексаплоидной пшеницы ( Triticum aestivum L.) по этому биохимическому маркеру. Исследовали зрелые семена сортов пшеницы селекции Московского НИИ сельского хозяйства «Немчиновка» (Московская обл.) - Злата, Любава, Агата, Лиза (яровые) и Мера (озимый); селекции Агрофизического НИИ (АФИ, г. Санкт-Петербург) - линии АФИ91 и АФИ177 (яровые), а также рекомбинантных инбредных линий картирующей популяции ITMI - 7, 10, 29, 32, 44, 47, 57, 83, 88, 89 и 115 (яровые). Семена размалывали в фарфоровой ступке, муку просеивали через сито. После экстракции ферментов образцы центрифугировали и отбирали надосадочную жидкость, затем осуществляли вертикальный нативный электрофорез. В качестве разделяющего использовали 8 % полиакриламидный гель, в качестве концентрирующего - 4 % полиакриламидный гель. Молекулярными маркерами служили окрашенные стандарты Page Ruler Prestained Protein Ladder («Thermo Scientific», Литва). По окончании электрофореза гель обрабатывали реактивом на неспецифическую эстеразу. Сканировали полученные электрофореграммы, оценивали индивидуальный электрофоретический профиль каждого образца, рассчитывали гетерозиготность и ее дисперсию. Эстеразный комплекс в исследованных семенах пшеницы был представлен 10 формами. У сортов Злата, Любава, Агата, Лиза и Мера выявили от 9 до 10 изоформ с разной электрофоретической подвижностью, у АФИ91 обнаружено 7, в АФИ177 - 8, у линий ITMI - от 7 до 10 изоформ эстераз. Для всех образцов было характерно наличие изоформ эстераз Est-8, Est-9 и Est-10. Гетерогенность отмечали только по качественному и количественному составу эстераз с большей молекулярной массой - Est-1, Est-2, Est-3, Est-4, Est-5, Est-6 и Est-7. Каждый из 18 сортов и линий обладал отличным от других образцов генотипом. Средняя гетерозиготность (H) образцов по 10 выявленным локусам, кодирующим изоформы эстераз, составила 0,924; дисперсия гетерозиготности для изученного набора образцов Var(H) = 0,0004. В результате исследований выделены наиболее перспективные селекционные родительские формы - сорта Злата, Мера и линии АФИ91, АФИ177, ITMI7, ITMI44, ITMI83 и ITMI115. Наличие 10 изоформ делает эстеразы гексаплоидной пшеницы удобным биохимическим маркером и позволяет проводить как физиолого-биохимические, так и генетико-селекционные исследования образцов гексаплоидной мягкой пшеницы.
Гексаплоидная мягкая пшеница, зрелые семена, изоферментный анализ, эстеразы
Короткий адрес: https://sciup.org/142213941
IDR: 142213941 | УДК: 633.111.1:577.152.31:577.151.64 | DOI: 10.15389/agrobiology.2016.3.327rus
Текст научной статьи Изоферментный анализ эстераз в зрелых семенах гексаплоидной мягкой пшеницы (Triticum aestivum L.)
Классическая фенотипическая оценка селекционно значимого материала становится все менее выгодной с увеличением числа коллекций генетических ресурсов растений, поскольку требует временных и денежных затрат, а полученные для генотипа результаты варьируют при выращивании в различных условиях. Анализ на основании полиморфного электрофоретического спектра белковых изоферментных маркеров — это более дешевый подход, который с успехом может применяться для экс-пресс-оценки генетических ресурсов растений, установления филогенетических отношений между разными видами и выявления дрейфа генов или иных генетико-эволюционных процессов на популяционном уровне (1-5).
Эстеразы — большая группа ферментов (не менее 20), катализирующих расщепление сложноэфирных связей. В широком смысле к эстеразам относятся липазы, фосфатазы, сульфатазы и собственно эстеразы. Последние включают в себя многочисленные специфические ферменты: холинэстеразу, хлорофиллазу, танназу, пектазу и др. Обычно эстеразы разделяют на четыре типа: холинэстеразы предпочитают заряженные субстраты (эфиры холина с уксусной, пропионовой или масляной кислотами), этот тип эстераз наиболее часто идентифицируется при традиционном электрофоретическом анализе; ацетилэстеразы используют в качестве субстратов алифатические углеводороды, в частности уксусную кислоту; арил-эстеразы ингибируются сероводородными компонентами, субстратами для них служат ароматические углеводороды; карбоксилэстеразы предпочитают эфиры алифатических (достаточно «длинных») соединений, ингибируются, как и холинэстеразы, фосфорорганическими веществами (6-9).
Эстеразы растений (EC 3.1.1.X) — это разновидность эстераз, присутствующих у сои, пшеницы, сорго, риса и других видов (6, 10, 11). Они катализируют превращение карбоксильных эфиров в биоактивные кислоты и спирты, благодаря чему играют ключевую роль во многих биологических процессах, например в активации сигнальных молекул (12) и регуляции биоактивности эндогенных продуктов (13), включая гидролитическое расщепление эфирных связей пестицидов и других ксенобиотиков (6). Так, гербициды тиазопир, диклофопметил и бромоксинилоктаноат, а также фунгицид бинапакрил инактивируются действием эстераз (6, 14).
Гексаплоидная пшеница (Triticum aestivum L.) — один из возделываемых видов, для которых компонентный состав эстераз был изучен, но генетический контроль их активности до сих пор полностью не установлен. Описаны четыре блока гомеологичных локусов, контролирующие эстеразы в различных органах и тканях пшеницы. Гены эстераз незрелых семян, корней, листовых пластинок и зеленых листьев растений картированы на 3AS, 3BS и 3DS плечах хромосом (15-17). Локусы эстераз этиолированных колеоптилей идентифицированы на 3AL, 3BL, 3DL, 7BS и 7DS (18), этиолированных листьев — на 6AL, 6BL и 6DL (18-20), зрелых семян — на длинных плечах хромосом 3AL, 3BL и 3DL (20-22). Эстеразы пшеницы в соответствии со структурной композицией были описаны как мономеры или димеры (15, 18, 19).
Отсутствие эпистатических взаимодействий и кодоминантный характер наследования изоформ эстераз делают их значимыми для быстрого и доступного изучения процессов биохимической адаптации к изменяющимся условиям окружающей среды. Этот тип биохимических маркеров удобен и для решения практических задач селекции в качестве средства, способного ускорить и упростить процесс отбора селекционно значимого материала.
В настоящей работе мы впервые провели изоферментный анализ ряда линий и сортов яровой и озимой пшеницы, что позволило определить возможные родительские формы для последующего получения исходного и гибридного материала в селекционных программах, направленных на повышение устойчивости и продуктивности у Triticum aestivum L.
Целью работы была оценка изоферментного профиля эстераз, выделенных из зрелых семян, и установление полиморфизма между образцами перспективного селекционного материала у гексаплоидной пшеницы по этому биохимическому маркеру.
Методика. Материалом для исследований служили зрелые семена сортов пшеницы (Triticum aestivum L.) селекции Московского НИИ сель-328
ского хозяйства «Немчиновка» — Злата, Любава, Агата, Лиза (яровые) и Мера (озимый); селекции Агрофизического НИИ (АФИ) — линии АФИ91 и АФИ177 (яровые), а также рекомбинантных инбредных линий картирующей популяции ITMI — 7, 10, 29, 32, 44, 47, 57, 83, 88, 89 и 115 (яровые).
Семена тщательно размалывали в фарфоровой ступке, муку просеивали через сито (менее 200 мкм). Экстракцию ферментов проводили при температуре 8-12 ° C в течение ночи, соотношение мука:буфер — 1:2. В качестве буфера использовали 0,005 М Трис-HCl (рН 8,3), 0,001 M MgCl2, 10 % глицерин. Экстракты центрифугировали 5 мин при 15000 об/мин и отбирали надосадочную жидкость. Затем осуществляли вертикальный нативный электрофорез (23). В качестве разделяющего использовали 8 % полиакриламидный гель, в качестве концентрирующего — 4 % полиакриламидный гель. Электрофорез проводили в камере Mini-PROTEAN Tetra System («Bio-Rad Laboratories, Inc.», США) при 50 мА/ч и температуре 5-8 ° C. Молекулярными маркерами служили предокрашенные стандарты Page Ruler Prestained Protein Ladder («Thermo Scientific», Литва).
На одну дорожку геля вносили 10-15 мкл раствора белка (без красителей и других агентов), по окончании электрофореза гель обрабатывали реактивом на неспецифическую эстеразу. Для этого гель выдерживали в свежеприготовленном растворе красителя в течение 5-10 мин до появления коричневых зон. Краситель для изоформ эстеразы готовили следующим образом: 0,1 г 1-нафтилацетата («Sigma-Aldrich», Швейцария) растворяли в 5 мл 70 % этанола, затем медленно, при перемешивании вливали в 15 мл 0,1 М фосфатного буфера (рН 6,0); 0,25 г синего прочного Fast Blue RR («Aldrich», США) растворяли в 4 мл пропанола и 15 мл 0,1 М фосфатного буфера (рН 6,0). Оба раствора смешивали (1:1), фильтровали через складчатый бумажный фильтр и сразу же использовали для проявления зон с эстеразной активностью (24). После того как зоны становились темными, избыток красителя удаляли, промывая гель 7 % уксусной кислотой.
Полученные электрофореграммы сканировали на Epson Expression 10000 XL («GE Healthcare», США). Индивидуальный электрофоретический профиль каждого образца оценивали с помощью программы Phoretix 1D («TotalLab, Ltd.», Великобритания).
Расчет гетерозиготности и ее дисперсии проводили по формулам, предложенным М. Nei (25) и Б. Вейр (26), базируясь на описанных нами ранее алгоритмах количественной оценки степени полиморфизма (27).
Результаты. Получение и описание линий картирующей популяции ITMI были опубликованы нами ранее (28-30). Описание и характеристики сортов Злата, Любава, Агата, Лиза и Мера приведены в соответствующих базах данных Государственного реестра РФ.
Линия АФИ91 была получена посредством индивидуального отбора в комбинации скрещивания сортов яровой мягкой пшеницы Asakaze ko-mugi х Ленинградская ранняя. Линия скороспелая, низкорослая, полуос-тистистая. Форма куста эректоидная. Листовая пластинка длинная и узкая. Колос пирамидальный, плотность средняя, длина колоса средняя, число зерен в колосе малое. Масса зерна с колоса изменяется от малой до средней, зерно мелкое, удлиненное, окраска красная. Линию АФИ177 получили индивидуальным отбором в комбинации скрещивания сортов яровой мягкой пшеницы Asakaze komugi х Sonora 64. Линия скороспелая, полу-карликовая, полуостистистая. Форма куста наклонная. Листовая пластинка средняя по длине и ширине. Колос пирамидальный, плотность средняя, длина колоса средняя, число зерен в колосе среднее. Масса зерна с колоса изменяется от малой до средней, зерно среднее, окраска красная.
Эстеразный комплекс в исследованных семенах пшеницы был представлен 10 формами (рис.). У сортов Злата, Любава, Агата, Лиза и Мера выявили от 9 до 10 изоформ с разной электрофоретической подвижностью. Сорта Агата и Лиза имели схожий изоферментный профиль, но активность Est-2 у сорта Агата оказалась более выражена.
АФИ91 АФИ177 Злата Любава Агата Лиза Мера 115
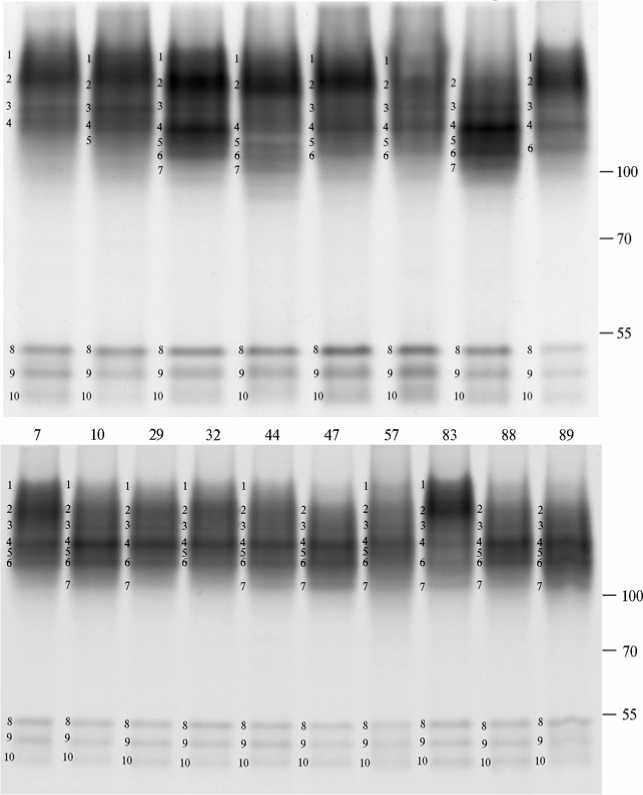
Электрофоретические профили изоформ эстераз у изученных сортов гексаплоидной мягкой пшеницы Triticum aestivum L. и рекомбинантных инбредных линий картирующей популяции ITMI. Номерам линий ITMI соответствуют цифры над дорожками; цифры справа — маркеры молекулярной массы, кДа; вдоль дорожек пронумерованы изоформы эстераз.
У АФИ91 было обнаружено 7, в АФИ177 — 8, у линий ITMI — от 7 до 10 изоформ эстераз. Изоферментные профили линий ITMI отличались друг от друга. Только профили линий ITMI10 и ITMI29 оказались схожи как по качественному, так и по количественному составу, однако они отличались от профилей линий АФИ91 и АФИ177, а также от профилей у всех изученных сортов, что позволяет использовать их для анализа при скрещиваниях с любым из исследованных сортов или с линиями АФИ91 или АФИ177.
Для всех образцов было характерно наличие изоформ эстераз Est-8, Est-9 и Est-10. Гетерогенность отмечали только по качественному и количественному составу эстераз с большей молекулярной массой — Est-1, Est-330
-
2, Est-3, Est-4, Est-5, Est-6 и Est-7.
Эстеразы Est-2 и Est-4 присутствовали у всех изученных образцов, однако активность Est-2 оказалась выше у сортов Злата, Любава, Агата и линий ITMI7, ITMI83, ITMI115, а активность Est-4 — у сортов Злата, Мера и линий ITMI10, ITMI29, ITMI32, ITMI44, ITMI47, ITMI88, ITMI89. Изоформа Est-6 была обнаружена у всех оцененных сортов и всех линий ITMI, за исключением линий АФИ91 и АФИ177. Максимальную активность Est-6 выявили у сорта Мера и линии ITMI89, у остальных сортов и линий ITMI она варьировала от низкой до средней. Изоформа Est-1 присутствовала у всех образцов, за исключением сорта Мера и линий ITMI47, ITMI88 и ITMI89. Изоформа Est-7 была обнаружена у сортов Злата, Любава и Мера, а также у линий ITMI10, ITMI29, ITMI44, ITMI47, ITMI57, ITMI83 и ITMI89. Наличие изоформы Est-3 отмечали у всех образцов, кроме сорта Любава, активность Est-3 была наибольшей у сорта Мера. Эстераза Est-5 отсутствовала только у линий АФИ91, ITMI29, ITMI83 и ITMI115, ее максимальную активность отмечали у сортов Злата, Мера и линии ITMI89.
Интересно, что сорт Мера, единственный озимый среди изученных образцов, обладал девятью из десяти изоформ эстераз. При этом активность изоформ со средней молекулярной массой у него оказалась выше, чем активность изоформ с низкой и высокой молекулярной массой. То есть сорт Мера можно использовать в качестве родительской формы при проведении скрещивания с любым из оцененных по изоформентному спектру эстераз образцов. Линии АФИ91 и АФИ177 также могут служить родительскими формами в скрещиваниях друг с другом или с остальными исследованными линиями и сортами.
Сорта Злата и Любава обладали уникальным электрофоретическим спектром изоформ эстераз. Сорт Злата отличается от сорта Любава наличием изоформы Est-3 и более высокой активностью Est-4. Сорта Агата и Лиза хоть и характеризовались схожими спектрами, но у сорта Агата была отмечена более высокая активность Est-2. Линии ITMI обладали схожими профилями изоформ эстераз, однако отличались друг от друга и от линий АФИ91 и АФИ177, а также от всех сортов. Линии ITMI7, ITMI83, ITMI89 и ITMI 115 имели явно выраженные отличительные морфологические характеристики (30), что позволяет использовать их в качестве исходного материала при проведении генетико-селекционных скрещиваний.
Каждый из 18 сортов и линий обладал отличным от других образцов генотипом. Мы оценили их гетерозиготность по 10 выявленным локусам, кодирующим изоформы эстераз. Средняя гетерозиготность (H) составила 0,924; дисперсия гетерозиготности для изученного набора образцов Var(H) = 0,0004.
Поскольку размер выборки был относительно мал и полиморфными оказались только 5 изоформ эстераз, для расчета гетерозиготности мы использовали подход, предложенный М. Nei (25) и Б. Вейр (26), который позволил более точно оценить среднюю гетерозиготность и ее дисперсию. Выявленная гетерозиготность рассматривалась как средняя порция локусов с двумя различными аллелями в одном локусе у одной особи и могла быть определена как наблюдаемая гетерозиготность, характеризующая часть генов, по которым популяция гетерозиготна (27). Основное отличие использованных нами статистических подходов заключается в том, что авторы (25, 26) вывели единообразно распределяемое минимальное отклонение несмещенной оценки гетерозиготности (Н) в соответствии с его точным значением дисперсии Var(H). Для этого они получили формулы, позволяющие рассчитать любой многочлен в наборе переменных, распределенных мультино-331
миально, что дает возможность рассматривать выявляемую гетерозиготность как меру информационного полиморфизма, служащего важным компонентом при составлении планов селекционных программ и одним из ключевых информационно-статистических показателей при их выполнении. Однако обнаружение любых селекционно значимых свойств зависит от генотипа образцов, а следовательно, информационный полиморфизм есть не что иное, как отражение выявляемой на фенотипическом и(или) биохимическом уровне активности и распределения в геноме генетических детерминант (генов, локусов хромосом), определяющих проявление изучаемых признаков.
Ранее было показано, что гены, кодирующие изоформы эстераз, располагаются в хромосоме 3 всех трех геномов гексаплоидной пшеницы (20-22). Из этого следует, что в процессе эволюции происходила дифференциация этой части геномов. Можно предположить как минимум два мутационных события, повлиявших на распределение изоформ эстераз среди гомологичных хромосом. Это может быть также отнесено к гибридным формам изоферментов (димеры, тримеры или тетрамеры и т.п.), поскольку получение таких форм обусловлено только наличием различных вариантов полимеров в тканях и органах растений (15, 18, 19). Тот факт, что выявленные изоферменты кодируются гомеологичными хромосомами (и, возможно, их плечами), хорошо согласуется с гипотезой о сравнительно недавней дифференциации геномов А, В и D у пшеницы (21). Переход от тетраплоидности к гексаплоидным формам, подтверждаемый аддитивностью изоформ эстераз, приводит к обобщающему заключению о возможности использовать систему изоформ эстераз в организации и увеличении пластичности полиплоидов и их адаптации к различным условиям окружающей среды. Это согласуется с наблюдением, что полиплоиды способны более эффективно колонизировать ареалы по сравнению с близкородственными диплоидными видами (3, 6).
Таким образом, полученные результаты позволили выделить из исследованного материала наиболее перспективные селекционные родительские формы — сорта Злата, Мера и линии АФИ91, АФИ177, ITMI7, ITMI44, ITMI83 и ITMI115, которые различались не только по фенологическим и фенотипическим показателям, но и по спектрам изоформ эстераз. При этом существует возможность определения полиморфизма не только у исходных форм, но и у предполагаемых потомков, полученных в результате скрещиваний. Наличие десяти изоформ делает эстеразы гексаплоидной пшеницы удобным биохимическим маркером и позволяет проводить как физиолого-биохимические, так и генетико-селекционные исследования. Более детальная интерпретация полученных изоферментных профилей станет возможной после сравнительного анализа изоферментных профилей у диплоидных доноров геномов изученных форм.
Выражаем благодарность НВ. Кочериной за помощь при проведении статистических расчетов гетерозиготности и ее дисперсии.
Список литературы Изоферментный анализ эстераз в зрелых семенах гексаплоидной мягкой пшеницы (Triticum aestivum L.)
- Boulter D., Thurmann D.A., Turner B.L. The use of disc electrophoresis of plant proteins in systematics. Taxon, 1966, 15: 135-142 ( ) DOI: 10.2307/1217532
- Shaw C.R. Isozymes: classification, frequency, and significance. Int. Rev. Cytol., 1969, 25: 297-332 ( ) DOI: 10.1016/S0074-7696(08)60206-5
- Hart G.E. Genetics and evolution of multilocus isozymes in hexaploid wheat. Isozymes Curr. Top. Biol. Med. Res., 1983, 10: 365-380.
- Brown A.H., Clegg M.T. Isozyme assessment of plant genetic resources. Isozymes Curr. Top. Biol. Med. Res., 1983, 11: 285-295.
- Nevo E. Plant genetic resources: prediction by isozyme markers and ecology. Isozymes Curr. Top. Biol. Med. Res., 1987, 16: 247-267.
- Cummins I., Burnet M., Edwards R. Biochemical characterization of esterases active in hydrolysing xenobiotics in wheat and competing weeds. Physilologia Plantarum, 2001, 113: 477-485.
- Carvalho V.M., Marques R.M., Lapenta A.S., Machado M.F.P.S. Functional classification of esterases from leaves of Aspidosperma polyneuron M. Arg. (Apocynaceae). Genet. Mol. Biol., 2003, 26(2): 195-198 ( ) DOI: 10.1590/S1415-47572003000200013
- Hou C.-J., He K., Yang L.-M., Huo D.-Q., Yang M., Huang S., Zhang L., Shen C.-H. Catalytic characteristics of plant-esterase from wheat flour. World J. Microbiol. Biotechnol., 2012, 28: 541-548 ( ) DOI: 10.1007/s11274-011-0845-9
- Rejon J.D., Zienkiewicz A., Rodríguez-García M.I., Castro A.J. Profiling and functional classification of esterases in olive (Olea europaea) pollen during germination. Ann. Bot., 2012, 110(5): 1035-1045 ( ) DOI: 10.1093/aob/mcs174
- Gershater M.C., Cummins I., Edwards R. Role of a carboxylesterase in herbicide bioactivation in Arabidopsis thaliana. J. Biol. Chem., 2007, 282: 21460-21466 ( ) DOI: 10.1074/jbc.M701985200
- Cummins I., Edwards R. Purification and cloning of an esterase from the weed black-grass (Alopecurus myosuroides), which bioactivates aryloxyphenoxypropionate herbicides. Plant J., 2004, 39: 894-904 ( ) DOI: 10.1111/j.1365-313X.2004.02174.x
- Stuhlfelder C., Mueller M.J., Warzecha H. Cloning and expression of a tomato cDNA encoding a methyl jasmonate cleaving esterase. Eur. J. Biochem., 2004, 271: 2976-2983 ( ) DOI: 10.1111/j.1432-1033.2004.04227.x
- Dogru E., Warzecha H., Seibel F., Haebel S., Lottspeich F., Stockigt J. The gene encoding polyneuridine aldehyde esterase of monoterpenoid indole alkaloid biosynthesis in plants is an ortholog of the alpha/betahydrolase super family. Eur. J. Biochem., 2000, 267: 1397-1406 ( ) DOI: 10.1046/j.1432-1327.2000.01136.x
- Feng P.C.C., Ruff T.G., Kosinski W.G. Metabolic deactivation of the herbicide thiazopyr by animal liver esterases. Xenobiotica, 1995, 25: 27-35 ( ) DOI: 10.3109/00498259509061830
- Barber H.N., Driscoll C.J., Vickery R.S. Enzymic markers for wheat and rye chromosomes. In: Proc. 8th Int. Wheat Genet. Symp. Aust. Acad. Sci., Canberra, 1968: 116-122.
- Barber H.N., Driscoll C.J., Long P.M., Vickery R.S. Gene similarity of the Triticinae and the study of segmental interchanges. Nature, 1969, 22: 897-898 ( ) DOI: 10.1038/222897a0
- Bergman J.W. Chromosome locations of genes controlling esterase and malate dehydrogenase isozymes in Triticum. PhD Diss. North Dakota State University, Fargo, 1972.
- Jaaska V. Electrophoretic survey of seedling esterased in wheats in relation to their phylogeny. Theor. Appl. Genet., 1980, 56: 273-284 ( ) DOI: 10.1007/BF00282570
- May C.E., Vickery R.S., Driscoll C.S. Gene control in hexaploid wheat. In: Proc. 4th Int. Wheat Genet. Symp. Columbia, 1973: 843-849.
- Nakai I. Isoenzyme variations in Aegilops and Triticum. 3. Chromosomal basis of the esterase isozyme production in indifferent organs of Chinese Spring wheat. Bot. Mag., 1976, 89: 219-234 ( ) DOI: 10.1007/BF02488344
- Cubadda R., Bozzini A., Quatrucci E. Genetic control of esterases in common wheat. Theor. Appl. Genet., 1975, 45: 290-293 ( ) DOI: 10.1007/BF00276681
- Ainsworth C.C., Gale M.D., Baird S. The genetic control of grain esterases in hexaploid wheat. Theor. Appl. Genet., 1984, 68: 219-226 ( ) DOI: 10.1007/BF00266893
- Davis B.J. Disc electrophoresis. II. Method and application to human serum proteins. Annals of the NY Academy of Sciences, 1964, 121: 404-427 ( ) DOI: 10.1111/j.1749-6632.1964.tb14213.x
- Meon S. Protein, esterase and peroxidase patterns of phytophtora isolates from Cocoa in Malaysia. J. Islamic Acad. Sci., 1988, 1(2): 154-158.
- Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89: 583-590.
- Вейр Б. Анализ генетических данных. М., 1995: 132-135.
- Чесноков Ю.В., Почепня Н.В., Козленко Л.В., Ситников М.Н., Митрофанова О.П., Сюков В.В., Кочетков Д.В., Ловассер У., Бёрнер А. Картирование QTL, определяющих проявление агрономически и хозяйственно ценных признаков у яровой мягкой пшеницы (Triticum aestivum L.) в различных экологических регионах России. Вавиловский журнал генетики и селекции, 2012, 16(4/2): 970-998.
- Чесноков Ю.В., Гончарова Э.А., Ситников М.Н., Кочерина Н.В., Ловассер У., Бёрнер А. Картирование QTL водного режима у яровой мягкой пшеницы. Физиология растений, 2014, 61(6): 855-863 ( ) DOI: 10.7868/S0015330314060049
- Chesnokov Yu.V., Sitnikov M.N., Schumlyanskaya N.V., Kocherina N.V., Goncharova E.A., Kozlenko L.V., Syukov V.V., Kochetkov D.V., Lohwasser U., Börner A. Catalogue of recombinant inbred lines of mapping population ITMI of soft spring wheat Triticum aestivum L. (ecological and geographical trials and QTL mapping). IPK, VIR, St. Petersburg-Gatersleben, 2014.
- Чесноков Ю.В., Артемьева А.М. Оценка меры информационного полиморфизма генетического разнообразия. Сельскохозяйственная биология, 2015, 50(5): 571-578 ( , 10.15389/agrobiology.2015.5.571eng) DOI: 10.15389/agrobiology.2015.5.571rus


