Изучение генетического разнообразия современных сортов яблони ( Malus domestica Borkh.) отечественной селекции с использованием микросателлитных локусов
Автор: Супрун И.И., Ушакова Я.В., Токмаков С.В., Дюрель Ч.Э., Денанс К., Ульяновская Е.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярная структура генома и селекция
Статья в выпуске: 1 т.50, 2015 года.
Бесплатный доступ
Микросателлитные ДНК-маркеры, основанные на анализе простых повторяющихся повторов (SSR - simple sequence repeats), признаются одной из наиболее эффективных ДНК-маркерных систем, используемых в селекции и генетике культурных растений. С помощью SSR-маркеров были построены первые наиболее насыщенные генетические карты яблони. Кроме того, выполнен широкий перечень исследований, направленных на изучение генетического разнообразия в коллекциях сортов, диких форм, межвидовых гибридов яблони. C целью изучения генетической структуры рабочей коллекции современных сортов яблони отечественной селекции был оценен полиморфизм 12 микросателлитных локусов в выборке из 31 сорта яблони селекции Северо-Кавказского зонального НИИ садоводства и виноградарства (СКЗНИИСиВ, г. Краснодар) и Всероссийского НИИ селекции плодовых культур (ВНИИСПК, г. Орел) из коллекции генетических ресурсов СКЗНИИСиВ. Использовали SSR-маркеры, рекомендованные Европейским консорциумом Fruitbreedomics для изучения генетического разнообразия яблони: CH01f03b, CH01h01, CH01h10, CH02c06, CH02d08, CH04e05, CH05f06, CH01f02, CH02c11, Hi02c07, CH02c09 и CH03d07. По данным SSR-анализа был выявлен уровень полиморфизма от 5 до 10 аллелей на локус (наибольший - по CH02c11, наименьший - по CH01f03b) при среднем значении 7,75 аллеля на локус. Суммарно по 12 проанализированным локусам идентифицировали 93 аллеля. Все изученные сорта обладали уникальным набором аллелей. Сравнение с данными о генетическом разнообразии мирового генофонда яблони позволяет утверждать, что изученная выборка современных сортов яблони отечественной селекции характеризуется высоким уровнем полиморфизма SSR-локусов. Величины ожидаемой (H e) и наблюдаемой (H o) гетерозиготности варьировали в пределах 0,548-0,897 для H o и 0,602-0,827 для H e при среднем показателе H o = 0,786 и H e = 0,755. Значение PIC (polymorphism information content) составило от 0,571 до 0,806. При этом по 9 локусам из 12 изученных названный показатель изменялся в пределах 0,712-0,806. Результаты UPGMA-анализа согласуются с данными по генетической разнородности сортов из изученной выборки. Построение дендрограммы позволило выделить пять основных кластеров. Распределение сортов по кластерам в большинстве случаев прямо соотносится с их генеалогией. Так, сорт Свежесть, вошедший в отдельный кластер № 1, и сорта Имрус и Зимнее утро, сформировавшие кластер № 5, происходят от сортов, не представленных в родительских формах ни у одного из остальных сортов в изученной выборке. Сорта Солнышко, Строевское, Юбилей Москвы, Афродита и Старт селекции ВНИИСПК, выделенные в кластер № 3, имеют одну общую родительскую форму. Cложная структура дендрита, полученная при выполнении кластеризации по результатам SSR-анализа, может быть следствием большого числа уникальных аллелей у изучаемых генотипов, что, в свою очередь, обусловлено высоким уровнем генетического разнообразия внутри выборки. В то же время факт объединения сортов с одинаковой генеалогией в одни кластеры подтверждает значительное генетическое сходство внутри групп таких сортов. Результаты исследования дают возможность оценить уровень генетического полиморфизма в изученной выборке, кроме того, данные SSR-генотипирования могут быть использованы в дальнейшем для подтверждения генеалогии сортов и гибридов при возникновении спорных вопросов, а также при сортовой идентификации.
Яблоня, ssr-маркеры, генотипирование, полиморфизм, генетическое разнообразие
Короткий адрес: https://sciup.org/142134838
IDR: 142134838 | УДК: 634.11:575.174.015.3:577.21 | DOI: 10.15389/agrobiology.2015.1.37rus
Текст научной статьи Изучение генетического разнообразия современных сортов яблони ( Malus domestica Borkh.) отечественной селекции с использованием микросателлитных локусов
Применение технологий ДНК-маркерного анализа в работе с генетическими ресурсами культурных растений позволяет значительно расширить область исследований — от оценки генетического разнообразия и вопросов паспортизации сортов до защиты авторских прав селекционеров и определения генетической чистоты селекционного материала.
ДНК-маркеры должны обладать определенными свойствами и от-
Работа выполнялась при поддержке РФФИ (проект ¹ 13-04-02089_а). The work was partly funded by the EU seventh Framework Programme through the FruitBreedomics Project («Integrated approach for increasing breeding efficiency in fruit tree crops»; Number 255582).
вечать ряду требований, к которым, в частности, относится высокий уровень полиморфизма, кодоминантный характер наследования, оптимальная частота встречаемости в геноме, равномерное распределение по хромосомам, легкая оценка параметров маркера, высокая воспроизводимость, возможность автоматизации оценки и легкого обмена данными между лабораториями (1). Микросателлитные ДНК-маркеры, основанные на анализе простых повторяющихся повторов (SSR — simple sequence repeat), в полной мере соответствуют этим требованиям и на сегодняшний день признаются одной из наиболее эффективных ДНК-маркерных систем, используемых в селекции и генетике культурных растений, в том числе яблони (2-4).
С помощью SSR-маркеров были построены первые наиболее полные генетические карты яблони. Так, C. Maliepaard с соавт. (5), использовав мультиаллельные маркеры, в том числе SSR, на основе гибридной популяции, созданной при скрещивании сортов Прима и Фиеста, разработали одну из первых карт генома яблони с 17 группами сцепления. Идентификация новых SSR-локусов в геноме яблони позволила в дальнейшем создать более насыщенные генетические карты (6-9). Информация об идентифицированных SSR-локусах и их геномной локализации служит ценной научной базой при выполнении генетических исследований яблони — картировании генов хозяйственно ценных признаков, анализе генетической структуры коллекций генетических ресурсов, паспортизации генофонда, изучении генетического разнообразия в пределах вида Malus ½ domestica Borkh.
В настоящее время выполнен широкий спектр исследований, направленных на оценку генетического полиморфизма в коллекциях генетических ресурсов (как современных сортов, так и аборигенного генофонда). Проводились работы по выяснению вопросов, касающихся генеалогии местных сортов, формирования автохтонной генплазмы яблони, а также ее генетической взаимосвязи с мировым генофондом (10-15), ДНК-паспортизации подвоев (16, 17). При этом в ряде случаев была выявлена уникальность аборигенного генофонда. Результаты, полученные A. Gharghani с соавт. (13), показали, что аборигенные иранские сорта занимают промежуточное положение между сортами яблони европейской селекции и видом Malus orientalis Uglitz. A. Patocchi с соавт. (18) провели масштабное исследование, в котором с использованием 88 микросателлитных ДНК-мар-керов был оценен полиморфизм в общей сложности порядка 2 000 тысяч образцов из европейских коллекций генетических ресурсов (сорта, гибридные популяции). На основании этих данных были рекомендованы SSR-маркеры, наиболее перспективные для анализа геномного полиморфизма яблони (18).
При изучении генетического разнообразия крупных генетических коллекций яблони, включающих, помимо сортов, дикие формы и межвидовые гибриды, SSR-маркеры также позволяют объективно оценивать филогенетические взаимосвязи на внутри- и межвидовом уровне (19, 20).
Анализ полиморфизма SSR-локусов при изучении генетического разнообразия позволяет составлять ДНК-паспорта генотипов. SSR-фин-герпринты могут эффективно использоваться для идентификации образцов коллекций, выявления дублей и др.
Учитывая высокую актуальность изучения разнообразия в генофонде яблони и перспективность использования микросателлитных маркеров при решении подобных задач, мы выполнили SSR-генотипирование сортов современной селекции из коллекции генетических ресурсов Севе- ро-Кавказского зонального НИИ садоводства и виноградарства (СКЗНИИ-СиВ) и оценили генетический полиморфизм в современном отечественном генофонде яблони.
Методика . Материалом для исследований послужила выборка сортов яблони современной селекции (оригинаторы: СКЗНИИСиВ и Всероссийский НИИ селекции плодовых культур — ВНИИСПК, г. Орел), сохраняемых в коллекции генетических ресурсов СКЗНИИСиВ ( n = 31).
В работе использовали 12 SSR-маркеров: CH01f03b, CH01h01, CH01h10, CH02c06, CH02d08, CH04e05, CH05f06, CH01f02, CH02c11, Hi02c07, CH02c09 и CH03d07. Перечисленные SSR-маркеры рекомендованы Европейским консорциумом Fruitbreedomics для изучения генетического разнообразия яблони (последовательность праймеров доступна в базе данных .
Для экстракции ДНК использовали CTAB-метод (21). ПЦР проводили по стандартным методикам с предварительной оптимизацией параметров. ПЦР-смесь (конечный объем 25 мкл) включала 50-70 мкг ДНК, 0,05 мM dNTPs, по 0,3 мкM каждого праймера, 2,5 мкл 10½ реакционного буфера, 2,5 мM MgCl2, 1 ед. Taq-полимеразы. Режим амплификации: начальная денатурации — 5 мин при 95 °С; денатурация — 10 с при 95 °С, отжиг праймеров — 30 с при 58 °С, элонгация — 30 с при 72 °С (30 циклов); заключительная элонгация — 3 мин при 72 °С. Реакцию проводили в амплификаторе Eppendorf Mastercycler gradient (Германия), фрагментный анализ SSR-локусов — на автоматическом генетическом анализаторе ABI prism 3130 (США). Результаты фрагментного анализа обрабатывали в программе GeneMapper v. 4.1.
При оценке данных микросателлитного анализа матрицу генетических дистанций строили с использованием коэффициентов (индексов) подобия по M. Nei и W. Li (22). Кластерный анализ выполняли методом попарного невзвешенного кластирования с арифметическим усреднением (UPGMA) с использованием FreeTree Application v. 0.9.1.50 (ZDAT v.o.s.). Для графического построения дендрограммы применили программу TreeView (Win32) v. 1.6.6. Фактическую (Ho) и ожидаемую (He) гетерозиготность рассчитывали с помощью программы GenAlEx v. 6.3. Значение PIC (polymorphism information content) определяли с помощью программы PIC-calculator в режиме online .
Результаты . При апробации отобранных микросателлитных маркеров использовали электрофорез в 8 % неденатурирующем полиакриламидном геле. После оптимизации параметров ПЦР микросателлитные ДНК-маркеры были сгруппированы в мультиплексные наборы (4 маркера в наборе) для одновременного анализа по нескольким локусам. При этом размеры амплифицируемых фрагментов не перекрывались, а каждый из маркеров содержал специфическую флуоресцентную метку (FAM, TAMRA, R6G, ROX).
При выполнении фрагментного анализа были получены четкие воспроизводимые результаты. В качестве примера на рисунке 1 (визуализация результатов в рабочем окне программы GeneMapper v. 4.1) приведены данные для сорта Красный янтарь. На электрофореграмме два пика присутствовали у маркеров, по которым выявлялись два продукта, то есть соответствующие локусы гетерозиготны. В обсуждаемом случае это маркеры CH01h01, CH01f03b и CH02c06. Один пик по локусу CH01h10 на электрофореграмме свидетельствовал об его гомозиготности.
Из использованных в работе 12 маркеров наибольший полиморфизм выявили по локусу CH02c11 (10 аллелей), наименьший — по локу- су CH01f03b (5 аллелей). Суммарно по 12 изученным локусам обнаружили 93 аллеля. Все изученные сорта обладали уникальным набором аллелей (полученные SSR-фингерпринты представлены в базе данных, проходящей в настоящее время регистрацию в качестве объекта интеллектуальной собственности).
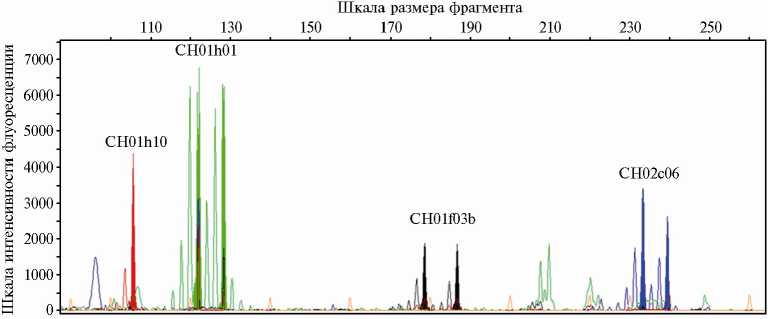
Рис. 1. Фрагментный электрофоретический анализ ДНК у сорта яблони Красный янтарь по мультиплексному набору, включающему SSR-маркеры CH01h10, CH01h01, CH01f03b и CH02c06 (коллекция Северо-Кавказского зонального НИИ садоводства и виноградарства, г. Краснодар)
Характеристика полиморфизма микросателлитных SSR-маркеров, использованных при оценке генетического разнообразия современных сортов яблони ( Malus ½ domestica Borkh.) отечественной селекции из коллекции ( n = 31, коллекция СевероКавказского зонального НИИ садоводства и виноградарства, г. Краснодар)
|
Маркер |
Диапазон размеров амплифициро-ванных фрагментов, п.н. |
Число выявленных аллелей |
PIC |
H o |
H e |
|
CH01f03b |
141-182 |
5 |
0,690 |
0,710 |
0,736 |
|
CH01h01 |
118-137 |
6 |
0,712 |
0,742 |
0,754 |
|
CH01h10 |
94-120 |
7 |
0,572 |
0,548 |
0,602 |
|
CH02c06 |
204-256 |
9 |
0,806 |
0,897 |
0,827 |
|
CH02d08 |
215-258 |
9 |
0,745 |
0,800 |
0,776 |
|
CH04e05 |
178-230 |
9 |
0,772 |
0,867 |
0,798 |
|
CH05f06 |
171-189 |
7 |
0,678 |
0,871 |
0,723 |
|
CH01f02 |
173-210 |
8 |
0,726 |
0,839 |
0,763 |
|
CH02c11 |
219-241 |
10 |
0,727 |
0,806 |
0,752 |
|
Hi02c07 |
104-166 |
9 |
0,752 |
0,897 |
0,781 |
|
CH02c09 |
235-259 |
7 |
0,737 |
0,774 |
0,772 |
|
CH03d07 |
188-228 |
7 |
0,747 |
0,679 |
0,776 |
|
П р и м еч ани е. PIC — polymorphism information content (информативность маркера), H |
o — наблюдае- |
||||
|
мая гетерозиготность, |
He — ожидаемая гетерозиготность. |
||||
Сравнение общего числа выявленных аллелей, полученного по результатам нашего исследования, с результатами анализа генетического разнообразия мирового генофонда яблони позволяет утверждать, что полиморфизм SSR-локусов в изученной выборке сортов яблони отечественной селекции весьма высок. В работе S. Pereira-Lorenzo с соавт. (14) при анализе 66 испанских сортов яблони по 10 SSR-локусам выявили суммарно 122 аллеля. При этом для группы из 27 аборигенных сортов этот показатель равнялся 75 (14). В финском и шведском генофонде яблони (суммарно 101 генотип) по 9 локусам обнаружили 105 аллелей. При этом показатели ожидаемой (He) и наблюдаемой (Ho) гетерозиготности варьировали в диапазоне соответственно 0,31-0,88 и 0,41-0,88 (23), в то время как в нашем исследовании они изменялись в пределах 0,548-0,897 для Ho и 0,6020,827 для He (табл.). Средняя величина показателя Ho в нашем исследовании составила 0,786, He — 0,755, что превысило показатели, полученные при анализе скандинавской генплазмы яблони (He = 0,72; Ho = 0,74), где 40
среднее число аллелей на локус составляло 11,6 (23). По результатам нашего исследования этот показатель в выборке из 31 сорта отечественной селекции равнялся 7,75. A. Pina с соавт. (24), изучая генетическое разнообразие аборигенного генофонда яблони на северо-востоке Испании (область Арагон), при анализе 20 SSR-локусов в выборке из 130 генотипов выявили средний показатель 12,4 аллеля. В своем исследовании авторы сопоставили результаты с данными по полиморфизму SSR-локусов у 21 сорта мировой селекции из разных регионов мира и определили у последних среднее число аллеей на локус, равное 8,2 (24).
Полученные нами значения PIC, варьирующие в диапазоне от 0,572 до 0,806 (см. табл.), свидетельствуют о высоком уровне информативности большинства изученных SSR-маркеров. По SSR-локусу CH01h10, у которого было выявлено 7 аллелей, наименьшая среди использованных маркеров величина PIC (0,572) в значительной степени обусловлена преобладанием одного из аллелей (с размером 100 п.н.) над другими (его обнаружили у 60 % изученных генотипов), а также наличием двух редких аллелей (118 и 120 п.н.), каждый из которых был идентифицирован только у одного образца.
На основании данных SSR-генотипирования была выполнена оценка степени генетического сходства изученных сортов с применением метода попарного невзвешенного кластирования с арифметическим усреднением (UPGMA) (рис. 2).
По степени генетического сходства выделились пять основных кластеров: ¹ 1 — сорт Свежесть; ¹ 2 — сорта Нимфа, Талида, Персиковое, Маяк станичный, Ноктюрн, Любава, Василиса, Кубань, Кармен, Болотов-ское, Юнона, КВ Зарево, Золотая корона, Дин Арт, Апорт АСС, Зори Кубани, Талисман, Амулет, Красный янтарь, Фортуна, Рассвет; ¹ 3 — Солнышко, Строевское, Юбилей Москвы, Афродита, Старт; ¹ 4 – Кубанское румяное, Золотое летнее; ¹ 5 – Имрус, Зимнее утро. При этом в кластер ¹ 2 вошли четыре субкластера (2a-2d) и сорта Талида и Нимфа.
Результаты UPGMA-анализа в целом отражают генетическую разнородность сортов из изученной выборки. Это выражается, с одной стороны, в наличии кластеров, объединяющих несколько сортов на одном уровне или включающих всего два либо даже один генотип (сорт Свежесть), с другой — в формировании кластеров со сложной структурой (например, ¹ 2).
Кластеризация сортов во многом согласуется с их происхождением. К примеру, сорт Свежесть [Антоновка краснобочка ½ PR12T67 (Уэлси ½ F2 M . floribunda )], выделенный в кластер ¹ 1, и сорта Имрус (Антоновка обыкновенная ½ OR18T13) и Зимнее утро (Либерти ½ Скарлет Стайма-ред), образующие кластер ¹ 5, имеют происхождение от сортов, не представленных в родительских формах ни у одного из остальных сортов изученной выборки.
Достоверность данных, полученных при построении дендрограммы, также подтверждается объединением в один кластер сортов Афродита, Старт, Солнышко, Юбилей Москвы и Строевское, которые имеют общую родительскую форму 814 — [F2 ( M . floribunda 821 ½ M . domestica) ] ½ Голден Делишес (25, 26), что служит наиболее вероятной причиной их обособленности от большинства изученных генотипов по результатам кластеризации. Сорта Талисман, Амулет, Красный янтарь, Фортуна, Рассвет (субкластер 2d) происходят из гибридной комбинации Редфри ½ Папи-ровка тетраплоидная.
Cложная структура дендрита, получаемая при выполнении класте- ризации, по результатам SSR-анализа может быть следствием наличия большого числа уникальных и редких аллелей у изучаемых генотипов, что, в свою очередь, обусловлено высоким уровнем генетического разнообразия внутри выборки (27).
Свежесть
Нимфа
Талида Персиковое
Маяк станичный
Ноктюрн
Любава
Василиса
Кубань
Кармен Болотовское
Юнона
КВ Зарево Золотая корона Дин Арт Апорт АСС Зори Кубани Талисман
Амулет
Красный янтарь
Фортуна
Рассвет
Солнышко
Строевское
Юбилей Москвы
Афродита
Старт
Кубанское румяное
Золотое летнее
Имрус
Зимнее утро

№2
№3
№4
№5
Рис. 2. Дендрограмма генетического сходства между изученными современными сортами яблони ( Malus ½ domestica Borkh.) отечественной селекции, построенная по методу UPGMA: ¹¹ 1-5 — кластеры, 2a, 2b, 2c, 2d — субкластеры (коллекция Северо-Кавказского зонального НИИ садоводства и виноградарства, г. Краснодар).
Таким образом, результаты проведенного исследования подтверждают эффективность SSR-маркеров для оценки генетического разнообразия генофонда яблони. На основании данных, полученных при анализе полиморфизма SSR-локусов у 31 сорта яблони современной отечественной селекции, можно говорить о высоком уровне полиморфизма в изу- ченной выборке генотипов. В то же время факт объединения сортов с одинаковой генеалогией в одни кластеры подтверждает значительное генетическое сходство внутри групп таких сортов. В этой связи мы рекомендуем не использовать сорта, вошедшие в один кластер, в качестве родительских пар при гибридизации, если необходимо получить наибольшую генетическую изменчивость в гибридных популяциях. Данные выполненного SSR-генотипирования могут учитываться при подтверждении генеалогии сортов и гибридов, в случае если одна или обе их родительские формы входят в изученную выборку генотипов.
Список литературы Изучение генетического разнообразия современных сортов яблони ( Malus domestica Borkh.) отечественной селекции с использованием микросателлитных локусов
- Хавкин Э.Е. Молекулярные маркеры в растениеводстве. Сельскохозяйственная биология, 1997, 5: 3-19.
- Diwan N., Cregan P.B. Automated sizing of fluorescent-labeled simple sequence repeat (SSR) markers to assay genetic variation in soybean. Theor. Appl. Genet., 1997, 95: 723-733 ( ) DOI: 10.1007/s001220050618
- Thomas M.R., Scott N.S. Microsatellite repeats in grapevine reveal DNA polymorphism when analysed as sequence-tagged sites (STSs). Theor. Appl. Genet., 1993, 86: 985-990 ( ) DOI: 10.1007/BF00211051
- Morgante M., Oliveri A.M. PCR-amplified microsatellite markers in plant genetics. Plant J., 1993, 3: 175-182 ( ) DOI: 10.1046/j.1365-313X.1993.t01-9-00999.x
- Maliepaard C., Alston H.F., Van Arkel G. Aligning male and female linkage maps of apple (Malus pumila Mill.) using multi-allelic markers. Theor. Appl. Genet., 1998, 97: 60-73 ( ) DOI: 10.1007/s001220050867
- Liebhard R., Gianffranceschi L., Koller B., Ryder C.D. Development and characterization of 140 new microsatellites in apple (Malus domestica Borkh.). Molecular Breeding, 2002, 10: 217-241 ( ) DOI: 10.1023/A:1020525906332
- Liebhard R., Koller B., Gianffranceschi L., Gessler C. Creating a saturated reference map for the apple (Malus domestica Borkh.) genome. Theor. Appl. Genet., 2003, 106: 1497-1508 ( ) DOI: 10.1007/s00122-003-1209-0
- Silfverberg-Dilworth E. Microsatellite markers spanning the apple (Malus * domestica Borkh.) genome. Tree Genetics &Genomes, 2006, 2: 202-224 ( ) DOI: 10.1007/s11295-006-0045-1
- Fernandez-Fernandez F., Eans K.M., Clarke J.B., Govan C.L. Development of an STS map of an interspecific progeny of Malus. Tree Genetic & Genomic, 2008, 4: 469-479 ( ) DOI: 10.1007/s11295-007-0124-y
- Muzher B.M., Younis R.A.A., El-Halabi O., Ismail O.M. Genetic identification of some Syrian local apple (Malus sp.) cultivars using molecular markers. Research Journal of Agriculture and Biological Sciences, 2007, 3(6): 704-713.
- Pereira-Lorenzo S., Ramos-Cabrer A.M., Diaz-Hernandez M.B. Evaluation of genetic identity and variation of local apple cultivars (Malus domestica Borkh.) from Spain using microsatellite markers. Genet. Resour. Crop. Evol., 2007, 54: 405-420 ( ) DOI: 10.1007/s10722-006-0003-7
- Fua G., Simon S., Pojskic N., Kurtovic M., Peji I. Genetic assessment of apple germplasm in Bosnia and Herzegovina using microsatellite and morphologic markers. Scientia Horticulturae, 2010, 126: 164-171 ( ) DOI: 10.1016/j.scienta.2010.07.002
- Gharghani A., Zamani Z., Talaie A., Oraguzie N.C. Genetic identity and relationships of Iranian apples (Malus * domestica Borkh.) cultivars and landraces, wild apple species and representative old apple cultivars based on SSR markers. Genet. Resour. Crop. Evol., 2009, 56: 829-842 ( ) DOI: 10.1007/s10722-008-9404-0
- Pereira-Lorenzo S., Ramos-Cabrer A.M., Gonzalez-Diaz A.J., Diaz-Hernandez M.B. Genetic assessment of local apple cultivars from La Palma, Spain, using simple sequence repeats (SSRs). Scientia Horticulturae, 2008, 117: 160-166 ( ) DOI: 10.1016/j.scienta.2008.03.033
- Савельев Н.И., Юшков А.Н., Шамшин И.Н. Применение метода молекулярных маркеров для изучения генетического разнообразия плодовых культур. Вестник Мичуринского государственного аграрного университета, 2011, 2(1): 8-12.
- Супрун И.И., Алексеев Я.И., Малюченко О.П., Бабаков А.В. Генотипирование подвоев яблони отечественной селекции с использованием мультиплексного STR-анализа. Садоводство и виноградарство, 2012, 4: 20-23.
- Oraguzie N.C., Yamamoto T., Soejima J., Suzuki T. DNA fingerprinting of apple (Malus spp.) rootstocks using Simple Sequence Repeats. Plant Breed., 2005, 124: 197-202 ( ) DOI: 10.1111/j.1439-0523.2004.01064.x
- Patocchi A., Fernandez-Fernandez F., Evans K., Gobbin D. Development and test of 21-multiplex PCRs composed of SSRs spanning most of the apple genome. Tree Genetics &Genomes, 2009, 5: 211-223 ( ) DOI: 10.1007/s11295-008-0176-7
- Richards C.M., Volk G.M., Reilley A.A., Henk A.D. Genetic diversity and population structure in Malus sieversii, a wild progenitor species of domesticated apple. Tree Genetics & Genomes, 2009, 5: 339-347 ( ) DOI: 10.1007/s11295-008-0190-9
- Gross B.L., Henk A.D., Forsline P.L., Richards C.M., Volk G.M. Identification of interspecific hybrids among domesticated apple and wild relatives. Tree Genetics & Genomes, 2012, 8(6): 1223-1235 ( ) DOI: 10.1007/s11295-012-0509-4
- Murray M.G., Thompson W.F. Rapid isolation of high molecular weight plant DNA. Nucl. Acids Res., 1980, 10: 4321-4325 ( ) DOI: 10.1093/nar/8.19.4321
- Nei M., Li W.-H. Mathematical model for studying genetic variation in terms of restriction endonucleases. PNAS USA, 1979, 76: 5269-5273 ( ) DOI: 10.1073/pnas.76.10.5269
- Garkava-Gustavsson L., Mujajub C., Sehic J., Zborowskaya A., Gunter M.B. Genetic diversity in Swedish and Finnish heirloom apple cultivars revealed with SSR markers. Scientia Horticulturae, 2013, 162: 43-48 ( ) DOI: 10.1016/j.scienta.2013.07.040
- Pina A., Urrestarazu J., Pilar E. Analysis of the genetic diversity of local apple cultivars from mountainous areas from Aragon (Northeastern Spain). Scientia Horticulturae, 2014, 174: 1-9 ( ) DOI: 10.1016/j.scienta.2014.04.037
- Седов Е.Н., Жданов В.В. Устойчивость яблони к парше. Орел, 1983.
- Седов Е.Н., Седышева Г.А., Жданов В.В., Ульяновская Е.В., Серова З.М. Результаты селекции иммунных к парше триплоидных сортов яблони. Вестник ВОГиС, 2009, 13(4): 793-785.
- Урбанович О.Ю., Козловская З.А., Хацкевич А.А., Картель Н.А. Анализ полиморфизма SSR-локусов видов Malus. Известия Национальной академии наук Беларуси, 2010, 1: 12-17.


