Эффект SNP G.73467314G>A (SSC7) на признаки продуктивности и состояние конечностей свиней
Автор: Гетманцева Л.В., Костюнина О.В., Бакоев С.Ю., Форнара М.С., Карпушкина Т.В., Бакоев Н.Ф., Свеженцева Н.А., Колосова М.А.
Журнал: Вестник аграрной науки @vestnikogau
Рубрика: Сельскохозяйственные науки
Статья в выпуске: 4 (73), 2018 года.
Бесплатный доступ
Увеличение объёмов производства высококачественной конкурентоспособной и экологически безопасной продукции животноводства приобретает стратегическое значение для обеспечения продовольственной безопасности РФ. Повышенный интерес к улучшению продуктивных качеств свиней оказал значительное влияние на развитие молекулярно-генетических исследований в данном направлении. Целью данной работы было разработать методику для тестирования SNP g.73467314G>A и оценить его эффект на откормочные и мясные качества, а также на состояние конечностей у свиней крупной белой породы и ландрас. Материалом для исследования служили пробы биологического материала (ушные выщипы) хряков крупной белой породы (n=144) и породы ландрас (n=120), принадлежащие племенному предприятию ООО «Знаменский СГЦ». В результате разработанной тест-системы методом пиросеквенирования были определены три генотипа АА, AG и GG для варианта g.73467314G>A (SSC7) и установлена статистически значимая ассоциация (Р=0.003) между генотипами и состоянием задних конечностей у свиней породы ландрас. Наилучшие оценки задних конечностей имели свиньи, обладающие генотипом АА. Отсутствие связи между генотипом АА и признаками откормочной и мясной продуктивности позволит проводить отбор с учетом этого генотипа, не затрагивая ростовые и мясные показатели свиней. В исследуемой выборке свиней крупной белой породы частота минорного аллеля А составила 0.05, что свидетельствует об очень низком уровне полиморфизма. Таким образом, полученные результаты в нашей работе позволяют предположить, что вариант g.73467314G>A можно рассматривать в качестве генетического маркера состояния конечностей у свиней породы ландрас. Разработанная методика тестирования позволит провести дальнейшие исследования на большем поголовье свиней различных пород и кроссов, разводимых в племенных хозяйствах РФ.
Свиньи, откормочная и мясная продуктивность, конечности, тест-система
Короткий адрес: https://sciup.org/147230617
IDR: 147230617 | УДК: 619:575.22:[636.4.03+619:611.71:636.4] | DOI: 10.15217/issn2587-666X.2018.4.65
Текст научной статьи Эффект SNP G.73467314G>A (SSC7) на признаки продуктивности и состояние конечностей свиней
Вве^ение. В современных условиях эффективное развитие свиноводства основано на интенсивных технологиях, позволяющих рационально использовать производственные мощности и значительно повысить продуктивность ^ивотных. Повышенный интерес к ростовым качествам свиней повлиял на развитие молекулярно-генетических исследований в данном направлении [1-3]. На сегодняшний день в базе данных Pig QTLdb (Pig Quantitative Trait Locus Database) идентифицировано 584 локуса для среднесуточного прироста свиней. Наибольшее количество локусов определено на хромосомах SSC1 (99 QTL), SSC4 (71 QTL) и SSC7 (57QTL).
В последнее время так^е мо^но отметить рост исследований, направленных на поиск локусов, ассоциированных с репродуктивными показателями [4-6]. По результатам полногеномных исследований на начало 2018 г. в базе данных «PigQTLdb» зарегистрировано 204 локуса для общего количества поросят при ро^дении, из них 157 локусов обнару^ены у свиней китайской породы эрхуалян и 47 у свиней европейских пород. В работе He L.C. с соавторами [7] у свиней породы эрхуалян значимые SNPs, ассоциированные с общим количеством поросят при ро^дении, определены на хромосомах SSC2 (rs81367039), SSC7 (rs80891106), SSC8 (rs81399474), SSC12 (rs81434499), SSC14 (rs80938898). Интересно отметить, что SNP rs80891106 (g.73467314G>A), располо^енный на SSC7 в позиции 73,5 Мbp был обнару^ен в области хромосомы, где ранее не были определены гены-кандидаты или QTL для плодовитости свиней. Сопоставив локализацию SNP rs80891106 с данными по раннее аннотированным QTL на SSC7, мы определили, что он располо^ен в локусах, идентифицированных для среднесуточного прироста (QTL, регион 0,0120,9 Mbp) [8], толщины шпика (50,3-120,7 и 55,9-101,2) [9, 10] и массы окорока (30,8-94,4) [11].
Цель иссле^ований – разработать методику для тестирования SNP g.73467314G>A и оценить его эффект на откормочные и мясные качества, а так^е на состояние конечностей у свиней крупной белой породы и ландрас, разводимых в племенном хозяйстве РФ.
Услови^, материалы и мето^ы . Исследования были выполнены в лаборатории молекулярных основ селекции Федерального государственного бюд^етного научного учре^дения «Федеральный научный центр ^ивотноводства – ВИЖ имени академика Л. К. Эрнста» в рамках выполнения задания Федерального агентства научных организаций (Ф^НО) №^^^^-^18-118021590138-1. Материалом для исследования слу^или пробы биологического материала (ушные выщипы) хряков крупной белой породы (n=144) и породы ландрас (n=120), принадле^ащие племенному предприятию ООО «Знаменский СГЦ». Все исследуемые ^ивотные были одного года ро^дения и имели одинаковые условия содер^ания и кормления. При дости^ении 100 кг свиней оценивали по следующим признакам: среднесуточный прирост (г), толщина шпика (мм), глубина мышечного глаза (мм), оценка передних и задних конечностей. Экстерьерную оценку ^ивотных проводили на ровной площадке, оценивали общий вид, конституцию, пропорциональность телосло^ения, и отдельные стати. Передние и задние конечности у свиней дол^ны быть прямыми, отвесно и широко поставленными, обеспечивать хорошую подви^ность ^ивотных. Узкая, иксообразная, слоновая или саблистая постановка ног считается большим пороком.
Для проведения молекулярно-генетических исследований геномную ДНК выделяли при помощи набора «ДНК-экстран-2» (НПО «Синтол», Россия) согласно рекомендациям производителя. Специфические олигонуклеотидные праймеры для идентификации SNP g.73467314G>A методом пиросеквенирования подбирали с помощью программ Primer-BLAST (NCBI). По результатам молекулярно-генетических исследований рассчитывали частоту аллелей и генотипов. Для расчета эффекта SNP на признаки продуктивности использовали однофакторный дисперсионный анализ. В качестве фактора выступал SNP g.73467314G>A, число градаций соответствовало числу генотипов. Согласно данному анализу, эффект существует, если F-рассч.>F-табл. при соответствующей степени достоверности.
Результаты и обсу^^ение. Разработанная тест-система предусматривает амплификацию фрагмента длиной 332 п.н., содер^ащего SNP g.73467314G>A с последующим пиросеквенированием. Полученные результаты показали, что в исследуемой выборке свиней крупной белой породы частота минорного аллеля ^ составила 0,05, что свидетельствует об очень низком уровне полиморфизма, практически об его отсутствии (табл. 1). Более 90% свиней крупной белой породы имели генотип GG. У свиней породы ландрас наоборот, минорным определен аллель G, но с достаточно высокой частотой (0,34), что проявилось в наличии трех генотипов ^^, AG и GG с частотами 45,0; 41,7 и 13,3% соответственно.
Tаблица 1 – Частоты аллелей и генотипов SNP g.73467314G>A у свиней крупной белой породы и ландрас
|
Порода |
Частота аллелей |
Частота генотипов, % |
|||
|
A |
G |
AA |
AG |
GG |
|
|
Крупная белая |
0,05 |
0,95 |
0,00 |
9,70 |
90,30 |
|
Ландрас |
0,66 |
0,34 |
45,00 |
41,70 |
13,30 |
По результатам однофакторного дисперсионного анализа статистически значимый эффект варианта g.73467314G>A установили только на состояние задних конечностей у свиней породы ландрас, уровень достоверности которого составил P=0,003 (табл. 2). Эффект генотипов на другие исследуемые признаки при уровнях достоверности Р≤0,05 в выборках свиней крупной белой породы и ландрас не установлен.
В изучаемой выборке свиньи породы ланрас, имеющие генотип ^^, обладали лучшей оценкой задних конечностей, по сравнению с ^ивотными с генотипом GG. Согласно полученным данным, мо^но предполо^ить, что поло^ительный эффект генотипа ^^ проявляется только в породе ландрас. Следует отметить, у свиней крупной белой породы генотип ^^ не был определен, а генотип AG имел очень низкую частоту, что возмо^но связано с особенностями генетических структур свиней крупной белой породы и ландрас.
Tаблица 2 – Эффект SNP g.73467314G>A на признаки продуктивности и состояние конечностей свиней
|
Признаки |
Генотипы |
P |
||
|
AA | |
AG 1 |
GG |
||
|
Крупная белая порода (n=144) |
||||
|
Среднесуточный прирост |
- |
870,42±18,69 |
922,29±10,38 |
0,115 |
|
Tолщина шпика |
- |
12,07 ± 0,59 |
10,66 ± 0,30 |
0,093 |
|
Глубина мышечного глазка |
- |
66,28 ± 2,87 |
62,45 ± 0,59 |
0,071 |
|
Оценка передних конечностей |
- |
3,71 ± 0,18 |
3,40 ± 0,06 |
0,110 |
|
Оценка задних конечностей |
- |
3,28 ± 0,18 |
3,40 ± 0,06 |
0,531 |
|
Ландрас (n= |
120) |
|||
|
Среднесуточный прирост |
902,62±15,32 |
904,36±17,37 |
832,00±29,61 |
0,086 |
|
Tолщина шпика |
12,86 ± 0,51 |
12,77 ± 0,45 |
13,14 ± 0,91 |
0,938 |
|
Глубина мышечного глазка |
59,84 ± 1,11 |
61,32 ± 1,34 |
59,56 ±0,56 |
0,549 |
|
Оценка передних конечностей |
3,44 ± 0,09 |
3,26 ± 0,09 |
3,14 ± 0,14 |
0,169 |
|
Оценка задних конечностей |
3,48 ± 0,09 |
3,17 ± 0,08 |
2,85 ± 0,14 |
0,003 |
Предполо^ить биологическую функцию исследуемого SNP g.73467314G>A (SSC7) довольно сло^но, т.к. он располо^ен в некодирующей области ДНК. По данным Pig QTLdb в непосредственной близости от него находятся гены RF00001(73234200..73234325) и ENSSSCG00000032058 (73861910..73911853), кодирующие рибосомную РНК и длинную некодирущую РНК соответственно.
На основании наших результатов, показывающих эффект SNP g.73467314G>A на состояние задних конечностей, мы проанализировали располо^ение локусов на SSC7, аннотированных раннее при изучении ассоциаций с постановкой конечностей и их дефектами. По данным базы Pig QTLdb на SSC7 было идентифицировано 5 локусов, ассоциированных с передними конечностями (FRLEGC) [12-14], 6 локусов с задними конечностями (HLEGC) [13-16], один локус с остеохондрозом (OSTEO) [15] и один с вро^денным недоразвитием мышц ног (SPLEG) (рис. 1). Ни один из этих локусов не находится в непосредственной близости (в районе 1 Mbp) от изучаемого нами SNP g.73467314G>A. В данном контексте следует подчеркнуть, что нами так^е не был установлен эффект SNP g.73467314G>A на толщину шпика и среднесуточный прирост, несмотря на располо^ение исследуемого полиморфизма в районе локусов, ассоциированных с этими признаками. Вероятно, это связано с особенностями генетической структуры и неравновесием по сцеплению у исследуемых популяций.
QTL Mapper и.2.019
о -rSW2564
-SW1873
-SW1354
■SWR1078
•SW1369
■SW472
■S0102
80 - SWR18O6
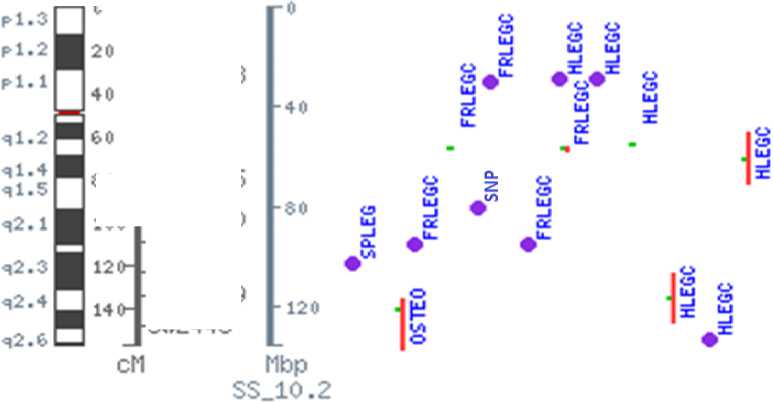
Рисунок 1 – Локусы, ассоциированные с состоянием и развитием конечностей свиней, располо^енные на SSC7 (по данным QTLdbPig): FRLEGC – постановка передних конечностей; HLEGC – постановка задних конечностей;
OSTEO – остеохондроз; SPLEG – вро^денное недоразвитие мышц;
SNP – вариант g.73467314G>A
ioo4TPS271O
-SW1083
-SW581
•TPS2699
•SW2446
Tаким образом, полученные результаты в нашей работе позволяют предполо^ить, что вариант g.73467314G>A мо^но рассматривать в качестве генетического маркера состояния конечностей у свиней породы ландрас. Разработанная методика тестирования позволит провести дальнейшие исследования на большем поголовье свиней различных пород и кроссов, разводимых в племенных хозяйствах РФ.
Выводы. На основе разработанной тест-системы методом пиросеквенирования были определены три генотипа ^^, AG и GG для варианта g.73467314G>A (SSC7) и установлена статистически значимая ассоциация (Р=0,003) ме^ду генотипами и состоянием задних конечностей у свиней породы ландрас. Наилучшие оценки задних конечностей имели свиньи, обладающие генотипом ^^. Отсутствие связи ме^ду генотипом ^^ и признаками откормочной и мясной продуктивности позволит проводить отбор с учетом этого генотипа, не затрагивая ростовые и мясные показатели свиней.
БИБЛИОГРAФИЯ
-
1. Genome-wide detection of genetic markers associated with growth and fatness in four pig populations using four approaches / Y. Guo, Y. Huang, L. Hou et al. // Genetics Selection Evolution. 2017. № 49. Р. 21.
-
2. Genome-wide association analyses reveal significant loci and strong candidate genes for growth and fatness traits in two pig populations / R.M. Qiao, J. Gao, Z.Y. Zhang et al. // Genet Sel Evol. 2015. № 47. Р. 17.
-
3. A genome-wide association study of social genetic effects in Landrace pigs / J.K. Hong, Y.D. Jeong, E.S. Cho et al. // Asian-Australas J Anim Sci. 2018. № 31(6). Р. 784-790.
-
4. Genome-wide association analysis reveals genomic regions on Chromosome 13 affecting litter size and candidate genes for uterine horn length in Erhualian pigs / X. Ma, P.H. Li, M.X. Zhu et al. // Animal. 2018. № 14. Р. 1-9.
-
5. Genome ‐ wide association study for reproductive traits in a Large White pig population / Y. Wang, X. Ding, Z. Tan et al. // Animal Genetics. 2018. № 49(2). Р. 127-131.
-
6. Single step genome-wide association studies based on genotyping by sequence data reveals novel loci for the litter traits of domestic pigs / P. Wu, Q. Yang, K. Wang et al. // Genomics. 2018. № 110(3). Р. 171-179.
-
7. Identification of new single nucleotide polymorphisms affecting total number born and candidate genes related to ovulation rate in Chinese Erhualian pigs / L.C. He, P.H. Li, X. Ma et al. // Animal Genetics. 2016. № 48. Р. 48-54.
-
8. Quantitative trait loci variation for growth and obesity between and within lines of pigs (Sus scrofa) / Y. Nagamine, C.S. Haley, A. Sewalem, P.M. Visscher // Genetics. 2003. № 164 (2). Р. 629-635.
-
9. A molecular genome scan analysis to identify chromosomal regions influencing geconomic traits in the pig. I. Growth and body composition / M. Malek, J.C. Dekkers, H.K. Lee et al. // Mammalian genome. 2001. № 12(8). Р. 630-636.
-
10. A genome scan reveals QTL for growth, fatness, leanness and meat quality in a Duroc-Pietrain resource population / G. Liu, D.G.J. Jennen, E. Tholen et al. // Animal genetics. 2007. № 38 (3). Р. 241-252.
-
11. Detection of quantitative trait loci for carcass composition traits in pigs / D. Milan, J.P. Bidanel, N. Iannuccelli et al. // Sel Evol. 2002. № 34(6). Р. 705-728.
-
12. Genome-wide association study for conformation traits in three Danish pig breeds / T.H. Le, O.F. Christensen, B. Nielsen, G. Sahana. Genet Sel Evol. 2017. № 49. Р. 12.
-
13. A whole genome scan for quantitative trait loci for leg weakness and its related traits in a large F2 intercross population between White Duroc and Erhualian / Y.M. Guo, H.S. Ai, J. Ren et al. // Anim Sci. 2009. № 87 (5). Р. 1569-1575.
-
14. Genome-Wide Association Study Identifies Loci for Body Composition and Structural Soundness Traits in Pigs / B. Fan, S.K. Onteru, Z.-Q. Du et al. // PLoS ONE. 2011. № 6(2). Р. e14726.
-
15. Detection of quantitative trait loci for locomotion and osteochondrosis-related traits in Large White x Meishan pigs / G.J. Lee, A.L. Archibald, G.B. Garth et al. // Anim. Sci. 2003. № 76. Р. 155-165.
-
16. Identification of genetic markers associated with fatness and leg weakness traits in the pig / B. Fan, S.K. Onteru, M.T. Nikkila et al. // Animal genetics. 2009. № 40(6). Р. 967-970.
-
17. Genome-wide association study identifies candidate genes for piglet splay leg syndrome in different populations / X. Hao, G. Plastow, C. Zhang et al. // BMC genetics. 2017. № 18(1). Р. 64.
Список литературы Эффект SNP G.73467314G>A (SSC7) на признаки продуктивности и состояние конечностей свиней
- Genome-wide detection of genetic markers associated with growth and fatness in four pig populations using four approaches / Y. Guo, Y. Huang, L. Hou et al. // Genetics Selection Evolution. 2017. № 49. Р. 21.
- Genome-wide association analyses reveal significant loci and strong candidate genes for growth and fatness traits in two pig populations / R.M. Qiao, J. Gao, Z.Y. Zhang et al. // Genet Sel Evol. 2015. № 47. Р. 17.
- A genome-wide association study of social genetic effects in Landrace pigs / J.K. Hong, Y.D. Jeong, E.S. Cho et al. // Asian-Australas J Anim Sci. 2018. № 31(6). Р. 784-790.
- Genome-wide association analysis reveals genomic regions on Chromosome 13 affecting litter size and candidate genes for uterine horn length in Erhualian pigs / X. Ma, P.H. Li, M.X. Zhu et al. // Animal. 2018. № 14. Р. 1-9.
- Genome-wide association study for reproductive traits in a Large White pig population / Y. Wang, X. Ding, Z. Tan et al. // Animal Genetics. 2018. № 49(2). Р. 127-131.


