Эффлюкс механизм и гены резистентности к тетрациклину у бактерий
Автор: Чижкова А.П.
Журнал: Форум молодых ученых @forum-nauka
Статья в выпуске: 12-4 (28), 2018 года.
Бесплатный доступ
В данной статье рассматриваются эффлюкс механизм и гены ответственные за резистентность к тетрациклину. В настоящее время идентифицировано 46 различных генов (tet) устойчивости к тетрациклину. Большинство генов (tet) кодируют один из двух механизмов резистентности к тетрациклинам: либо эффлюкс, либо рибосомную защиту.
Детерминанты устойчивости к тетрациклину, эффлюкс насосы, суперсемейство mfs
Короткий адрес: https://sciup.org/140281559
IDR: 140281559
Текст научной статьи Эффлюкс механизм и гены резистентности к тетрациклину у бактерий
В 1945 году впервые был выделен антибиотик хлортетрациклин из культуральной жидкости лучистого гриба Streptomyces aureofaciens. В 1948 году препарат начали использовать в клинической практике, а в 1952 году химическим путём, посредством восстановительного дегалоидирования хлортетрациклина, был получен полусинтетический антибиотик тетрациклин [1].
Тетрациклины проникают в бактериальные клетки путем пассивной диффузии. Тетрациклин действует путем связывания с 30S рибосомальной субъединицей, приводя к ингибированию синтеза белка. Гены, определяющие устойчивость к тетрациклину у грамотрицательных бактерий, часто обнаруживаются на транспозоне Tn10, передаваемом между ними крупными конъюгативными плазмидами [2].
Спустя год после получения тетрациклина, в 1953 году были выделены бактерии резистентные к этому антибиотику [1].
Несмотря на большую клиническую устойчивость, тетрациклины по-прежнему остаются важными препаратами для лечения различных инфекций человека, вызванных грамотрицательными и грамположительными бактериями, наряду с атипичными патогенами, включая микоплазмы, нематоды и паразитических простейших [3].
Механизм и гены антибиотикорезистентности к тетрациклину
В настоящий момент известно 46 различных генов резистентности к тетрациклину [4]. Большинство генов tet кодируют один из двух механизмов резистентности к тетрациклинам: либо отток антибиотика, либо рибосомную защиту. Рибосомная защита включает синтез белка, подобный фактору элонгации G. Он взаимодействует с рибосомой, не препятствуя синтезу белка, но и не давая тетрациклину ингибировать этот синтез [2].
Эффлюкс насосы и рибосомные защитные белки являются наиболее распространёнными типами клинической резистентности к тетрациклину и обнаруживаются у большинства патогенных бактерий человека [5].
Эффлюкс насосы являются наиболее часто встречаемы среди грамотрицательных бактерий, а рибосомные защитные механизмы устойчивости более распространены у грамположительных бактерий [6].
Эффлюкс гены грамотрицательных бактерий часто ассоциируются с конъюгативными плазмидами (гены tetA–tetE, tetG и tetH ), в то время как у грамположительных бактерий эти гены часто обнаруживают на небольших мобилизуемых плазмидах с молекулярной массой 2,7 и 2,8 МДа или хромосоме (гены tetK и tetL ). У грамположительных микроорганизмов резистентность к тетрациклину обычно ассоциируется с плазмидами [2].
Эффлюкс насосы подразделяются на 5 суперсемейств:
-
1. ABC (ATP binding cassette);
-
2. SMR (the Small Multidrug Resistance);
-
3. MFS (the Мajor Facilitator Superfamily);
-
4. RND (Resistance Nodulation Division);
-
5. MATE (the Multidrug And Toxic compound Extrusion).
Они различаются по аминокислотной последовательности, энергетическим ресурсам, затрачиваемым на экспорт антибиотика, и по субстратной специфичности [7].
К эффлюкс генам выводящим тетрациклины из бактериальных клеток относятся: otrB, otrC, tcr3, tetA, tetA(P), tetB, tetC, tetD, tetE, tetG, tetH, tetI, tetJ, tetK, tetL, tetV, tetY, tetZ, tet(30), tet(31), tet(33), tet(35), tet(38), tet(39), tet(40), tet(41), tet(42) [3, 5].
Большинство эффлюкс генов tet кодирует приблизительно 46 кДа эффлюкс белков, которые связанны с мембранной клетки [1].
Гены резистентности к тетрациклину, кодирующие эффлюкс насосы, принадлежат к суперсемейству MFS. Эффлюкс насосы разделяют на 28 классов, присутствующих в бактериях, они делятся на семь групп, которые основаны на гомологии последовательностей. Самая большая группа -первая, содержащая 12 трансмембранных спиралей. Она включает в себя следующие гены: tetA, tetB, tetC, tetD, tetE, tetG, tetH, tetZ, tetl и tet30 [3].
Все гены tet ответственные за эффлюкс, кодируют мембранноассоциированные белки, которые экспортируют тетрациклин из клетки. Выведение тетрациклина и клетки снижает внутриклеточную концентрацию препарата и, таким образом, защищает рибосомы (рис. 1)
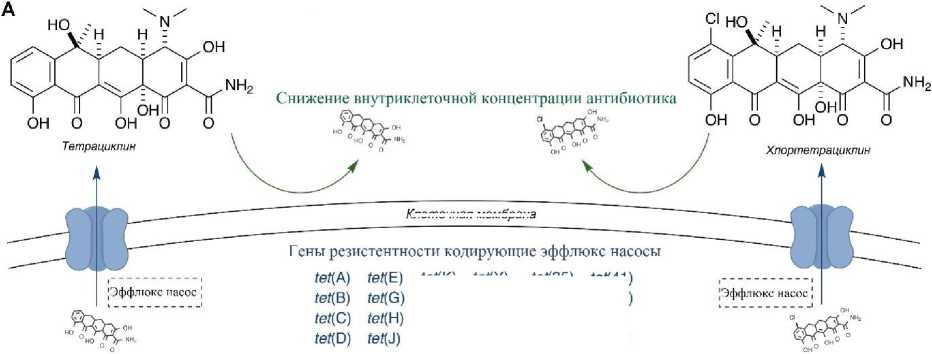
Клеточная мембрана
Рис. 1. Эффлюкс механизм резистентности к тетрациклину [5].
tef(K) fef(Y) tef(L) fef(Z) ?еИ(Р) fef(31) tef(V) te«33)
tef(35)
fef(38)
/ef(39) fef(40)
fe/(41) tef(42) tor(3) otrtC)
В 2007 году ученые изучили встречаемость генов устойчивости к тетрациклину среди кишечной палочки, выделенной от пациентов, и обнаружили, что гены tetA и tetB имели наибольшую частоту встречаемости среди изученных детерминант [8].
В 2016 году Sahar Nouri Gharajalar и Vahideh Hamidi Sofiani провели исследования по изучению распространенности детерминант устойчивости к тетрациклину у кишечной палочки, выделенных из мочи пациентов. Из 50 протестированных изолятов кишечной палочки устойчивость к тетрациклину была выявлена у 62% штаммов. ПЦР-анализ показал, что 36% изолятов содержали ген tetB и 32% бактерий содержали ген tetA. Среди всех изолятов распространенность генов tetG, tetZ, tetC, tetE, tetH и tetD составила 16%, 14%, 12%, 12%, 11% и 8% соответственно [9].
Заключение
Бактериальная резистентность к тетрациклину вследствие приобретения новых генов и / или мутации существующих уже генов увеличилась в течение последних 30 лет. Окружающая среда, несомненно, является важным резервуаром для генов устойчивости. Приобретенные бактериями гены часто связаны с мобильными элементами, которые обеспечивают распространение этих генов в различных бактериальных популяциях и экосистемах.
Если общее использование тетрациклинов, и всех антибиотиков в целом, не изменится, то это может привести к снижению эффективности современных методов лечения.
Список литературы Эффлюкс механизм и гены резистентности к тетрациклину у бактерий
- van Hoek, Angela & Mevius, Dik & Guerra, Beatriz & Mullany, Peter & Roberts, Adam & Aarts, Henk. Acquired Antibiotic Resistance Genes: An Overview // Frontiers in microbiology. - 2011. - 2. 203. URL: (Дата обращения 16.10.2018). DOI: 10.3389/fmicb.2012.00384
- Супотницкий М.В. Механизмы развития резистентности к антибиотикам у бактерий // Биопрепараты. - 2011. - № 2. - с. 4-44.
- Chopra, I. Tetracycline antibiotics: mode of action, applications, molecular biology, and epidemiology of bacterial resistance / Chopra, M. Roberts // Microbiol. Mol. Biol. Rev. - 2001. - 1. Vol. 65, -P. 232 - 260 URL: https://mmbr.asm.org/content/mmbr/65/2/232.full.pdf (Дата обращения 30.11.2018).
- M. C. Roberts and S. Schwarz. Tetracycline and phenicol resistance genes and mechanisms: Importance for agriculture, the environment, and humans // Journal of Environmental Quality. - 2016. - Vol. 45, № 2. - P. 581. URL: https://www.researchgate.net/publication/284755235 (Дата обращения 28.10.2018).
- Markley J.L., Wencewicz T.A. Tetracycline-Inactivating Enzymes // Front. Microbiol. 2018. - Vol. 9:1058. P. 1 - 27. URL: (Дата обращения 01.12.2018). DOI: 10.3389/fmicb.2018.01058
- Bryan A, Shapir N, Sadowsky MJ. Frequency and distribution of tetracycline resistance genes in genetically diverse, nonselected, and nonclinical Escherichia coli strains isolated from diverse human and animal sources // Appl Environ Microbiol. - 2004. - 70(4) P. 2503 - 2507. URL: (Дата обращения 01.12.2018). DOI: 10.1128/AEM.70.4.2503-2507.2004
- Марданова, А. М. Эффлюкс-системы SERRATIA MARCESCENS / А.М. Марданова, Л.М. Богомольная, Ю.Д. Романова, М.Р. Шарипова // Микробиология. - 2014. - Т.83. - № 1. - С.3-14.
- Tuckman M, Petersen PJ, Howe AY, Orlowski M, Mullen S, Chan K, et al. Occurrence of tetracycline resistance genes among Escherichia coli isolates from the phase 3 clinical trials for tigecycline. Antimicrob Agents Chemother. - 2007.- 51(9):3205-11. (Дата обращения 20.12.2018). DOI: 10.1128/AAC.00625-07
- Nouri Gharajalar S, Hamidi Sofiani V. Patterns of Efflux Pump Genes Among Tetracycline Resistance Uropathogenic Escherichia coli Isolates Obtained From Human Urinary Infections. Jundishapur J Microbiol. - 2017. - 10(2):e40884. - P. 1-5. URL: http://jjmicrobiol.com/en/articles/56732.html (Дата обращения 20.12.2018).


