Экспрессия H/ACA мякРНК в клеточных линиях с хромосомными нарушениями после облучения
Автор: Расторгуева Евгения Владимировна, Погодина Евгения Сергеевна, Юрова Елена Валерьевна, Белобородов Евгений Алексеевич, Сугак Дмитрий Евгеньевич, Саенко Юрий Владимирович, Фомин Александр Николаевич
Журнал: Ульяновский медико-биологический журнал @medbio-ulsu
Рубрика: Физиология
Статья в выпуске: 4, 2022 года.
Бесплатный доступ
Семейство H/ACA мякРНК участвует в биогенезе псевдоуридина, препятствуя генетическим изменениям в клетках, делая их более устойчивыми, через стабильность рибосомальной РНК. Поэтому изучение экспрессии H/ACA мякРНК в клеточных линиях при хромосомных нарушениях после облучения представляет особый интерес. Цель исследования - изучение влияния хромосомных нарушений на экспрессию H/ACA мякРНК в радиорезистентной К562 и радиочувствительной HL-60 клеточных линиях после радиационного воздействия. Материалы и методы. Клеточные линии К562 и HL-60 облучали в дозе 4 Гр. Экспрессию H/ACA мякРНК анализировали с помощью NGS-секвенирования через 1, 4 и 24 ч после облучения. Результаты. Выявили отличия экспрессии H/ACA мякРНК по хромосомам в изучаемых клеточных линиях, а также влияние хромосомных аномалий на экспрессию H/ACA мякРНК после радиационного воздействия. Изменение количества копий нормальных хромосом ведет к незначительным изменениям экспрессии H/ACA мякРНК. Наличие маркерных хромосом приводит к нарушению экспрессии H/ACA мякРНК, что делает невозможным использование H/ACA мякРНК, локализованных в аномальных хромосомах, в качестве маркеров радиорезистентности. Кроме того, при наличии маркерных хромосом снижается количество экспрессирующихся H/ACA мякРНК в K562, несмотря на большее количество генетического материала.
Семейство h/aca мякрнк, раковые клетки, маркерные хромосомы
Короткий адрес: https://sciup.org/14126330
IDR: 14126330 | УДК: 577.218 | DOI: 10.34014/2227-1848-2022-4-149-159
Текст научной статьи Экспрессия H/ACA мякРНК в клеточных линиях с хромосомными нарушениями после облучения
Введение. Все большее значение в патофизиологических процессах приобретают малые некодирующие РНК, одними из которых являются малые ядрышковые РНК (мяк(мяо)РНК). Литературные данные [1] свидетельствуют о том, что мякРНК обладают опухоль-супрессивными или онкогенными функциями при различных типах рака. МякРНК участвуют во многих биологических процессах рака, включая инвазию супрессоров роста и гибель клеток, активацию инвазии и метастазирования, а также ангиогенез и устойчивую пролиферативную передачу сигналов. Регуляция экспрессии мякРНК может способствовать достижению цели лечения рака. Например, опосредованное антисмысловыми олигонуклеотидами подавление экспрессии SNORA23 уменьшало рост опухоли, диссеминацию и метастазирование в печень при аденокарциноме протоков поджелудочной железы у мышей [2]. Wenjing Zhu et al. идентифицировали сигнатуру из девяти мякРНК в качестве биомаркеров для прогнозирования рака яичников [3]. Возможно, в ближайшем будущем мякРНК станут одними из мишеней для лечения рака [1].
В своей предыдущей работе [4] мы исследовали влияние хромосомных нарушений на экспрессию мякРНК в радиорезистентной и радиочувствительной клеточных линиях после радиационного воздействия. Здесь мы сфокусировались на одном из семейств мякРНК. Большинство мякРНК можно разделить на два семейства: C/D и H/ACA, названные в честь консервативных консенсусных частей, которые они содержат. Дополнительные РНК, такие как микроРНК (miRNAs), которые могут быть обнаружены в ядрышках, не рассматриваются как мякРНК. Некоторые малые РНК тельца Кахаля (скаРНК), обычно участвующие в направлении модификаций малых ядерных РНК (снРНК) сплайсосомы, содержат C/D, H/ACA или оба типа консервативных блоков, а фрагмент РНК теломеразного ком- плекса (hTR/TERC), участвующий в поддержании концов хромосом, содержит блок H/ACA [5]. Эти мякРНК широко распространены в различных хромосомах, при этом хромосома 1 содержит наибольшее количество мякРНК (188 мякРНК), за ней следует хромосома 15, в которой находится 183 мякРНК [6]. Биогенез C/D- и H/ACA-блоков мякРНК включает биосинтез и сборку малых ядрышковых рибонуклеопротеиновых частиц (мякРНП) с последующей их локализацией в тельцах Кахаля и ядрышках [1].
Основная функция H/ACA мякРНК – превращение специфических остатков уридина в псевдоуридин на рибосомальной РНК (рРНК). Псевдоуридин стабилизует рРНК, тем самым потенциально влияя на структуру, белковый состав и функцию рибосомы [5, 7].
Возможно, экспрессия H/ACA мякРНК и биогенез H/ACA мякРНП изменяются в ответ на повышенную потребность в синтезе белка (при повышенных требованиях к клеточному росту). Например, несколько генов [7, 8], содержащих H/ACA мякРНК, включая DKC1, кодирующий псевдоуридинсинтазу дискерин, являются прямыми мишенями хорошо охарактеризованного онкогена и фактора транскрипции Myc [8]. Мутации дискерина и последующее снижение псевдоуридилирования рРНК характерны для Х-сцепленного врожденного дискератоза (рибосомопатии), при этом у пациентов наблюдается явное увеличение восприимчивости к раку [9]. Мутанты дис-керина и нарушение псевдоуридилирования снова связаны со сниженной IRES-зависимой трансляцией p53 мРНК. Дискерин также является частью теломеразного РНП, поскольку РНК TERC разделяет блок H/ACA с одноименным семейством мякРНК, а активация теломеразы является общим признаком многих видов рака. Отдельные мякРНК, особенно U3 или U8, необходимы для онкогенеза, их истощение приводит к противоопухолевому ответу посредством повышения уровня p53 и апоптоза, а затем к нарушению биогенеза рибосом [5].
Исследования показывают, что мякРНК играют критическую роль в микроокружении опухоли [10]. Псевдоуридин стабилизирует РНК. Наше исследование может показать, меняется ли экспрессия семейства H/ACA мякРНК после радиационного воздействия и играют ли роль данные нуклеиновые кислоты в изменении радиочувствительности опухолевых клеток при хромосомных нарушениях.
Цель исследования. Изучение влияния хромосомных нарушений на экспрессию H/ACA мякРНК в радиорезистентной К562 и радиочувствительной HL-60 клеточных линиях после радиационного воздействия.
Материалы и методы. Объектами исследования являлись радиочувствительная клеточная линия промиелоцитарного лейкоза человека HL-60 и радиорезистентная клеточная линия хронической миелоидной лейкемии человека К562 (Институт цитологии РАН, г. Санкт-Петербург). Клетки культивировались при температуре 37 °С, содержании 5 % CO 2 и 98 % влажности. В экспериментах использовалась среда RPMI-1640 с L-глутамином («ПанЭко», Россия), содержащая 50 мкг/мл гентамицина («ПанЭко», Россия) и 10 % эмбриональную бычью сыворотку (PAA Laboratories GmbH, Австрия).
Клеточные линии подвергались γ-облуче-нию в дозе 4 Гр (энергия фотонов 10 МэВ) линейным ускорителем Elekta Synergy (Elekta, Швеция). Облучение происходило при комнатной температуре и длилось 55 с. Перед облучением клетки переносились в 6-луночные планшеты. Транспортировка планшетов с клеточными культурами до места облучения и обратно производилась в термостатном контейнере при температуре 37 °С. Все эксперименты проводились во время логарифмической фазы роста клеток. Манипуляции с контрольной группой были аналогичны манипуляциям с экспериментальной группой, за исключением облучения.
Выживаемость клеток оценивалась с использованием смеси флуоресцентных красителей – пропидиум йодида и акридинового оранжевого. Анализ окрашенных клеток проводился с помощью методов флуоресцентной микроскопии [12].
Тотальная РНК выделялась из клеток набором Absolutely RNA miRNA Kit (Agilent Technologies, США) через 1, 4 и 24 ч после облучения. Качество выделенной РНК оценивалось по соотношению 18S/28S РНК капиллярным электрофорезом Agilent 2100 Bioanalyzer
(Agilent Technologies, США). Для эксперимента отбирались образцы с индексом сохранности РНК RIN>8,0. Библиотеки кДНК из мякРНК готовились с использованием набора NEBNext Small RNA Library Prep Set (NEB, Великобритания). Очистка библиотек кДНК мякРНК осуществлялась в 6 % полиакриламидном геле с помощью электрофореза. Из геля вырезались участки кДНК соответствующих мякРНК между 145 и 160 п.н. и выделялись кДНК. Во всех полученных библиотеках мякРНК количество кДНК измерялось флуорометром Qubit (Invitrogen, США). Из каждой библиотеки отобрались эквимолярные количества (2 нМ) образца для пула библиотек, который секвенировался с помощью системы высокопроизводительного секвенирования MiSeq System (Illumina, США) с использованием набора для одноконцевого чтения 150 п.н.
После секвенирования получились файлы формата FASTQ. Обработка данных осуществлялась с использованием сервиса GenXPro omiRas. В результате формировались таблицы с данными, которые включали название H/ACA мякРНК и нормализованное на 105 прочтений количество H/ACA мякРНК. При обработке полученных данных вычислялось количество дифференциально экспрессирующихся H/ACA мякРНК и количество одноименных дифферен- циально экспрессирующихся H/ACA мякРНК в двух клеточных линиях через 1, 4 и 24 ч после облучения. Величина экспрессии H/ACA мякРНК выражалась через log2 отношения нормализованной экспрессии мякРНК в опыте к аналогичному показателю в контроле (log2fc).
Определение локализации H/ACA мякРНК в хромосомах проводилось с использованием базы данных National Center for Biotechnology Information Search data base .
Каждый эксперимент проводился в трех повторах. Результаты представлялись как среднее значение и стандартное отклонение (М±SD). Для оценки статистической значимости различий применялся t-критерий Стьюдента для парных переменных. Различия между группами считались статистически значимыми при р<0,05.
Результаты. На рис. 1 показан процент живых клеток после однократного облучения в дозе 4 Гр раковых клеточных линий HL-60 и K562 от общего числа. Доля живых клеток в клеточной линии HL-60 в контроле составила 94 %. После радиационного воздействия данный показатель снизился до 79 %. В радиоре-зистентной клеточной линии K562 доля выживших клеток от общего количества клеток снизилась с 92,5 % до 88 %.
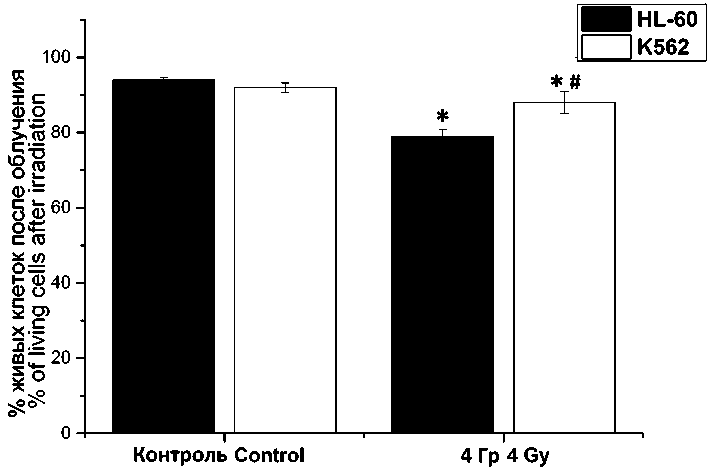
Рис. 1. Процент живых клеток в контроле и после радиационного облучения в дозе 4 Гр в клеточных линиях HL-60 и K562.
* – статистически значимые отличия по отношению к контролю;
# – статистически значимые отличия по отношению к клеточной линии HL-60 (р<0,05)
Fig. 1 . Percentage of living cells in the control and after radiation exposure (4 Gy) in HL-60 and K562 cell lines. * – the differences are statistically significant compared to the control;
-
# – the differences are statistically significant compared to HL-60 cell line, p<0.05
Количество типов экспрессирующихся мякРНК, локализованных в каждой хромосоме, в каждой точке эксперимента представлено на рис. 2. В клеточной линии K562 наибольшее количество экспрессирующихся РНК локализовано в 1, 3, 17-й хромосомах, а в клеточной линии HL-60 – в 1-й и 11-й хромо- сомах. В обеих клеточных линиях есть хромосомы, в которых мякРНК не экспрессируются, например 15, 21 и 22-я. Распределение экспрессирующихся H/ACA мякРНК по хромосомам в клеточных линиях различается и изменяется в течение эксперимента.
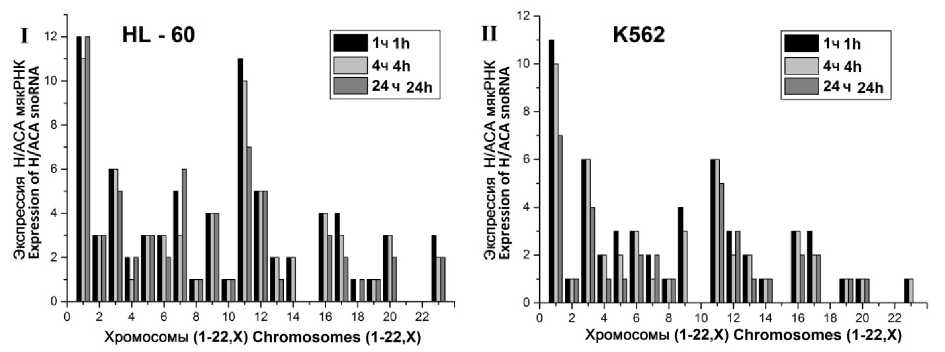
Рис. 2. Количество экспрессирующихся H/ACA мякРНК в зависимости от хромосомной локализации в клеточных линиях HL-60 (I) и К562 (II) через 1, 4 и 24 ч после облучения
Fig. 2. The number of expressed H/ACA snoRNAs depending on the chromosomal localization in HL-60 (I) and K562 (II) cell lines (1, 4 and 24 hours after irradiation)
На рис. 3 представлен разброс значений экспрессии (выраженных через log2fc) H/ACA мякРНК, распределенных в соответствии с их локализацией в хромосомах, в клеточных линиях HL-60 и К562. В клеточной линии K562 во всех точках эксперимента (рис. 3, I–III) H/ACA мякРНК имеют более широкий разброс значений log2fc (от -1 до +1 через 1 ч после облучения, от -1 до +2 через 4 ч, от -2 до +1 через 24 ч), что говорит о значительных отличиях в экспрессии H/ACA мякРНК после радиационного воздействия в сравнении с контрольной группой. Клеточная линия HL-60 характеризуется меньшим разбросом значений log2fc, их основное количество сосредоточено в интервале от -1 до +1,5 через 1, 4 и 24 ч после облучения (рис. 3, IV–VI).
Таким образом, изменения экспрессии H/ACA мякРНК в клеточных линиях К562 и HL-60 характерны для всех хромосом. Наибольшие отличия в значениях log2fc экспрессирующихся мякРНК зафиксированы в кле- точной линии К562 на протяжении всего эксперимента.
На рис. 4 представлен кластерный анализ экспрессии H/ACA мякРНК, локализованных в хромосомах 3, 5 и 11 в клеточных линиях K562 и HL-60: I и II – кластеризация экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 3, в клеточных линиях K562 и HL-60 соответственно; III – кластеризация экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 5, в клеточной линии K562 (трисомия хромосомы 5); IV – кластеризация экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 5, в клеточной линии HL-60 (моносомия хромосомы 5); V – кластеризация экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 11, в клеточной линии K562 (трисомия хромосомы 11, маркерная хромосома); VI – кластеризация экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 11, в клеточной линии HL-60 (две нормальные хромосомы 11).
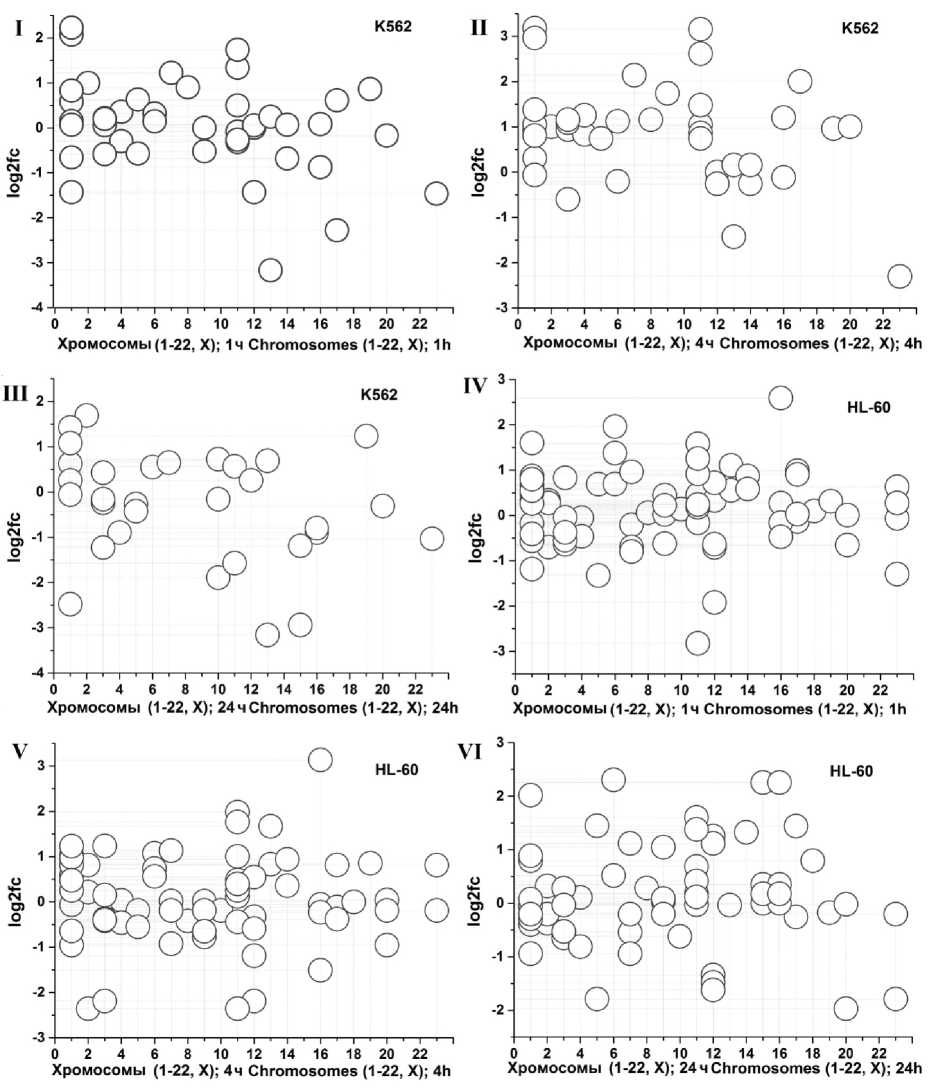
Рис. 3 . Анализ экспрессии H/ACA мякРНК в зависимости от хромосомной локализации после радиационного воздействия в дозе 4 Гр в сравнении с контролем
Fig. 3 . Dynamics of H/ACA snoRNA expression depending on chromosomal localization after radiation exposure (4 Gy) compared to the control
Как видно из рис. 4, экспрессия H/ACA мякРНК в хромосоме 3 в рассматриваемых клеточных линиях, которые имеют по две нормальные хромосомы из каждой пары, примерно одинаковая. В хромосоме 5, в которой у клеточной линии K562 выявлена трисомия, а у клеточной линии HL-60 – моносомия, виды и кластеризация H/ACA мякРНК также имеют лишь незначительные отличия.
Однако в хромосоме 11 кластеризация и виды экспрессирующихся H/ACA мякРНК достаточно сильно отличаются. В К562 – 9 H/ACA мякРНК (помимо общих H/ACA мякРНК экспрессируется has-sno-U97), а в HL-60 –
12 H/ACA мякРНК (отличается от К562 экспрессией has-sno-ACA52, has-sno-ACA32, has-sno-ACA40, has-sno-U90). Наблюдаемые различия в кластеризации и типах экспрессирующихся H/ACA мякРНК говорят о влиянии хромосомных мутаций на экспрессию генов.
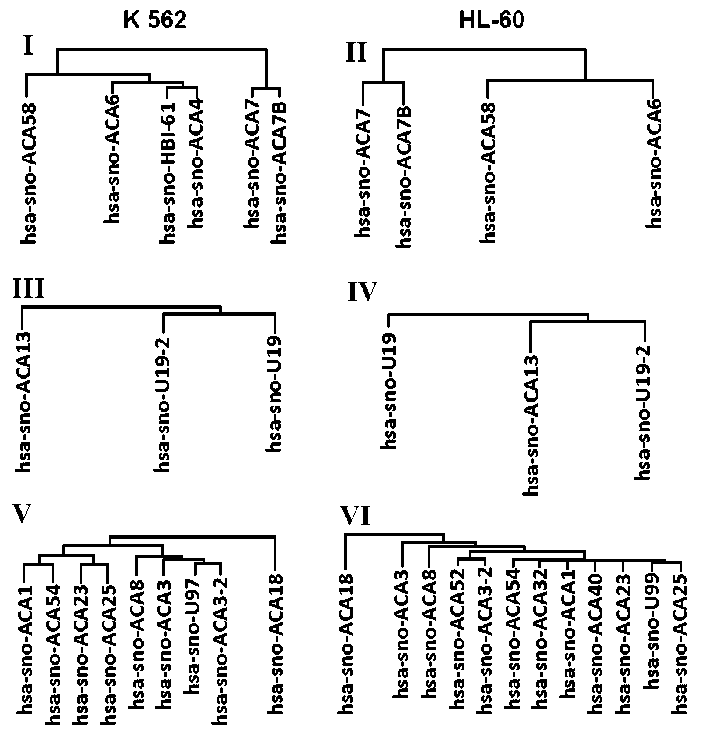
Рис. 4. Кластерный анализ экспрессии H/ACA мякРНК, локализованных в хромосомах 3 (I, II), 5 (III, IV) и 11 (V, VI), в клеточных линиях K562 и HL-60
Fig. 4 . Cluster analysis of H/ACA snoRNA expression localized in 3 (I, II), 5 (III, IV), and 11 (V, VI) chromosomes, K562 and HL-60 cell lines
Обсуждение. В данной работе были использованы две раковые клеточные линии лейкемии, которые имеют разную чувствительность к радиационному воздействию и разный уровень хромосомных аномалий. Клеточная линия HL-60 характеризуется небольшим количеством геномных аномалий (моносомия хромосом 5, 15 и Х, трисомия хромосомы 18) и обладает умеренной радиочувствительностью [12]. Клеточная линия К562 имеет 14 геномных мутаций (моносомия хромосом 7, 12, 13, 17, 19, Х, трисомия хромосом 1, 4, 5,
-
8, 11, 15, 16, нулисомия хромосомы 9) и маркерную хромосому 21 и является менее радиочувствительной [13]. Подобные различия в геноме будут влиять на экспрессию всех генов. По данным наших экспериментов, около половины всех H/ACA мякРНК в клетках К562 и HL‑60 изменяют свою экспрессию после радиационного воздействия в дозе 4 Гр, литературные данные также свидетельствуют о том, что измененная экспрессия – это часть сложной реакции в клетках после облучения [14]. Анализ экспрессии мякРНК с учетом хромосом-
- ной локализации после радиационного воздействия показал, что изменения в количестве экспрессирующихся H/ACA мякРНК характерны для большинства хромосом (рис. 2). Наибольшие отличия в профиле экспрессии H/ACA мякРНК наблюдаются в радиорези-стентной клеточной линии К562 (рис. 2).
В нашей работе было показано, что клеточная линия К562 экспрессирует меньшее количество H/ACA мякРНК по сравнению с клеточной линией HL-60 (рис. 2 и 3), хотя и имеет большее количество генетического материала. Увеличение количества генетического материала в клеточной линии К562 связано с наличием большого числа трисомий и маркерной хромосомы. Однако эти хромосомы, появившиеся в результате геномных мутаций и хромосомных аберраций, обладают нарушенной функциональной целостностью, что в конечном итоге сказывается на снижении их функциональной активности [15]. Показано, что H/ACA мякРНК участвуют в биогенезе псевдоуридина, который в свою очередь препятствует генетическим изменениям в клетках, делает их более устойчивыми через стабильность рибосомальной РНК [16]. Следовательно, увеличение количества экспрессирующихся H/ACA мякРНК можно связать с генетически более стабильной клеточной линией HL-60.
На рис. 4 (I и II) показаны экспрессирующиеся H/ACA мякРНК, локализованные в хромосоме 3 клеточных линий K562 и HL-60. Обе клеточные линии имеют по две нормальные копии хромосомы 3. Кластеризация H/ACA мякРНК в обеих клеточных линиях схожа, при этом экспрессируются одинаковые H/ACA мякРНК. Аналогичные результаты получены нами и для других нормальных хромосом клеточных линий К562 и HL-60 (хромосомы 14 и 20). Данные результаты говорят о том, что экспрессия H/ACA мякРНК, локализованных в нормальных хромосомах родственных клеточных линий, может иметь лишь незначительные отличия.
Количество, кластеризация и типы экспрессирующихся H/ACA мякРНК, локализованных в хромосоме 5, в двух исследуемых клеточных линиях одинаковы. Экспрессия H/ACA мякРНК при наличии трисомии и мо- носомии отличается незначительно (рис. 4). Это обусловлено отсутствием функциональных перестроек внутри хромосомы при данных видах хромосомных аномалий. Поэтому можно предположить, что регуляция экспрессии генов должна быть такая же, как и у нормальных хромосом.
Для дальнейшего анализа была выбрана хромосома 11, которая представлена в клеточной линии HL-60 двумя нормальными копиями, а в K562 тремя нормальными копиями хромосомы 11 и маркерной хромосомой. В наших экспериментах было продемонстрировано, что при наличии дополнительной маркерной хромосомы экспрессия H/ACA мякРНК и кластеризация существенно отличаются по сравнению с нормальной хромосомой 11 (рис. 4, V и VI). Подобная картина наблюдается и при сравнении экспрессии H/ACA мякРНК других нормальных хромосом клеточной линии HL-60 c аномальными хромосомами клеточной линии К562 (хромосомы 2, 6, 17, 19, 22). О маркерных хромосомах и их влиянии на экспрессию генов у людей известно немного. Исследования сперматозоидов с маркерными хромосомами показали, что данное нарушение приводит к изменению экспрессии генов и бесплодию [17], у пациентов с маркерными хромосомами наблюдаются отклонения в развитии и различные патологии [18]. Приведенные выше данные свидетельствуют о том, что маркерные хромосомы оказывают значительное влияние на экспрессию H/ACA мякРНК и экспрессию генов в целом. Но из-за большого разнообразия маркерных хромосом соотнести определенную маркерную хромосому с конкретным нарушением или заболеванием достаточно трудно.
Заключение. Проанализировав динамику экспрессии семейства H/ACA мякРНК в раковых клеточных линиях К562 и HL-60, можно сделать вывод, что характер и изменение экспрессии в ответ на ионизирующее облучение в наших экспериментах могут зависеть от наличия хромосомных аномалий. Трисомии и моносомия не оказывают такого существенного влияния на экспрессию H/ACA мякРНК, как наличие маркерных хромосом. Маркерные хромосомы приводят к нарушению экспрессии H/ACA мякРНК, что делает невоз- можным использование H/ACA мякРНК, локализованных в аномальных хромосомах, в качестве маркеров радиорезистентности.
Данное исследование позволяет ближе подойти к пониманию значения нарушений экспрессии H/ACA мякРНК для использования их в качестве маркеров для лечения онкологических заболеваний человека и будет способствовать дальнейшему развитию персонализированной медицины.
Работа выполнена при поддержке Министерства образования и науки РФ в рамках государствен ного задания № 0830-2020-0008.
Список литературы Экспрессия H/ACA мякРНК в клеточных линиях с хромосомными нарушениями после облучения
- Liang J., Wen J., Huang Z., Chen X.P., Zhang B.X., Chu L. Small Nucleolar RNAs: Insight Into Their Function in Cancer. Front Oncol. 2019; 9: 587.
- Cui L., Nakano K., Obchoei S. Small Nucleolar Noncoding RNA SNORA23, Up-Regulated in Human Pancreatic Ductal Adenocarcinoma, Regulates Expression of Spectrin Repeat-Containing Nuclear Envelope
- 2 to Promote Growth and Metastasis of Xenograft Tumors in Mice. Gastroenterology. 2017; 153 (1): 292–306.e2.
- Zhu W., Zhang T., Luan S. Identification of a novel nine-SnoRNA signature with potential prognostic and therapeutic value in ovarian cancer. Cancer Med. 2022; 11 (10): 2159–2170.
- Rastorgueva E., Liamina D., Panchenko I. The effect of chromosome abnormalities on expression of SnoRNA in radioresistant and radiosensitive cell lines after irradiation. Cancer Biomark. 2022; 34 (4): 545–553.
- Abel Y., Rederstorff M. SnoRNAs and the emerging class of sdRNAs: Multifaceted players in oncogenesis. Biochimie. 2019; 164: 17–21.
- Dsouza V.L., Adiga D., Sriharikrishnaa S., Suresh P.S., Chatterjee A., Kabekkodu S.P. Small nucleolar RNA and its potential role in breast cancer - A comprehensive review. Biochim Biophys Acta Rev Cancer. 2021; 1875 (1): 188501.
- McMahon M., Contreras A., Holm M. A single H/ACA small nucleolar RNA mediates tumor suppression downstream of oncogenic RAS. Elife. 2019; 8: e48847.
- McMahon M., Contreras A., Ruggero D. Small RNAs with big implications: new insights into H/ACA snoRNA function and their role in human disease. Wiley Inter discip Rev RNA. 2015; 6 (2): 173–189.
- Garus A., Autexier C. Dyskerin: an essential pseudouridine synthase with multifaceted roles in ribosome biogenesis, splicing, and telomere maintenance. RNA. 2021; 27 (12): 1441–1458.
- Cai C., Peng Y., Shen E. Identification of tumour immune infiltration-associated snoRNAs (TIIsno) for predicting prognosis and immune landscape in patients with colon cancer via a TIIsno score model. EBio Medicine. 2022; 76: 103866.
- Mascotti K., McCullough J., Burger S.R. HPC viability measurement: Trypan blue versus acridine orange and propidium iodide. Transfusion. 2000; 40: 693–696.
- Liang J.C., Ning Y., Wang R.Y. Spectral karyotypic study of the HL-60 cell line: detection of complex rearrangements involving chromosomes 5, 7, and 16 and delineation of critical region of deletion on 5q31.1. Cancer Genet Cytogenet. 1999; 113 (2): 105–109.
- Naumann S., Reutzel D., Speicher M., Decker H.J. Complete karyotype characterization of the K562 cell line by combined application of G-banding, multiplex-fluorescence in situ hybridization, fluorescence in situ hybridization, and comparative genomic hybridization. Leuk Res. 2001; 25 (4): 313–322.
- Chaudhry M.A. Expression pattern of small nucleolar RNA host genes and long non-coding RNA in X-rays-treated lymphoblastoid cells. Int J Mol Sci. 2013; 14 (5): 9099–9110.
- De Falco G., Ambrosio M.R., Fuligni F. Burkitt lymphoma beyond MYC translocation: N-MYC and DNA methyltransferases dysregulation. BMC Cancer. 2015; 15: 668.
- Barozzi C., Zacchini F., Asghar S., Montanaro L. Ribosomal RNA Pseudouridylation: Will Newly Available Methods Finally Define the Contribution of This Modification to Human Ribosome Plasticity? Front Genet. 2022; 13: 920987.
- Slimani W., Jelloul A., Al-Rikabi A. Small supernumerary marker chromosomes (sSMC) and male infertility: characterization of five new cases, review of the literature, and perspectives. J Assist Reprod Genet. 2020; 37 (7): 1729–1736.
- An N., Yu Y., Xi Q. Molecular Characterization of Mosaicism for a Small Supernumerary Marker Chromosome Derived from Chromosome Y in an Infertile Male with Apparently Normal Phenotype: A Case Report and Literature Review. Biomed ResInt. 2019; 2019: 9398275.


