Экспрессия микроРНК в опухолевой ткани у больных раком гортани
Автор: Никитина Екатерина Геннадьевна, Черемисина Ольга Владимировна, Бычков Вячеслав Алексеевич, Кульбакин Денис Евгеньевич, Чойнзонов Евгений Лхамацыренович, Стегний Владимир Николаевич, Литвяков Николай Васильевич
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 2 (68), 2015 года.
Бесплатный доступ
Изучен аберрантный паттерн экспрессии микроРНК-18а, -21, -155, -200а, -200с, -205, -221, -494 в опухолевой ткани плоскоклеточной карциномы гортани относительно прилежащей нормальной ткани у 46 пациентов. Установлено, что клинико-морфологические параметры не оказывают влияния на экспрессию анализируемых микроРНК. Показана гиперэкспрессия онкогенных микроРНК-21, -155, -205 и гипоэкспрессия онкосупрессорной микроРНК-200а в опухолевой ткани относительно прилежащей неизмененной ткани. Представленные результаты позволяют предполагать существенную роль этих микроРНК в канцерогенезе опухолей гортани.
Рак гортани, микрорнк, экспрессия microrna
Короткий адрес: https://sciup.org/14056526
IDR: 14056526 | УДК: 616.22-006.6,
Текст научной статьи Экспрессия микроРНК в опухолевой ткани у больных раком гортани
МикроРНК постранскрипционно регулируют генную экспрессию, связываясь с 3’-UTR мРНК генов-мишеней по принципу комплементарности, ингибируя или полностью инактивируя мРНК [3, 31]. Благодаря такому механизму микроРНК участвуют в регуляции многих клеточных процессов, включая развитие, дифференцировку, апоптоз, пролиферацию, ответ на стрессовое воздействие, метаболизм и секрецию инсулина [2].
В опухоли часто наблюдается аберрантная экспрессия микроРНК, которая свидетельствует о важной роли этих молекул в процессе канцерогенеза [12]. Аберрантная экспрессия микроРНК может быть следствием различных причин, таких как делеции, амплификации хромосомных локусов микроРНК, мутации или нарушение регуляции транскрипционных факторов, регулирующих экспрессию микроРНК. Влияние на микроРНК могут оказывать и эпигенетические механизмы [17, 23]. Экспрессия микроРНК отличается значительной тканевой специфичностью, и аберрантный паттерн экспрессии микроРНК различается в зависимости от тканевого происхождения опухоли. Описание этого аберрантного паттерна важно с точки зрения понимания механизмов канцерогенеза, поиска новых мишеней для таргетной терапии и маркеров для диагностики, прогноза и мониторинга заболевания. Одной из малоизученных локализаций в этом отношении являются плоскоклеточные карциномы гортани, которые по частоте заболеваемости занимают второе место в структуре опухолей головы и шеи
[8]. Среди микроРНК, для которых было проведено исследование аберрантного паттерна экспрессии в опухоли гортани, были выбраны те молекулы, относительно которых в литературе представлены данные, доказывающие их значительную роль в канцерогенезе при других локализациях. Это микроРНК-21, -18а, -200а, -200с, -205, -221, -494. Было показано, что они играют одну из ключевых ролей в регуляции процессов клеточного цикла, апоптоза, роста, инвазии, пролиферации клеток при опухолях репродуктивных органов у женщин (рак молочной железы, рак яичников), глиобластомах и многих других локализациях [9, 19, 20, 24].
Целью исследования явилось изучение аберрантного паттерна экспрессии микроРНК -18а, -21, -155, -200а, -200с, -205, -221, -494 в опухолевой ткани плоскоклеточной карциномы гортани относительно нормальной прилежащей ткани и связи этого показателя с клинико-морфологическими характеристиками опухолевого процесса.
Материал и методы
В исследование включены 46 больных раком гортани (табл. 1), мужского пола, в возрасте от 45 до 64 лет (в среднем – 56 ± 10 лет), проходивших обследование и лечение в Томском НИИ онкологии. Работа проведена с соблюдением принципов добровольности и конфиденциальности в соответствии с «Основами законодательства РФ об охране здоровья граждан» (Указ президента РФ 39 от 24.12.93 № 2288), получено разрешение
таблица 1
Клинико-морфологические характеристики пациентов
В работе был использован парный операцион-ный/биопсийный материал (опухолевая и прилежащая нормальная ткань) от пациентов с первичным диагнозом плоскоклеточного рака гортани (РГ), не получавших на момент исследования специального лечения. Биопсийный материал из опухолевой и неизмененной ткани получали при видеоларингоскопии. Диагноз у всех пациентов был морфологически верифицирован. Биологический материал помещали в пробирки типа Eppendorf с консервирующим раствором RNAlater (Sigma, USA), пробирки выдерживали сутки при 4°С и потом хранили при –80°С до выделения РНК. Тотальная фракция РНК из ткани была выделена набором реагентов mirVana™ (Ambion, USA). Количество РНК и чистоту выделения оценивали на спектрофотометре NanoDrop 2000 (Thermo-Scientific, USA) (концентрация РНК варьировала от 30 до 800 нг/ мкл, А260/280=1,90). Значение RIN (RNA Integrity Number) колебалось от 8,0 до 9,4 (оценивали на приборе TapeStation 2200 Agilent Technologies, USA). Мультиплексная обратно транскриптазная ПЦР (ОТ-ПЦР) проводилась в объеме 20 мкл, в состав которого входили 100–500 нг РНК-матрицы, 0,5 нM микроРНК-специфичного праймера особой шпилевидной конструкции (hsa-miR-18a-5p, hsa-miR-21-5p, hsa-miR-155-5p, hsa-miR-200a-3p, hsa-miR-200c-3p, hsa-miR-205-5p, hsa-miR-221-3p, hsa-miR-494-3p) [18], 1 ед. ингибитора РНКаз, 50 ед. MMLV ревертазы («Сибэнзим», Новосибирск), 1x MMLV буфера и 1 мM каждого dNTP. Протокол реакции: 38 циклов при 16°C в течение 20 сек, при 42°C в течение 20 сек, при 50°C в течение 1 сек, с заключительным этапом прогрева смеси при 85°C в течение 1 мин. При каждой постановке были включены отрицательные контроли, не содержащие РНК и/или несущие в составе геномную ДНК. Все реакции ПЦР в режиме реального времени проведены в триплете согласно протоколу [18]. В качестве гена-рефери выбрана микроРНК-103 [26], калибровка выполнена относительно прилежащей к опухоли нормальной ткани у каждого пациента, уровень экспрессии микроРНК рассчитан согласно методу Pfaffl [27]. В качестве результата использовано логарифмически трансформированное по основанию е нормализованное значение уровня экспрессии (ln(fold+const)).
Корреляционный анализ, а также обработка данных с применением t-критерия Уэлша и Стьюдента проводились в программе Statistica 8.0. Для всех статистических подсчетов применен критерий множественности сравнения Бенджамини – Хох-берга (FDR – false discovery rate). Все значения р, удовлетворяющие условию FDR ≤ 5 %, принимались как статистически значимые.
Результаты исследования
На первом этапе было изучено возможное влияние на экспрессию микроРНК таких параметров, как стадия распространенности процесса, статус лимфогенного метастазирования и степень дифференцировки опухоли путем сравнительной оценки показателей уровня экспрессии микроРНК в подгруппах. Ввиду малочисленности выборки были объединены пациенты с I–II и III–IV стадиями опухолевого процесса. Сравнение этих групп не выявило значимых различий в уровне экспрессии анализируемых микроРНК. Не обнаружено влияния лимфогенного метастазирования и степени дифференцировки опухолевой ткани на экспрессию анализируемых микроРНК (табл. 2). Хотя для микроРНК-200с показано снижение уровня экспрессии у пациентов без метастазирования в сравнении с подгруппой лиц с лимфогенными метастазами, для микроРНК-18а наблюдается тенденция к повышению уровня экспрессии от низкой к высокой степени дифференцировки опухоли. Однако с учетом поправки FDR данные значения р не являются статистически значимыми (табл. 2). Таким образом, клинико-морфологические параметры не оказывали статистически значимого влияния на экспрессию микроРНК в опухолевой ткани гортани. Это позволило анализировать аберрантный паттерн экспрессии микроРНК в общей группе.
Установлено, что для некоторых микроРНК выявлен аберрантный уровень экспрессии в опухолевой ткани относительно прилежащей нормальной ткани пациентов. Показана статистически значимая гиперэкспрессия микроРНК-21, -155 и -205 (р=0,00005, р=0,00008 и р=0,00085 соответственно). Частота встречаемости случаев с гиперэкспрессией по этим микроРНК в 2,3–4,8
таблица 2
Уровень экспрессии микрорнК в опухоли относительно прилежащей нормальной ткани пациентов в зависимости от клинико-морфологических параметров
|
Параметры |
МикроРНК |
-18а |
-21 |
-155 |
-200а |
-200с |
-205 |
-221 |
-494 |
|
Распространенность первичной опухоли |
Т |
0,20 ± 0,21 |
0,64 ± 0,27 |
0,67 ± 0,33 |
-1,05 ± 0,30 |
-0,22 ± 0,19 |
0,50 ± 0,15 |
-0,70 ± 0,32 |
-0,29 ± 0,39 |
|
Т |
0,26 ± 0,15 |
1,27 ± 0,33 |
0,84 ± 0,17 |
-0,69 ± 0,36 |
-0,01 ± 0,30 |
0,81 ± 0,39 |
0,06 ± 0,31 |
0,09 ± 0,33 |
|
|
р |
0,15 |
0,66 |
0,45 |
0,56 |
0,46 |
0,09 |
0,46 |
0,83 |
|
|
FDR |
58,3 |
75,2 |
119,7 |
74,2 |
74,1 |
74,2 |
91,3 |
82,9 |
|
|
Лимфогенное метастазирование |
N0 |
0,16 ± 0,14 |
0,85 ± 0,24 |
0,72 ± 0,21 |
-0,82 ± 0,28 |
-0,35 ± 0,20 |
0,63 ± 0,25 |
-0,27 ± 0,24 |
-0,11 ± 0,32 |
|
N 1-2 |
0,26 ± 0,23 |
1,82 ± 0,57 |
1,04 ± 0,37 |
-0,82 ± 0,50 |
0,87 ± 0,46 |
0,53 ± 0,49 |
-0,35 ± 0,76 |
0,04 ± 0,25 |
|
|
р |
0,696 |
0,123 |
0,449 |
0,993 |
0,018 |
0,858 |
0,923 |
0,717 |
|
|
FDR |
139,2 |
49,2 |
119,8 |
99,3 |
14,5 |
114,5 |
105,5 |
114,8 |
|
|
Степень дифференцировки опухоли |
Низкая |
0,29 ± 0,18 |
0,87 ± 0,68 |
0,61 ± 0,39 |
-0,60 ± 0,51 |
-0,65 ± 0,62 |
0,37 ± 0,61 |
0,15 ± 0,11 |
-1,14 ± 1,05 |
|
Умеренная |
0,31 ± 0,15 |
0,97 ± 0,27 |
0,74 ± 0,22 |
-0,91 ± 0,31 |
-0,1 ± 0,221 |
0,69 ± 0,28 |
-0,40 ± 0,31 |
0,07 ± 0,28 |
|
|
Высокая |
0,68 ± 0,57 |
0,34 ± 0,68 |
0,62 ± 0,70 |
-0,94 ± 0,70 |
0,19 ± 0,20 |
0,47 ± 0,38 |
-0,38 ± 0,30 |
-0,06 ± 0,53 |
|
|
р1 |
0,01 |
0,89 |
0,77 |
0,6 |
0,41 |
0,63 |
0,1 |
0,27 |
|
|
FDR, % |
12 |
89,4 |
88,5 |
95,9 |
82,8 |
84,6 |
39,7 |
72,9 |
|
|
р2 |
0,14 |
0,59 |
0,99 |
0,7 |
0,23 |
0,9 |
0,14 |
0,39 |
|
|
FDR, % |
57,6 |
94,8 |
99,2 |
93,7 |
61,9 |
102,5 |
112,8 |
77,3 |
|
|
р3 |
0,53 |
0,39 |
0,87 |
0,97 |
0,32 |
0,64 |
0,97 |
0,83 |
|
|
FDR, % |
141,8 |
156,9 |
115,9 |
97,3 |
257 |
127,7 |
110,7 |
132,4 |
Примечание: р – уровень значимости рассчитан по t-критерию Уэлша; р1 – «низкий» vs «умеренный», р2 – «низкий» vs «высокий», р3 – «умеренный» vs «высокий»; FDR – поправка Бенджамини–Хохберга (FDR – false discovery rate).
таблица 3
Уровень экспрессии микрорнК в опухоли относительно прилежащей нормальной ткани
|
МикроРНК |
Уровень экспрессии |
р |
FDR |
Число случаев с гиперэкспрессией |
Число случаев с гипоэкспрессией |
|
-18а (n=45) |
0,23 ± 0,83 |
0,071 |
11,39 |
27 (61 %) |
18 (39 %) |
|
-21 (n=46) |
0,98 ± 1,49 |
0,00005 |
0,04 |
38 (83 %) |
8 (17 %) |
|
-155 (n=46) |
0,76 ± 1,19 |
0,00008 |
0,03 |
34 (74 %) |
12 (26 %) |
|
-200а (n=46) |
-0,85 ± 1,61 |
0,0008 |
0,22 |
14 (30 %) |
32 (70 %) |
|
-200с (n=46) |
-0,11 ± 1,24 |
0,569 |
65,05 |
21 (45 %) |
25 (54 %) |
|
-205 (n=44) |
0,66 ± 1,43 |
0,003 |
0,75 |
37 (81 %) |
9 (19 %) |
|
-221 (n=43) |
-0,31 ± 1,48 |
0,174 |
23,24 |
25 (54 %) |
21 (46 %) |
|
-494 (n=43) |
-0,08 ± 1,64 |
0,762 |
76,21 |
22 (43 %) |
26 (56 %) |
Примечание: р – уровень значимости рассчитан по t-критерию Стьюдента (t-for single means); FDR – поправка Бенджамини–Хохберга.
раза выше, чем гипоэкспрессии. Кроме повышенной экспрессии, для микроРНК-200а отмечается гипоэкспрессия (р=0,0008), и частота случаев с гипоэкспрессией микроРНК-200а в 4,3 раза выше, чем частота гиперэкспрессии (табл. 3).
Для микроРНК, как и для многих молекулярных регуляторов клетки и организма в целом, характерно образование сложной системы взаимодействий друг с другом [14, 32]. На основе корреляционного анализа была показана такая сеть взаимодействий для изученных микроРНК (рис. 1). Выявлена положительная корреляция для микроРНК-21 и -155 (r=0,31, р=0,00001), микроРНК-200с и -18а (r=0,38, р=0,001), 200с и -205 (r=0,34, р=0,025), -200а и -221 (r=0,35, р=0,021) (рис. 1), а также наблюдается тенденция к положительной корреляции между микроРНК-21 и -200с (r=0,31, р=0,036), однако значение р не удовлетворяет 5 % поправке FDR.
Обсуждение
Полученные нами результаты по гиперэкспрессии микроРНК-21, -155 и -205 в опухоли гортани согласуются с данными литературы. J. Ren et al. [29] и P. Cao et al. [5] выявили гиперэкспрессию микроРНК-21 при раке гортани. Результаты по микроРНК-155 при злокачественных опухолях головы и шеи частично соотносятся с данными литературы, имеются отдельные работы, в которых показаны как гиперэкспрессия [6, 16], так и отсутствие значимых изменений ее экспрессии в опухолевой ткани [5]. В целом данные относительно экспрессии микроРНК-205 при раке гортани противоречивы. P. Cao et al. [5] выявили значимую гиперэкспрессию микроРНК-205, однако L. Tian et al. [30] в исследованиях in vitro (культура Hep-2 клеток) и in vivo (n=30) обнаружили гипоэкспрессию этой микроРНК при раке гортани, что авторы
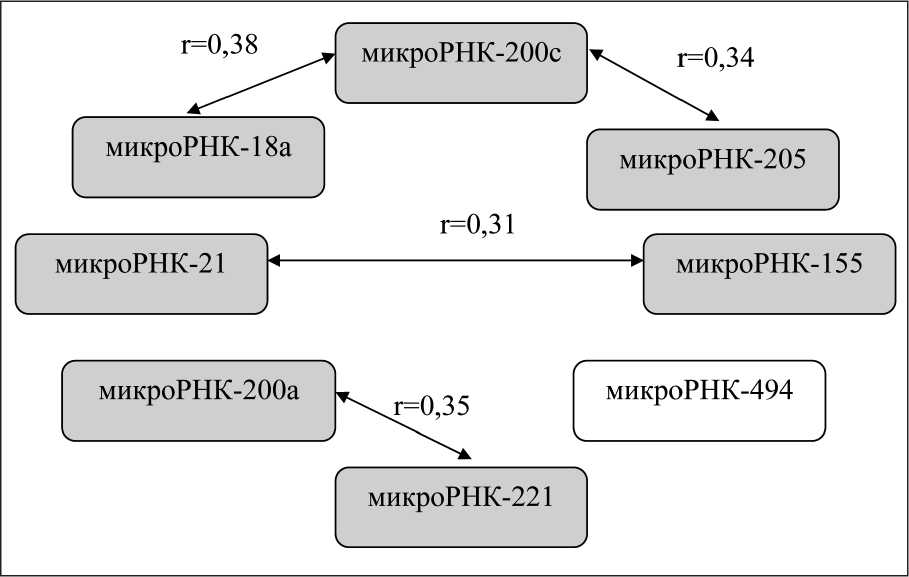
Рис. 1. Корреляционные связи между показателями экспрессии изучаемых микроРНК в опухолевой ткани у больных РГ
объясняют возможными влияниями выбора гистотипов опухоли, ее подтипов, микроокружения и регуляции генов-мишеней. Результаты исследования и данные литературы позволяют предполагать, что микроРНК-21, -155 и -205 играют онкогенную роль при опухолях гортани, и повышение их экспрессии связано с регуляцией различных процессов, задействованных в канцерогенезе.
Онкогенную функцию этих микроРНК подтверждает список генов-мишеней, экспрессию которых регулируют данные микроРНК. В настоящее время для микроРНК-21 валидированы следующие мишени: PDCD4 (programmed cell death protein 4), RECK (reversion inducing cysteine-rich protein with kazal motifs), maspin (mammary serine protease inhibitor), NFIB (nuclear factor I), TPM1 (Tropomyosin 1), SPRY2 (Sprouty2), PTEN (phosphatase and tensin homologue), Tap63 (transformation-related protein 63), лиганд Fas, Cdc25a (cyclin-dependent kinases 25), HNRPK (Heterogeneous nuclear ribonucleoprotein K). Все перечисленные гены-мишени микроРНК-21 принимают непосредственное участие в трансформации клеток [15, 20].
МикроРНК-155 принимает участие в регуляции нескольких ключевых процессов канцерогенеза через ингибирование генов-мишеней, задействованных в TGF-β/Smad-индуцированном эпителиально-мезенхимальном переходе, гликолизе, JNK2/STAT3 пути, NF-kB и AKT путях. Представлены убедительные доказательства того, что мишенями этой микроРНК являются: FOXO3a (Forkhead box O3), C/EBPβ (CCAAT-enhancer-binding proteins), которая, в свою очередь, активирует экспрессию hexokinase 2, STAT3 (Signal Transducer and Activator of Transcription), действие которого проявляется через репрессию гена SOCS1 (suppressor of cytokine signaling 1), CDC73 (Cell division cycle 73) и ряд других [7].
Относительно микроРНК-205 отмечается ее дуалистическая роль в канцерогенезе. Онкосу- прессорную роль микроРНК-205 осуществляет путем подавления пролиферации при ЗНО, репрессируя экспрессию таких генов-мишеней, как ErbB3 (Receptor tyrosine-protein kinase erbB-3), E2F1(Transcription factor E2F1) и гены MAPK- и AR-сигнальных путей, PKCε (Protein kinase C epsilon type), ZEB1/2, ММР2 и ММР9, VEGF-A (Vascular endothelial growth factor). Имеются сообщения, свидетельствующие и об онкогенной роли микроРНК-205, которая участвует в малигнизации, прогрессии опухоли и развитии устойчивости к противоопухолевой терапии, репрессируя гены PTEN, CYR61 (Cysteine-rich angiogenic inducer 61) и CTGF (connective tissue growth factor), SHIP2 (enzyme phosphatidylinositol-3,4,5-trisphosphate 5-phosphatase), DHFR (Dihydrofolate reductase) [28].
Наши результаты относительно гипоэкспрессии микроРНК-200а при раке гортани согласуются и дополняют полученные ранее данные других авторов, которые показали онкосупрессорную роль этой микроРНК в канцерогенезе опухолей разных локализаций [13]. Для микроРНК-200а валидированы такие гены-мишени, как: ZEB1 и 2 (Zinc finger E-box-binding homeobox 1 и 2) [25], ERRFI-1 (ERBB receptor feedback inhibitor 1) [1], CTNNB1 (cadherin-associated protein) [22] и ряд других. Эти данные позволили заключить, что микроРНК-200а играет ключевую роль в эпителиально-мезенхимальном переходе и процессе приобретения клеткой эмбрионального фенотипа [4, 19].
Результаты корреляционного анализа могут свидетельствовать о наличии связи между этими микроРНК, в основе которой лежит их вовлеченность в процессы внутриклеточной регуляции общих генов-мишеней. Такие гены-мишени найдены для всех связанных пар. МикроРНК-21 и -155 объединяет в первую очередь их взаимодействие с сигнальным белком и активатором транскрипции STAT3 [21]. На микроРНК-18а и -200с оказывает влияние репрограммирующий фактор c-Myc [10]. МикроРНК-200с и -205 играют определенную роль в эпителиально-мезенхимальном переходе и процессе приобретения клеткой эмбрионального фенотипа посредством регуляции экспрессии генов ZEB1 и ZEB2 [13]. Пары микроРНК-221 и 200а могут быть связаны через ген р27Kip1, который играет существенную роль в регуляции перехода от G1 к S-фазе клеточного цикла [11].
Заключение
Таким образом, установлены гиперэкспрессия онкогенных микроРНК-21, -155, 205 и гипоэк- спрессия онкосупрессорной микроРНК-200а в опухолевой ткани гортани пациентов с первичным диагнозом рака гортани, что согласуется с данными литературы, полученными для этих микроРНК на примере других локализаций. Эти данные позволяют предположить значительную роль микроРНК21, -155, -200а и -205 в канцерогенезе опухолей гортани, что, вероятно, связано с их участием в регуляции многих генов-мишеней и клеточных процессов, задействованных в малигнизации.
Работа выполнена в рамках программы повышения конкурентоспособности Национального исследовательского Томского государственного университета.
Список литературы Экспрессия микроРНК в опухолевой ткани у больных раком гортани
- Adam L., Zhong M., Choi W., Qi W., Nicoloso M., Arora A., Calin G., Wang H., Siefker-Radtke A., McConkey D. miR-200 expression regulates epithelial-to-mesenchymal transition in bladder cancer cells and reverses resistance to epidermal growth factor receptor therapy//Clin. Cancer Res. 2009. Vol. 15 (16). P. 5060-5072 DOI: 10.1158/1078-0432.CCR-08-2245
- Ambros V. The functions of animal microRNAs//Nature. 2004. Vol. 431. (7006). P. 350-355.
- Bartel D.P. MicroRNAs: genomics, biogenesis, mechanism, and function//Cell. 2004. Vol. 116 (2). P. 281-297.
- Bracken C.P., Gregory P.A., Kolesnikoff N., Bert A.G., Wang J., Shannon M.F., Goodall G.J. A double-negative feedback loop between ZEB1-SIP1 and the microRNA-200 family regulates epithelial-mesenchymal transition//Cancer Res. 2008. Vol. 68, № 19. P. 7846-7854 DOI: 10.1158/0008-5472.CAN-08-1942
- Cao P., Zhou L., Zhang J., Zheng F., Wang H., Ma D., Tian J. Comprehensive expression profiling of microRNAs in laryngeal squamous cell carcinoma//Head Neck. 2013. Vol. 35 (5). P. 720-728 DOI: 10.1002/hed.23011
- Chang S.S., Jiang W.W., Smith I., Poeta L.M., Begum S., Glazer C., Shan S., Westra W., Sidransky D., Califano J.A. MicroRNA alterations in head and neck squamous cell carcinoma//Intern. J. Cancer. 2008. Vol. 123 (12). P. 2791-2797 DOI: 10.1002/ijc.23831
- Chen Z., Ma T., Huang C., Hu T., Li J. The Pivotal Role of microRNA-155 in the Control of Cancer//Journal of Cellular Physiology. 2014. Vol. 229 (5). P. 545-550 DOI: 10.1002/jcp.24492
- Chu E.A., Kim Y.J. Laryngeal cancer: diagnosis and preoperative work-up//Otolaryngol. Clin. North Am. 2008. Vol. 41 (4). P. 673-695 DOI: 10.1016/j.otc.2008.01.016
- Cochrane D.R., Howe E.N., Spoelstra N.S., Richer J.K. Loss of miR-200c: a marker of aggressiveness and chemoresistance in female reproductive cancers//J. Oncol. 2009. Vol. 2010: 821717 DOI: 10.1155/2010/821717
- Concepcion C.P., Bonetti C., Ventura A. The miR-17-92 family of microRNA clusters in development and disease//Cancer J. 2012. Vol. 18(3). P. 262-267 DOI: 10.1097/PPO.0b013e318258b60a
- Di Martino M.T., Gullà A., Cantafio M.E.G., Lionetti M., Leone E., Amodio N., Guzzi P.H., Foresta U., Conforti F., Cannataro M. In vitro and in vivo anti-tumor activity of miR-221/222 inhibitors in multiple myeloma//Oncotarget. 2013. Vol. 4 (2). P. 242-255.
- Esquela-Kerscher A., Slack F.J. Oncomirs -microRNAs with a role in cancer//Nature Rev. Cancer. 2006. Vol. 6 (4). P. 259-269.
- Feng X., Wang Z., Fillmore R., Xi Y. MiR-200, a new star miRNA in human cancer//Cancer lett. 2014. Vol. 344 (2). P. 166-173 DOI: 10.1016/j.canlet.2013.11.004
- Gurtan A.M., Sharp P.A. The role of miRNAs in regulating gene expression networks//J. Mol. Biol. 2013. Vol. 425 (19). P. 3582-3600 DOI: 10.1016/j.jmb.2013.03.007
- Hong L., Han Y., Zhang Y., Zhang H., Zhao Q., Wu K., Fan D. MicroRNA-21: a therapeutic target for reversing drug resistance in cancer//Expert Opin. Ther. Targets. 2013. Vol. 17 (9) P. 1073-1080 DOI: 10.1517/14728222.2013.819853
- Hui A.B., Lenarduzzi M., Krushel T., Waldron L., Pintilie M., Shi W., Perez-Ordonez B., Jurisica I., O’Sullivan B., Waldron J. Comprehensive MicroRNA profiling for head and neck squamous cell carcinomas//Clin. Cancer Res. 2010. Vol. 16 (4). P. 1129-1139 DOI: 10.1158/1078-0432.CCR-09-2166
- Iorio M.V., Croce C.M. MicroRNA dysregulation in cancer: diagnostics, monitoring and therapeutics. A comprehensive review//EMBO Mol. Med. 2012. Vol. 4 (3). P. 143-159 DOI: 10.1002/emmm.201100209
- Iyevleva A.G., Kuligina E.S., Mitiushkina N.V., Togo A.V., Miki Y., Imyanitov E.N. High level of miR-21, miR-10b, and miR-31 expression in bilateral vs. unilateral breast carcinomas//Breast Cancer Res. Treat. 2012. Vol. 131 (3). P. 1049-1059 DOI: 10.1007/s10549-011-1845-z
- Korpal M., Lee E.S., Hu G., Kang Y. The miR-200 family inhibits epithelial-mesenchymal transition and cancer cell migration by direct targeting of E-cadherin transcriptional repressors ZEB1 and ZEB2//J. Biol. Chem. 2008. Vol. 283 (22). P. 14910-14914 DOI: 10.1074/jbc.C800074200
- Krichevsky A.M., Gabriely G. miR-21: a small multi-faceted RNA//J. Cell. Mol. Med. 2009. Vol. 13 (1). P. 39-53 DOI: 10.1111/j.1582-4934.2008.00556.x
- Löffler D., Brocke-Heidrich K., Pfeifer G., Stocsits C., Hackermüller J., Kretzschmar A.K., Burger R., Gramatzki M., Blumert C., Bauer K., Cvijic H., Ullmann A.K., Stadler P.F., Horn F. Interleukin-6 -dependent survival of multiple myeloma cells involves the Stat3-mediated induction of microRNA-21 through a highly conserved enhancer//Blood. 2007. Vol. 110 (4). P. 1330-1333.
- Mongroo P.S., Rustgi A.K. The role of the miR-200 family in epithelial-mesenchymal transition//Cancer Biol. Ther. 2010. Vol. 10 (3). P. 219-222.
- Nikitina E., Urazova L., Stegny V. MicroRNAs and human cancer//Exp. Oncol. 2012. Vol. 34 (1). P. 2-8.
- O’Donnell K.A., Wentzel E.A., Zeller K.I., Dang C.V., Mendell J.T. c-Myc-regulated microRNAs modulate E2F1 expression//Nature. 2005. Vol. 435 (7043). P. 839-843.
- Park S.-M., Gaur A.B., Lengyel E., Peter M.E. The miR-200 family determines the epithelial phenotype of cancer cells by targeting the E-cadherin repressors ZEB1 and ZEB2//Genes Dev. 2008. Vol. 22. (7). P.894-907 DOI: 10.1101/gad.1640608
- Peltier H.J., Latham G.J. Normalization of microRNA expression levels in quantitative RT-PCR assays: identification of suitable reference RNA targets in normal and cancerous human solid tissues//RNA. 2008. Vol. 14 (5). P. 844-852 DOI: 10.1261/rna.939908
- Pfaffl M.W. A new mathematical model for relative quantification in real-time RT-PCR//Nucleic Acids Res. 2001. Vol. 29 (9). P. e45-e45.
- Qin A.-Y., Zhang X.-W., Liu L., Yu J.-P., Li H., Wang S.-Z.E., Ren X.-B., Cao S. MiR-205 in cancer: An angel or a devil?//Eur. J. Cell Biol. 2013. Vol. 92. (2). P. 54-60.
- Ren J., Zhu D., Liu M., Sun Y., Tian L. Downregulation of miR-21 modulates Ras expression to promote apoptosis and suppress invasion of Laryngeal squamous cell carcinoma//Eur. J. Cancer. 2010. Vol. 46 (18). P. 3409-3416 DOI: 10.1016/j.ejca.2010.07.047
- Tian L., Zhang J., Ge J., Xiao H., Lu J., Fu S., Liu M., Sun Y. MicroRNA-205 suppresses proliferation and promotes apoptosis in laryngeal squamous cell carcinoma//Med. Oncol. 2014. Vol. 31 (1). P. 785 DOI: 10.1007/s12032-013-0785-3
- Zamore P.D., Haley B. Ribo-gnome: the big world of small RNAs//Science. 2005. Vol. 309 (5740). P. 1519-1524.
- Zhou J.-J., Zheng S., Sun L.-F., Zheng L. MicroRNA regulation network in colorectal cancer metastasis//World J. Biol. Chem. 2014. Vol. 5 (3). P. 301 DOI: 10.4331/wjbc.v5.i3.301


