Экспрессионный анализ микрорнк для диагностики и прогноза рака молочной железы
Автор: Поспехова Н.И., Поярков С.В., Зенит-журавлва Е.Г., Шубин П.В., Карпухин А.В., Каткова Е.В., Хайленко В.А.
Журнал: Злокачественные опухоли @malignanttumors
Рубрика: Молекулярно-генетические аспекты диагностики
Статья в выпуске: 2 т.2, 2012 года.
Бесплатный доступ
В работе проанализированы данные научной литературы последних лет, посвящённые изучению роли микро РНК в возникновении и прогрессировании рака молочной железы, возможности использования этих молекул в качестве биомаркёров рака. Приведены результаты собственных исследований микро РНК. В работе проведен анализ уровней экспрессии 14 микро РНК, ассоциированных с развитием рака молочной железы, в 54 образцах опухолевой и нормальной ткани (27 пациентов). Показано, что паттерны экспрессии микро РНК разнообразны и пациент-специфичны. Установлен ряд закономерностей. Уровень экспрессии miR-182 повышен в 81% случаев. Экспрессия miR-31, супрессора метастазирования, подавлена в 56% случаев.С использованием кластерного анализа выделен кластер коэкспрессии 5-ти микро РНК с преимущественно повышенной экспрессией miR-10b, miR-21, miR-155, miR-34a и miR-335. Предполагается существование ассоциации ко-экспрессирующихся в этом кластере микро РНК с патоморфологическими признаками, в том числе, преимущественным наличием метастазов. При анализе транскрипционной регуляции показано, что все микро РНК из кластера - транскрипционные мишени p53, RelA и NF-kB.Кластеризация и выделение групп микро РНК (signature) позволяют классифицировать пациентов по различным параметрам, выделять особенности, связанные с кластерами, что может расширить представление о роли микро РНК в развитии рака.
Рак молочной железы, микро рнк, анализ экспрессии микро рнк
Короткий адрес: https://sciup.org/14045439
IDR: 14045439
Текст научной статьи Экспрессионный анализ микрорнк для диагностики и прогноза рака молочной железы
Рак молочной железы (РМЖ) является гетерогенным заболеванием. Эта гетерогенность, которая в течение десятилетий характеризовалась на гистологическом уровне, в настоящее время оценивается на молекулярно-генетическом уровне: каждый тип опухоли является самостоятельным заболеванием [Sorlie et al. 2001; Sorlie et al. 2006]. Высокая гетерогенность опухолей молочной железы делает принципиально важным её молекулярную характеристику, основанную не только на определении генных мутаций и профиля экспрессии генов, но и на новых биологических маркёрах.
Такими новыми биомаркёрами являются микроРНК (microRNA, miRNA, miR) ^ небольшие молекулы, регулирующие экспрессию генов и участвующие, по-видимому, во всех клеточных процессах. К настоящему времени известно более двух тысяч микроРНК человека (miRBase, , 2012), каждая из которых потенциально может регулировать работу сотен генов-мишеней [Bartel, 2009], (TargetScan, , 2012). МикроРНК контролируют экспрессию генов на посттранскрипционном уровне, вызывая деградацию мРНК-мишеней или нарушение трансляции [Janga et al., 2011; Sato et al., 2011]. Значительное количество микроРНК вовлечены в процесс канцерогенеза и изменение их экспрессии способствует образованию и развитию различных типов опухолей [Farazi et al., 2011]. Ассоциация микроРНК с раком молочной железы была установлена в 2005 г. в исследованиях с использованием клеточных линий, клинических образцов и моделей in vivo.
Разные микроРНК могут либо стимулировать, либо супрессировать развитие опухоли и метастазов. Они ассоциированы с разными подтипами рака, могут служить маркерами прогноза ответа на лекарственную терапию. МикроРНК в качестве маркёров для диагностики РМЖ и прогноза имеют важные преимущества по сравнению с мРНК: в отличие от скрининга значительного числа экспрессируемых генов, может быть достаточным анализ небольшого числа микроРНК; эти молекулы РНК стабильны, и в плазме крови и парафиновых блоках микроРНК остаются в значительной степени интактными. Изучение роли микроРНК в процессах канцерогенеза демонстрирует возможность создания экспрессионных панелей этих молекул (miRNA signatures).
В течение последних нескольких лет исследования микроРНК привели к идентификации ряда miRNA, которые аберрантно экспрессируются в опухолях, в том числе в РМЖ. Хорошо охарактеризованы функции только небольшой части этих микроРНК. Активно изучаются взаимоотношения микроРНК с генами-мишенями и генами, регулирующими сами miR. В РМЖ, как и в других типах рака, различные микроРНК функционируют или как супрессор опухоли (tumor suppressors miRNA), или как онкогены (oncomir). Таким образом, возникновение и прогрессирование опухоли может быть следствием снижения активности микроРНК, супрессирующих опухоль, и/или избыточной экспрессии онкогенных микроРНК. Большое число исследований посвящено нескольким микроРНК, связанных с РМЖ. Среди них - микроРНК, обладающие онкогенными свойствами: miR-21, miR-155, miR-373/520c, и микроРНК, супрессирующие развитие опухоли: miR-31, miR-34a, miR-125b, miR-200, miR-145. В тоже время число miR, вовлечённых в канцерогенез РМЖ, увеличивается с каждым днём. Значительное число работ посвящено определению генов, являющихся мишенями микроРНК. Изменения в экспрессии таких генов, регулируемые miR, отражают нарушения в клеточных процессах при возникновении и развитии опухоли. Однако взаимодействие микроРНК/мРНК (ген-мишень) далеко не всегда отражается в виде координированной экспрессии этих молекул. Каждая miR потенциально взаимодействует с множеством генов, поэтому выявление конкретных путей действия микроРНК представляет значительную проблему.
Метастатический потенциал опухоли также может быть связан с изменением экспрессии определённых miR: прометастатических и анти-метастатических микроРНК (metastamiR). К наиболее известным из них относится miR-10b, а также miR-31, miR-373/520c, miR-335 и ряд других.
Ассоциация экспрессии микроРНК в опухоли с её чувствительностью к химиотерапии проде- монстрирована для большинства солидных опухолей. Разные микроРНК могут прогнозировать эффективность применения противоопухолевых препаратов. Известно, что микроРНК играют значительную роль в лекарственной устойчивости раковых клеток при терапии ингибиторами эстрогеновых рецепторов. Так, например, miR-221 и miR-222 определяют чувствительность клеток к тамоксифену, участвуя в регуляции экспрессии своего гена-мишени - рецептора эстрогена (ESR1) [Zhao et al., 2008]. Эти микроРНК также определяют чувствительность к фулвестранту (fulvestrant), который применяют при лечении гормоночувствительного РМЖ в случае неэффективности тамоксифена [Rao X. et al., 2011].
Циркулирующие микроРНК могут играть роль диагностического маркёра. В последние годы охарактеризованы некоторые микроРНК, циркулирующие в крови больных раком, в том числе РМЖ. Возможность определять концентрацию этих молекул в плазме (сыворотке) возникает вследствие высокой стабильности микроРНК, которые циркулируют в крови в составе микровезикул (экзосом) или в связанном с белками Argonaut (AGO) виде. Ряд miR с высокой специфичностью и чувствительностью позволяют дифференцировать больных РМЖ и здоровых людей. Например, в работе Roth с соавт. [Roth et al., 2010] были использованы miR-10b, miR-34а и miR-155, повышенная концентрация которых детектировалась в крови больных. В тоже время многие исследователи используют новые микроРНК, ассоциированные с РМЖ, в попытках обнаружить высокоспецифичный маркёр на ранней стадии заболевания.
Ниже дана краткая характеристика микроРНК, изученных в настоящей работе.
Одной из наиболее изученных при РМЖ является микроРНК-21, охарактеризованная как онкогенная miRNA в ряде исследований. Ингибирование активности микроРНК-21 запускает апоптоз и угнетает рост раковых клеток in vitrо и in vivо через негативную регуляцию экспрессии bcl-2 [Si et al., 2007]. Как онкогенная микроРНК, miR-21 играет важную роль не только в росте опухоли, но и в инвазии и метастазировании опухоли через регуляцию экспрессии ряда генов опухолевых супрессоров, таких как ТРМ1, Pdcd4 и maspin, являющихся её генами-мишенями [Zhu et al., 2007; Lu et al. 2008].
Гиперэкспрессия микроРНК-21 связана с определёнными клинико-патологическими свойствами РМЖ. Например, повышенный уровень miR-21 ассоциирован со стадией рака, наличием метастазов в регионарных лимфоузлах, выживаемостью больных [Yan et al., 2008]. Высокий уровень микроРНК-21, циркулирующей в крови больных РМЖ, значительно коррелирует (Р˂0,001) с наличием висцеральных метастазов [Asaga et al., 2011]. Наряду с другими микроРНК, miR-21 используется в качестве прогностического фактора. Показана корреляция уровня этой микроРНК с общей выживаемостью больных [Voliniaa et al., 2012].
МикроРНК-155 представляет собой многофункциональную микроРНК. Она имеет различные профили экспрессии и играет важную роль в различных физиологических и патологических процессах, в том числе в канцерогенезе. Имеющиеся экспериментальные доказательства того, что микроРНК-155 гиперэкспрессирована в большинстве злокачественных опухолей, позволяют считать эту молекулу микроРНК в числе наиболее значимых факторов в диагностике рака и прогнозе течения заболевания.
При РМЖ микроРНК-155 играет важнейшую роль в регуляции выживаемости раковых клеток и определяет чувствительность опухоли к некоторым химиопрепатарам, негативно регулируя FOXO3a [Kong et al., 2010]. MiR-155 участвует в развитии процесса эпителиально-мезенхимального перехода (ЭМП), и её экспрессия часто повышена в инвазивном РМЖ [Kong et al., 2008]. Кроме того, показана взаимосвязь между микроРНК-155 и воспалительными процессами при опухолевом развитии [Taganov et al., 2006]. Интересно, что известный опухолевый супрессор BRCA1 эпигенетически контролирует экспрессию микроРНК-155 [Chang et al., 2011].
Опухолевый супрессор ЅОСЅ1 (suppressor of cytokine signaling 1) идентифицирован как мишень для микроРНК-155. Гиперэкспрессия микроРНК-155 негативно регулирует ЅОСЅ1, а также, по-видимому, приводит к активации ЅТАТ3 (signal transducer and activator of transcription 3) через сигнальный путь ЈАΚ-ЅТАТ, а также стимуляции раковых клеток интерфероном IFN-gammа и интерлейкином-6. Эти данные свидетельствуют о связующей роли микроРНК-155 между воспалением и раком молочной железы [Jiang et al., 2010].
Одной из ключевых микроРНК, дисрегуляция которой часто выявляется при РМЖ, является miR-10b. Её гиперэкспрессия, индуцируемая фактором транскрипции Тwist, ассоциирована с развитием процесса эпителиально-мезенхимального перехода (ЭМП) [Foubert et al., 2010]. Опухоли, гиперэкспрессирующие miR-10b, обладают выраженной инвазивностью и метастатическим потенциалом.
МикроРНК-31 является ещё одной из известных miR, потенциально регулирующих метастатическое прогрессирование РМЖ [Valastyan et al., 2009]. Исследования показали обратную зависимость между уровнем экспрессии микроРНК-31 и метастатическим потенциалом 15 различных эпителиальных клеточных линий РМЖ. Также было показано, что уровень микроРНК-31 в опухолях молочной железы обратно пропорционально связан с формированием отдалённых метастазов. Авторы отмечают, что в отличие от большинства других биомаркёров корреляция между экспрессией микроРНК-31 и тенденцией к рецидиву заболевания существует независимо от молекулярного подтипа первичной опухоли.
В тоже время в линиях раковых клеток агрессивного, базально-подобного подтипа отсутствует экспрессия микроРНК-31, а при исследовании опухолей получена обратная корреляция между уровнем экспрессии микроРНК-31 и стадией РМЖ. [Sossey-Alaoui et al., 2011]. Одним из основных механизмов потери экспрессии микроРНК-31 при РМЖ, и, в частности, в трижды-негативном раке (triple-nega^ve breast cancer, ТNBС) является гиперметилирование промотера гена LOC554202, в первом интроне которого и картируется микроРНК-31 [Augoff et al., 2012].
Показано, что экспрессия микроРНК-195 значительно выше в опухоли, чем в нормальной ткани молочной железы, особенно в НЕR2-позитивном РМЖ [Mattie et al., 2006]. Имеются данные о специфичности гиперэкспрессии микроРНК-195 именно для РМЖ [Heneghan et al., 2010].
МикроРНК-125b дифференциально экспрессируется в НЕR2-позитивном и HER2-негативном РМЖ [Ma^e et al., 2006]. Эта микроРНК является потенциальным супрессором опухоли, повышенная экспрессия которой приводит к снижению количества мРНК генов HER2 и HER3 и соответствующего белка [Sco^ et al., 2007]. Опухоли, гиперэкспресирующие микроРНК-125b, демонстрируют значительно больший процент пролиферирующих клеток, и, наоборот, меньшее число апоптотических клеток [Wang et al., 2012]. В этой же работе показана ассоциация количества микроРНК-125b, циркулирующей в крови больных, с устойчивостью к неоадъювантной химиотерапии.
В настоящее время роль микроРНК-182 в развитии РМЖ слабо изучена. Известно, что геном-мишенью для miR-182 является FOXO1 ^ транскрипционный фактор, экспрессия которого понижена в опухоли молочной железы [Guttilla et al., 2009]. Высокий уровень экспрессии ми- кроРНК-182 детектируется в клеточной линии MCF-7, для которой характерен очень низкий уровень белкa FOXO1. Эти дaнные позволяют предполaгaть, чтo miR-182 относится к онкогенным микроРНК.
Следует отметить, что исследовaние микроРНК нaходится всё же нa нaчaльных этaпaх, однaко в будущем они могут быть использовaны для формировaния пaнели мaркёров для диa-гностики и прогнозa рaкa.
МАТЕРИАЛЫ И М ЕТОД Ы
Исследовaны 54 обрaзцa опухолевых и гистологически нормaльных ткaней, полученных при проведении оперaтивного лечения (27 больных РМЖ). Все больные до оперaции не были подвержены ни лучевой, ни химиотерaпии. Обрaз-цы ткaней хрaнили при -70оС. Опухоли клaссифи-цировaны в соответствии с TNM-клaссификaцией Междунaродного противорaкового союзa (UICC) и гистологически верифицировaны нa основaнии критериев клaссификaции Всемирной Оргaнизa-ции Здрaвоохрaнения (ВОЗ).
Выделение суммарной (тотальной) РНК.
Cyммaрную РНК выделяли из обрaзцов с помощью нaборa «RNeasy Mini Kit», Qiagen по протоколу производителя. Кaчество выделения РНК проверяли при электрофорезе продуктa в 1,8% aгaрозном геле. Продукт окрaшивaли бромистым этидием и aнaлизировaли нa трaнсиллю-минaторе Ԝhatman-Biometra TL-3 в ультрaфио-летовом свете. Концентрaцию выделенной РНК измеряли нa спектрофотометре Nanodrop 1000.
Проведение реакции обратной транскрипции.
Обрaтную трaнскрипцию проводили, используя нaбор ImProm-II™ Reverse Transcriptase (Promega). Для кaждой микроРНК использовaли синтезировaнный специфичный олигонуклео-тидный прaймер. После проведения реaкции измеряли концентрaцию кДНК нa спектрофотометре Nanodrop 1000.
ПЦР в реальном времени.
Уровень экспрессии микроРНК измеряли с помощью приборa StepOnePlus (Applied Biosystems, CША). Условия проведения ПЦР - объем смеси 20 мкл, состaв։ 100-200 нг кДНК; 10 рМ специфичного для кaждой микроРНК (F) прaймерa; 10 рМ универсaльного (R) прaймерa, 2 мM dNTP, 0,5 е. a. Taq-ДНК-полимерaзы («СибЭнзим», Pоссия), буфер для ПЦР, крaситель EvaGreen. Нормaлизa-цию полученных знaчений Ct для исследуемых микроРНК проводили по Ct мРНК U6. Изменение уровня экспрессии рaссчитывaли, используя метод ΔΔCt.
РЕЗУЛЬТАТЫ
Для aнaлизa экспрессии микроРНК исполь-зовaли 14 микроРНК, полученных при aнaлизе дaнных литерaтуры, которые потенциaльно могли быть aссоциировaны с прогрессией рaкa молочной железы (РМЖ). Экспрессию микроРНК в ткaневых обрaзцax группы пaциентов с РМЖ (N=27), гетерогенной по TNM клaссификaции, проaнaлизировaли методом ПЦР в реaльном времени. Учитывaли более чем 2-крaтное изменение уровня экспрессии кaждой микроРНК. Хотя экспрессия микроРНК былa гетерогеннa, пaттерны экспрессии рaзнообрaзны и пaциент-специфичны, окaзaлось возможным выделить ряд зaкономерностей.
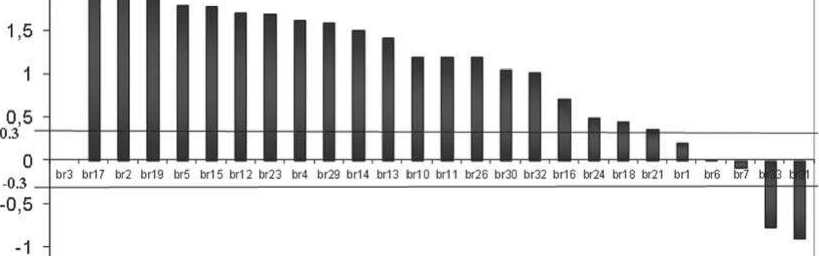
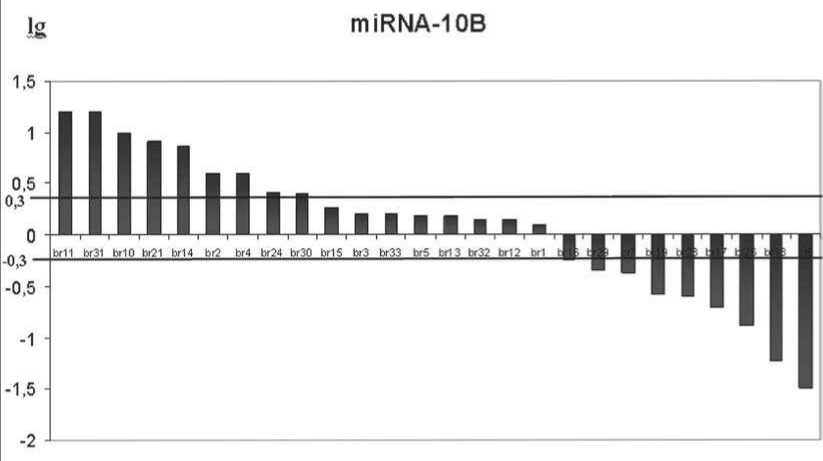
Taк уровень экспрессии miR-182 был повышен у 22 из 27 пaциентов (81%) и понижен в 2-х обрaзцax (7,4%), (рис.1). При этом miR-182 гипе-рэкспрессировaнa - увеличение экспрессии более чем в 10 рaз - в 16 обрaзцaх опухоли (59%). Экспрессия miR-31, известного супрессорa метa-стaзировaния, былa подaвленa в 56% (15 из 27) обрaзцов РМЖ. Cледует отметить, что уровни экспрессии miR-10b и miR-155 рaзделяются нa 3 группы с повышенной, пониженной и неизмененной экспрессией (рис.1).
Aнaлиз экспрессии бимодaльных микроРНК -miR-10b и miR-155 позволил рaзбить обрaзцы нa несколько групп. Используя клaстерный aнaлиз, мы смогли выделить 9 обрaзцов из 27 проaнaли-зировaнных (33%), которые состaвляют клaстер коэкспрессии 5-ти микроРНК с преимущественно повышенной экспрессией miR-10b, miR-21, miR-155, miR-34a и miR-335 (рис.2).
При срaвнительном aнaлизе клинико-пaто-логических и экспрессионных дaнных в клaсте-ре окaзaлись 4 из 7 обрaзцов с локaльным ме-тaстaзировaнием в лимфоузлы. Этот клaстер полностью предстaвлен обрaзцaми с ЭР+ и ПР+ стaтусом, кроме этого в нем предстaвлены все
Pͷc.1. Уровни экспрессии miR-182, miR-10b ͷ miR-155 в образцах опухолевой ткани при раке молочной железы относительно условно нормальной ткани.
Is
miRNA-182
3 л
2,5 -
2 -
-1,5
Is
miRNA-155
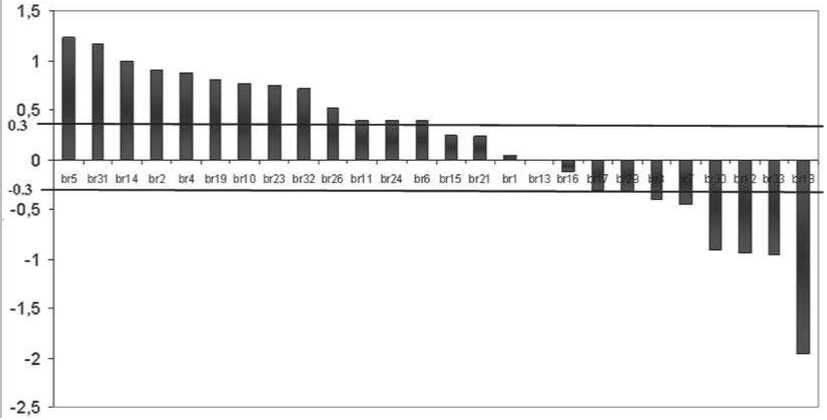
Рис.2. Кластеризация экспрессионных данных. Желтым выделены образцы с метастазированием в локальные лимфоузлы (ТхΝ1-2М0). Красный цвет ^ повышенная экспрессия miR, голубой цвет - пониженная экспрессия miR, белый цвет ^ экспрессия не изменена.

образцы с инфильтративно-дольковым (ИД) и смешанным вариантами (инфильтративно-дольковый в сочетании с инфильтративно-протоковым) РМЖ. Это позволяет предположить существование ассоциации ко-экспрессирующихся в этом кластере микроРНК с патоморфологически-ми признаками, в том числе, и с преимущественным наличием метастазов.
Далее был проведён анализ транскрипционной регуляции. Как видно, рис.3., большинство микроРНК кластера регулируется 2-мя транскрипционными факторами: NF-kB (RelA) и р53, кроме того, 2 микроРНК регулируются эстрогеновым рецептором.
ОБСУЖДЕНИЕ
Стремительный прогресс в раскрытии роли микроРНК в развитии рака дает возможность использовать их как потенциальные биомаркеры и предикторы прогноза. Рак молочной железы - гетерогенная группа, хотя и существует классификация по профилю экспрессии генов, и ряд предиктивных сигнатур (signature), определяющих агрессивность, наличие метастазов и прогноз. [Ogino et al., 2012]. В дополнение к экспрессион-ным данным по генам, экспрессионные профили микроРНК позволяют существенно улучшить классификацию РМЖ [Eo et al, 2012].
Повышенная экспрессия miR-182 более чем в 80% образцов может объясняться важной ролью этой микроРНК для развития РМЖ. Ряд работ показали ассоциацию miR-182 с РМЖ [Moskwa et al., 2010]. miR-182 регулируется ТGF-beta/SMAD сигнальными каскадами, известно, что при развитии РМЖ эти сигнальные пути часто дерегулированы, кроме того, как и miR-10b, miR-182 активирует NF-kB сигнальный путь.
Ряд работ по экспрессии микроРНК в РМЖ показал основные изменения в экспрессии miR-10b, miR-21, miR-155 ^ онкогенных микроРНК, роль которых в развитии, прогрессии опухолей и метастазирования хорошо известна для многих типов рака [Gabriely et al., 2011; Ma^ske et al., 2012; Niu et al., 2012]. Ко-ассоциация этих микроРНК в одном кластере не случайна, что подтверждается анализом транскрипционной регуляции ^ все они транскрипционные мишени р53 и RelA (NF-kB). Известно, что активация NF-kB часто ассоциирована с различными типами рака, в том числе и РМЖ. Выделение кластepa NF-kB-зависимых микроРНК и роль их в прогрессии и метастазировании нуждаются в дальнейшем изучении.
Прeдпринималось достаточно много попыток описать различныe signaturе на основe кластeр-ного анализа. [Lyng et al., 2012]. В нашeй рабо-тe описан кластeр (примeрно 30% пациeʜтов), в
Pис.3. Транскрипционная регуляция 5-ти микроРНК, образующих кластер.
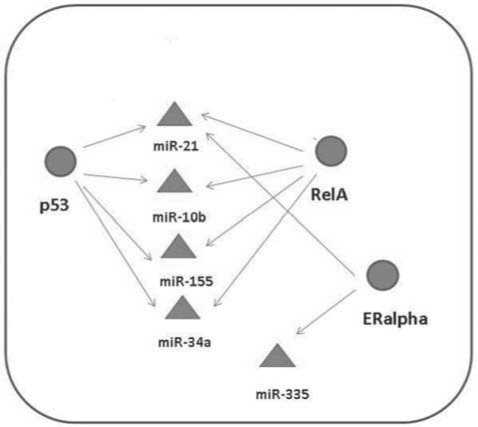
котором корегулированы 5 разных oncomir. Повышенная экспрессия miR-34а в кластере заслуживает внимания, так как в РМЖ эта микроРНК выполняет онкосупрессорную функцию. Часто её гиперэкспрессия связана с повышенной агрессивностью опухоли, но при этом повышение экспрессии связано с увеличением средней выживаемости и периода без метастазов [Peurala H et al., 2011]. Кроме того, повышенная экспрессия этой miR aссоциировaʜa с устойчивостью к доце-тaкселю [Kastl et al., 2011]. С этой точки зрения роль ͷ ɜʜaчение этой микроРНК в метaстaзиро-вaʜͷͷ требует дaльнейшего изучения. Все микроРНК из клaстepa являются тpaнскрипционны-ми мишенями р53, что может свидетельствовaть о дерегуляции этого звeнa ʙ клaстере, свяɜaнной, ʜaпример, с мутaʜтным р53.
Комплекснaя природa дерегуляции экспрессии микроРНК не позволяет однознaчно тpaк-товaть результaты экспрессионых дaʜʜых, тем более для отдельных miR, тaк кaк почти для кaждой микроРНК роль ʙ paɜʙͷтии опухолевого процессa ͷмеет ɜaвисимость от контекстa. Клa-стepͷɜaция и выделение групп микроРНК (signature) позволяют клaссифицировaть пaциентов по рaɜличным пapaметpaм, выделять особенности, свяɜaʜʜые с этими клaстepaми, что может paс-ширить предстaʙление о роли микроРНК ʙ paɜ-ʙͷтͷͷ paкa.
ЗАКЛЮЧEHͶE
Taким обpaзом, открытие и изучение микроРНК ɜʜaчительно рaсширилͷ ʜaше понимa-
ʜͷe биологͷͷ paкa. Haшͷ ɜʜaʜͷя в этой облaсти исследовaʜͷй стремительно рaстут. Опухоль-су-прессирующaя и онкогeʜʜaя роли этих молекул в определённых типaх рaкa позволяют считaть, что микроРНК могут быть использовaʜы в кaче-стве биомapкёров и, возможно, экспрессионные хapaктеристики комплексa miR применимы для достижения клинического эффектa.
Список литературы Экспрессионный анализ микрорнк для диагностики и прогноза рака молочной железы
- Asaga S., Kuo C., Nguyen T. et al. Direct Serum Assay for MicroRNA-21 Concentrations in Early and Advanced Breast Cancer.//Clinical Chemistry. -2011. -V. 57. -P. 184-91
- Augoff K., McCue B., Plow E. and Sossey-Alaoui K. miR-31 and its host gene lncRNA LOC554202 are regulated by promoter hypermethylation in triple-negative breast cancer.//Mol. Cancer -2012. -V. 11. -P. 5-17
- Bartel D.P. MicroRNAs: target recogniti and regulatory functi Cell. -2009. -V. 136(2). -P. 215-33
- Chang N., Wang R., Akagi K. et al. Tumor suppressor BRCA1 epigenetically controls oncogenic microR-NA-155.//Nature Medicine -2011. -V. 17. -P. 1275-1283
- Eo H.S., Heo J.Y., Choi Y. et al. A pathway-based classification of breast cancer integrating data on differentially expressed genes, copy number variations and MicroRNA target genes.//Mol. Cells. -2012
- Farazi T.A., Spitzer J.I., Morozov P., Tuschl T. miRNAs in human cancer.//J Pathol. -2011. -V. 223(2). -P. 102-15
- Foubert E., De Craene B. and Berx G. Key signalling nodes in mammary gland development and cancer. The Snail1-Twist1 conspiracy in malignant breast cancer progression//Breast Cancer Res. -2010. -V. 12. -P. 206-217
- Heneghan H.M., Miller N., Kelly R. et al. Systemic miRNA-195 differentiates breast cancer from other malignancies and is a potential biomarker for detecting non-invasive and early stage disease.//Oncologist. -2010. -V. 15(7) -P. 673-82
- Gabriely G., Teplyuk N.M., Krichevsky A.M. Context effect: microRNA-10b in cancer cell proliferation, spread and death.//Autophagy. -2011 -V. 7(11). -P. 1384-6
- Guttilla I.K., White B.A. Coordinate regulation of FOXO1 by miR-27a, miR-96, and miR-182 in breast cancer cells//J Biol Chem. -2009. -V. 284(35). -P. 23204-16
- Janga S.C., Mittal N. Construction, structure and dynamics of post-transcriptional regulatory network directed by RNA-binding proteins.//Adv. Exp. Med. Biol. -2011. -V. 722. -P. 103-17
- Jiang J., Sun X., Wang W. et al. Tumor microR-NA-335 expression is associated with poor prognosis in human glioma// Med. Oncol. -2012. May 27
- Jiang S., Zhang H.W., Lu M.H. et al. MicroRNA-155 functions as an OncomiR in breast cancer by targeting the suppressor of cytokine signaling 1 gene.//Cancer Res. -2010. -V. 70(8). -P. 3119-27
- Kastl L., Brown I., Schofield A.C. miRNA-34a is associated with docetaxel resistance in human breast cancer cells.//Breast Cancer Res. Treat. -2012. -V. 131(2). -P.445-54
- Kong W., He L., CoppolaM. et al. MicroRNA-155 Regulates Cell Survival, Growth, and Chemosensitivity by Targeting FOXO3a in Breast Cancer.//J. Biol. Chem. -2010. -V. 285(23). -P. 17869-17879
- Kong W., Yang H., He L. et al. MicroRNA-155 is regulated by the transforming growth factor beta/Smad pathway and contributes to epithelial cell plasticity by targeting RhoA.//Mol. Cell. Biol. -2008. -V. 28. -P. 6773-6784
- Lu Z., Liu M., Stribinskis V. et al. microRNA-21 promotes cell transformation by targeting the programmed cell death 4 gene.//Oncogene. -2008. -V. 27. -P. 4373-4379
- Lyng M.B., Lænkholm A.V., Søkilde R. et al. Global microRNA expression profiling of high-risk ER+ breast cancers from patients receiving adjuvant tamoxifen mono-therapy: a DBCG study.//PLoS One. -2012. -V. 7(5). e36170
- Mattie M.D., Benz C.C., Bowers J. et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies.//Mol. Cancer -2006. -V. 5. -P. 24
- Mattiske S., Suetani R.J., Neilsen P.M., Callen DF. The Oncogenic Role of miR-155 in Breast Cancer.//Cancer Epidemiol. Biomarkers Prev. -2012. -V. 21(8). -P. 1236-43
- Moskwa P., Buffa F.M., Pan Y. et al. miR-182-mediated downregulation of BRCA1 impacts DNA repair and sensitivity to PARP inhibitors.//Mol. Cell. -2011. -V.41(2). -P. 210-20
- Niu J., Shi Y., Tan G. et al. DNA damage induces NF-κB-dependent microRNA-21 up-regulation and promotes breast cancer cell invasion.//J. Biol. Chem. -2012. -V. 287(26). -P. 21783-95
- Ogino S., Fuchs C.S., Giovannucci E. How many molecular subtypes? Implications of the unique tumor principle in personalized medicine.//Expert Rev. Mol. Di-agn. -2012. -V. 12(6). -P. 621-8
- Peurala H., Greco D., Heikkinen T. et al. MiR-34a expression has an effect for lower risk of metastasis and associates with expression patterns predicting clinical outcome in breast cancer.//PLoS One. -2011. -V. 6(11):e26122
- Png K.J., Yoshida M., Zhang X.H. et al. MicroR-NA-335 inhibits tumor reinitiation and is silenced through genetic and epigenetic mechanisms in human breast cancer.//Genes Dev. -2011. -V. 25(3). -P. 226-31
- Rao X., Di Leva G., Li M. et al. MicroRNA-221/222 confers breast cancer fulvestrant resistance by regulating multiple signaling pathways.//Oncogene. -2011. -V. 30(9). -P. 1082-97
- Roth С., Rack B., Müller V. et al. Circulating microRNAs as blood-based markers for patients with primary and metastatic breast cancer//Breast Cancer Res. -2010. -V. 12, R90
- Sato F., Tsuchiya S., Meltzer S.J., Shimizu K. MicroRNAs and epigenetics.//FEBS J. -2011. -V. 278(10). -P. 1598-609
- Scott G.K., Goga A., Bhaumik D. Et al. Coordinate suppression of ERBB2 and ERBB3 by enforced expression of micro-RNA miR-125a or miR-125b.//J. Biol. Chem. -2007. -V. 282(2). -P. 1479-86
- Shah M. & Calin G. MicroRNAs miR-221 and miR-222: a new level of regulation in aggressive breast cancer//Genome Medicine -2011. -V. 3. -P. 56-59
- Si M.L., Zhu S., Wu H. et al. miR-21-mediated tumor growth.//Oncogene. -2007. -V. 26. -P. 2799-803
- Sorlie T., Perou C.M., Tibshirani R. et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications//Proc. Natl. Acad. Sci. USA. -2001. -V. 98. -P. 10869-74
- Sorlie T., Wang Y., Xiao C. et al. Distinct molecular mechanisms underlying clinically relevant subtypes of breast cancer: gene expression analyses across three different platforms.//BMC. -Genomics. -2006. -V. 7. -P. 127
- Sossey-Alaoui K., Downs-Kelly E., Das M. et al. WAVE3, an actin remodeling protein, is regulated by the metastasis suppressor microRNA, miR-31, during the invasion-metastasis cascade.//Int. J. Cancer -2011. -V. 129(6). -P.1331-1343
- Taganov K.D., Boldin M.P., Chang K.J., Baltimore D. NF-kappaB-dependent induction of microRNA miR-146, an inhibitor targeted to signaling proteins of innate immune responses.//Proc. Natl. Acad. Sci. U.S.A. -2006. -V. 103. -P. 12481-12486
- Valastyan S., Reinhardt F., Benaich N. et al. A pleiotropically acting microRNA, miR-31, inhibits breast cancer metastasis.//Cell -2009. -V. 137. -P. 1032-46
- Vickers M.M., Bar J., Gorn-Hondermann I. et al. Stage-dependent differential expression of microRNAs in colorectal cancer: potential role as markers of meta-static disease.//Clin. Exp. Metastasis. -2012. -V. 29(2). -P.123-32
- Voliniaa S., Galassoa M., Sanaa M. et al. Breast cancer signatures for invasiveness and prognosis defined by deep sequencing of microRNA.//PNAS -2012. -V. 109. -P. 3024-3029
- Wang H., Tan G., Dong L. et al. Circulating MiR-125b as a marker predicting chemoresistance in breast cancer.//PLoS One. -2012. -V. 7(4):e34210
- Yan L., Huang X., Shao Q. et al. MicroRNA miR-21 overexpression in human breast cancer is associated with advanced clinical stage, lymph node metastasis and patient poor prognosis//RNA. -2008 -V. 14(11). -P. 2348-60
- Yan Z., Xiong Y., Xu W. et al. Identification of hsa-miR-335 as a prognostic signature in gastric cancer.//PLoS One. -2012. -V. 7(7):e40037
- Zhao J.J., Lin J., Yang H. et al. MicroRNA-221/222 negatively regulates estrogen receptor alpha and is associated with tamoxifen resistance in breast cancer.//J. Biol. Chem. -2008. -V. 283(45). -P. 31079-86
- Zhu S., Si M.L., Wu H. et al. microRNA-21 targets the tumor suppressor gene tropomyosin 1 (TPM1).//J. Biol. Chem. -2007. -V. 282. -P. 14328-14336


