Клонирование гена DREB1 и создание его ДНК-маркера, дифференцирующего DREB1 мягкой пшеницы и ее дикорастущих сородичей
Автор: Почтовый А.А., Крупин П.Ю., Дивашук М.Г., Кочешкова А.А., Соколов П.А., Карлов Г.И.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярные технологии
Статья в выпуске: 3 т.53, 2018 года.
Бесплатный доступ
Ген DREB кодирует транскрипционный фактор DREB (dehydration-responsive elementbinding), участвующий в ответе растения на засуху, засоление, высокую температуру. Транскрипционные факторы DREB при абиотическом стрессе индуцируют экспрессию множества генов, связанных с зависимой и не зависимой от абсцизовой кислоты передачей сигнала. Эволюционно многие дикорастущие виды формировались в экстремальных эколого-географических условиях, поэтому могут служить источниками новых генетических вариантов DREB для селекции пшеницы на устойчивость к стрессам. Изучение ортологов DREB у дикорастущих сородичей пшеницы позволит расширить арсенал генов, используемых для ее селекционного улучшения методами отдаленной гибридизации, а разработка и использование молекулярных маркеров дадут возможность направленно переносить эти гены в геном мягкой пшеницы. Среди всего разнообразия генов, кодирующих DREB белки, наибольший интерес представляет DREB1, который связан с устойчивостью растений к различным абиотическим факторам...
Мягкая пшеница, пырей, псевдорогнерия, дазипирум, гены устойчивости, гены-ортологи, засуха, засоление, полимеразная цепная реакция, молекулярные маркеры, секвенирование
Короткий адрес: https://sciup.org/142216552
IDR: 142216552 | УДК: 633.11:577.21 | DOI: 10.15389/agrobiology.2018.3.499rus
Текст научной статьи Клонирование гена DREB1 и создание его ДНК-маркера, дифференцирующего DREB1 мягкой пшеницы и ее дикорастущих сородичей
Существенные потери в аграрном секторе обусловлены такими абиотическими факторами, как засоленность почв и дефицит влаги. Толерантность растений к этим стрессам носит комплексный характер, то есть
Работа выполнена при финансовой поддержке РНФ в рамках выполнения гранта по Соглашению ¹ 1616-00097 от 27 января 2016 года.
представляет собой результат взаимодействия множества генов и биохимических факторов. Ключевую роль в устойчивости растений играют различные транскрипционные факторы (1, 2). Среди них выделяется группа DREB (dehydration-responsive element binding) из семейства AP2/ERF белков. Транскрипционные факторы DREB в ответ на абиотический стресс индуцируют экспрессию множества генов, связанных с зависимым от абсцизовой кислоты (АБК) и АБК-независимым путем передачи сигнала (35). Транскрипционный фактор DREB специфически связывается с регуляторной последовательностью Dre (5´-TACCGACAT-3´), которую впервые выявили в промоторе гена rd29A (6). Белок DREB содержит щелочной N-концевой аминокислотный регион, действующий как сигнал ядерной локализации. Справа от N-концевого участка расположен ДНК-связываю-щий домен AP2, который состоит из трехцепочечных β -складчатостей и одной α -спирали, почти параллельной β -складчатости (7). Ключевыми для связывания с GCC-боксом ДНК служат семь остатков аминокислот в AP2 домене (8). К ДНК-связывающему домену AP2/ERF прилегает консервативный регион с высоким содержанием серина и треонина (ST-богатый регион), который может служить сайтом фосфорилирования (9). У DREB имеется кислый С-концевой участок, который, как предполагают, обладает транскрипционной активационной активностью (10, 11).
Среди всего разнообразия генов, кодирующих DREB белки, наибольший интерес представляет DREB1 , который связан с устойчивостью растений к различным абиотическим факторам, таким как засуха, засоление, низкие температуры (12-14). Гены DREB1 секвенированы у многих видов растений, в том числе у мягкой пшеницы (15-17). B. Wei с соавт. (17) локализовали гены-гомологи TaDREB1 мягкой пшеницы на хромосомах 3-й гомеологичной группы и выявили полиморфизм по ДНК-последовательностям. Различные однонуклеотидные замены (single nucleotide polymorphisms, SNPs) в DREB1 у пшеницы ассоциированы с устойчивостью к засухе и засолению (18, 19). Кроме мягкой пшеницы, гены DREB были охарактеризованы у родственных ей дикорастущих видов Aegilops tauschii , Leymus chinensis , Elymus spicatus (20-22).
Использование подходов генетической инженерии позволило продемонстрировать роль генов DREB в усилении устойчивости к абиотическому стрессу. В частности, ген дикого ячменя Hordeum spontaneum был встроен в геном Paspalum notatum (23). В этом и других подобных опытах полученные трансгенные растения отличались от контрольных повышенной соле- и засухоустойчивостью (24-27).
Использование генетического потенциала дикорастущих видов, близкородственных пшенице, также может быть полезно для ее генетического улучшения, в том числе повышения устойчивости к биотическим и абиотическим факторам. Эволюционно многие виды формировались в экстремальных эколого-географических условиях (засуха, засоление) (28, 29). Поэтому близородственные неокультуренные сородичи пшеницы могут служить источниками новых генетических вариантов DREB для селекции на устойчивость к стрессам.
В отдаленной гибридизации пшеницы широко используются алло-полиплоидные виды — пырей средний Thinopyrum intermedium (геномная конституция JrJvsSt, 2 n = 6½ = 42) и пырей понтийский Th. ponticum (геномная конституция JJJJsJs, 2 n = 10x½ = 70) (29, 30). Они несут ценные гены устойчивости к таким факторам, как засоление, засуха и пониженные температуры (31, 32), прорастание на корню (33). Созданы формы пшеницы с генетическим материалом пырея, обладающие устойчивостью 500
к листовой (34-36), стеблевой ржавчине (37), фузариозу (38), вирусу полосатой мозаики (39) и другим заболеваниям. Хромосомы пырея понтийского и пырея среднего относительно легко конъюгируют и обмениваются участками (40). По современным представлениям, донорами субгеномов этих видов служат Dasypyrum villosum (V, 2 n = 2½ = 14), Th. bessarabicum (Jb, 2 n = 2½ = 14) и Pseudoroegneria spicata (St, 2 n = 2x = 14); два первых вида также широко используются в отдаленной гибридизации пшеницы (41-43).
В нашей работе были впервые получены последовательности ДНК генов-ортологов DREB1 у представителей родов Thinopyrum , Dasypyrum , Pseudoroegneria , а также разработан CAPS-маркер P18_ Fok I, которые позволяет различать ген пшеницы TaDREB1 и ген перечисленных видов.
Цель нашего исследования состояла в секвенировании и анализе генов-ортологов DREB1 диких видов, родственных пшенице, и создании ПЦР-маркера, использование которого дало бы возможность различать ген DREB1 диких видов и мягкой пшеницы.
Методика . В работе использовали образцы Thinopyrum bessarabicum (Savul. & Rayss) A. Love (PI 531711), Th. intermedium (Host) Barkworth & D.R. Dewey (PI 401200), Th. ponticum (Podp.) Z.-W. Liu & R.-C. Wang (PI 508561), Pseudoroegneria spicata (Pursh) A. Love (PI 537371), P. stipifolia (Czern. ex Nevski) A. Love (PI 325181), Dasypyrum villosum ( L.) Borbas (W6 21717), семена которых получили из Germplasm Research International Network (GRIN, США), и Th. ponticum (1158A/19), полученный из Главного ботанического сада (ГБС) РАН им. Н.В. Цицина (г. Москва, Россия). Картирование гена DREB1 проводили на наборе линий мягкой пшеницы Chinese Spring, дополненных хромосомами Th. elongatum . Также использовали 10 образцов пшенично-пырейных гибридов (ППГ), содержащих пырейный и пшеничный хроматин, из коллекции ГБС РАН им. Н.В. Цицина — 5542, 2087, 548, 1674, 4082, ЗП26, М3202, 4044, 4015, М12. Контролем служили сорта мягкой пшеницы Triticum aestivum L. Немчиновская 24, Айвина, а также сорта мягкой пшеницы Тулайковская золотистая, Тулайковская 10, Тулайковская 100, Тулайковская 110 с замещенной хромосомой от пырея среднего 6J(6D) (35).
ДНК выделяли из молодых листьев по методу R. Bernatzky с соавт. (44). В ПЦР использовали праймеры P18F (5´-CCC AAC CCA AGT GAT AAT AAT CT-3´), P18R (5´-TTG TGC TCC TCA TGG GTA CTT-3´), P20F (5´-TCG TCC CTC TTC TCG CTC CAT-3´), P20R (5´-GCG GTT GCC CCA TTA GA CAT AG-3´), P21F (5´-CGG AAC CAC TCC CTC CAT CTC-3´), P21R (5´-CGG TTG CCC CAT TAG ACG TAA-3´), P22F (5´-CTG GCA CCT CCA TTG CCG CT-3´), P25F (5´-CTG GCA CCT CCA TTG CTG CC-3´), PRa (5´-AGT ACA TGA ACT CAA CGC ACA GGA CAA C-3´), разработанные B. Wei (17). ПЦР проводили на амплификаторе DNA Engine Tetrad 2 («Bio-Rad», США) по протоколу: 5 мин при 95 ° C (1 цикл); 30-45 с при 95 ° C, 30-60 с при б0 ° C, 40-120 с при 72 ° C (34 цикла); 10 мин при 72 ° C (1 цикл). Образцы хранили при 10 ° C. Продукты ПЦР разделяли в 1,5 % агарозном геле с буфером ТВЕ при напряженности поля 6 В/см.
Для клонирования продуктов амплификации их очищали с помощью набора GeneJET™ PCR Purification Kit («Fermentas», Литва) согласно инструкции производителя. Очищенную ДНК лигировали в вектор pGEM®-T Easy («Promega», США). Вектор трансформировали в Escherichia coli штамм DH10B («Life Technologies», США). Трансформацию проводили на электро-пораторе GenePulser («Bio-Rad», США). Рекомбинантные клоны выявляли методом бело-голубой селекции.
Клоны для секвенирования отбирали с помощью ПЦР с прайме- рами М13, фланкирующими область вставки. Секвенирование осуществляли на приборе ABI-3130XL («Applied Biosystems», США). Выравнивание нуклеотидной и аминокислотной последовательностей выполняли в программе ClustalW2 (45). Для получения гипотетических аминокислотных последовательностей применили ресурс ExPASy (46). Сигнал ядерной локализации находили с помощью алгоритма cNLS Mapper (47). Для определения GCC-связывающих боксов и AP2-связывающго домена воспользовались базой данных Conserved Domain Database (48).
Эндонуклеазу рестрикции (Fok1) подбирали на основании анализа нуклеотидного выравнивания региона, амплифицируемого с праймером P18. Рестрикцию ПЦР-продуктов (10 мкл) проводили в течение 12 ч при 37 ° С, рестрикты разделяли электрофорезом в агарозном 2 % геле.
Результаты. Привлечение генетических ресурсов третичного пула трибы Triticeae играет важную роль в селекционном улучшении пшеницы. В этом отношении перспективны дикорастущие представители родов Thin-opyrum , Dasypyrum , Pseudoroegneria. Они характеризуются соле- и засухоустойчивостью, устойчивостью к вирусным и грибным фитопатогенам, вредителям и другими хозяйственно значимыми признаками (28, 31, 49). Гены DREB1 неокультуренных близкородственных пшенице видов важны в селекции пшеницы на повышенную толерантность к засолению и засухе.
В результате клонирования ПЦР-продуктов, амплифицированных с помощью различных комбинаций праймеров, и их последующего секвенирования мы получили 30 уникальных нуклеотидных последовательностей для шести изучаемых видов дикорастущих злаков: Th. bessarabicum (Thbe1, Thbe2, Thbe3), Th. intermedium (Thin1, Thin2, Thin4, Thin5, Thin6, Thin7), Th. ponticum (Thpo1, Thpo2, Thpo3, Thpo4, Thpo5, Thpo6, Thpo7, Thpo8 — c образца PI 508561; Thpo9, Thpo10, Thpo11, Thpo12, Thpo13, Thpo14, Thpo15, Thpo16 — с образца 1158А/19), P. spicata (Pssp1, Pssp2), P. stipifola (Psst), D. villosum (Davi). Все последовательности были получены с праймерами P18F/R, кроме Thbe1 (P20F + PRa — полноразмерная последовательность гена), Thbe2, Thbe3, Thpo1, Thpo2, Thin1, Pssp1, Davi (P21F + PRa).

Рис. 1. Фрагменты выравненных последовательностей гена-ортолога DREB1 у Thinopyrum bessa- rabicum (строки 1-3), Pseudoroegneria stipifolia (строка 4), P. spicata (строки 5-6), Dasypyrum villosum (строка 7) , Th. intermedium (строки 8-13) , Th. ponticum (строки 14-29) и TaDREB трех субгеномов мягкой пшеницы (три нижние строки). Цветом выделены наиболее крупные инде-лы: четыре индела на участке 299-327 (выделено желтым); регион 682-689 (выделено сиреневым); регион 698-705 (выделено зел¸ным); регион 1266-1268 (выделено голубым); регион 1316-1324 (выделено красным).
Выравнивание последовательностей ДНК с генами DREB1 мягкой пшеницы выявило гомологию (92-98 %) между ними. Основные различия между сиквенсами изучаемых видов и последовательностями мягкой пше- ницы составляли многочисленные SNPs и инсерции/делеции (инделы) (рис. 1). К наиболее крупным относились четыре индела в позициях 299327 у Th. bessarabicum (1 п.н., 6 п.н., 11 п.н. и 3 п.н.) и индел у D. villosum (см. рис. 1, выделено желтым), у всех изучаемых видов — 682-689 (см. рис. 1, выделено сиреневым), 698-705 (выделено зеленым), 1266-1268 (см. рис. 1, выделено голубым) и 1316-1324 (см. рис. 1, выделено красным). Несмотря на значительное количество полученных нами последовательностей ДНК, полиморфизм гена-ортолога DREB1 не ограничивается выявленными SNPs и инделами, поскольку даже внутри одного образца дикорастущего вида может наблюдаться внутрипопуляционное разнообразие.
Полученные ДНК-последовательности были транслированы in silico в гипотетические последовательности аминокислот (АК). Ни одна из них не содержала стоп-кодонов. Все предсказанные АК-последовательности имели одинаковую структурную организацию, характерную для DREB-белка. В N-концевом домене находился сигнал ядерной локализации (NLS). Рядом с AP2 ДНК-связывающим доменом располагалась консервативная ST-богатая область. C-концевой домен был обогащен глутаминовой и аспарагиновой кислотами, что свидетельствует о наличии транскрипционного активационного домена.
Thbe3
Psspl
Pssp2 Davi Thinl
Thin2
Thin4 Thin5
Thin?
Thpo4
Thpo? ThpoS ThpoS Thpo10
Thpo13
ThpolS
Taest_B
I RHR jw
R RjW R
R Rjw R R
R r|w
R R
R R
R R
R R
R R j
R RjW
R RjW R R
R RjW R R W
R RjW R . F
R R jH
R В
R В|»
R R
R RjW
R rIw
R rEw YRGVRqRTW
gkwv; gkwv; gkwv; gkwv; gkwv; gkwv; gkwv; GKWV, gkwv; gkwv; gkwv; GKWV, gkwv; gkwv; gkwv; gkwv, gkwv; GKWV, GKWV, gkwv; gkwv; gkwv; gkwv; GKWV, gkwv; gkwv; gkwv; gkwv; gkwv; GKWV, GKWV, gkwv;
i
iLGlFPTi lgIfpt; lgsfpt;
so
AARAYDDAARAMYGAKARVNFSEQSP]
E
8:
s
l:
i
8:
8:
8:
E
F ■
i^lgIfpt; lRlgsfpt; ■lPlgsfpt;
IHLG|FPTj I^LGeEPTi i^lgbfpt; iblgIfpt;
IMLGeFPTj I№LG|FPTj iBlghfpt; wlgIfpt; iBlgIfpt; lRlgsfpti i^lgIppt; lRlgsfpt; тйъсЛррт; iSlgsfpt; iklghfpt;
sli
I * 8 8 I I I я 8
AARAYDDAARAMYGAKARVNFSEQSP] aarayddaaramygakarvnfseqs pi lARAYDDAARAM YGAKARVNFSEQS Pi
AARAYDDAARAMYGAKARVNFSEQSPl AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSP]
§
AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSPl
■aarayddaaramygakarvnfseqs Pl
aarayddaaramygakarvnfseqspi AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQS Pl aarayddaaramygakarvnfseqspi AARAYDDAARAMYGAKARVNFSEQSP] aARA YDDAARAMYGAKARVl^SEQS P] AARAYDDAARAMYGAKARVNFSEQSP) AARAYDDAARAMYGAKARVNFSEQSP]
AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSPl
|Lg| fptaBaarayddaaram YGAKARVNFSEQS Pl
iJ lgIfpt, i№lgIfpt; TgLGSFPT; iBlgIfpt; mlgIfpt; LgLGSFPT; lRlgsfpt; wlgIfpt; iklgIfpti
AARAYDDAARAMYGAKARVNFSEQSP] aarayddaaramygakarvnfseqspi AARAYDDAARAMYGAKARVNFSEQSPJ aarayddaaramygakarvnfseqs pi AARAYDDAARAMYGAKARVNFSEQSP] aarayddaaramygakarvnfseqspi
AARAYDDAARAMYGAKARVNFSEQS Pl AARAYDDAARAMYGAKARVNFSEQSP] AARAYDDAARAMYGAKARVNFSEQSP]
IGKWVAEIREPNBGNRI,wLGsFPTAVBAZlR?lYDCMMMYGaKARVN5SECSPB
Рис. 2. Выравнивание гипотетических аминоксилотных последовательностей AP2 ДНК-связы- вающего домена белка DREB1 Thinopyrum bessarabicum (строки 1-3), Pseudoroegneria stipifolia (строка 4) , P. spicata (строки 5-6) , Dasypyrum villosum (строка 7) , Th. intermedium (строки 813) , Th. ponticum (строки 14-29) и субгеномов A, B и D мягкой пшеницы (три нижние строки) . Стрелками указаны полиморфные аминокислоты в AP2-домене; зеленым цветом выделены аминокислотные остатки, которые взаимодействуют с GCC-боксом, красным — замена по одному такому аминокислотному остатку; звездочками указаны аминокислоты, важные для DRE-специфического связывания.
В функционировании DREB-белка особая роль принадлежит AP2 ДНК-связывающему домену, полиморфизмы внутри этого региона могут оказывать критическое влияние на активность DREB-белка. Во всех гипотетических АК-последовательностях встречался AP2 ДНК-связывающий домен, состоящий из 59 остатков аминокислот (рис. 2). Сравнение АК-последовательностей AP2 ДНК-связывающего домена у изученных образцов показало наличие полиморфизмов по отдельным аминокислотам (single amino acid polymorphism, SAP). Поскольку дикие виды способны занимать различные эколого-географические ниши и проявляют значительное популяционное разнообразие, следует предположить, что SAP-полиморфизм по белку DREB1 может быть шире, чем выявлено нами. В полученных нами последовательностях присутствовали все консервативные аминокислоты AP2-домена в DREB-белков злаков (50), исключение составила последовательность Thpo11: у нее в положениях 25-27 специфичный мотив WLG (25-27) был замещен на RLG (замена триптофана на аргинин) (см. рис. 2). Выявленная нами замена W(R) произошла в одной из семи консервативных аминокислот GCC-связывающего бокса в AP2 домене. Наличие подобной замены в AP2 домене может привести к нарушению пространственной конфигурации белка, что, в свою очередь, скажется на ДНК-связывающей способности (51).
Сравнительный анализ ДНК-последовательностей гена DREB1 шести видов дикорастущих злаков ( Th. intermedium , Th. ponticum , D. villosum , P. spicata , P. stipifolia , Th. bessarabicum ) с ДНК-последовательностями гена DREB1 мягкой пшеницы выявил SNPs, характерные для каждого вида. Это позволило разработать ПЦР-маркер, который может быть использован для идентификации большинства генов DREB1 изученных нами видов в генетическом бэкграунде мягкой пшеницы.
Мы использовали праймеры P18F/R, предложенные B. Wei с соавт. (17), для амплификации консервативной области гена TaDREB1 мягкой пшеницы, кодирующей ДНК-связывающий домен AP2. На основании сравнения нуклеотидных последовательностей генов-ортологов DREB1 , полученных с парой праймеров P18F/R, у пяти изученных видов — Th. intermedium , Th. ponticum , D. villosum , P. spicata , Th. bessarabicum и последовательностей генов DREB1 мягкой пшеницы было выявлено три сайта рестрикции для эндонуклеазы FokI (рис. 3, выделены бирюзовым цветом). Отсутствие одного из сайтов рестрикции и наличие двух других было характерно для большинства последовательностей гена DREB1 у дикорастущих видов, что отличало их от последовательностей гена DREB1 субгеномов A и D мягкой пшеницы. У гена DREB1 субегенома B присутствовал лишь один сайт рестрикции FokI, кроме того, в той же области находилась крупная делеция (более 35 п.н.). Это различие позволило нам создать CAPS (cleaved amplified polymorphic sequences) маркер P18_FokI, который в большинстве случаев может дифференцировать гены-ортологи DREB1 пшеницы и дикорастущих злаков.

Рис. 3. Фрагменты выравненных последовательностей гена-ортолога DREB1 у Thinopyrum bessa- rabicum (строки 1-3), Pseudoroegneria stipifolia (строка 4), P. spicata (строки 5-6), Dasypyrum villosum (строка 7) , Th. intermedium (строки 8-13) , Th. ponticum (строки 14-29) и TaDREB трех субгеномов мягкой пшеницы (три нижние строки) . Бирюзовым цветом показаны сайты рестрикции для эндонуклеазы FokI.
С помощью разработанного нами маркера P18_FokI была установ- лена хромосомная локализация гена DREB1 в геноме J. Для этого мы использовали серию дополненных линий пшеницы с хромосомами пырея удлиненного Th. elongatum (2n = 2½ = 14, геном JeJe). Контролем служил пырей бессарабский Th. bessarabicum, несущий геном JbJb (2n = 2½ = 14), высокогомологичный геному Th. elongatum. В свою очередь, в коллекциях генетических ресурсов под обозначением Th. elongatum нами были обнаружены только представители полиплоидного вида Th. ponticum (2n = 10½ = 70, геномный состав JJJJJJJsJsJsJs), который ранее называли восточноевропейской разновидностью Th. elongatum (52, 53), что нередко для генетических коллекций видов с затрудненной ботанической идентификацией (54).
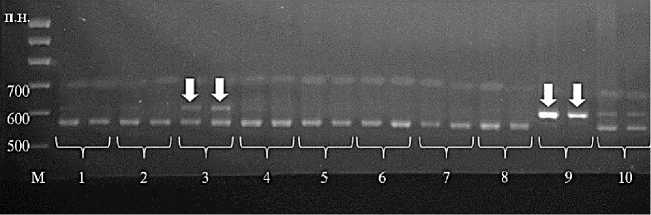
Рис. 4. Электрофореграмма, показывающая результаты анализа дополненных линий мягкой пшеницы Chinese Spring c хромосомами Thinopyrum elongatum с помощью CAPS маркера P18_ FokI, ассоциированного с геном DREB1 : 1 — CS + 1Je, 2 — CS + 2Je, 3 — CS + 3Je, 4 — CS + 4Je, 5 — CS + 5Je, 6 — CS + 6Je, 7 — CS + 7Je, 8 — Triticum aestivum , сорт Айвина, 9 — Th. bessarabicum ; М — маркер длины фрагментов ДНК (GeneRuler 100 bp DNA Ladder, «Thermo Fisher Scientific», США). Стрелкой указан дополнительный фрагмент ~ 600 п.н., амплифицируемый с гена-ортолога DREB1 пырейного происхождения .
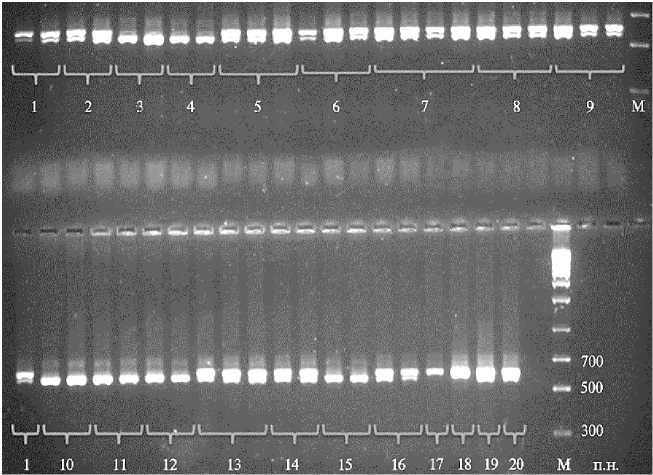
Рис. 5. Электрофореграмма, показывающая результаты анализа образцов пшенично-пырейных гибридов и сортов с помощью CAPS маркера P18_FokI для гена DREB1 : 1 — 5542, 2 — 2087, 3 — Тулайковская 110, 4 — Немчиновская 24, 5 — 548, 6 — 1674, 7 — 4082, 8 — ЗП26, 9 — М3202, 10 — Айвина, 11 — Тулайковская 10, 12 — Тулайковская 100, 13 — 4044, 14 — 4015, 15 — Тулайковская золотистая, 16 — М12, 17 — Thinopyrum intermedium , 18 — Th. ponticum , 19 — Pseudoroegneria spicata , 20 — Th. intermedium ; М — маркер длины фрагментов ДНК (Fisher BioReagents™ exACTGene™ DNA Ladders, «Thermo Fisher Scientific», США).
С участием маркера P18_FokI у пшеницы был выявлен фрагмент размером около 570 п.н., у пырея бессарабского — около 600 п.н. (рис. 4, указан стрелкой). У всех дополненных линий мягкой пшеницы c хромосомами пырея удлиненного был обнаружен фрагмент ∼ 570 п.н., специфичный для пшеницы, и только у одной линии CS + 3E, дополненной хромосомой 3Je, — фрагмент ∼ 600 п.н., специфичный для пырея. То есть ген-ортолог DREB1 у Th. elongatum был локализован в гомеологичной 3-й группе, как и у мягкой пшеницы, что подтверждает корректную работу разработанного нами маркера.
Для апробации полученного маркера P18_FokI мы провели последовательно ПЦР и рестрикцию у 10 образцов пшенично-пырейных гибридов. С помощью маркера P18_FokI у всех исследуемых образцов ППГ выявили два фрагмента — размером ∼ 570 п.н. и ∼ 600 п.н., то есть образцы несли DREB1 ген как пшеничного, так и пырейного типа (рис. 5). У всех контрольных образцов мягкой пшеницы, включая сорта мягкой пшеницы с замещенной хромосомой от пырея среднего 6J(6D) — Тулайковская золотистая, Тулайковская 10, Тулайковская 100, Тулайковская 110, фрагмент ∼ 600 п.н. отсутствовал. Следовательно, фрагмент ∼ 600 п.н. пырейного типа специфичен для гена DREB1 , локализованного в 3-й гомеологичной группе.
Маркер-опосредованный отбор способствует повышению результативности селекции. Использование молекулярных маркеров позволяет с наименьшими затратами определить ценные аллели в расщепляющей популяции, быстро и точно идентифицировать любой ген при интрогрессии от линии-донора в геном реципиента, обеспечивает удобный инструмент при бэкроссировании (55). С помощью разработанного нами CAPS-маркера P18_FokI, ассоциированного с геном-ортологом DREB1 , можно увеличить эффективность переноса этого гена из дикорастущих злаков в геном мягкой пшеницы, изучить влияние чужеродного DREB1 гена на устойчивость мягкой пшеницы к засухе, засолению, низким температурам и в перспективе отобрать ценные селекционные формы.
Итак, мы получили ДНК-последовательности генов, ортологичных гену пшеницы DREB1 , у видов Thinopyrum intermedium, Th. ponticum, Th. bes-sarabicum , Dasypyrum villosum , Pseudoroegneria spicata, P. stipifola . Исследование этих нуклеотидных последовательностей выявило полиморфизм между образцами изучаемых видов — наличие однонуклеотидных замен (SNPs) и инделов. Анализ гипотетических аминокислотных последовательностей, кодируемых выявленными генами-ортологами DREB1 , показал консервативность: только в одном сиквенсе обнаружена замена W(R), важная для функционирования AP-2 домена. Создан CAPS-маркер P18_FokI, с помощью которого можно отличать ген-ортолог DREB1 изученных дикорастущих видов от DREB1 мягкой пшеницы. С помощью маркера P18_FokI ген-ортолог DREB1 картирован на хромосоме 3Je пырея удлиненного. У 10 пшенично-пырейных гибридов обнаружен ген-ортолога DREB1 пырейного происхождения.
Авторы выражают признательность В.И. Белову и В.П. Упелниеку (ГБС РАН им. Н.В. Цицина) за предоставленные образцы пшенично-пырейных гибридов, а также доктору J. Raupp (Kansas State University, США) за образцы линий мягкой пшеницы, дополненных хромосомами Thinopyrum elongatum (созданы Jan Dvorak, UC Davis, США).
Список литературы Клонирование гена DREB1 и создание его ДНК-маркера, дифференцирующего DREB1 мягкой пшеницы и ее дикорастущих сородичей
- Chen W.J., Zhu T. Networks of transcription factors with roles in environmental stress response. Trends Plant Sci., 2004, 9(12): 591-596 ( ) DOI: 10.1016/j.tplants.2004.10.007
- Xu Z.S., Chen M., Li L.C., Ma Y.Z. Functions of the ERF transcription factor family in plants. Botany, 2008, 86(9): 969-977 ( ) DOI: 10.1139/B08-041
- Wang J.W., Yang F.P., Chen X.Q., Liang R.Q., Zhang L.Q., Geng D.M., Zhang X.D., Song Y.Z., Zhang G.S. Induced expression of DREB transcriptional factor and study on its physiological effects of drought tolerance in transgenic wheat. ActaGeneticaSinica, 2006, 33(5): 468-476() DOI: 10.1016/S0379-4172(06)60074-7
- Agarwal P.K., Agarwal P., Reddy M.K., Sopory S.K. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants. Plant Cell Rep., 2006, 25(12): 1263-1274 () DOI: 10.1007/s00299-006-0204-8
- Xu Z.S., Ni Z.Y., Li Z.Y., Li L.C., Chen M., Gao D.Y., Yu X.D., Liu P., Ma Y.Z. Isolation and functional characterization of HvDREB1-a gene encoding a dehydration-responsive element binding protein in Hordeum vulgare. J. Plant Res., 2009, 122(1): 121-130 () DOI: 10.1007/s10265-008-0195-3
- Yamaguchi-Shinozaki K., Shinozaki K. A novel cis-acting element in an Arabidopsis gene is involved in responsiveness to drought, low-temperature, or high-salt stress. The Plant Cell Online, 1994, 6(2): 251-264 () DOI: 10.1105/tpc.6.2.251
- Magnani E., Sjölander K., Hake S. From endonucleases to transcription factors: evolution of the AP2 DNA binding domain in plants. Plant Cell, 2004, 16(9): 2265-2277 ( ) DOI: 10.1105/tpc.104.023135
- Liu Y.,Zhao T.J.,Liu J.M.,Liu W.Q.,Liu Q.,Yan Y.B.,Zhou H.M. The conserved Ala37 in the ERF/AP2 domain is essential for binding with the DRE element and the GCC box. FEBS Lett., 2006, 580(5): 1303-1308 () DOI: 10.1016/j.febslet.2006.01.048
- Lata C.,Prasad M. Role of DREBs in regulation of abiotic stress responses in plants. J. Exp.Bot., 2011, 62(14): 4731-4748 () DOI: 10.1093/jxb/err210
- Stockinger E.J.,Gilmour S.J.,Thomashow M.F.Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit. PNASUSA, 1997, 94(3): 1035-1040 () DOI: 10.1073/pnas.94.3.1035
- Wang S.X.,Wang Z.Y.,Peng Y.K. Dehydration responsive element binding (DREB) transcription activator and its function in plant tolerance to environmental stresses. Plant Physiology Communications, 2004, 40(1): 7-13.
- Liu Q.,Kasuga M.,Sakuma Y.,Abe H.,Miura S.,Yamaguchi-Shinozaki K.,Shinozaki K.Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression, respectively, in Arabidopsis. The Plant Cell Online, 1998, 10(8): 1391-1406 () DOI: 10.1105/tpc.10.8.1391
- Akhtar M.,Jaiswal A.,Taj G.,Jaiswal J.P.,Qureshi M.I.,Singh N.K. DREB1/CBF transcription factors: their structure, function and role in abiotic stress tolerance in plants. J.Genet., 2012, 91(3): 385-395 () DOI: 10.1007/s12041-012-0201-3
- Kidokoro S.,Watanabe K.,Ohori T.,Moriwaki T.,Maruyama K.,Mizoi J.,Myint P.S.H.N.,Fujita Y.,Sekita S.,Shinozaki K,Yamaguchi-Shinozaki K. Soybean DREB1/CBF-type transcription factors function in heat and drought as well as cold stress-responsive gene expression. Plant J., 2015, 81(3): 505-518 () DOI: 10.1111/tpj.12746
- Shen Y.G.,Zhang W.K.,He S.J.,Zhang J.S.,Liu Q.,Chen S.Y. An EREBP/AP2-type protein in Triticumaestivum was a DRE-binding transcription factor induced by cold, dehydration and ABA stress. Theor. Appl. Genet., 2003, 106(5): 923-930 () DOI: 10.1007/s00122-002-1131-x
- Khan M.S. The role of DREB transcription factors in abiotic stress tolerance of plants. Biotechnol. Biotec. Eq., 2011, 25(3): 2433-2442 () DOI: 10.5504/BBEQ.2011.0072
- Wei B.,Jing R.,Wang C.,Chen J.,Mao X.,Chang X.,Jia J. Dreb1 genes in wheat (TriticumaestivumL.): development of functional markers and gene mapping based on SNPs. Mol.Breeding, 2009, 23(1): 13-22 () DOI: 10.1007/s11032-008-9209-z
- Chen J.,Jing R.,Yuan H.,Wei B.,Chang X. Single nucleotide polymorphism of TaDREB1gene in wheat germplasm. Scientia AgriculturaSinica, 2005, 38(12): 2387-2394.
- Mondini L.,Nachit M.,Porceddu E.,Pagnotta M.A. Identification of SNP mutations in DREB1, HKT1, and WRKY1 genes involved in drought and salt stress tolerance in durum wheat (Triticumturgidum L. vardurum). OMICS: A Journal of Integrative Biology, 2012, 16(4): 178-187 () DOI: 10.1089/omi.2011.0081
- Suneja Y. Physio-biochemical responses and allelic diversity for water deficit tolerance related traits in Aegilopstauschii and Triticumdicoccoides. Ph.D. Thesis. Ludhiana, India, 2014.
- Xianjun P., Xingyong M., Weihong F., Man S., Liqin C., Alam I., Lee B.H., Dongmei Q., Shihua S., Gongshe L. Improved drought and salt tolerance of Arabidopsis thaliana by transgenic expression of a novel DREB gene from Leymuschinensis. Plant Cell Rep., 2011, 30(8):1493-502 ( ) DOI: 10.1007/s00299-011-1058-2
- Почтовый А.А., Дивашук М.Г., Карлов Г.И. Секвенированиеианализгеновdreb1уThinopyrum, DasypyrumиElymusspicatus. Мат. XVМолодежной науч. конф. «Биотехнология в растениеводстве, животноводстве и ветеринарии». М., 2015: 35-36.
- James V.A.,Neibaur I.,Altpeter F. Stress inducible expression of the DREB1A transcription factor from xeric, Hordeumspontaneum L. in turf and forage grass (PaspalumnotatumFlugge) enhances abiotic stress tolerance. Transgenic Res., 2008, 17(1): 93-104( ) DOI: 10.1007/s11248-007-9086-y
- Dubouzet J.G.,Sakuma Y.,Ito Y.,Kasuga M.,Dubouzet E.G.,Miura S.,Seki M.,Shinozaki K.,Yamaguchi-Shinozaki K. OsDREB genes in rice, Oryza sativa L., encode transcription activators that function in drought-, high-salt-and cold-responsive gene expression. Plant J., 2003, 33(4): 751-763( ) DOI: 10.1046/j.1365-313X.2003.01661.x
- Bhatnagar-Mathur P.,Devi M.J.,Reddy D.S.,Lavanya M.,Vadez V.,Serraj R.,Yamaguchi-Shinozaki K.,Sharma K.K. Stress inducible expression of At DREB1A in transgenic peanut (Arachishypogaea L.) increases transpiration efficiency under water-limiting conditions. Plant Cell, 2007, 26: 2071-2082( ) DOI: 10.1007/s00299-007-0406-8
- Gao S.Q.,Chen M.,Xia L.Q.,Xiu H.J.,Xu Z.S.,Li L.C.,Zhao C.P.,Cheng X.G.,Ma Y.Z. A cotton (Gossypiumhirsutum)DREB-binding transcription factor gene, GhDREB, confers enhanced tolerance to drought, high salt, and freezing stresses in transgenic wheat. Plant Cell Rep., 2009, 28(2): 301-311 ( ) DOI: 10.1007/s00299-008-0623-9
- Wang X.,Chen X.,Liu Y.,Gao H.,Wang Z.,Sun G. CkDREB gene in Caraganakorshinskii is involved in the regulation of stress response to multiple abiotic stresses as an AP2/EREBP transcription factor. Mol. Biol. Rep., 2011, 38(4): 2801-2811( ) DOI: 10.1007/s11033-010-0425-3
- Wang R.R.-C., Jensen K.B. Wheatgrass and wildrye grasses (Triticeae). Genetic Resources, Chromosome Engineering, and Crop Improvement, 2009, 5: 48-49.
- Wang R.R.C. Agropyron andpsathyrostachys. In: Wild crop relatives: genomic and breeding resources. Springer, Berlin, Heidelberg, 2011: 77-108 ( ) DOI: 10.1007/978-3-642-14228-4_2
- Chen Q. Detection of alien chromatin introgression from Thinopyruminto wheat using S genomic DNA as a probe-a landmark approach for Thinopyrum genome research. Cytogenet. Genome Res., 2005, 109(1-3): 350-359 ( ) DOI: 10.1159/000082419
- Tabaei-Aghdaei S.R.,Harrison P.,Pearce R.S. Expression of dehydration-stress-related genes in the crowns of wheatgrass species having contrasting acclimation to salt, cold and drought. Plant Cell Environ., 2000, 23(6): 561-571 ( ) DOI: 10.1046/j.1365-3040.2000.00572.x
- Крупин П.Ю., Дивашук М.Г., Баженов М.С., Гриценко Л.А., Тараканов И.Г., Упелниек В.П., Белов В.И., Почтовый А.А., Старикова Е.В., КхуатТхиМайЛ., Климуши-наМ.В.,Давыдова А.Н., Карлов Г.И. Полиморфизмреакциипроростковпшенично-пырейныхгибридовназасоление. Сельскохозяйственнаябиология, 2013, 5: 44-53 ( ) DOI: 10.15389/agrobiology.2013.5.44rus
- Kocheshkova A.A., Kroupin P.Yu., Bazhenov M.S., Karlov G.I., Pochtovyy A.A., Upelniek V.P.,Belov V.I., Divashuk M.G. Pre-harvest sprouting resistance and haplotype variation of ThVp-1 gene in the collection of wheat-wheatgrass hybrids. PLoS ONE, 2017, 12(11): e0188049( ) DOI: 10.1371/journal.pone.0188049
- Крупин П.Ю., Дивашук М.Г., Белов В.И., Жемчужина А.И., Коваленко Е.Д., Упелниек В.П., Карлов Г.И. Исследование промежуточных пшенично-пырейных гибридов на устойчивость к листовой ржавчине. Сельскохозяйственная биология, 2013, 1: 68-73 ( ) DOI: 10.15389/agrobiology.2013.1.68rus
- Salina E.A., Adonina I.G., Badaeva E.D., Kroupin P.Yu., Stasyuk A.I., Leonova I.N., Shishkina A.A., Divashuk M.G., Starikova E.V., Khuat T.M.L., Syukov V.V., Karlov G.I. AThinopyrum intermedium chromosome in bread wheat cultivars as a source of genes conferring resistance to fungal diseases.Euphytica, 2015, 204(1): 91-101( ) DOI: 10.1007/s10681-014-1344-5
- Сибикеев С.Н., Бадаева Е.Д., Гультяева Е.И., Дружин А.Е., Шишкина А.А., Драгович А.Ю.,Крупин П.Ю., Карлов Г.И., Кхуат Т.М., Дивашук М.Г. Сравнительныйанализ 6Agi и 6Agi2 хромосомAgropyron intermedium (host) Beauvусортовилиниймягкойпшеницыспшенично-пырейнымизамещениями. Генетика, 2017, 53(3): 298-309.
- Liu W., Danilova T.V., Rouse M.N., Bowden R.L., Friebe B., Gill B.S., Pumphrey M.O. Development and characterization of a compensating wheat -Thinopyrum intermediumRobertsonian translocation with Sr44 resistance to stem rust (Ug99). Theor. Appl. Genet., 2013, 126(5): 1167-1177( ) DOI: 10.1007/s00122-013-2044-6
- Shen X.,Ohm H. Fusarium head blight resistance derived from Lophopyrumelongatum chromosome 7E and its augmentation with Fhb1 in wheat. Plant Breeding, 2006, 125(5): 424-429 ( ) DOI: 10.1111/j.1439-0523.2006.01274.x
- Friebe B.,Qi L.L.,Wilson D.L.,Chang Z.J.,Seifers D.L.,Martin T.J.,Fritz A.K.,Gill B.S Wheat-Thinopyrum intermedium recombinants resistant to wheat streak mosaic virus and Triticum mosaic virus. Crop Sci., 2009, 49(4): 1221-1226 () DOI: 10.2135/cropsci2008.09.0513
- Chen Q., Conner R.L., Laroche A., Ahmad F. Molecular cytogenetic evidence for a high level of chromosome pairing among different genomes in Triticumaestivum-Thinopyrum intermedium hybrids. Theor. Appl. Genet., 2001, 102(6): 847-852 ( ) DOI: 10.1007/s001220000496
- De Pace C.,Vaccino P.,Cionini P.G.,Pasquini M.,Bizzarri M.,Qualset C.O.Dasypyrum. In: Wild crop relatives: genomic and breeding resources. Springer, Berlin, Heidelberg, 2011: 185-292 ( ) DOI: 10.1007/978-3-642-14228-4_4
- Ceoloni C.,Kuzmanović L.,Gennaro A.,Forte P.,Giorgi D.,Grossi M.R.,Bitti A.Genomes, chromosomes and genes of the wheatgrass genus Thinopyrum: the value of their transfer into wheat for gains in cytogenomic knowledge and sustainable breeding. In: Genomics of Plant Genetic Resources/R. Tuberosa, A. Graner, E. Frison (eds.). Springer, Dordrecht, 2014: 333-358 ( ) DOI: 10.1007/978-94-007-7575-6_14
- Divashuk M.G.,Khuat T.M.,Kroupin P.Y.,Kirov I.V.,Romanov D.V.,Kiseleva A.V.,Khrustaleva L.I.,Alexeev D.G.,Zelenin A.S.,Klimushina M.V.,Razumova O.V.,Karlov G.I.Variation in copy number of Ty3/gypsy centromeric retrotransposons in the genomes of Thinopyrum intermedium and its diploid progenitors. PLoS ONE, 2016, 11(4): e0154241( ) DOI: 10.1371/journal.pone.0154241
- Bernatzky R.,Tanksley S.D. Toward a saturated linkage map in tomato based on isozyme and random cDNA sequences. Genetics, 1986, 112(1): 887-898.
- Goujon M., McWilliam H., Li W., Valentin F., Squizzato S., Paern J., Lopez R. A new bioinformatics analysis tools framework at EMBL-EBI. Nucleic Acids Res., 2010, 38(Web Server issue):W695-W699 ( ) DOI: 10.1093/nar/gkq313
- Artimo P.,Jonnalagedda M.,Arnold K.,Baratin D.,Csardi G.,Castro E.,Duvaud S.,Flegel V.,Fortier A.,Gasteiger E.,Grosdidier A.,Hernandez C.,Ioannidis V.,Kuznetsov D.,Liechti R.,Moretti S.,Mostaguir K.,Redaschi N.,Rossier G.,Xenarios I.,Stockinger H. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Res., 2012, 40(1): W597-W603 ( ) DOI: 10.1093/nar/gks400
- Kosugi S.,Hasebe M.,Tomita M.,Yanagawa H.Systematic identification of cell cycle-dependent yeast nucleocytoplasmic shuttling proteins by prediction of composite motifs. PNASUSA, 2009, 106(25):10171-10176 ( ) DOI: 10.1073/pnas.0900604106
- Marchler-Bauer A.,Bo Y.,Han L.,He J.,Lanczycki C.J.,Lu S.,Gwadz M. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures. Nucleic Acids Res., 2017, 45(D1): D200-D203 ( ) DOI: 10.1093/nar/gkw1129
- Luo P.G.,Luo H.Y.,Chang Z.J.,Zhang H.Y.,Zhang M.,Ren Z.L. Characterization and chromosomal location of Pm40 in common wheat: a new gene for resistance to powdery mildew derived from Elytrigiaintermedium. Theor. Appl. Genet., 2009, 118(6): 1059-1064 ( ) DOI: 10.1007/s00122-009-0962-0
- Filiz E., Tombuloğlu H. In silico analysis of DREB transcription factor genes and proteins in grasses. Appl.Biochem.Biotechnol., 2014, 174(4):1272-1285 ( ) DOI: 10.1007/s12010-014-1093-x
- Allen M.D.,Yamasaki K.,Ohme-Takagi M.,Tateno M.,Suzuki M.A novel mode of DNA recognition by a b-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA. The EMBO Journal, 1998, 17(18): 5484-5496 ( ) DOI: 10.1093/emboj/17.18.5484
- Цицин Н.В. Многолетняя пшеница. М., 1978.
- The PLANTS Database.Режимдоступа:https://plants.usda.gov/java/.Дата обращения:01.12.2017.
- KhuatThi Mai L.,Divashuk M.G.,Kroupin P.Y.,Nguyen A.P.,Kiseleva A.V.,Karlov G.I.Differences in ploidy level and genome constitution revealed by cytogenetic analysis of Pseudoroegneria germplasm accessions: case study. ИзвестияТСХА, 2015, 2(4): 29-35.
- Collard B.C.Y.,Mackill D.J.Marker-assisted selection: an approach for precision plant breeding in the twenty-first century. Philos. T. Roy. Soc. B, 2008, 363(1491): 557-572 ( ) DOI: 10.1098/rstb.2007.2170


