Компьютерное предсказание патогенных свойств мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом
Автор: Алемасов Н.А., Иванисенко Н.В., Иванисенко В.А.
Журнал: Международный журнал гуманитарных и естественных наук @intjournal
Рубрика: Биологические науки
Статья в выпуске: 10-1 (25), 2018 года.
Бесплатный доступ
Внутриклеточная агрегация белков является одной из причин многих нейродегенеративных заболеваний. В частности, мутации в гене SOD1 связаны с наследственной формой бокового амиотрофического склероза (БАС). Ранее нами была предложена регрессионная модель, предсказывающая дожитие пациентов с БАС, которые являются носителями мутаций в белке SOD1. Эта модель была построена на основе анализа стабильности водородных связей, возникающих в мутантах SOD1 при моделировании методом молекулярной динамики (МД). В настоящей работе данная регрессионная модель была улучшена с помощью учёта новой характеристики водородных связей, отражающей показатель консервативности водородной связи в пространстве конформационных состояний белка. Расчёт конформационной консервативности водородных связей, осуществлённый с помощью эластичных сетевых (ЭС) моделей позволил увеличить коэффициент корреляции между предсказанными с помощью новой модели и наблюдаемыми значениями дожития пациентов с мутациями в SOD1 (R = 0.92).
Боковой амиотрофический склероз, водородные связи, регрессионные модели, молекулярная динамика, эластичные сетевые модели
Короткий адрес: https://sciup.org/170184758
IDR: 170184758 | DOI: 10.24411/2500-1000-2018-10053
Текст научной статьи Компьютерное предсказание патогенных свойств мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом
Боковой амиотрофический склероз (БАС) – неизлечимое нейродегенеративное заболевание. Выделяется две его формы: наследственная и спорадическая [1]. Известно, что второй по распространённости и наиболее изученной причиной наследственной формы БАС являются мутации в гене SOD1, кодирующем фермент супер-оксиддисмутазу-1 [2]. Одной из гипотез относительно механизма возникновения заболевания на молекулярном уровне является агрегация белка SOD1, вызванная его неверной укладкой в результате мутаций [2].
Известно, что водородные связи играют важную роль, в частности, в поддержании структуры белков [3]. В одном из in silico исследований структуры и динамики SOD1 уже анализировались водородные связи и сеть, которую они образуют в этом белке [4]. Ранее нами с помощью метода молекулярной динамики (МД) было исследовано 39 мутаций в белке SOD1 [5]. Были обнаружены водородные связи, доля времени существования которых в траектории МД (или стабильность) достоверно коррелирует с дожитием пациентов с БАС. На основе найденных водородных связей была построена регрессионная модель, позволяющая предсказать дожитие пациентов с мутациями в SOD1.
Настоящее исследование направлено на расширение ранее созданной модели с учётом крупномасштабной динамики белка SOD1. Для этого проведено моделирование динамики структуры белка с использованием эластичных сетевых моделей (ЭС) [6]. На основе результатов анализа структуры и динамики мутантов SOD1 предложена комплексная регрессионная модель, учитывающая как конформации, полученные с помощью МД, так и конформации, полученных в данной работе с помощью ЭС. Для построения регрессионной модели проводился расчёт конформационной консервативности водородных связей, как отношение числа конформаций, в которых связь присутствовала, к общему числу полученных в ЭС конформаций. Анализ конформационной консервативности водородных связей, позволил улучшить точность расширенной модели по сравнению с ранее созданной моделью. Коэффициент корреляции между предсказанными и наблюдаемыми значениями дожития пациентов, носителей мутаций в SOD1, составил R = 0.92.
Материалы и методы
В работе исследована структура белка SOD1 дикого типа и 35 его мутантных форм, взятые из базы данных ALSOD . Получение пространственных структур мутантных белков SOD1 производилось с помощью моделирования мутаций в структуре PDBID 2V0A с использованием FoldX [7]. Проверка и исправление полученных структур мутантов, включая удаление клэшей и исправление неоптимальных конформаций также проводилась с использованием FoldX.
Построение ЭС для белка дикого типа и его мутантов проводилось с помощью системы ElNemo с параметрами по- умолчанию [8]. В рамках каждой такой модели был получен ансамбль из 11 конформаций для каждой из 5 нетривиальных мод колебаний с наивысшим показателем коллективности. Таким образом, для белка дикого типа и каждого из изучаемых мутантов были рассчитаны 55 конформаций, представляющих крупномасштабные флуктуации структуры этих молекул. С помощью пакета программ AMBER [9] структура, соответствующая каждой конформации впоследствии минимизировалась с целью избежать физически недостижимых торсионных углов, длин связей и других параметров SOD1.
Водородные связи в каждой из 55 конформаций белка находились с помощью утилиты cpptraj из пакета программ AMBER. Статистический анализ стабильности водородных связей проводился с помощью библиотек numpy, scipy и sklearn для языка Python.
Результаты и обсуждение
Следует заметить, что характер движений атомов молекул в моделях ЭС и МД различается. Различия состоят в том, что в моделировании ЭС все конформации молекулы отличаются и соответствуют позициям атомов вдоль выбранной моды колебаний в некоторый промежуток времени. В МД атомы движутся неподалёку от некоторого локального минимума и могут принимать одну и ту же конформацию не один раз. По этой причине доля существования водородной связи среди конформаций белка в ЭС названа конформационной консервативностью, в то время как в нашей предыдущей работе [5] доля конформаций белка в траектории МД, в которых связь была сформирована называлась стабильностью.
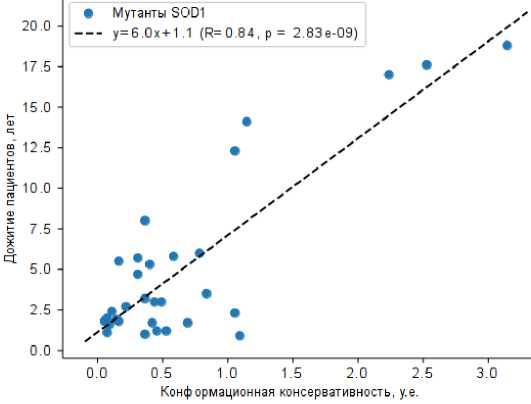
Рис. 1. Корреляция между функцией конформационной консервативности водородных связей, найденных в рамках моделирования эластичных сетей для мутантов SOD1 и дожитием пациентов с БАС – носителей соответствующих мутациями в белке.
Показатель, характеризующий патогенность водородных связей в МД рассчитывался с помощью суммарной стабильности Om,K, описанной нами ранее в работе [5]. Для оценки патогенности водородных связей в ЭС применялась суммарная конформационная консервативность Cu,N, которая рассчитывается на основе
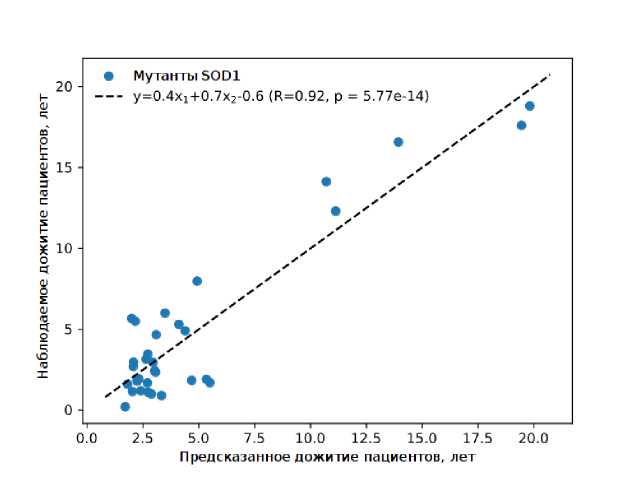
Рис. 2. Корреляция между предсказанным временем жизни пациентов с БАС с мутациями в SOD1, полученным с помощью двухфакторной линейной регрессии на основе конформационной консервативности патологичных водородных связей в ЭС и стабильностей патологичных водородных связей в МД
конформационной консервативности отдельных водородных связей аналогично Om,K. Регрессионная модель включала два независимых фактора – Om,K и Cu,N.
Коэффициент корреляции между функцией конформационной консервативности Cu,N и дожитием пациентов с мутациями в SOD1 составил R = 0.84 (p = 2.8*10-9) (см. рис. 1).
Конформационная консервативность и стабильность водородных связей, соответственно, в ЭС и МД являются различными по своей природе. Поэтому на основе данных двух независимых характеристик была построена множественная регрессия. Коэффициент корреляции для данной регрессии оказался выше (R = 0.92, p = 5.8*10-14), чем для регрессий на основе каждого из параметров в отдельности (см. рис. 2). Таким образом, комбинация результатов моделирования ЭС и МД позволила получить более точную регрессионную модель для предсказания дожития пациентов с мутациями в SOD1, страдающих БАС.
Заключение
Учёт показателя консервативности водородных связей в пространстве конформационных состояний белка SOD1
позволил улучшить созданную ранее модель, которая была построена на использовании информации о стабильности водородных связей. Коэффициент корреляции между предсказанными с помощью новой модели и наблюдаемыми значениями дожития пациентов с мутациями в SOD1 составил R = 0.92, что позволяет объяснить более 84% (R2 = 0,846) дисперсии в дожитии пациентов, носителей рассматриваемых мутаций в SOD1.
Список литературы Компьютерное предсказание патогенных свойств мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом
- Kiernan M.C. et al. Amyotrophic lateral sclerosis // Lancet. 2011. Vol. 377. №9769. P. 942-955.
- Tokuda E., Furukawa Y. Copper Homeostasis as a Therapeutic Target in Amyotrophic Lateral Sclerosis with SOD1 Mutations // Int. J. Mol. Sci. Multidisciplinary Digital Publishing Institute, 2016. Vol. 17, № 5. P. 636.
- Fernández A., Scheraga H.A. Insufficiently dehydrated hydrogen bonds as determinants of protein interactions. // Proc. Natl. Acad. Sci. U. S. A. 2003. Vol. 100, № 1. P. 113-118.
- Khare S.D., Dokholyan N. V. Common dynamical signatures of familial amyotrophic lateral sclerosis-associated structurally diverse Cu, Zn superoxide dismutase mutants. // Proc. Natl. Acad. Sci. U. S. A. 2006. Vol. 103, № 9. P. 3147-3152.
- Alemasov N.A. et al. Dynamic properties of SOD1 mutants can predict survival time of patients carrying familial amyotrophic lateral sclerosis // J. Biomol. Struct. Dyn. Taylor & Francis, 2017. Vol. 35, № 3. P. 645-656.
- Tirion M. Large Amplitude Elastic Motions in Proteins from a Single-Parameter, Atomic Analysis. // Phys. Rev. Lett. American Physical Society, 1996. Vol. 77, № 9. P. 1905-1908.
- Guerois R., Nielsen J.E., Serrano L. Predicting Changes in the Stability of Proteins and Protein Complexes: A Study of More Than 1000 Mutations // J. Mol. Biol. 2002. Vol. 320, № 2. P. 369-387.
- Suhre K., Sanejouand Y.-H. ElNemo: a normal mode web server for protein movement analysis and the generation of templates for molecular replacement. // Nucleic Acids Res. Oxford University Press, 2004. Vol. 32, № Web Server issue. P. W610-4.
- Salomon-Ferrer R., Case D.A., Walker R.C. An overview of the Amber biomolecular simulation package // Wiley Interdiscip. Rev. Comput. Mol. Sci. 2013. Vol. 3, № 2. P. 198-210.


