Метагеномная характеристика ризосферного эффекта при выращивании злаковых культур в черноземной и дерново-подзолистой почв
Автор: Зверев А.О., Першина Е.В., Проворов Н.А., Андронов Е.Е., Серикова Е.Н.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Растение и почва
Статья в выпуске: 5 т.51, 2016 года.
Бесплатный доступ
Ризосферный эффект (изменение состава сообщества микроорганизмов под действием корневой экссудации растений) широко освещен в научной литературе. Убедительно показана его зависимость от таких внешних факторов, как тип почвы, вид и сорт растения и т.д. Задачей настоящей работы стало изучение влияния типа почвы и вида растения на ризосферный эффект при помощи современных методик высокопроизводительного секвенирования. Эта проблема неоднократно рассматривалась иностранными коллегами, однако подобное исследование на российских почвах и культурах, используемых в отечественной агропромышленности, было поведено впервые. В работе использовались две контрастные по агрохимическим показателям почвы - чернозем (Воронежская обл.) и дерново-подзолистая почва (Псковская обл.). Семена ржи (Secale cereale L., к-6469 по каталогу ВИР, местный сорт, используемый в Псковской области) и пшеницы (Triticum aestivum L., сорт Волшебница, к-54609 по каталогу ВИР) (получены из коллекции ВИР, г. Санкт-Петербург) выращивали в вегетационном домике на обеих почвах в течение 42 сут. Используя NGS-секвенирование V4 вариабельного участка гена 16S-рДНК, про-анализировали состав сообществ микроорганизмов исходных почв и сформированных на них ризосфер. Несмотря на небольшую длительность эксперимента, в обеих почвах был выявлен выраженный ризосферный эффект. Наиболее сильным фактором оказался тип почвы. Сообщества исходных почв, а также различные ризосферные сообщества, сформированные на этих почвах, достоверно отличались друг от друга. Для обеих почв показано сохранение этой закономерности при формировании ризосферных сообществ микроорганизмов у ржи и пшеницы. Вид растения был вторым по значимости (после типа почвы) фактором, определяющим таксономический состав ризосферного микробиома. У ржи сообщества ризосферы в целом оказались несколько ближе к сообществам исходных почв, чем у пшеницы. Также сообщества ризосфер ржи на дерново-подзолистой почве, по данным кластерного анализа, близки по структуре к сообществам исходной почвы. Таксономический микробиомный анализ сообществ на уровне фил позволил выявить группы, в наибольшей степени ответственные за ризосферный эффект. Формирование ризосферных сообществ сопровождалось увеличением количества последовательностей, характерных для класса Betaproteobacteria, при снижении числа представителей филы Verrucomicrobia. Сочетание особенностей дерново-подзолистой почвы и выращиваемой на ней пшеницы приводит к значительным изменениям в сообществе. По результатам всех анализов эти сообщества существенно отличаются от сообществ исходной почвы и ризосферы ржи на дерново-подзолистой почве. Возможно, это связано с увеличением доли бактерий из рода Flavobacterium (фила Bacteroidetes) в указанных сообществах. Таким образом, при помощи метода высокопроизводительного секвенирования, обладающего высокой разрешающей способностью и возможностью исследовать в том числе некультивируемые формы микроорганизмов, было наглядно показано наличие ризосферного эффекта при выращивании культурных сортов растений, а также продемонстрированы особенности взаимодействия отдельных формирующих его факторов. Однако для подтверждения ризосферного эффекта, а также более подробного исследования механизмов, лежащих в его основе, необходимо дополнить проведенный нами таксономический анализ исследованиями, характеризующими связь структуры ризосферного микробиома с составом растительных экссудатов. Для этого запланировано проведение ряда модельных экспериментов по внесению в почву веществ, входящих в состав корневых экссудатов исследуемых сортов ржи и пшеницы.
Ризосферный эффект, ризосферный микробиом, ризосфера ржи, ризосфера пшеницы, метагеномный анализ
Короткий адрес: https://sciup.org/142213968
IDR: 142213968 | УДК: 573.22:581.55 | DOI: 10.15389/agrobiology.2016.5.654rus
Текст научной статьи Метагеномная характеристика ризосферного эффекта при выращивании злаковых культур в черноземной и дерново-подзолистой почв
Ризосфера растений представляет собой особую нишу, в которой формируется специфическое для каждого вида растений микробное сообщество (1-4). Структура этого сообщества во многом определяется составом растительных экссудатов, выполняющих как роль субстрата, так и ре-
∗ Работа поддержана Российским научным фондом, грант 14-26-00094.
гуляторные функции (3-6). Благодаря экссудации растение активно взаимодействует с почвенной микробиотой, формируя микробное окружение, обеспечивающее растению ряд адаптивных преимуществ: защиту от патогенов, минеральное питание, адаптацию к абиотическим стрессам, регуляцию развития (3-6). Развитие растения, рост его корней и корневая экссудация — сильный биотический фактор, влияющий на формирование ризосферного микробиома (6-9). Качественное и количественное изменение состава сообщества микроорганизмов под его действием и получило название ризосферного эффекта (6, 8). Показано, что он проявляется неодинаково на разных типах почв (10, 11), в разные фазы развития (12), а также у разных видов растений и даже сортов (10-14). Существенные различия в ризосферном эффекте выявлены также при сравнении культурных растений с исходными дикими формами (14, 15).
Долгое время ризосферный эффект изучали с использованием классических микробиологических методов, в результате чего был накоплен богатый научный опыт относительно как физиологических, так и генетических свойств основных представителей микробиома ризосферы (2, 6, 8). Однако, как известно, в этих исследованиях была охвачена лишь малая часть разнообразия (16, 17). Современные молекулярные методики, в частности высокопроизводительное секвенирование, сделали возможным более детальное изучение микробиома ризосферы, включая не только культивируемых, но и некультивируемых его представителей. Этот метод активно используется зарубежными учеными (18-22), но в отечественных почвенных исследованиях подобного рода работ явно недостаточно. Применение высокопроизводительного секвенирования при изучении ризосферного эффекта позволило убедительно показать роль типа почвы, продолжительности роста и сорта растения (10, 12-14) в определении таксономического состав микробиомов ризосферы. Во всех исследованиях отмечалось, что наибольшее влияние на ризосферный эффект оказывает именно тип почвы (10, 13, 22). Ризосферный эффект анализировали у ряда растений — от модельных объектов, таких как Arabidopsis sp. (17, 18, 21), до видов, имеющих важное сельскохозяйственное значение, например у риса ( Oryza sativa ) (23) и салата-латука ( Lactuca sativa ) (22). Ризосферный эффект в этих случаях проявлялся неодинаково для разных растений. Стоит также отметить, что в большей части сообщений ризосферный эффект изучали для типов почв, близких по структуре и генезису (11, 17, 21).
Поскольку проявление ризосферного эффекта зависит от особенностей растений и типов почв, нашей основной задачей стало расширение круга разнообразия исследуемых объектов. В представленной работе впервые выполнено исследование ризосферного эффекта на распространенных в сельском хозяйстве России объектах (сорта ржи и пшеницы) и часто встречающихся контрастных почвах (чернозем и дерново-подзолистая).
В задачи работы входила оценка ризосферного эффекта в модельном опыте по культивированию растений ржи и пшеницы в почвах разного типа с выявлением характерных таксономических групп бактерий.
Методика . Почву для экспериментов отбирали на участках сельскохозяйственного назначения (разделительные кромки полей, свободные от посевов в течение последних 50 лет) с глубины 3-15 см. Дерновоподзолистая почва была предоставлена Псковским НИИ сельского хозяйства и совхозом «Родина» (Псковская обл., координаты точки обора — 57 ° 50'44,2"N, 28 ° 12'03,7"E). Чернозем получали из Воронежской области (заповедник «Каменная степь», 510 ° 01'41,6"N, 400 ° 43' 39,3"E). Почву просеивали на грохоте с ячейкой 5 мм, подсушивали и расфасовывали в пла-655
стиковые сосуды (по 5,0 кг для чернозема и 5,5 кг для дерново-подзолистой) и увлажняли из расчета 75 % максимальной влагоемкости.
Через 1 сут в каждый сосуд на глубину 3-5 см правильными рядами вносились семена из расчета 25 шт/сосуд. Использовали семена ржи (к6469 по каталогу ВИР, местный сорт, засеваемый только в Псковской области) и пшеницы (сорт Волшебница, к-54609 по каталогу ВИР). На один сорт использовалось по два сосуда с каждым типом почв. Эксперимент проводился в течение 42 сут (с 23 сентября по 4 ноября 2014 года) в теплице, крытой полиэтиленовой пленкой (торцевую стену для обеспечения газообмена закрывали сеткой) при поддержании постоянной влажности почвы (75 % от полной влагоемкости). Средняя дневная температура за время эксперимента — 13 ° С, ночная — 4 ° С. По завершении эксперимента из каждого сосуда отбирали 2 образца корневой массы. Только что отделенные от почвы корни делили на две примерно равные части, помещались во флаконы с водой (50 мл), интенсивно встряхивались в течение 1 мин до получения однородной почвенной суспензии. По 2 мл суспензии отбирали в микропробирку, центрифугировали, осадок использовали для выделения ризосферной ДНК.
ДНК выделяли по методике, разработанной во Всероссийском НИИ сельскохозяйственной микробиологии (25). Концентрация полученного препарата ДНК в среднем составляла 18 нг/мл. Очищенную ДНК использовали в качестве матрицы в ПЦР с универсальными праймерами к вариабельному участку V4 гена 16S-рРНК — F515 GTGCCAGCMGCCGCGG-TAA и R806 GGACTACVSGGGTATCTAAT (26) с добавлением олигонук-леотидных идентификаторов для каждой пробы и служебных последовательностей, необходимых для пиросеквенирования. NGS-секвенирование (next-generation sequencing — секвенирование нового поколения) проводили на приборе GS Junior («Roche», США) согласно рекомендациям производителя.
Данные обрабатывали в программе QIIME 1.8.0 (27). Секвенированные последовательности гена 16S-рРНК анализировали в несколько этапов. На первом осуществляли проверку качества последовательностей: из анализа исключали последовательности длиной менее 200 нуклеотидов, имеющие параметр качества прочтения (quality score) менее 25, содержащие неправильно прочитанные последовательности праймеров и мультиплексных идентификаторов, а также протяженные гомопо-лимерные повторы (более 8 нуклеотидов) и неидентифицированные нуклеотиды. После исключения всех небактериальных и химерных последовательностей полученные библиотеки были нормализованы по числу последовательностей в библиотеке наименьшего размера. В результате всех проведенных процедур отобрали 19440 последовательностей (по 810 в каждой библиотеке). Последовательности со сходством более 97 % объединяли в операционные таксономические единицы (operational taxonomic units, OTUs) с использованием алгоритма denovo (в основе — метод «uclust»). Из каждой OTU выбирали одну последовательность для составления набора репрезентативных последовательностей. На следующем этапе проводили классификацию репрезентативных последовательностей при помощи RDP naive Bayesianr RNA Classifier и выравнивание по алгоритму PyNast (27), матрицей для выравнивания служил специально сконструированный набор последовательностей Greengenes coreset (28). После выравнивания последовательности использовались для построения матрицы дистанций и филогенетического древа.
Для характеристики биоразнообразия и выполнения сравнительного анализа сообществ рассчитывали параметры а - и p -разнообразия. 656
α-Разнообразие оценивали с использованием индексов видового богатства (число OTU в образце) и индекса Шеннона (Shannon, Н). Достоверность различий по индексам α-разнообразия между микробиомами определяли по t-тесту. Для оценки β -разнообразия применяли метод Weighted unifrac, позволяющий выявить процент сходств между всеми парами сравниваемых микробиомов (29). Результаты представляли с использованием методов многомерной статистики PCoA (principal components analysis, анализ главных компонент), данные визуализировали в программе Emperor (входит в QIIME) . При подсчете индексов разнообразия и проведения кластерного анализа использовался критерий Bray-Curtiss, вычисления осуществляли в программе PAST (30). Статистическую поддержку кластеров рассчитывали по методу bootstrap (1000 перестановок).
Различия между образцами по частоте таксонов определяли с помощью точного теста Фишера с поправкой на множественные сравнения по процедуре Бенджамини-Хохберга на 5 % уровне значимости.
Результаты . Использованные нами праймеры сконструированы на основе анализа нуклеотидных последовательностей как бактерий, так и архей и позволяют амплифицировать фрагмент гена 16S-рРНК длиной примерно 400 п.н. В работе были проанализированы микробиомы сообществ в шести вариантах: дерново-подзолистая почва (ДП), чернозем (ЧЗ), ризосфера ржи в дерново-подзолистой почве (рДП), ризосфера пшеницы в дерново-подзолистой почве (пДП), ризосфера ржи в черноземе (рЧЗ) и ризосфера пшеницы в черноземе (пЧЗ).
Индексы разнообразия. Индексы разнообразия, рассчитанные для сообществ почвы и ризосферных сообществ, различались незначительно. Достоверные различия по величине индексов Chao-1 и Шеннона показаны для сообщества ризосферы пшеницы на дерново-подзолистой почве (табл.). Видно, что значения обоих индексов оказались достоверно ниже, чем у сообществ дерново-подзолистой почвы или ризосферы ржи на той же почве.
Индексы α -разнообразия микробиомов почвенных и ризосферных сообществ в зависимости от типа почвы и вида растений ( Х ± х , пшеница Triticum aestivum L. и рожь Secale cereale L., вегетационный опыт)
Индексы разнообразия
|
Микробиом |
видовое богатство (Species richness) |
Chao-1 |
Shannon (Н) |
Ч е р н о з е м (Воронежская обл.)
|
Без растений Ризосфера: |
277±35 |
360±41 |
4,94±0,17 |
|
пшеницы |
219±21 |
310±31 |
4,78±0,13 |
|
ржи |
297±23 |
426±18 |
4,96±0,04 |
|
Дерново-подзолистая почва |
(Псковская обл.) |
||
|
Без растений Ризосфера: |
287±33 |
401±28 |
5,20±0,11 |
|
пшеницы |
248±26 |
335±31 |
4,78±0,07 |
|
ржи |
290±22 |
393±10 |
5,09±0,05 |
П р и м е ч а н и е. Использованы сорта ржи (к-6469 по каталогу ВИР, местный сорт из Псковской области) и пшеницы (сорт Волшебница, к-54609 по каталогу ВИР).
Обнаруженный эффект особенно интересен в связи с имеющимися данными литературы: ранее сообщалось, что достоверно индексы разнообразия ризосферных сообществ не отличаются от индексов разнообразия сообществ исходных почв как в случае разных почв и сортов (9, 10), так и при анализе ризосфер у растений разного возраста (9).
Кластерный анализ и анализ главных компонент (PCoA). На дендрограмме (рис. 1) отчетливо выделялись «черноземный кластер» (включающий исходный чернозем и ризосферы обоих растений, сформи-
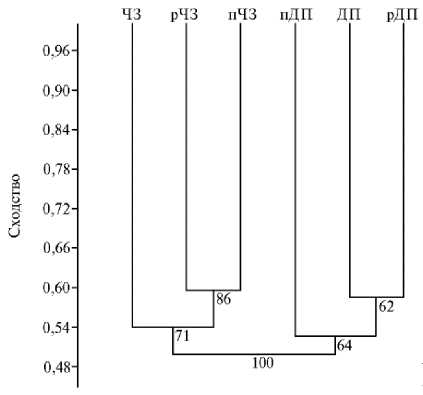
Рис. 1. Кластерный анализ микробиомов почв и ризосферных сообществ, сформированных на этих почвах: ЧЗ и ДП — чернозем и дерново-подзолистая почва; рЧЗ и рДП — ризосферы ржи ( Secale cereale L., к-6469 по каталогу ВИР, местный сорт из Псковской области), пЧЗ и пДП — ризосферы пшеницы ( Triticum aesivum L., сорт Волшебница). Вегетационный опыт. Дендрограмма сходства построена с использованием меры сходства Bray-Curtiss, bootstrap = 1000.
рованные на этой почве) и «дерново-подзолистый кластер». В первом наблюдалось выраженное разделение сообществ на две обособленные группы, соответствующие исходной почве и ризосфере. В этом случае сообщества ризосфер

Рис. 2. Анализ главных компонент (principal components analysis, PCoA) почвенных и образованных на их основе ризосферных микробных сообществ: А — в сравнении с исходной почвой, Б — в исходных почвах и ризосферах на их основе; 1 — исходная почва, 2 — ризосфера ржи ( Secale cereale L., к-6469 по каталогу ВИР, местный сорт из Псковской области), 3 — ризо-
РСЗ (7,07 %)
РС2 (21.43 %)
сфера пшеницы ( Triticum aesivum L., сорт Волшебница); 4 — чернозем, 5 — ризосферы на черноземе, 6 — дерново-подзолистая почва, 7 — ризосферы на дерново-подзолистой почве (вегетационный опыт).
ржи и пшеницы на черноземе по таксономической структуре были более сходны друг с другом, чем с сообществом исходной почвы. В «дерновоподзолистом кластере» наблюдается другая тенденция: в отдельную кладу попала исходная почва и ризосфера ржи на дерново-подзолистой почве. Эти данные хорошо соотносятся с показателями индексов разнообразия, дополнительно свидетельствуя о выраженном ризосферном эффекте при культивировании пшеницы. Однако наблюдаемый эффект, связанный со снижением разнообразия в ризосфере пшеницы на дерново-подзолистой почве, может рассматриваться пока лишь в качестве тенденции, поскольку соответствующий кластер дендрограммы имел относительно низкую статистическую поддержку (не более 64 %). Тем не менее, он воспроизводился при анализе главных компонент.
На графике, представляющем результаты анализа PCoA, можно наблюдать выраженные различия в структуре кластеров, соответствующих чернозему и дерново-подзолистой почве: в первом случае мы наблюдали значительный разброс по повторностям опыта, во втором случае разброс практически отсутствовал (рис. 2). Причина может заключаться в высокой гетерогенности почвы. В то же время не исключено, что глубина секвенирования была недостаточной для сообщества. Также на графике видно, что сообщества ризосфер ржи на дерново-подзолистой почве оказались близки по структуре к сообществам исходной почвы, а отличия демонстрировали лишь сообщества ризосфер пшеницы, составляющие отдельную группу (см. рис. 2, А). Черноземные сообщества не демонстрировали отчетливо обособленных групп. Тем не менее, заметно, что сообщества ризосфер ржи в целом были ближе к сообществам исходных почв, чем сообщества ризосфер пшеницы.
Таксономический состав сообществ. Анализ, проведенный на уровне фил (классов для филы Proteobacteria ), выявил достоверные различия (p < 0,05) в численности последовательностей между вариантами опыта (рис. 3).
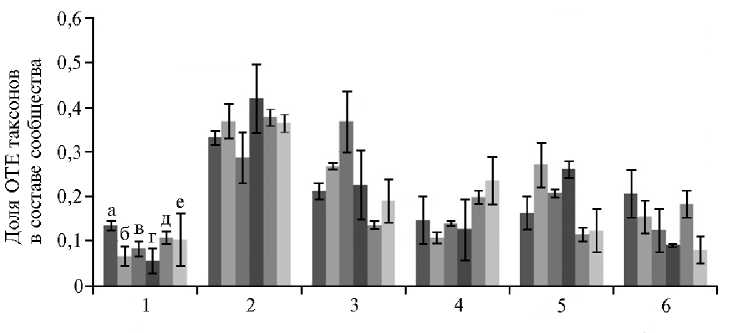
Рис. 3. Таксономический состав (представлен частично) почвенных и образованных на их основе ризосферных микробных сообществ: ОТЕ — оперативные таксономические единицы; 1 — Acidobacteria , 2 — Actinobacteria , 3 — Bacteroidetes , 4 — Verrucomicrobia , 5 — Betaproteobacteria , 6 — Gammaproteobacteria ; а — рожь ( Secale cereale L., к-6469 по каталогу ВИР, местный сорт из Псковской области), дерново-подзолистая почва; б — рожь, чернозем; в — пшеница ( Triticum aesivum L., сорт Волшебница), дерново-подзолистая почва; г — пшеница, чернозем; д — контроль, дерново-подзолистая почва; е — контроль, чернозем. Вегетационный опыт (p < 0,05).
Во всех ризосферных сообществах отмечалось увеличение числа последовательностей, принадлежащих классу Betaproteobacteria (недосто- верно для сообществ ДП-рДП), а также снижение числа представителей филы Verrucomicrobia (недостоверно для сообществ рДП—ДП и пЧЗ—ЧЗ). Достоверно большее количество последовательностей, принадлежащих классу Gammaproteobacteria, отличало сообщества дерново-подзолистой почвы от сообществ чернозема, однако в сообществах ризосфер этих почв такая тенденция утрачивалась. В сообществах ризосферы пшеницы на дерновоподзолистой почве наблюдали повышение доли представителей филы Bacteroidetes. Их оказалось достоверно меньше в сообществе ризосферы ржи в дерново-подзолистой почве, еще меньше — в самой дерново-подзолистой почве. Также было показано уменьшение числа представителей филы Actinobacteria в ризосфере пшеницы на дерново-подзолистой почве в сравнении с исходной почвой. Сообщества ризосферы ржи на дерново-подзолистой почве отличались от сообществ ризосферы пшеницы на той же почве достоверно большим количеством представителей филы Actinobacteria.
Из данных литературы известно, что на разных жизненных стадиях растения преимущество получают разные группы микроорганизмов. При изучении корневой экссудации арабидопсиса основные различия найдены в четырех филах — Acidobacteria , Actinobacteria , Bacteroidetes и Cyanobacteria , причем для численности представителей филы Bacteroidetes показана положительная корреляция с количеством аминокислот, секретируемых корнями этого растения, и обратная — с фенольными соединениями (11).
По всей видимости, именно увеличение числа представителей Bac-teroidetes в ризосферном сообществе пшеницы на дерново-подзолистой почве по сравнению с исходной почвой привело к выраженной разнице в показателях α -разнообразия. Статистический анализа показал, что различия в составе сообществ в этом случае связаны с увеличением (более чем в 12,6 раза, p < 0,05) численности микроорганизмов из рода Flavobacterium .
Более подробный статистический анализ таксономической структуры микробных сообществ позволил также выявить группы микроорганизмов, обусловившие различия в формировании ризосферных микробиомов в других вариантах опыта. В частности, по сравнению с исходной почвой (ЧЗ) в ризосфере рЧЗ существенно увеличилась доля родов Pedo-bacter (в 47,6 раза) и Chitinophaga (в 76,9 раза), в ризосфере пЧЗ — рода Pedobacter (в 10,0 раза) и Kaistobacter (в 14,2 раза). В ризосфере рДП (по сравнению с ДП) отмечали увеличение доли родов Pseudomonas (в 48,6 раза) и Achromobacter (в 25,0 раза), в ризосфере пДП — Mesorhizobium (в 40,0 раза) и Chitinophaga (53,7 раза). Таким образом, на разных почвах состав ризосферного сообщества был неодинаковым. Эти результаты согласуются с данными литературы о том, что ризосферы растений, культивируемых на почвах разных типов, существенно различаются по составу на родовом уровне. Так, при выращивании салата на трех разных почвах показано увеличение количества представителей семейств Sphingomonas и Rhizobium ( α -протеобактерии), Pseudomonas ( γ -протеобактерии), Variovorax ( β -протеобактерии) и Flavobacterium ( Bacteroidetes ), однако для каждой почвы также характерны уникальные семейства, изменяющие свою численность только в ризосферах на этом типе почвы (11).
Стоит подчеркнуть, что в настоящее время не существует единой признанной в мире методики как для разделения сообществ ризосферы и ризопланы, так и для выделения ДНК из почвы и ее дальнейшего анализа. Поэтому полученные нами данные достаточно сложно сравнивать с описанными в литературе. Заметим, например, что более тщательный анализ сообществ прикорневой зоны, разделяющей ризосферу и ризоплану, свидетельствует, что сообщество ризосферы имеет сходный таксономический состав с сообществом исходных почв, главные же различия наблюдаются в 660
ризоплане и сводятся к уменьшению доли фил Acidobacteria , Planctomycetes , и G emmatimonadetes (24). Поэтому уже сейчас понятно, что для более полной оценки ризосферного эффекта требуется разработка универсальной методики отбора проб с включением дополнительных методов, позволяющих более точно локализовать отдельных представителей ризосферного сообщества (например, технологии FISH — fluorescence in situ hybridization и др.).
Таким образом, несмотря на относительно короткую продолжительность эксперимента (42 сут), сообщества исходных почв и ризосфер сильно различались, то есть в обеих почвах был выявлен выраженный ризосферный эффект. На формирование ризосферного сообщества существенно влияет как тип почвы, так и вид растения. Наиболее сильным фактором оказался тип почвы: и ризосферные сообщества, сформированные в различных типах почвы, и исходные почвенные сообщества достоверно отличаются друг от друга. Для обеих почв показано сохранение этих различий при формировании разных ризосферных сообществ. Вид растения был вторым по значимости (после типа почвы) фактором, определяющим таксономический состав ризосферного микробиома. Сообщества ризосферы ржи в целом оказываются несколько ближе к сообществам исходных почв, чем сообщества ризосфер пшеницы.
Таксономический анализ сообществ на уровне фил позволил выявить группы, в наибольшей степени ответственные за ризосферный эффект: формирование ризосферных сообществ сопровождалось увеличением количества последовательностей класса Betaproteobacteria при снижении количества представителей филы Verrucomicrobia .
Сочетание особенностей дерново-подзолистой почвы и выращиваемой на ней пшеницы приводило к значительным изменениям в сообществе. По результатам всех анализов эти сообщества существенно отличаются от сообществ исходной почвы и сообществ ризосферы ржи на дерново-подзолистой почве. Подобное может быть связано с увеличением в таких сообществах доли бактерий из рода Flavobacterium (фила Bacteroidetes ).
Итак, при помощи метода высокопроизводительного секвенирования, обладающего высокой разрешающей способностью и возможностью исследовать в том числе некультивируемые формы микроорганизмов, было показано наличие ризосферного эффекта в почве при выращивании культурных сортов растений. Однако для подтверждения наличия этого эффекта, а также для более подробного исследования механизмов, лежащих в его основе, необходимо дополнить проведенный нами таксономический анализ исследованиями, характеризующими связь структуры ризосферного микробиома с составом растительных экссудатов. Для этого запланированы модельные эксперименты по внесению в почву компонентов растительных экссудатов исследуемых сортов ржи и пшеницы.
Список литературы Метагеномная характеристика ризосферного эффекта при выращивании злаковых культур в черноземной и дерново-подзолистой почв
- Костычев С.П. Физиология растений. Ч. 2. М.-Л., 1933.
- Генкель П.А. Физиология растений с основами микробиологии. М., 1962.
- Philippot L., Raaijmakers J.M., Lemanceau P., van der Putten W.H. Going back to the roots: the microbial ecology of the rhizosphere. Nat. Rev. Microbiol., 2013, 11(11): 789-799 ( ) DOI: 10.1038/nrmicro3109
- Rovira A.D. Plant root exudates. Bot. Rev., 1969, 35: 35-57.
- Hale M.G., Moore L.D., Griffin G.J. Root exudates and exudation. In: Interaction between non-pathogenic soil microorganisms and plants/Y.R. Dommergues, S.V. Krupa (eds.). Amsterdam, 1978: 163-203.
- Тихонович И.А., Проворов Н.А. Симбиозы растений и микроорганизмов: молекулярная генетика агросистем будущего. СПб, 2009.
- Кравченко Л.В., Шапошников А.И., Макарова Н.М., Азарова Т.С., Львова К.А., Костюк И.И., Тихонович И.А. Видовые особенности состава корневых выделений растений и его изменение в ризосфере под влиянием почвенной микрофлоры. Сельскохозяйственная биология, 2011, 3: 71-75.
- Шапошников А.И., Белимов А.А., Кравченко Л.В., Виванко Д.М. Взаимодействие ризосферных бактерий с растениями: механизмы образования и факторы эффективности ассоциативных симбиозов (обзор). Сельскохозяйственная биология, 2011, 3: 16-22.
- Bais H.P., Weir T.L., Perry L.G., Gilroy S., Vivanco J.M. The role of root exudates in rhizosphere interactions with plants and other organisms. Annu. Rev. Plant Biol., 2006, 57: 233-266 (doi: 10.1146/annurev.arplant.57.032905.105159).
- Wieland G., Neumann R., Backhaus H. Variation of microbial communities in soil, rhizosphere, and rhizoplane in response to crop species, soil type, and crop development. Appl. Environ. Microbiol., 2001, 67(12): 5849-5854 ( ) DOI: 10.1128/AEM.67.12.5849-5854.2001
- Schreiter S., Ding G.C., Heuer H., Neumann G., Sandmann M., Grosch R., Kropf S., Smalla K. Effect of the soil type on the microbiome in the rhizosphere of field-grown lettuce. Front. Microbiol., 2014, 5: 144 ( ) DOI: 10.3389/fmicb.2014.00144
- Chaparro J.M., Badri D.V., Vivanco J.M. Rhizosphere microbiome assemblage is affected by plant development. ISME J., 2014, 8(4): 790-803 ( ) DOI: 10.1038/ismej.2013.196
- Winston M.E., Hampton-Marcell J., Zarraonaindia I., Owens S.M., Moreau C.S., Gilbert J.A., Hartsel J.A., Kennedy S.J., Gibbons S.M. Understanding cultivar-specificity and soil determinants of the Сannabis microbiome. PLoS ONE, 2014, 9(6): e99641 ( ) DOI: 10.1371/journal.pone.0099641
- Bulgarelli D., Garrido-Oter R., Münch P.C., Weiman A., Dröge J., Pan Y., McHardy A.C., Schulze-Lefert P. Structure and function of the bacterial root microbiota in wild and domesticated barley. Cell Host Microbe, 2015, 17(3): 392-403 ( ) DOI: 10.1016/j.chom.2015.01.011
- Coleman-Derr D., Desgarennes D., Fonseca-Garcia C., Gross S., Clingenpeel S., Woyke T., North G., Visel A., Partida-Martinez L.P., Tringe S.G. Plant compartment and biogeography affect microbiome composition in cultivated and native Agave species. New Phytol., 2016, 209(2): 798-811 ( ) DOI: 10.1111/nph.13697
- Handelsman J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol. Mol. Biol. Rev., 2004, 68: 669-685 ( ) DOI: 10.1128/MMBR.68.4.669-685.2004
- Lundberg D.S., Lebeis S.L., Paredes S.H., Yourstone S., Gehring J., Malfatti S., Tremblay J., Engelbrektson A., Kunin V., delRio T.G., Edgar R.C., Eickhorst T., Ley R.E., Hugenholtz P., Tringe S.G., Dangl J.L. Defining the core Arabidopsis thaliana root microbiome. Nature, 2012, 488(7409): 86-90 ( ) DOI: 10.1038/nature11237
- Micallef S.A., Shiaris M.P., Colón-Carmona A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot., 2009: 60(6): 1729-1742 ( ) DOI: 10.1093/jxb/erp053
- van der Heijden M.G., Schlaeppi K. Root surface as a frontier for plant microbiome research. PNAS USA, 2015, 112(8): 2299-2300 ( ) DOI: 10.1073/pnas.1500709112
- Bonito G., Reynolds H., Robeson M.S. 2nd, Nelson J., Hodkinson B.P., Tuskan G., Schadt C.W., Vilgalys R. Plant host and soil origin influence fungal and bacterial assemblages in the roots of woody plants. Mol. Ecol., 2014, 23(13): 3356-3370 ( ) DOI: 10.1111/mec.12821
- Bodenhausen N., Horton M.W., Bergelson J. Bacterial communities associated with the leaves and the roots of Arabidopsis thaliana. PLoS ONE, 2013, 8(2): e56329 ( ) DOI: 10.1371/journal.pone.0056329
- Neumann G., Bott S., Ohler M.A., Mock H.P., Lippmann R., Grosch R., Smalla K. Root exudation and root development of lettuce (Lactucasativa L. cv. Tizian) as affected by different soils. Front. Microbiol., 2014, 5: 2 ( ) DOI: 10.3389/fmicb.2014.00002
- Donn S., Kirkegaard J.A., Perera G., Richardson A.E., Watt M. Evolution of bacterial communities in the wheat crop rhizosphere. Environ. Microbiol., 2015, 3: 610-621 ( ) DOI: 10.1111/1462-2920.12452
- Edwards J., Johnson C., Santos-Medellín C., Lurie E., Podishetty N.K., Bhatnagar S., Eisen J.A., Sundaresan V. Structure, variation, and assembly of the root-associated microbiomes of rice. PNAS USA, 2015, 112(8): E911-E920 ( ) DOI: 10.1073/pnas.1414592112
- Андронов Е.Е., Пинаев А.Г., Першина Е.В., Чижевская Е.П. Научно-методические рекомендации по выделению высокоочищенных препаратов ДНК из объектов окружающей среды. СПб, 2011.
- Bates S.T., Berg-Lyons D., Caporaso J.G., Walters W.A., Knight R., Fierer N. Examining the global distribution of dominant archaeal populations in soil. ISME J., 2011, 5: 908-917 ( ) DOI: 10.1038/ismej.2010.171
- Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336 ( ) DOI: 10.1038/nmeth.f.303
- DeSantis T.Z., Hugenholtz P., Larsen N., Rojas M., Brodie E.L., Keller K., Huber T., Dalevi D., Hu P., Andersen G.L. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Appl. Environ. Microbiol., 2006, 72(7): 5069-5072 ( ) DOI: 10.1128/AEM.03006-05
- Lozupone C.A., Knight R. Global patterns in bacterial diversity. PNAS USA, 2007, 104(27): 11436-11440 ( ) DOI: 10.1073/pnas.0611525104
- Hammer O., Harper D., Ryan P. PAST: Paleontological Statistics software package for education and data analysis. Paleontologia Electronica, 2001, 4(1): art. 4 (9pp).


