МЕТОДИКА ОЦЕНКИ КАЧЕСТВА ГЕНОМНОЙ СБОРКИ НА ОСНОВЕ АНАЛИЗА ЧАСТОТНОСТИ K-МЕРОВ В СЕКВЕНАТОРЕ ПАРАЛЛЕЛЬНОГО СЕКВЕНИРОВАНИЯ
Автор: А. Г. Бородинов, В. В. Манойлов, И. В. Заруцкий, А. И. Петров, В. Е. Курочкин
Журнал: Научное приборостроение @nauchnoe-priborostroenie
Рубрика: Системный анализ приборов и измерительных методик
Статья в выпуске: 1 т.32, 2022 года.
Бесплатный доступ
В настоящее время в связи с развитием приборостроения для проведения генетического анализа существует острая необходимость в разработке методик оценки качества геномной сборки. Подсчет встречаемости различных k-меров часто возникает в задачах сборки генома. В данной работе на основе анализа различных программных средств выбраны программы, которые позволяют оценить качество геномной сборки. С помощью выбранных программ обработаны данные, полученные на отечественном секвенаторе параллельного секвенирования Нанофор СПС. На основе результатов обработки этих данных произведена оценка качества геномной сборки по методике анализа k-меров для прибора Нанофор СПС
K-мер, NGS-методы, биоинформаика, сборка генома
Короткий адрес: https://sciup.org/142231103
IDR: 142231103 | УДК: 543.51+ 681.2–5 | DOI: 10.18358/np-32-1-e107
Текст научной статьи МЕТОДИКА ОЦЕНКИ КАЧЕСТВА ГЕНОМНОЙ СБОРКИ НА ОСНОВЕ АНАЛИЗА ЧАСТОТНОСТИ K-МЕРОВ В СЕКВЕНАТОРЕ ПАРАЛЛЕЛЬНОГО СЕКВЕНИРОВАНИЯ
K -мер — это просто последовательность из k символов в строке (или нуклеотидов в последовательности ДНК в задаче секвенирования). Разложение последовательности на ее k -меры позволяет анализировать этот набор фрагментов фиксированного размера, а не последовательность целиком, и это может быть более эффективным подходом. Простой пример: чтобы проверить, происходит ли последовательность S из организма A или из организма B, предполагая, что геномы A и B известны и достаточно разные, мы можем проверить, содержит ли S больше k -меров, присутствующих в A или в B.
Практически любой геном содержит повторяющиеся области, однако, начиная с определенного значения k , k -меры определенным образом однозначно идентифицируют его; если мы посчитаем количество появлений k -мер для достаточно большого k (ограниченного сверху длиной чтения), оказывается, что большинство из них находятся в геноме в единственном экземпляре. Например, если порядок длины генома сравним с человеческим, вероятность встретить случайную подстроку длины 14 хотя бы один раз составляет 0.975893 [1]. Для k = 20 эта же вероятность составляет 0.000909.
Подсчет встречаемости различных k-меров часто возникает в задачах сборки генома. Распределение частот встречаемости используется для процедуры корректирования ридов, что подразумевает разделение содержащихся k-меров на "доверенные" и "ошибочные" [1]. Подобная информация генома используется некоторыми программами сборки генома для определения того, является ли рассматриваемый участок повтором или нет.
В настоящее время в связи с развитием приборостроения для проведения генетического анализа существует острая необходимость в разработке методик оценки качества геномной сборки. Такие методики позволят оценить достоверность проведения генетического анализа в существующих и вновь разрабатываемых приборах. В данной работе на основе анализа различных программных средств выбраны программы, которые позволяют оценить качество геномной сборки в секвенаторах параллельного секвенирования. С помощью выбранных программ обработаны данные, полученные на отечественном секвенаторе параллельного секвенирования Нанофор СПС.
АНАЛИЗ ПРОГРАММНЫХ СРЕДСТВ ОЦЕНКИ КАЧЕСТВА СБОРКИ ГЕНОМА
Поскольку количество k -мер растет экспоненциально для значений k , подсчет k -мер для больших значений k является вычислительно сложной задачей. Хотя достаточно простые реализации работают для малых значений k , их необходимо адаптировать для приложений с высокой пропускной способностью или когда k велико. Для решения этой проблемы были разработаны различные инструменты:
-
• Jellyfish использует многопоточную хеш-таблицу без блокировок для подсчета k -мер и имеет реализации на Python, Ruby и Perl [2];
-
• KMC — это инструмент для подсчета k -мер, который использует многодисковую архитектуру для оптимизации скорости [3];
-
• Gerbil использует подход хеш-таблицы, но с дополнительной поддержкой ускорения графического процессора [4];
-
• K-mer Analysis Toolkit (KAT) использует модифицированную версию Jellyfish для анализа количества k -мер [5].
В качестве основного инструмента работы с k -мерами был выбран KAT (K-mer Analysis Tookit), представляющий эффективный набор средств для быстрого подсчета, сравнения и анализа спектров k -мер произвольной длины из данных генетических последовательностей.
Основным методом анализа при работе с k -мерами является проверка качества сборки генома путем сравнения характеристик k -меров совокупности анализируемых р и дов с референтным образцом или с собранным геномом (при сборке de novo ). Инструмент KAT hist — это графическое представление набора данных, показывающее, сколько коротких последовательностей фиксированной длины ( k -мер) появляется определенное количество раз. Частота встречаемости нанесена на ось х , а число k -меров на оси у . Пример 31-mer spectrum of S.cerevisae S288C WGS приведен на рис. 1.
Инструмент KAT comp генерирует матрицу с k-мерным набором последовательностей частот k-меров на одной оси, а частотой встречаемости k-меров другого набора на другой оси. При сравнении набора ридов со сборкой KAT сначала вычисляет свойства и состав k-меров сборки. При представлении в виде стоковых гистограмм спектр к-меров для ридов разбивается по числу копий к-меров для сборки. Кроме того, KAT предоставляет инструмент sect для отслеживания покрытия k-мерами, исходя из рассчитанных спектров k-меров для совокупности ридов и референса. Это может помочь идентифицировать такие артефакты сборки, как события сворачивания и разворачивания, или обнаруживать повторяющиеся области в последовательности ДНК.
KAT также включает инструмент hist для вычисления спектра из одного набора последовательностей и инструмент gcp для анализа гуанин-цитозин содержания (GC-контента) в зависимости от частоты k -меров. Инструмент filter можно использовать для выделения последовательностей из полного набора в соответствии либо с покрытием k -мерами или GC-содержанием для заданного набора. Эти инструменты могут использоваться для различных задач, включая обнаружение и извлечение загрязняющих веществ (contaminant detection) как в необработанных р и дах, так и в сборках (assemblies), анализ смещения по GC-составу и согласованность между парноконцевыми (paired end) р и дами с чувствительностью по концентрациям примесей от 0.1 ppm.
1 000 000
900 000 ■
800 000 ■
700 000 -. * •
600 000 ■ •'
Е• g 500 000
400 000 •
300 000 ■,
200 000-• юоооо ■ ,•
01-------------1-------------1-------------1-------------1f-
0 5 10 15 2025
30 35 40 45 50 55 60
Freq
Рис. 1. Графическое представление набора данных KAT hist
KAT прост в использовании, обеспечивает высокую скорость анализа. Время получения результатов анализа составляет не более минуты.
МЕТОДИКИ РАБОТЫ С K -МЕРАМИ
В работе [1] предложен метод оценки качества геномной сборки, заключающийся в установлении соответствия между уникальными k -мерами в собранном геноме и к -мерами в р и дах. Процедура выглядит следующим образом.
-
1. Построение гистограммы встречаемости k -меров для р и дов.
-
2. Выбор окрестности пика уникальных k -меров на гистограмме встречаемости.
-
3. Построение гистограммы встречаемости k -меров для каждой сборки.
-
4. Расчет меры Q как доли различных k -меров, взятых из окрестности пика на гистограмме встречаемости k -меров в чтениях.
-
5. Выбор сборки с максимальным значением Q в качестве наилучшей.
В работе [6] предложен метод исправления ошибок, оптимизированный для работы с чтениями, содержащими как ошибки замены, так и ошибки вставки и удаления. Поскольку ошибки происходят с небольшой частотой, вероятность того, что один и тот же k -мер будет прочитан несколько раз с одинаковым набором ошибок, очень мала. Из этого вытекает, что те k -меры, которые встречаются в наборе чтений мало раз, являются ошибочными, остальные же являются реальными подстроками генома (рис. 2).
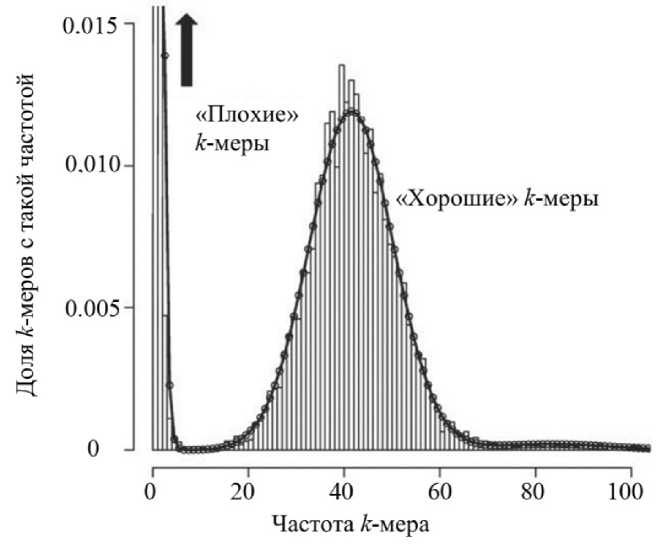
Рис. 2. Распределение частот к -меров в р и дах [6]
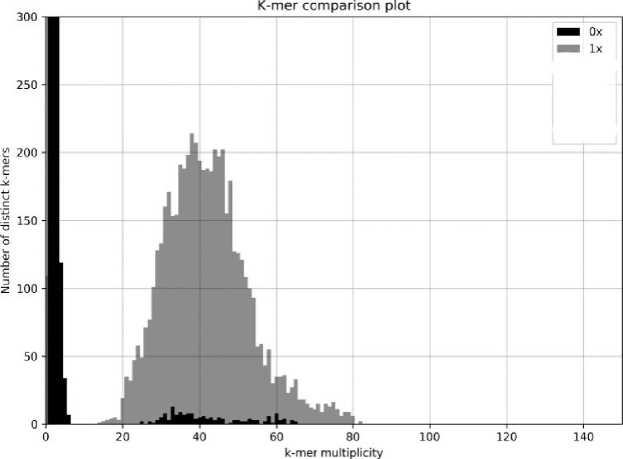
Рис. 3. Типичный k -mer comparison plot секвенирования Phix 174 на Illumina Miseq
ИСПОЛЬЗОВАНИЕ ПРОГРАММЫ KAT ДЛЯ ОБРАБОТКИ ДАННЫХ
СЕКВЕНАТОРА НАНОФОР СПС
Для обработки данных секвенатора Нанофор СПС была использована опция программы КАТ "K-mer comparison plot". По сути мы представляем, сколько элементов каждой частоты в спектре ридов оказались не включены в референтный ге- ном (в нашем случае Phix174), включены один раз, включены дважды и т.д.
На рис. 3, 4 представлены k-mer comparison plot, полученные соответственно для приборов Illumina и Нанофор СПС. Показательно, что для сходных характеристик проточных ячеек запуск Нанофор СПС обеспечивает больший уровень покрытия р и дами референсной последовательности (центр тяжести k -меров с уникальным покрытием).
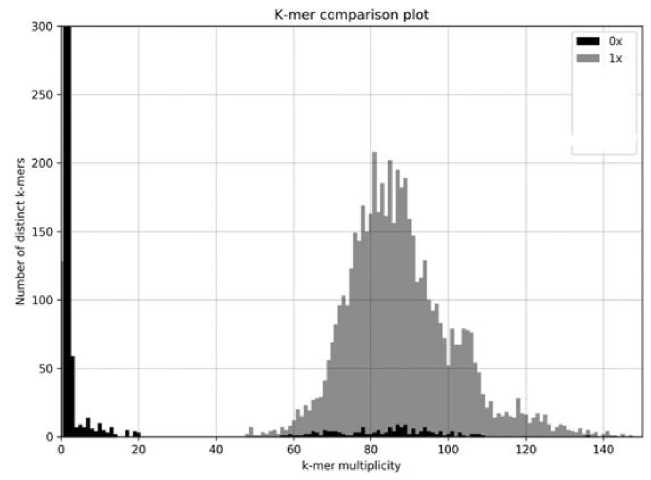
Рис. 4. Типичный k -mer comparison plot секвенирования Phix 174 на Нанофор СПС
ЗАКЛЮЧЕНИЕ
Проекты сборки генома обходятся дорого как по времени, так и по вложенным средствам. В этом случае выявление проблем с экспериментальными данными, обаруженных уже после сборки, может стать настоящей неудачей. С помощью K-mer Analysis Toolkit (KAT) исследователи могут получить доступ к качественным критериям и подтвердить свои результаты на более ранних этапах.
K -меры представляют собой небольшие фрагменты исходного генома с фиксированным числом оснований ДНК. Компьютер может эффективно работать с большим количеством k -меров, а затем идентифицировать связи между этими фрагментами, чтобы создать представление об исходном геноме. Основанные на k -мерах методы обычно используются для эффективного создания геномных сборок. KAT построен для изучения и сравнения наборов данных секвенирования с использованием основных свойств каждого отдельного k -мера, таких как частота встречаемости и нуклеотидный состав.
В первую очередь KAT может анализировать данные секвенирования для определения уровней случайных ошибок, систематических ошибок и контаминации. Информация, полученная в ходе этого анализа, может помочь исследователям решить, следует ли продолжать выполнение последующих задач, таких как сборка генома. Затем KAT может перепроверить проведенную сборку генома, определив полноту и точность сборки без каких-либо внешних справочных данных.


