Микробиом почв Кингисеппского месторождения фосфоритов при разных типах горнотехнической и биологической рекультивации
Автор: Кимеклис А.К., Дмитракова Я.А., Першина Е.А., Иванова Е.А., Зверев А.О., Гладков Г.В., Кичко А.А., Андронов Е.Е., Абакумов Е.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Микробные сообщества почв
Статья в выпуске: 1 т.55, 2020 года.
Бесплатный доступ
Микробный состав нарушенных почвенных покровов после рекультивации может указывать на степень восстановления и характер протекающих в них процессов, а также на пригодность таких почв для дальнейшего использования в сельскохозяйственном обороте. Кингисеппский карьер по добыче фосфоритов разрабатывался в 1960-е годы, а в конце 1970-х была проведена его горнотехническая рекультивация. Уникальность этого объекта в том, что мониторинг почвенных показателей здесь ведется уже на протяжении 29 лет, а при рекультивации использовались три разные древесные породы - ель, лиственница и сосна, причем на участке с елью глыбистую массу разравнивали с внесением торфяно-минеральной смеси, тогда как при высаживании лиственницы и сосны использовали минеральный субстрат без торфа. Однако анализ микробного состава почвенного покрова на рекультивационных площадках до сих пор не проводился. В представленном исследовании мы показали, что структура изученного почвенного микробиома не зависела от физико-химических параметров почв, разнообразие почвенного микробиома не коррелировало с основными минеральными элементами питания, а доминирующая растительная порода существенно не повлияла на структуру микробиома...
Месторождение фосфоритов, горнотехническая рекультивация, метагеном, микробиом почв, биологическая активность, высокопроизводительное секвенирование, 16s ррнк, почвообразование
Короткий адрес: https://sciup.org/142223777
IDR: 142223777 | УДК: 631.618:631.46:577.2 | DOI: 10.15389/agrobiology.2020.1.137rus
Текст научной статьи Микробиом почв Кингисеппского месторождения фосфоритов при разных типах горнотехнической и биологической рекультивации
Повсеместное увеличение территорий, где отсутствует почвенно-
Работа подержана грантом РНФ ¹ 17-16-01030.
растительный покров, — актуальная экологическая проблема современности. Сформировавшиеся карьерно-отвальные комплексы нуждаются в восстановлении и дорогостоящей рекультивации, в то же время это хорошие площадки для исследований почвообразования и процессов первичной сукцессии (1). Такие наблюдения важны в прикладном аспекте, поскольку рекультивированные почвы снова могут обрести сельскохозяйственное значение (2), а с точки зрения фундаментальной науки изучение педогенеза позволит приблизиться к пониманию эволюции почв.
Как правило, при восстановлении экосистем основное внимание уделяется изучению физико-химических параметров почв (3) или растительному покрову (4, 5). Однако в итоге ключевым фактором в процессах почвообразования остается почвенный микробиом (6, 7). Наиболее широкое представление о микробиоме дают методы высокопроизводительного секвенирования (8-10), позволяющие наиболее полно описать микробный состав образцов (11). При этом состав микробиома может меняться в значительной степени в зависимости от различных биотических и абиотических воздействий (12). К одним из самых сильных факторов, влияющих на микробиом почвы, относится pH и влажность почвы (13, 14). Немаловажная роль отводится разнообразию растительного покрова (15-17). В частности, ранее мы показали, что при относительно небольшом разбросе значений физико-химических характеристик в пределах одного типа почв формирование определенного профиля почвенного микробиома в значительной степени определяется структурой и составом растительного фитоценоза (18), причем различия наблюдались преимущественно на низком таксономическом уровне (порядки, семейства и роды).
Таким образом, вопрос о том, как происходит реабилитация почвенного покрова и как при этом изменяется его микробиом, остается актуальным. В настоящее время формирование и разнообразие микробных сообществ на нарушенных территориях в большей степени связывают именно с определяющей ролью растительных сообществ (19). Поскольку процесс реабилитации нарушенных почвенных покровов занимает продолжительное время, для почвенных исследований необходимо выбирать объекты, за которыми ведется долгосрочный мониторинг. Примером может служить Кингисеппский карьер, где в 1960-е годы добывали фосфориты, а в конце 1970-х была проведена горнотехническая рекультивация. Уникальностью этого объекта в том, что мониторинг почвенных показателей здесь осуществляется уже на протяжении 29 лет, а рекультивация проводилась с использованием трех разных древесных пород (20). Однако до сих пор не проводился анализ микробного состава на участках рекультивации, который позволил бы оценить степень восстановления почвенного покрова.
В настоящем исследовании мы показали, что на рекультивационных участках Кингисеппского месторождения фосфоритов структура почвенного микробиома не зависела от физико-химических параметров почв, разнообразие почвенного микробиома не коррелировало с основными минеральными элементами питания, а доминирующая растительная порода не оказала существенного влияния на структуру микробиома.
Целью нашей работы было описание количественного состава микробиома рекультивируемых почв и поиск закономерностей, связывающих формируемую структуру микробиома с физико-химическими почвенными параметрами и доминирующим типом древесной растительности.
Методика . Исследования проводили в 2016 году в карьере ¹ 3 ПО «Фосфорит» (Кингисеппское месторождение фосфоритов, запад Ленинградской обл., Кингисеппский р-н, между городами Кингисепп и Ивангород). 138
Пробы отбирали на трех участках (¹¹ 1, 2 и 3) площадью 20½40 м, подвергнутых в 1970-х годах горнотехнической рекультивации. Возраст участков составлял соответственно 37, 32 и 28 лет. В 2014 году выполняли геоботанические описания участков согласно доминантной схеме, в 2016 году на них изучали начальное (рецентное) почвообразование.
На каждом участке заложили по 3 почвенных разреза. При описании почвенных профилей применяли субстантивно-морфологический подход, основанный на идентификации почвенных таксонов по совокупности морфологических признаков. Из каждого горизонта отбирали по 3 пробы массой 100 г для последующих лабораторных анализов. Образцы из каждого верхнего органоминерального горизонта массой 15 г использовали для микробиологического исследования.
Гранулометрический состав определяли пипет-методом Качинского с пирофосфатной пептизацией микроагрегатов (21), содержание органического углерода — методом Тюрина по бихроматной окисляемости (22), рН водной вытяжки — потенциометрически в соотношении почва-вода 1:2,5. Концентрацию подвижных соединений фосфора и калия измеряли по Кирсанову в модификации ЦИНАО (23), обменный аммоний — по методу ЦИНАО (24), нитраты — ионометрически (25). Величину субстрат-индуци-рованного дыхания (СИД, измерение основано на регистрации дополнительного выделения СО2 в ответ на внесение субстрата — глюкозы) и базального дыхания (БД, определяется по той же методике, что и СИД, но без обогащения нативной почвы глюкозой) оценивали согласно описанию (26).
ДНК выделяли в 4 повторностях для каждого участка по разработанной ранее методике (27) с использованием в качестве абразивного материала стеклянных шариков разного диаметра. Почвенные образцы обрабатывали на гомогенизаторе Precellys 24 («Bertin Technologies», Франция). Чистоту и количество ДНК в препарате определяли электрофорезом в 1 % агарозе в ½0,5 TAE буфере (средняя концентрация ДНК в пробе — 50 нг/мл). Очищенные препараты ДНК использовали для количественной ПЦР (qPCR) и создания ампликонных библиотек согласно инструкции к протоколу секвенирования («Illumina, Inc.», США).
Для выявления в почве трех основных таксономических групп микроорганизмов проводили количественную ПЦР (qPCR) со следующими парами праймеров: к фрагменту 16S рДНК бактерий — EUB338 (5'-ACT-CCTACGGGAGGCAGCAG-3') и EUB518 (5'-ATTACCGCGGCTGCTGG-3') (28, 29), к фрагменту 16S рДНК архей — ARC915f (5'-AGGAATTGGCG-GGGGAGCAC-3') и ARC1059r (5'-GCCATGCACCWCCTCT-3') (30), к фрагменту ITS грибов — ITS1f (5'-TCCGTAGGTGAACCTGCGG-3') и 5.8S (5'-CGCTGCGTTCTTCATCG-3') (31). Для приготовления реакционной смеси использовали набор qPCRmix-HS SYBR («Евроген», Россия) согласно инструкции производителя. Стандартами служили серии 10-кратных разведений фрагментов гена 16S рРНК E . coli и H . pilori , а также фрагмента ITS1 S. cerevisiae . Каждая проба смеси для ПЦР, включая стандарты, была проанализирована в 3-кратной повторности. Измерения проводили на амплификаторе CFX96 («Bio-Rad», Германия) по следующему протоколу: 3 мин при 95 °С; 20 с при 95 °С, 20 с при 50 °С, 20 с при 72 °С (40 циклов). Для повторностей (как самой ПЦР, так и для разных образцов ДНК по одному участку) вычисляли средние значения ( M ) и ошибки среднего (±SEM). После пересчета с использованием стандартов результаты выражали как число рибосомальных оперонов на 1 г почвы.
При создании библиотек фрагментов гена 16S рРНК с каждым образцом почвенной ДНК проводили ПЦР с универсальными праймерами к вариабельному участку V4 — F515 (5'-GTGCCAGCMGCCGCGGTAA-3') и R806 (5'-GGACTACVSGGGTATCTAAT-3') на термоциклере T100 («BioRad», Германия) по следующему протоколу: 3 мин при 95 °С; 30 с при 95 °С, 30 с при 55 °С, 30 с при 72 °С (35 циклов) (32). Секвенирование и первичную обработку данных осуществляли на приборе Illumina MiSeq («Illumina, Inc.», США) в ЦКП «Геномные технологии и клеточная биология» (ФГБНУ ВНИИСХМ). Секвенированные последовательности гена 16S рРНК обрабатывали с помощью пакетов программ Trimmomatic (33) и QIIME (34). Из библиотек удаляли все служебные последовательности, осуществляли сборку парноконцевых прочтений, проверяли качество нуклеотидных последовательностей. Все небактериальные и химерные последовательности исключали, данные нормализовали. Последовательности с долей сходства более 97 % объединяли в операционные таксономические единицы (ОТЕ) при помощи алгоритма de novo, основанного на методе UCLUST (35). Из каждой ОТЕ выбирали одну последовательность для составления набора репрезентативных последовательностей. На следующем этапе репрезентативные последовательности классифицировали с помощью программы RDP naive Bayesian rRNA Classifier, затем выравнивали по алгоритму PyNast (36), используя в качестве матрицы базу данных Greengenes coreset (37). После выравнивания последовательностей строили матрицы дистанций в QIIME на основе евклидовых расстояний.
Для сравнительного анализа сообществ рассчитывали показатели а- и p-разнообразия. а-Разнообразие оценивали с использованием индексов видового богатства (richness) (число ОТЕ в образце, индекс обилия Chao 1, показатель филогенетического разнообразия Фейта PD) и индекса разнообразия Шеннона H (Shannon index) (38-40).
Достоверность различий между микробиомами по индексам а-разнообразия оценивали при помощи t -критерия Стьюдента. Для определения р-разнообразия применяли метод Weighted unifrac, позволяющий оценить процент сходств/различий между всеми парами сравниваемых микробиомов (41). Результаты анализировали методами многомерной статистики (анализ главных компонент) в программе Emperor (42). Для оценки корреляции состава и структуры бактериальных сообществ с содержанием базовых макроэлементов почвы в QIIME провели тест Мантеля (корреляция Пирсона, 100 пермутаций) для объединенных повторностей (43). Для оценки достоверности различий в представленности отдельных таксонов в анализируемых образцах в дополнение к программному обеспечению QIIME нами был написан скрипт с использованием U-критерия Манна-Уитни (Mann-Whitney U-test) с порогом значимости 0,05 на языке программирования Python (44).
В работе приведены средние значения показателей ( M ) и их стандартные ошибки (±SEM). Выявленные различия считали статистически значимыми при р < 0,05.
Результаты. Кингисеппский карьер предназначался для добычи содержащих фосфориты отложений, приуроченных к оболовым песчаникам нижнего ордовика, над которыми залегают известняки и доломиты, чуть выше которых расположены среднедевонские глины, мергели, пески, аргиллиты. Озерно-ледниковые пески и супеси, моренные суглинки и торфы представляют собой верхние четвертичные отложения (3). Все эти породы в той или иной степени слагают отвалы карьера. Месторождение фосфоритов разрабатывали открытым способом с 1960 годов. В результате образовались разновозрастные отвальные площади, на которых после нескольких лет са-мозарастания и спонтанного проседания пород применили горнотехниче- скую рекультивацию. Эта территория — одна из крупнейших по площади нарушения и масштабу рекультивационных работ на северо-западе России.
При горнотехнической рекультивации на участке ¹ 1 в 1979 году после разравнивания отвалов на глыбистый материал нанесли торфяноминеральную смесь (четвертичные суглинисто-песчаные породы с содержанием примесного торфа 20-30 %), затем применили биологическую рекультивацию (высадка саженцев ели). На участках ¹ 2 и ¹ 3 после разравнивания по поверхности отвалов распределили минеральный субстрат, состоящий из так называемой рыхлой породы, не содержащей примеси торфа. На участке ¹ 2 высадили лиственницу, ¹ 3 — сосну.
Почвы участков рекультивации представлены карбо-литоземами. В целом основные почвенные параметры оказались сходными на всех участках. Однако отметим, что содержание подвижного фосфора и нитратов более чем в 2 раза больше на участке ¹ 2 под лиственницей по сравнению с аналогичными горизонтами на других участках (табл. 1). Реакция рН водной вытяжки на участке ¹ 1 (под елью) слабокислая, тогда как на остальных — слабощелочная. По гранулометрическому составу репланто-земы характеризовались как средние суглинки.
1. Основные биохимические и гранулометрические характеристики почвы в различных фитоценозах на участках длительной рекультивации (карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год)
|
Показатель |
Реплантозем |
||
|
на торфяно-минеральной смеси под елью, 37 лет, участок ¹ 1 |
на минеральной породе |
||
|
под лиственницей, 32 года, участок ¹ 2 |
под сосной, 27 лет, участок ¹ 3 |
||
|
Тип горизонта |
АY ( |
АY |
АY |
|
Глубина, см |
3-18 |
1-18 |
1-10 |
|
рН вод. |
6,5 |
7,6 |
7,2 |
|
С орг. , % |
2,0 |
2,4 |
1,9 |
|
N общ. , % |
0,20 |
0,31 |
0,46 |
|
C/N |
11,6 |
8,9 |
4,8 |
|
N (NH 4 ), мг/кг |
31,1 |
27,2 |
27,4 |
|
N (NO 3 ), мг/кг |
0,24 |
0,82 |
0,37 |
|
P (Р 2 О 5 ), мг/кг |
2043,5 |
4198,5 |
1731,5 |
|
K (К 2 О), мг/кг |
338,3 |
242,7 |
214,7 |
|
Доля частиц, %: |
|||
|
< 0,001 мм |
15,0 |
22,0 |
24,3 |
|
< 0,01 мм |
33,2 |
36 |
36 |
№ участка
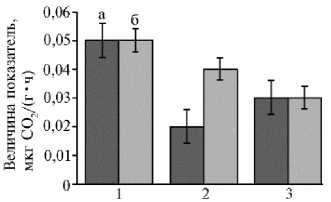
Рис. 1. Базальное (а) и субстрат-инду-цированное (б) почвенное дыхание органоминеральных горизонтов (AY) в различных фитоценозах на участках длительной рекультивации ( n = 3, M ±SEM, карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год). Описание участков по типу рекультивации см. в разделе «Методика».
На всех участках мы выявили очень низкие показатели базального и субстрат-индуцированного почвенного дыхания (рис. 1) с максимальными значениями 0,05 мкг СО2/(г•ч) при минимальном 0,02 мкг СО2/(г•ч). Дополнительную эмиссию СО2 наблюдали только на участке ¹ 2 (под лиственницей). В целом наименьшую дыхательную активность проявляли почвы, которые сформировались на отвалах под покровом сосны. Нужно отметить, что достоверных различий между двумя показателями не было, хотя для зрелых почв климаксных экосистем они обычно наблюдаются.
Для содержания микробной биомассы пределы варьирования составляли от 1,6±0,1 мкг С/г (под сосной, участок ¹ 3) до 2,4±0,09 мкг С/г (под елью, участок ¹ 1). Значения микробного метаболического коэффициента (отношение величины базального дыхания к содержанию углерода микробной биомассы) были крайне малы, максимальное значение 0,021±0,002 мкг СO2-С/(мг Смик.•ч) также зафиксировали на участке под елью, минимальное 0,010±0,005 мкг СO2-С/(мг Смик.•ч) — под лиственницей (участок ¹ 2).
Непосредственно после проведения горнотехнического этапа рекультивации происходило самозарастание поверхности пород типичными видами-эксплерентами — Tussilago farfara L., Chamaenerion angustifolium (L.) Scop., Calamagrostis epigeios (L.) Roth. К 2014 году на участке ¹ 1 сформировался ельник-мертвопокровник, где мы выявили 11 видов высших сосудистых растений (2 деревянистых и 9 травянистых видов) из 8 семейств. Общее проективное покрытие травянистой растительностью здесь составило 5 %. На участке ¹ 2 под лиственницей оно достигало 25 %. Всего на участке ¹ 2 произрастали 14 видов высших растений из 10 семейств (4 деревянистых и 10 травянистых видов). На участке ¹ 3 с высаженной сосной выявили 12 видов высших растений (3 деревянистых и 9 травянистых) при общем проективном травяном покрытии 25 %. Примерно одинаковое число видов приходилось на разнообразные жизненные формы травянистых растений — длинно- и коротко-корневищные, стержнекорневые, стержне-кистекорневые, дерновинные многолетние травы. Согласно индексу Шеннона H и обратному индексу Симпсона 1/D (табл. 2), наименьшим разнообразием характеризовался ельник мертвопокровный.
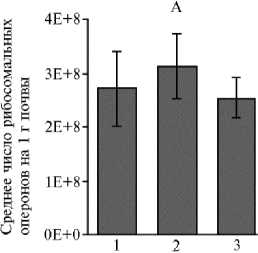
Рис. 2. Количественное определение трех групп микроорганизмов методом qPCR в образцах почвы из различных фитоценозов на участках длительной рекультивации: А — бактерии, Б — археи, В — грибы (для участков ¹¹ 1 и 2 n = 4, для участка ¹ 3 n = 3; 3 измерения для каждой повторности, M ±SEM; карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год). Описание участков по типу рекультивации см. в разделе «Методика».
2. Индексы разнообразия растительных сообществ в различных фитоценозах на участках длительной рекультивации (карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2014 год)
|
¹ участка |
Индекс Шеннона Н |
Индекс Симпсона (обратный) 1/D |
Число видов на участке всего |
1 1,3 2,1 11
2 2,0 4,2 14 22
3 1,9 4,2 12
П р и м еч а ни е. Описание участков по типу рекультивации см. в разделе «Методика».
Анализ представленности трех групп микроорганизмов методом qPCR на трех испытательных участках выявил некоторые различия по количеству рибосомальных оперонов бактерий и архей (рис. 2): их число оказалось наибольшим на участке ¹ 2 под лиственницей — соответственно (3,13±0,6)½108 и (2,06±0,55)½107. Однако для бактерий этот показатель достоверно не отличался от значений на других участках — (2,73±0,67)½108 на участке ¹ 1 и (2,55±0,37)½108 на участке ¹ 3, тогда как для архей он был достоверно (р < 0,05) выше, чем на двух других: (0,9±0,27)½107 на участке ¹ 1 и (1,52±0,39)½107 на участке ¹ 3. Количество грибов в почве разных участков статистически значимо не различалось и составляло для участка ¹ 1 (1,88±0,26)½107, ¹ 2 — (1,8±0,27)½107, ¹ 3 — (1,6±0,17)½107.
3. Индексы разнообразия почвенных микробиомов в различных фитоценозах на участках длительной рекультивации ( M ±SEM, карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год)
|
¹ участка |
Индекс Шеннона H |
Индекс Фейта PD |
Сhao 1 |
Число ОТЕ |
Покрытие библиотеки, % |
|
1 |
9,3±0,1 |
171,1±9,7 |
2559,5±181,9 |
2163,8±165,5 |
84,5 |
|
2 |
9,4±0,1 |
169,6±10,5 |
2368,8±117,8 |
1978,8±176,6 |
83,5 |
|
3 |
9,3±0,1 |
188,3±10,1 |
2672,3±208,7 |
2322,3±176,8 |
86,9 |
|
Примеча участка ¹ 3 |
н и е. ОТЕ — операционная таксономическая единица. Для участков ¹¹ n = 3. Описание участков по типу рекультивации см. в разделе «Методика». |
1 и 2 n = 4, для |
|||
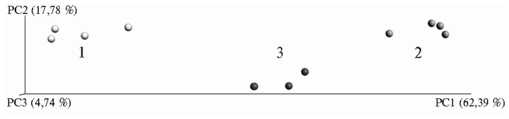
Рис. 3. Сравнение β -разнообразия почвенных метагеномов с использованием PCoA (Principal Components Analysis матрицы попарных сравнений, построенной методом weighted_unifrac) в различных фитоценозах на участках длительной рекультивации: 1, 2, 3 — соответственно участки ¹ 1, ¹ 2 и ¹ 3 (карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год). Оси представляют проекции многомерных данных с указанными значениями объясненной вариации (%), число окружностей соответствует числу повторностей. Описание участков по типу рекультивации см. в разделе «Методика».
Для анализа микробиома почв каждого из участков выделяли ДНК в 4 повторностях. Однако после секвенирования по участку ¹ 3 (сосна) удалось обработать только 3 повторности. Далее все измерения для участков ¹ 1 и ¹ 2 выполняли в 4 повтороностях, а для участка ¹ 3 — в 3.
Показатели α-раз-нообразия и численности микробиомов на всех участках были сопоставимы (табл. 3). На рисунке 3 приведен график β-разнообразия, представляющий трехмерную проекцию данных из матрицы попарных расстояний между микробиомами почвенных образцов, построенной с использованием метода weighted_unifrac. Видно, что микробиомы отчетливо дифференцируются в соответствии с участком отбора почвенных проб.
При анализе разнообразия доминирующих бактериальных фил —
Proteobacetria , Actinobacteria и Acidobacteria проявились несколько закономерностей (табл. 4). Наибольшее разнообразие имели актинобактерии в микробиомах, сформированных под ельником мертвопокровным (участок ¹ 1), причем число ОТЕ указанной группы бактерий значимо уменьшалось в ряду ель (участок ¹ 1) > сосна (участок ¹ 3) > лиственница (участок ¹ 2). Разнообразие протеобактерий, напротив, было максимальным в варианте с посадками лиственницы (участок ¹ 2) и минимальным в еловом фитоценозе (участок ¹ 1). Стоит отметить также достоверный (р < 0,05) минимум филогенетического разнообразия протеобактерий (индекс Фейта) в микробиоме елового леса, достоверный максимум (р < 0,05)
числа выявленных ОТЕ под высаженными соснами (участок ¹ 3) и максимум индекса Шеннона на участке ¹ 2 (лиственница) относительно участка ¹1. Последнее может свидетельствовать о наиболее выравненной представленности различных видов протеобактерий в фитоценозе, сформированным под покровом сосны. Для представителей филы Acidobacteria также сохранялась общая тенденция увеличения разнообразия и обилия на участке с сосной (участок ¹ 3). Здесь отмечали достоверные (р < 0,05) максимумы филогенетического разнообразия ацидобактерий (индекс Фейта и индекс Chao 1), характеризующие максимальное обилие (richness) этой группы на участке ¹ 3 по сравнению с участком ¹ 1.
4. Индексы α -разнообразия групп бактерий почвенных микробиомаов в различных фитоценозах на участках длительной рекультивации ( M ±SEM, карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год)
|
¹ участка Индекс Шеннона H |
Индекс Фейта PD Сhao 1 Число ОТЕ Actinobacteria |
|
1 7,2±0,1a 2 6,9±0,1b 3 6,8±0,1a |
23,9±0,5a 558,2±8,0a 311,9±5,1a 21,9±0,4a 379,5±23,4a 272,1±5,8a 23,1±0,7b 454,0±10,4a 291,5±6,4b Proteobacteria |
|
1 7,2±0,1a 2 7,7±0,1a 3 7,3±0,2b |
43,5±1,3ab 702,0±37,6a 528,9±18,2a 48,1±0,8a 742,6±31,7b 564,5±14,5b 49,7±1,0b 850,6±44,3c 595,1±14,3a Acidobacteria |
|
1 6,4±0,1a 2 6,0±0,3b 3 6,3±0,4c |
18,6±0,3a 343,7±16,9a 275,7±5,6a 19,3±0,2b 389,0±16,8b 275,9±5,3b 20,0±0,2a 455,3±21,4a 293,7±15,0c |
П р и м еч а ни е. ОТЕ — операционная таксономическая единица. Для участков ¹¹ 1 и 2 n = 4, для участка ¹ 3 n = 3. Описание участков по типу рекультивации см. в разделе «Методика». По столбцам в пределах таксономической группы одинаковые буквы (a, b, c) означают наличие статистически значимого различия (p < 0,05).
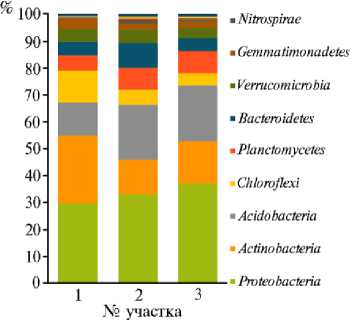
Рис. 1. Таксономическая структура почвенных микробиомов на уровне прокариотных фил в различных фитоценозах на участках длительной рекультивации (карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год). Для каждого участка повторности объединены. Указаны филы с представленностью > 1 %. Описание участков по типу рекультивации см. в разделе «Методика».
При анализе ампликонных библиотек гена 16S рРНК бактерий и архей с учетом нормализации идентифицировали 220910 нуклеотидных последовательностей. Доминирующими филами были Proteobacteria (29,6-37 %),
Actinobacteria (12,8-25,2 %) и Acidobacteria (12,3-20,8 %), далее следовали представители фил Chloroflexi (4,6-11,8 %), Planctomycetes (5,7-8,2 %), Bacterоidetes (4,9-9,2 %), Verrucomicrobia (3,9-5,0 %), Gemmatimonadetes (2,13,8 %), Nitrospirae (0,2-1,0 %); доля остальных оставляла менее 1 % (рис. 4). Стоит отметить сравнительно низкую представленность (0,1 %) и низкое разнообразие архей из групп Crenarchaeota и [ Parvarchaeota ]. В целом количественный состав фил на разных участках был схожим, но следует отметить, что на участке ¹ 1 число представителей групп Actinobacteria и Chloroflexi было выше относительно других, а группы Acidobacteria — ниже.
5. Тепловая карта (heatmap), отражающая средний процент представленности мажорных ОТЕ почвенных микробиомов в различных фитоценозах на участках длительной рекультивации (карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский
|
р-н, 2016 год) |
||||||||
|
¹ участка |
ОТЕ |
Фила |
Класс |
Порядок |
Семейство |
Род |
||
|
1 |
2 |
3 |
||||||
|
52697 Proteobacteria Alphaproteobacteria Rhizobiales Bradyrhizobiaceae – 9881* Proteobacteria Alphaproteobacteria Rhizobiales Hyphomicrobiaceae Rhodoplanes 39913 Proteobacteria Alphaproteobacteria Rhizobiales Hyphomicrobiaceae Rhodoplanes 39246 Proteobacteria Alphaproteobacteria Rhizobiales Hyphomicrobiaceae Rhodoplanes 36288* Proteobacteria Alphaproteobacteria Rhizobiales Rhizobiaceae – 2794 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas 54700 Proteobacteria Betaproteobacteria Burkholderiales Comamonadaceae – 44729 Proteobacteria Betaproteobacteria Burkholderiales Comamonadaceae – 9787 Proteobacteria Betaproteobacteria Burkholderiales Comamonadaceae Delftia 18503* Proteobacteria Betaproteobacteria IS-44 – – 18585 Proteobacteria Betaproteobacteria SC-I-84 – – 7276* Proteobacteria Betaproteobacteria SC-I-84 – – 29307 Proteobacteria Deltaproteobacteria Syntrophobacterales Syntrophobacteraceae – 33418 Proteobacteria GammaproteobacteriaPseudomonadales Pseudomonadaceae Pseudomonas 15473* Proteobacteria GammaproteobacteriaXanthomonadales Sinobacteraceae – 35545 Actinobacteria Actinobacteria Actinomycetales Microbacteriaceae – 46306 Actinobacteria Actinobacteria Actinomycetales Microbacteriaceae Agromyces 18325* Actinobacteria Actinobacteria Actinomycetales Micrococcaceae – 32722* Actinobacteria Actinobacteria Actinomycetales PromicromonosporaceaePromicromonospora 37122* Actinobacteria MB-A2-108 0319-7L14 – – 45581 Actinobacteria Thermoleophilia Gaiellales Gaiellaceae – 6537* Actinobacteria Thermoleophilia Gaiellales Gaiellaceae – 36062* Actinobacteria Thermoleophilia Solirubrobacterales – – 17100 Actinobacteria Thermoleophilia Solirubrobacterales Solirubrobacteraceae – 6252* Acidobacteria [Chloracidobacteria] RB41 – – 9076* Acidobacteria [Chloracidobacteria] RB41 – – 20749* Acidobacteria [Chloracidobacteria] RB41 Ellin6075 – 22604* Acidobacteria [Chloracidobacteria] RB41 Ellin6075 – 54563 Acidobacteria Acidobacteria -6 iii1-15 – – 52756 Acidobacteria Acidobacteria -6 iii1-15 – – 37100 Acidobacteria Acidobacteria -6 iii1-15 – – 43517 Acidobacteria Acidobacteria -6 iii1-15 mb2424 – 4897 Chloroflexi Chloroflexi [Roseiflexales] [Kouleothrixaceae] – 32350 Chloroflexi Chloroflexi [Roseiflexales] [Kouleothrixaceae] – 57060* Chloroflexi Chloroflexi [Roseiflexales] [Kouleothrixaceae] – 23485 Chloroflexi Ellin6529 – – – 46192* Chloroflexi Ellin6529 – – – 11945 Bacteroidetes [Saprospirae] [Saprospirales] Chitinophagaceae – 54731 Bacteroidetes [Saprospirae] [Saprospirales] Saprospiraceae – 49979 Bacteroidetes Cytophagia Cytophagales Cytophagaceae Adhaeribacter 9879 Verrucomicrobia [Spartobacteria] [Chthoniobacterales] [Chthoniobacteraceae] Chthoniobacter 24870 Verrucomicrobia [Spartobacteria] [Chthoniobacterales] [Chthoniobacteraceae] DA101 20531 Gemmatimonadetes Gemm-1 – – – 27129* GemmatimonadetesGemmatimonadetes N1423WL – – 28624 Planctomycetes Phycisphaerae CCM11a – – |
||||||||
|
— |
||||||||
П р и м е ч а н и е. ОТЕ — операционная таксономическая единица. К мажорным отнесены OTE с представленностью > 0,5 % хотя бы в одном из вариантов. Для каждой ОТЕ указаны фила и самый низко-разрешенный таксон. Звездочкой (*) отмечены ОТЕ, которые более чем в 5 раз различаются по представленности хотя бы между одной парой участков (p < 0,05). Прочерки означают, что ОТЕ не определена до отмеченного таксона. Описание участков по типу рекультивации см. в разделе «Методика».
Всего среди всех повторностей библиотек гена 16S рРНК для трех исследуемых участков выявили 5760 ОТЕ. Из них только 45 ОТЕ составляли более 0,5 % от всех последовательностей хотя бы для одной библиотеки (табл. 5). Они охватывали все 9 наиболее представленных фил бактерий. Таксономическое положение ОТЕ удалось определить в основном до уровней родов и семейств, но в некоторых случаях только до классов и по- рядков. Большинство ОТЕ принадлежали к таксонам филы Proteobacteria, ассоциированным с ризосферой. Самым многочисленным из них был род Pseudomonas, который преобладал на участке под сосной. Также распространены ОТЕ порядков Rhizobiales и Sphingomonadales, тесно ассоциированные с растениями в почве. Для одной из OTE семейства Rhizobiaceae превышение содержания на участке под елью относительно участка под сосной было 5-кратным. Представителей Sinobacteraceae в 5 раз больше под сосной, чем под елью. Род Rhodoplanes связывают с разложением древесины в лесных почвах (45). ОТЕ, принадлежащие к этим порядкам, чаще встречались на участке ¹ 1, который был рекультивирован ранее остальных.
Род Delftia , напротив, чаще находили в более молодых реплантозе-мах участков ¹ 2 и ¹ 3. Его наличие связывают с активными процессами биоремедиации в почвах (46, 47). Второй по численности представителей ОТЕ была фила Actinobacteria (классы Actinobacteria , Thermoleophillia и MBA2-108). Наиболее многочисленные ОТЕ принадлежали к семействам Mi-crococcaceae и Microbacteriaceae , члены которых, в том числе род Agromyces , относятся к биодеструкторами (48). Причем на участках под елью и лиственницей присутствие Micrococcaceae достоверно возрастало (в 10 раз) по сравнению с участком под сосной. На участке ¹ 1 содержание актинобактерий Promicromonospora было достоверно больше, чем на участках ¹ 2 и № 3 ( в 20 раз, p < 0,05). Среди ацидобактерий на всех участках доминировал класс Chloracidobacteria , вторым по численности был класс Acido-bacteria -6. ОТЕ порядка RB41, доминирующие на участках ¹ 2 и ¹ 3, в литературе часто описываются в метагеномах нарушенных и подверженных стрессу почв (49-51). Из представителей минорных фил нужно отметить ОТЕ классов Chloroflexi и Ellin6529, доминирующие на участке ¹ 1. Бактерии класса Ellin6529 способны фиксировать атмосферный азот (52). Также на участке ¹ 1 преобладали бактерии рода DA101 из филы Verru-comicrobia.
На обследованных участках мы не обнаружили взаимосвязи структуры почвенного микробиома с почвообразовательными процессами, в частности с разложением органических остатков в почве и их минерализацией. Тест Мантеля на основе матриц расстояний сообществ, полученных методом weighted_unifrac, не выявил достоверной корреляции состава микробима с pH или содержанием в почве основных биогенных элементов — C, N, P, K (табл. 6) .
6. Анализ корреляции β -разнообразия почвенного микробиома с основными химическими показателями почв в изученных фитоценозах на участках длительной рекультивации ( карьер ¹ 3 ПО «Фосфорит», Кингисеппское месторождение фосфоритов, Ленинградская обл., Кингисеппский р-н, 2016 год)
|
Показатель |
r |
p-value |
|
pH |
0,99064 |
0,1638 |
|
Содержание: |
||
|
C |
- 0,02333 |
1,0000 |
|
N общ. |
- 0,67638 |
0,6840 |
|
N (NH 4 ) |
0,76821 |
0,4973 |
|
N (NO 3 ) |
0,48269 |
0,8311 |
|
P (P 2 O 5 ) |
0,08478 |
1,0000 |
|
K (K 2 O) |
0,51337 |
0,6560 |
П р и м еч а ни е. Повторности по всем участкам объединены. Описание участков по типу рекультивации см. в разделе «Методика».
В карьере по добыче фосфорита в Кингисеппском районе сформировались лесные сообщества, типичные для таежной зоны Европейской части России. Органоминеральный горизонт участка ¹ 1 характеризовался более кислой реакцией, что может быть следствием внесения субстрата с примесью торфа на горнотехническом этапе рекультивации. Согласно данным о базальном и субстрат-индуцированном дыхании, микробиологическая активность почв низкая, что может свидетельствовать о пониженной устойчивости микробных сообществ.
На всех участках отмечалось довольно низкое α - разнообразие. Это может быть следствием низкой интенсивности почвообразования на щебнистых отвалах, что хорошо соотносится с данными для подзолистых и дерново-подзолистых почв Северо-Западного региона, полученными ранее (53). Показатели α - разнообразия были сходны для микробиомов всех участков, при этом на участке ¹ 3 (под сосной) зафиксировано достоверное увеличение видового и филогенетического разнообразия микробного сообщества. Но несмотря на то, что для участков индексы α - разнообразия в целом были сопоставимы, их значения существенным образом менялись для трех доминирующих фил в микробиомах почв, сформированных в условиях разных растительных сообществ (см. табл. 4). Так, для участка ¹ 1 было характерно относительное увеличение разнообразия, а также доли актинобактерий, в то время как разнообразие и обилие протеобакте-рий было максимальным в посадках сосны (участок ¹ 3). Актинобактерии и протеобактерии нередко оказываются антагонистами по представленности в почвенном микробиоме (54, 55). Интересен тот факт, что почва на участке ¹ 1 имели наименьшее значение рН, хотя актинобактерии в большинстве случаев распространены в почвах, где реакцией среды нейтральная или ближе к щелочной (56). Скорее всего, именно состав опада и доступность органического вещества для микробного разложения, которое, по всей видимости, различается из-за неодинакового внесения торфа на разных участках, оказывают в обсуждаемом случае более определяющее влияние на структуру микробиома, нежели рН. Вероятно, почва, сформированная под покровом ели, отличалась от почв на других участках наличием труднодоступных для микробного сообщества форм органического вещества, чем может объясняться относительное увеличение разнообразия и обилия актинобактерий, большинство которых гидролитики с олиготрофным типом питания (57). На участке ¹ 3, напротив, разнообразие высших растений их жизненных форм наибольшее. Это позволяет предположить более широкий спектр и большее разнообразие форм органических веществ, в том числе легкодоступных, что, в свою очередь, отражается на структуре микробиома, в частности растет число копиотроф-ных микроорганизмов, к которым относится большинство представителей филы Proteobacteria (58).
Таксономический анализ показал формирование на исследуемых территориях характерного (53) для закисленных дерново-подзолистых и подзолистых почв Северо-Запада микробного сообщества с преобладанием разнообразных групп ацидобактерий из порядков RB41, iii1-15, а также актинобактерий (семейства Gaiellaceae и Solirubrobacteriaceae). Присутствие этих групп, а также весомая доля бактерий из фил Bacteroidetes, Verruco-microbia, Planctomycetes (их представители в основном относятся к оли-готрофам) (59, 60), указывают на полноту углеродного цикла и стабилизацию состава микробного сообщества. Однако наблюдаемую стадию, судя по всему, следует рассматривать как предклимаксную, поскольку во всех проанализированных сообществах много копиотрофных форм бактерий, в частности, псевдомонад (в среднем 5,5 %) (61). Примечательно присутствие на всех участках протеобактерий (сем. Comаmonadaceae, р. Delftia). В литературе этот микроорганизм часто обнаруживают в сообщества загрязненных и техногенно нарушенных почв, а также в микробиомах активного ила (62). По-видимому, указанная бактерия принимает участие в почвообразовательном процессе на исследуемых рекультивируемых участках.
Итак, в результате проведенных исследований на участках рекультивации (ПО «Фосфорит», Ленинградская обл.) мы не выявили зависимости между структурой почвенного микробиома и физико-химическими параметрами почв. Кроме того, разнообразие почвенного микробиома не коррелирует с показателями по основным минеральным элементам питания. Фактор доминирующей растительной породы также не оказывает существенного влияния на структуру микробиома. Возможно, что в обсуждаемом случае определяющее значение для микроорганизмов имеют другие экологические факторы. Ими могут быть различия в составе растительного опада и подстилки и, как следствие, разнообразие химической природы органических субстратов. Анализ α - разнообразия и таксономического состава микробиомов на трех испытательных участках позволяет предположить, что мы наблюдаем предклимаксную стадию развития почвенного покрова. При этом структура почвы и повышенное содержание минерального фосфора оказываются слишком сильными абиотическими факторами, препятствующие полному восстановлению микробиома нарушенных почв.
Список литературы Микробиом почв Кингисеппского месторождения фосфоритов при разных типах горнотехнической и биологической рекультивации
- Дмитракова Я.А., Абакумов Е.В., Першина Е.А., Иванова Е.А., Андронов Е.Е. Динамика растительного сообщества и микробиома хроносерий посттехногенных почв в известняковом карьере в условиях рекультивации. Сельскохозяйственная биология, 2018, 53(3): 557-569 ( ). DOI: 10.15389/agrobiology.2018.3.557rus
- Bing-yuan H., Li-xun K. Mine land reclamation and eco-reconstruction in Shanxi province I: mine land reclamation model. The Scientific World Journal, 2014, 2014: 483862 ( ). DOI: 10.1155/2014/483862
- Сумина О.И. Формирование растительности на техногенных местообитаниях Крайнего Севера России. Автореф. канд. дис. СПб, 2011.
- Абакумов Е.В., Гагарина Э.И. Почвообразование в посттехногенных экосистемах карьеров на Северо-Западе Русской равнины. СПб, 2006.
- Valdez-Hernández M., Gil-Medina R., López-Martínez J.O., Torrescano-Valle N., Cabanillas-Terán N., Islebe G.A. Succession and the relationship between vegetation and soil in the Marl Quarries of the Yucatan Peninsula, Mexico. Forests, 2019(2), 10: 116 ( ). DOI: 10.3390/f10020116
- Fierer N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Reviews Microbiology, 2017, 15: 579-590 ( ).
- DOI: 10.1038/nrmicro.2017.87
- Sergaki C., Lagunas B., Lidbury I., Gifford M.L., Schäfer P. Challenges and approaches in microbiome research: from fundamental to applied. Frontiers in Plant Science, 2018, 9: 1205 ( ).
- DOI: 10.3389/fpls.2018.01205
- Torsvik V., Øvreas L. Microbial diversity and function in soil: from genes to ecosystems. Current Opinion in Microbiology, 2002, 5(3): 240-245 (
- DOI: 10.1016/s1369-5274(02)00324-7)
- Wei Y.J., Wu Y., Yan Y.Z., Zou W., Xue J., Ma W.R., Wang W., Tian G., Wang L.Y. High-throughput sequencing of microbial community diversity in soil, grapes, leaves, grape juice and wine of grapevine from China. PLoS One, 2018, 13(3): e0193097 ( ).
- DOI: 10.1371/journal.pone.0193097
- Soliman T., Yang S.Y., Yamazaki T., Jenke-Kodama H. Profiling soil microbial communities with next-generation sequencing: the influence of DNA kit selection and technician technical expertise. PeerJ, 2017, 5: e4178 ( ).
- DOI: 10.7717/peerj.4178
- Janssen P.H. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA Genes. Applied and Environmental Microbiology, 2006, 72(3): 1719-1728 ( ).
- DOI: 10.1128/AEM.72.3.1719
- Hua J., Feng Y., Jiang Q., Bao X., Yin Y. Shift of bacterial community structure along different coastal reclamation histories in Jiangsu, Eastern China. Scientific Reports, 2017, 7(1): 10096 ( ).
- DOI: 10.1038/s41598-017-10608-3
- Lauber C.L., Hamady M., Knight R., Fierer N. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale. Applied and Environmental Microbiology, 2009, 75(15): 5111-5120 ( ).
- DOI: 10.1128/AEM.00335-09
- Karimi B., Terrat S., Dequiedt S., Saby N.P.A., Horrigue W., Lelièvre M., Nowak V., Jolivet C., Arrouays D., Wincker P., Cruaud C., Bispo A., Maron P.-A., Bouré N.C.P., Ranjard L. Biogeography of soil bacteria and archaea across France. Science Advances, 2018, 4(7): eaat1808 ( ).
- DOI: 10.1126/sciadv.aat1808
- Valencia E., Gross N., Quero J.L., Carmona C.P., Ochoa V., Gozalo B., Delgado-Baquerizo M., Dumack K., Hamonts K., Singh B.K., Bonkowski M., Maestre F.T. Cascading effects from plants to soil microorganisms explain how plant species richness and simulated climate change affect soil multifunctionality. Global Change Biology, 2018, 24(12): 5642-5654 ( ).
- DOI: 10.1111/gcb.14440
- Hu L., Robert C.A.M., Cadot S., Zhang X., Ye M., Li B., Manzo D., Chervet N., Steinger T., van der Heijden M.G.A., Schlaeppi K., Erb M. Root exudate metabolites drive plant-soil feedbacks on growth and defense by shaping the rhizosphere microbiota. Nature Communications, 2018, 9(1): 2738 ( ).
- DOI: 10.1038/s41467-018-05122-7
- Hartman K., van der Heijden M.G.A., Wittwer R.A., Banerjee S., Walser J.C., Schlaeppi K. Cropping practices manipulate abundance patterns of root and soil microbiome members paving the way to smart farming. microbiome, 2018, 6(1): 14 ( ).
- DOI: 10.1186/s40168-017-0389-9
- Pershina E.V., Ivanova E.A., Korvigo I.O., Chirak E.L., Sergaliev N.H., Abakumov E.V., Provorov N.A., Andronov E.E. Investigation of the core microbiome in main soil types from the East European plain. Science of the Total Environment, 2018, 631-632: 1421-1430 ( ).
- DOI: 10.1016/j.scitotenv.2018.03.136
- Thavamani P., Samkumar R.A., Satheesh V., Subashchandrabose S.R., Ramadass K., Naidu R., Venkateswarlu K., Megharaj M. Microbes from mined sites: harnessing their potential for reclamation of derelict mine sites. Environmental Pollution, 2017, 230: 495-505 ( ).
- DOI: 10.1016/j.envpol.2017.06.056
- Abakumov E.V., Gagarina E.I., Lisitsyna O.V. Land reclamation in the Kingisepp area of phosphorite mining. Eurasian Soil Science. 2005, 38(6): 648-655.
- Растворова О.Г. Физика почв - практическое руководство. Л., 1983.
- Растворова О.Г., Андреев Д.П., Гагарина Э.И., Касаткина Г.А., Федорова Н.Н. Химический анализ почв. СПб, 1995.
- ГОСТ Р 54650-2011. Почвы. Определение подвижных соединений фосфора и калия по методу Кирсанова в модификации ЦИНАО. M., 2011.
- ГОСТ 26489-85. Почвы. Определение обменного аммония по методу ЦИНАО. М., 1985.
- ГОСТ 26951-86. Почвы. Определение нитратов ионометрическим методом. М., 1986.
- Ананьева Н.Д., Сусьян Е.А., Гавриленко Е.Г. Особенности определения углерода микробной биомассы методом субстрат индуцированного дыхания. Почвоведение, 2011, 11: 1327-1333.
- Андронов Е.Е., Петрова С.Н., Чижевская Е.П., Коростик Е.В., Ахтемова Г.А., Пинаев А.Г. Влияние внесения генетически модифицированного штамма Sinorhizobium meliloti Ach-5 на структуру почвенного сообщества микроорганизмов. Микробиология, 2009, 78(4): 525-534.
- Lane D.J. 16S/23S rRNA sequencing. In: Nucleic acid techniques in bacterial systematic /E. Stackebrandt, M. Goodfellow (eds.). John Wiley and Sons, New York, 1991: 115-175.
- Muyzer G., de Waal E.C., Uitterlinden A.G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
- Yu Y., Lee C., Hwang, S. Analysis of community structures in anaerobic processes using a quantitative real-time PCR method. Water Science and Technology, 2005, 52(1-2): 85-91 ( ).
- DOI: 10.2166/wst.2005.0502
- Gardes M., Bruns T.D. ITS primers with enhanced specificity for basidiomycetes - application to the identification of mycorrhizae and rusts. Molecular Ecology, 1993, 2(2): 113-118 ( ).
- DOI: 10.1111/j.1365-294X.1993.tb00005.x
- Bates S.T., Berg-Lyons D., Caporaso J.G., Walters W.A., Knight R., Fierer N. Examining the global distribution of dominant archaeal populations in soil. The ISME Journal, 2011, 5(5): 908-917 ( ).
- DOI: 10.1038/ismej.2010.171
- Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114-20 ( ).
- DOI: 10.1093/bioinformatics/btu170
- Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7: 335-336 ( ).
- DOI: 10.1038/nmeth.f.303
- Edgar R.C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461 ( ).
- DOI: 10.1093/bioinformatics/btq461
- Caporaso J.G., Bittinger K., Bushman F.D., DeSantis T.Z., Andersen G.L., Knight R. PyNAST: a flexible tool for aligning sequences to a template alignment. Bioinformatics, 2009, 26(2): 266-267 ( ).
- DOI: 10.1093/bioinformatics/btp636
- DeSantis T.Z., Hugenholtz P., Larsen N., Rojas M., Brodie E.L., Keller K., Huber T., Dalevi D., Hu P., Andersen G.L. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and Environmental Microbiology, 2006, 72(7): 5069-5072 ( ).
- DOI: 10.1128/AEM.03006-05
- Chao A. Non-parametric estimation of the number of classes in a population. Scandinavian Journal of Statistics, 1984, 11: 265-270.
- Shannon C.E., Weaver W. The mathematical theory of communication. Urbana, University of Illinois Press, 1949.
- Faith D.P. Conservation evaluation and phylogenetic diversity. Biological Conservation, 1992, 61(1): 1-10 (
- DOI: 10.1016/0006-3207(92)91201-3)
- Lozupone C., Knight R. UniFrac: a new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology, 2005, 71(12): 8228-8235 ( ).
- DOI: 10.1128/AEM.71.12.8228-8235.2005
- Vázquez-Baeza Y., Pirrung M., Gonzalez A., Knight R. EMPeror: a tool for visualizing high-throughput microbial community data. GigaScience, 2013, 2(1): 16 ( ).
- DOI: 10.1186/2047-217X-2-16
- Mantel N. The detection of disease clustering and a generalized regression approach. Cancer Research, 1967, 27(2 Part 1): 209-220.
- Mann H.B., Whitney D.R. On a test of whether one of two random variables is stochastically larger than the other. The Annals of Mathematical Statistics, 1947, 18(1): 50-60.
- Tláskal V., Zrůstová P., Vrška T., Baldrian P. Bacteria associated with decomposing dead wood in a natural temperate forest. FEMS Microbiology Ecology, 2017, 93(12): fix157 ( ).
- DOI: 10.1093/femsec/fix157
- Liu J., Xu G., Dong W., Xu N., Xin F., Ma J., Fang Y., Zhou J., Jiang M. Biodegradation of diethyl terephthalate and polyethylene terephthalate by a novel identified degrader Delftia sp. WL-3 and its proposed metabolic pathway. Letters in Applied Microbiology, 2018, 67(3): 254-261 ( ).
- DOI: 10.1111/lam.13014
- Liu Y., Tie B., Li Y., Lei M., Wei X., Liu X., Du H. Inoculation of soil with cadmium-resistant bacterium Delftia sp. B9 reduces cadmium accumulation in rice (Oryza sativa L.) grains. Ecotoxicology and Environmental Safety, 2018, 163: 223-229 ( ).
- DOI: 10.1016/j.ecoenv.2018.07.081
- Wang R., Zhang H., Sun L., Qi G., Chen S., Zhao X. Microbial community composition is related to soil biological and chemical properties and bacterial wilt outbreak. Scientific Reports, 2017, 7: 343 ( ).
- DOI: 10.1038/s41598-017-00472-6
- Awasthi M.K., Li J., Kumar S., Awasthi S.K., Wang Q., Chen H., Wang M., Ren X., Zhang Z. Effects of biochar amendment on bacterial and fungal diversity for co-composting of gelatin industry sludge mixed with organic fraction of municipal solid waste. Bioresource Technology, 2017, 246: 214-223 ( ).
- DOI: 10.1016/j.biortech.2017.07.068
- Shen Y., Ji Y., Li C., Luo P., Wang W., Zhang Y., Nover D. Effects of phytoremediation treatment on bacterial community structure and diversity in different petroleum-contaminated soils. International Journal of Environmental Research and Public Health, 2018, 15(10): 2168 ( ).
- DOI: 10.3390/ijerph15102168
- Jurburg S.D., Natal-da-Luz T., Raimundo J., Morais P.V., Sousa P., van Elsas J.D., Salles J.F. Bacterial communities in soil become sensitive to drought under intensive grazing. Science of The Total Environment, 2018, 618: 1638-1646 ( ).
- DOI: 10.1016/j.scitotenv.2017.10.012
- Lopes A.R., Bello D., Prieto-Fernández Á., Trasar-Cepeda C., Manaia C.M., Nunes O.C. Relationships among bulk soil physicochemical, biochemical, and microbiological parameters in an organic alfalfa-rice rotation system. Environmental Science and Pollution Research, 2015, 22(15): 11690-11699 ( ).
- DOI: 10.1007/s11356-015-4410-1
- Чирак Е.Л., Першина Е.В., Дольник А.С., Кутовая О.В., Василенко Е.С., Когут Б.М., Мерзлякова Я.В., Андронов Е.Е. Таксономическая структура микробных сообществ в почвах различных типов по данным высокопроизводительного секвенирования библиотек гена 16S-рРНК. Сельскохозяйственная биология, 2013, 3: 100-109 ( ).
- DOI: 10.15389/agrobiology.2013.3.100rus
- Ren C., Sun P., Kang D., Zhao F., Feng Y., Ren G., Han X., Yang G. Responsiveness of soil nitrogen fractions and bacterial communities to afforestation in the Loess Hilly Region (LHR) of China. Scientific Reports, 2016, 6: 28469 ( ).
- DOI: 10.1038/srep28469
- Wu B., Hou S., Peng D., Wang Y., Wang C., Xu F., Xu H. Response of soil micro-ecology to different levels of cadmium in alkaline soil. Ecotoxicology and Environmental Safety, 2018, 166: 116-122 ( ).
- DOI: 10.1016/j.ecoenv.2018.09.076
- Yun Y., Wang H., Man B., Xiang X., Zhou J., Qiu X., Duan Y., Engel A.S. The relationship between pH and bacterial communities in a single karst ecosystem and its implication for soil acidification. Frontiers in Microbiology, 2016, 7: 1955 ( ).
- DOI: 10.3389/fmicb.2016.01955
- Arocha-Garza H.F., Canales-Del Castillo R., Eguiarte L.E., Souza V., De la Torre-Zavala S. High diversity and suggested endemicity of culturable Actinobacteria in an extremely oligotrophic desert oasis. Journal of Life and Environmental Sciences, 2017, 5: e3247 ( ).
- DOI: 10.7717/peerj.3247
- Rosenfeld C.E., James B.R., Santelli C.M. Persistent bacterial and fungal community shifts exhibited in selenium-contaminated reclaimed mine soils. Applied and Environmental Microbiology, 2018, 84(16): e01394-18 ( ).
- DOI: 10.1128/AEM.01394-18
- Leff J.W., Fierer N. Bacterial communities associated with the surfaces of fresh fruits and vegetables. PLoS ONE, 2013, 8(3): e59310 ( ).
- DOI: 10.1371/journal.pone.0059310
- Masse J., Prescott C.E., Renault S., Terrat Y., Grayston S.J. Plant community and nitrogen deposition as drivers of alpha and beta diversities of prokaryotes in reconstructed oil sand soils and natural boreal forest soils. Applied and Environmental Microbiology, 2017, 83(9): e03319-16 ( ).
- DOI: 10.1128/AEM.03319-16
- Lladó S., Baldrian P. Community-level physiological profiling analyses show potential to identify the copiotrophic bacteria present in soil environments. PLoS ONE, 2017, 12(2): e0171638 ( ).
- DOI: 10.1371/journal.pone.0171638
- Hou Q., Wang C., Guo H., Xia Z., Ye J., Liu K., Yang Y., Hou X., Liu H., Wang J., Du B., Ding Y. Draft genome sequence of Delftia tsuruhatensis MTQ3, a strain of plant growth-promoting Rhizobacterium with Antimicrobial Activity. Genome Announcements, 2015, 3(4): e00822-15 ( ).
- DOI: 10.1128/genomeA.00822-15


