Моделирование и изучение антикоронавирусной активности производных 6,7-диметоксихиназолин-4(3H)-она в отношении SARS-CoV-2 методами in silico и in vitro
Автор: Чиряпкин А.С., Тюренков И.Н., Кодониди И.П., Шабанова Н.Б., Вдовенко-мартынова Н.Н.
Журнал: Бюллетень науки и практики @bulletennauki
Рубрика: Медицинские науки
Статья в выпуске: 8 т.9, 2023 года.
Бесплатный доступ
Пандемия новой коронавирусной инфекции SARS-CoV-2 (COVID-19) оказала существенную нагрузку на систему здравоохранения и выявила необходимость разработки эффективных лекарственных препаратов для лечения данного заболевания. Приводимые в статье результаты исследования посвящены разработке новых соединений с антикоронавирусным действием в отношении SARS-CoV-2, которые являются производными 6,7-диметоксихиназолин-4(3H)-она. In silico исследования осуществляли методом молекулярного докинга в свободно распространяемой программе Autodock 4.0. Виртуальные структуры моделируемых соединений были построены в программе HyperChem 8.0.4, а окончательная оптимизация геометрии виртуальных структур рассчитывалась в программе ORCA 4.1 с использованием метода теории функционала плотности (UB3LYP) и базового набора 6-311G**. Для прогнозирования противовирусной активности использовалась главная протеаза (идентификационный номер 7K6D) и папаиноподобная (идентификационный номер 7LBR) протеаза SARS-Cov-2. In vitro исследования проводили путем кратных разведений анализируемых соединений с использованием хромогенного субстрата. Из результатов следует, что новые синтезированные производные 6,7-диметоксихиназолин-4(3H)-она, следует рассматривать в качестве перспективных кандидатов для разработки новых противовирусных лекарственных средств. Наиболее выраженная противовирусная активность наблюдается у вещества 8c, содержащего остаток глицилтриптофана. Стоит отметить, то предложенный алгоритм поиска инновационных противовирусных препаратов in silico достаточно хорошо согласуется с результатами эксперимента in vitro .
Sars-cov-2, ковид-19, 6, 7-диметоксихиназолин-4(3h)-оны, антикоронавирусная активность, молекулярный докинг, лиганд-ферментативный комплекс, протеазы
Короткий адрес: https://sciup.org/14128692
IDR: 14128692 | УДК: 615.281.8:519.876.5 | DOI: 10.33619/2414-2948/93/06
Текст научной статьи Моделирование и изучение антикоронавирусной активности производных 6,7-диметоксихиназолин-4(3H)-она в отношении SARS-CoV-2 методами in silico и in vitro
Бюллетень науки и практики / Bulletin of Science and Practice
Появление новой коронавирусной инфекции (COVID-19/SARS-CoV-2) в декабре 2019 года потребовало реализацию задач по поиску и созданию новых противовирусных препаратов. Глобальное распространение SARS-CoV-2 в совокупности с высоким количеством смертей побудило Всемирную организацию здравоохранения объявить пандемию 12 марта 2020 года [1]. За время пандемии погибло около 20 миллионов человек. В настоящее время на фармацевтическом рынке отсутствуют высокоэффективные лекарственные средств против данного инфекционного заболевания, соответственно это подчеркивает актуальность проблемы по разработки высокоэффективных и безопасных веществ против инфекции SARS-CoV-2 [2]. Значимыми мишенями в поиске соединений для терапии SARS-CoV-2 являются главная и папаино-подобная протеазы COVID-19, ингибирование которых приводит к нарушению жизнедеятельности вируса и его гибели [3].
Конденсированные и бензаннелированные производные пиримидина широко используются в медицинской практике для лечения различных заболеваний. В частности пиримидиновое ядро содержится в структурах различных противовирусных лекарственных препаратов [4], что объясняет целесообразность поиска новых противовирусных биологически активных соединений среди производных пиримидина. Например, синтезирована серия производных индоло[3,2-c]изохинолина (δ-карболина), некоторые из которых обладают противомикробным действием, а дальнейшее изучение этих структур in silico позволило предположить возможность ими ингибировать главную протеазу вируса COVID-19 [5]. In vitro установлено, что новые аналоги пиримидиновых тиогликозидов обладают активностью против штаммов вируса SARS-COV-2 и птичьего гриппа H5N1 [6]. Пиридо[2,3-d]пиримидины могут более эффективно ингибировать главную протеазу SARS-COV-2, чем препарат лопинавир, который обычно используется в качестве ингибитора вирусных протеаз [7]. Новые гетероциклические конденсированные системы содержащие фрагменты пиримидина и бензимидазола можно рассматривать в качестве ингибиров главной протеазы и спайкового гликопротеина SARS-CoV-2 [8]. Согласно данным in silico некоторые представители синтезированных нафтопирано[2,3-d]пиримидин-11(12H)-онов, могут обладать высоким сродством к сайту связывания главной протеазы коронавируса, что обуславливает их фармакологическое действие [9]. Таким образом, конденсированные гетероциклические производные пиримидина являются перспективным классом органических соединений для поиска новых биологически активных соединений с антикоронавирусной активностью. Среди методов прогнозирования биологической активности соединений in silico выделяют наиболее значимый и широко используемый метод молекулярного докинга. В ходе такого вычислительного эксперимента представляется возможным предположить энергии взаимодействия виртуальных структур с биологической мишенью и следовательно более обоснованно построить алгоритм исследования соединений in vitro и in vivo [10].
Материал и методы исследования
Объектами виртуального исследования выступают производные 6,7-диметоксихиназолин-4(3H)-она с остатками нейроактивных аминокислот и дипептидов [11]. Структурные формулы изучаемых соединений приведены на Рисунке 1 под шифрами 1с-13с.
Молекулярный докинг проводился в свободно распространяемой программе Autodock 4.0 [12]. Молекулярное моделирование осуществлялось с учетом конформационной подвижности лигандов, торсионные углы которых были установлены и заданы в данной программе. Заряды всех атомов моделируемой системы рассчитаны алгоритмом гастейгера (Gasteiger algorithm). В программе был задан поиск 200 энергетически выгодных конформаций образования молекулярного комплекса изучаемых соединений с белковой мишенью с использованием ламарковской скоринговой функцией подсчета энергии взаимодействия (Lamarckian GA 4.2). Расстояние между точками сетки составляло 0,375 ангстрема. Трехмерные модели рецепторов и ферментов для вычислительных экспериментов брали из базы данных виртуальных структур RCSB Protein Data Bank ( [13].
Виртуальные структуры моделируемых соединений были построены в программе HyperChem 8.0.4, а затем геометрически оптимизированы способом молекулярной механики с использованием метода MM+ [14]. Окончательная оптимизация геометрии виртуальных структур рассчитывалась в программе ORCA 4.1 с использованием теории функционала плотности (UB3LYP) и базового набора 6-311G**.
Преобразование формата hin в pdb, необходимого для молекулярного моделирования, было выполнено в программе Open Babel 2.4.1 [15].
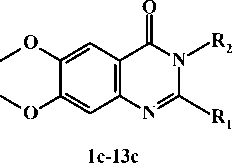
1с; R = -C H , R = -CH -COOH
2c; R = -C H , R =-CH -C(O)-NH-CH -COOH
5c; R = -C H , R = -CH -COOH
6c; R = -C H , R = -CH -C(O)-NH-CH -COOH
3c; R1 = -C2H5, R
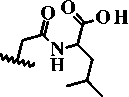
7c; R1 = -C3H7, R2
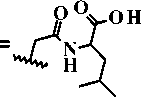
4c; R1 = -C2H5, R2 =
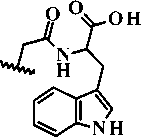
8c; R1 = -C3H7, R
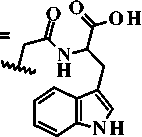
9c; R1 = -CH(CH3)2, R2 = -CH2-C(O)-NH-CH2-COOH
11c; R = -CH ; R = -CH COOH
12 с ; R = Ph; R = -CH COOH
13 с ; R = Ph; R = -C H COOH
10c; R1 = -CH(CH3)2, R2 =
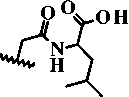
Рисунок 1. Структурные формулы прогнозируемых и далее синтезированных производных 6,7-диметоксихиназолин-4(3H)-она
Для прогнозирования противовирусной активности использовалась главная протеаза (идентификационный номер 7K6D [16]) и папаино-подобная (идентификационный номер 7LBR [17]) протеаза SARS-Cov-2. Областью вычислительного эксперимента для главной протеазы является куб, центр которого находится по следующим координатам: x = 9,664, y = 1,911, z = 22,528. Количество точек в измерениях по осям x, y и z равно 46. В случае папаиноподобной протеазы областью моделирования также является куб, центр которого находится по следующим координатам: x = 2,285, y = 14,435, z = 34,815. Количество точек в измерениях по осям x и z равно 46, а по оси y составляет 52.
Целенаправленный синтез наиболее перспективных прогнозируемых соединений выявленных in silico раннее был подробно описан в статье [11]. Соединения синтезировали взаимодействием соответствующих бензоксазинонов с глицином, глицилглицином, глициллейцином и глицилтриптофаном в среде ледяной уксусной кислоты при добавлении диметилформамида.
Оценку ингибирующей активности в отношении главной (Mpro) и папаино-подобной протеазы (PLpro) SARS-Cov-2 определяли путем проведения in vitro тестов [18, 19]. Для этого 20 мг исследуемого соединения растворяли в ДМСО до получения маточного раствора с концентрацией 1 мг/мл. Далее приготовляли серию двукратных разведений (растворитель — ДМСО) с конечным содержанием исследуемого соединения 500 µг/мл; 250 µг/мл; 125 µг/мл; 62,5 µг/мл; 31,25 µг/мл; 15,625 µг/мл и 7,8125 µг/мл. В микропланшет вносили 20 µМ Mpro или PLpro SARS-Cov-2 (Sigma-Aldrich) в объеме 10 µл, добавляли 10 µл раствора двукратных разведений исследуемого соединения. Полученную смесь инкубировали 10 минут при температуре 37 °С после чего в анализируемую среду вносили хромогенный субстрат и доводили до 200 µл буферным раствором 20 мМ HEPES рН 6,0 + 0,4 мМ ЭДТА+ 1 мМ дитиотреитол + 1% глицерин. Полученную смесь повторно инкубировали 60 минут в аналогичном температурном режиме. Параллельно ставили контрольную пробу и холостую пробу с водой для инъекций. Оптическую плотность полученных смесей регистрировали при 450 нм с использованием системы микропланшетного ридера Infinite F50 (Tecan, Австрия). Степень ингибирования ферментативной активности рассчитывали по разнице оптической плотности опытной/контрольной и холостой проб в %. На основании полученных результатов строили кривую зависимости «ингибирующая активность — logC» и рассчитывали коэффициент полуингибиции (IC50) в µМ/мл методом пробит-анализа. Все тесты выполнены в триплетном варианте с расчетом M±SEM (среднее ± стандартная ошибка среднего)
Результаты и обсуждение
С целью выявления достоверности вычислительного эксперимента связывания виртуальных структур с белковой мишенью осуществлялся расчет лиганд-ферментных комплексов классических ингибиторов протеаз и сравнение его с экспериментально полученными результатами рентгеноструктурного анализа описанными в научной литературе [16, 17]. В результате сравнительного анализа конформаций экспериментально определенных и теоретически рассчитанных лиганд-ферментных комплексов известных ингибиторов установлена достаточная достоверность используемого метода моделирования. Такой вывод вытекает из сравнения пространственное расположение референта телапревира в эксперименте in silico и экспериментально полученного рентгеноструктурным анализом, эта разница равняется 1,84 ангстремом (Рисунок 2), а для референтного вещества XR8-89 равняется 1,81 ангстрем (Рисунок 3). Эта разница меньше классически описанного предельного порога 3,0 ангстрем. Тем самым полученные результаты in silico эксперимента обладают достаточно высокой достоверностью.
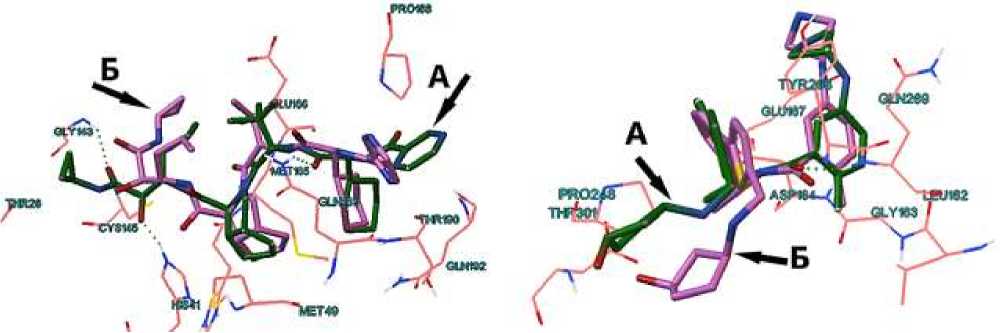
Рисунок 2. Расположение телапревира в активном сайте главной протезы SARS-Cov-2. А — рентгеноструктурный анализ (зеленый цвет), Б — молекулярный докинг (розовый цвет)
Рисунок 3. Расположение XR8-89 в активном сайте папаино-подобной протезы SARS-Cov-2. А — рентгеноструктурный анализ (зеленый цвет), Б — молекулярный докинг (розовый цвет)
Наибольшее сродство к активному сайту главной протеазы SARS-Cov-2 показали производные 6,7-диметоксихиназолин-4(3H)-она с остатками глициллейцина и глицилтриптофан: 3c, 4c, 7c и 8c (Таблица 1). Максимальной устойчивостью лиганд-ферментного комплекса обладает вещество 2-пропил-6,7-диметокси-3,1-бензоксазин-4-она содержащее фрагмент глицилтриптофана (-9,38 ккал/моль, 8с). Особо следует отметить, что для наиболее перспективных структур (3с, 7с и 8с) представляется возможным формирование межмолекулярной водородной связи с Glu 166. На Рисунке 4 приведена наиболее устойчивая конформация соединения-лидера 8с в активном сайте главной протеазой SARS-Cov-2.
Таблица 1
ЗНАЧЕНИЯ ЭНЕРГИЙ ЛИГАНД-ФЕРМЕНТНОГО КОМПЛЕКСА
И ДЛИН ВОДОРОДНЫХ СВЯЗЕЙ ПРОИЗВОДНЫХ
6,7-ДИМЕТОКСИХИНАЗОЛИН-4(3 H )-ОНА С ГЛАВНОЙ ПРОТЕАЗОЙ SARS-COV-2
|
Соединение |
Энергия докинга, ккал/моль |
Водородные связи |
||
|
Остаток аминокислоты |
Группа лиганда |
Длина водородной связи, Å |
||
|
1c |
-6,24 |
Gln192 |
COOH |
1,731 |
|
2c |
-7,75 |
Glu 166 |
COOH |
2,055 |
|
Gln 189 |
C=O |
2,197 |
||
|
3c |
-8,21 |
Glu 166 |
COOH |
2,198 |
|
4c |
-9,05 |
Gln 189 |
C=O |
1,901 |
|
Gln 192 |
COOH |
2,095 |
||
|
5c |
-6,20 |
Gln 189 |
C=O |
2,124 |
|
6c |
-7,22 |
— |
— |
— |
|
7c |
-8,59 |
Glu 166 |
COOH |
2,225 |
|
8c |
-9,38 |
Gly 143 |
OCH 3 |
2,189 |
|
Glu 166 |
C=O |
1,759 |
||
|
9c |
-7,22 |
Gln 189 |
COOH |
2,016 |
|
10c |
-7,89 |
Gln 189 |
C=O |
2,073 |
|
Glu 166 |
COOH |
1,173 |
||
|
11c |
-6,13 |
Gln 192 |
COOH |
2,227 |
|
12c |
-6,81 |
Arg 188 |
C=O |
2,185 |
|
13c |
-7,03 |
Gly 143 |
COOH |
2,252 |
|
Glu 166 |
-N= |
1,926 |
||
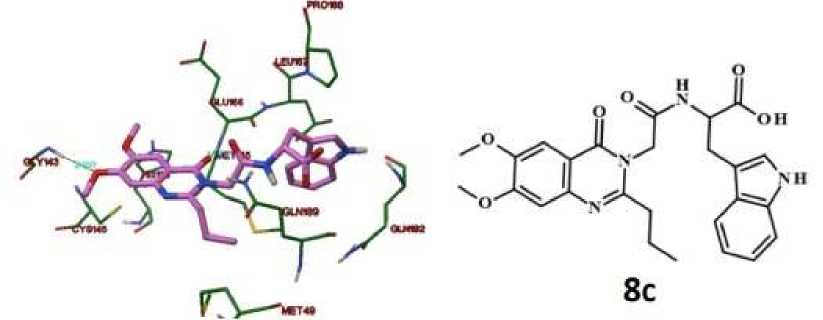
Рисунок 4. Расположение соединения 8с в активном сайте главной протеазы SARS-Cov-2
Как и в случае с главной протеазой наибольшее сродство к папаино-подобной протеазе SARS-Cov-2 прогнозируется для производных 6,7-диметоксихиназолин-4(3 H )-она с остатками глициллейцина и глицилтриптофана, вещества 4с, 7с и 8с (Таблица 2).
Таблица 2
ЗНАЧЕНИЯ ЭНЕРГИЙ ЛИГАНД-ФЕРМЕНТНОГО КОМПЛЕКСА
И ДЛИН ВОДОРОДНЫХ СВЯЗЕЙ ПРОИЗВОДНЫХ 6,7-ДИМЕТОКСИХИНАЗОЛИН-4(3 H )-ОНА С ПАПАИНО-ПОДОБНОЙ ПРОТЕАЗОЙ SARS-COV-2
|
Соединение |
Энергия докинга, ккал/моль |
Водородные связи |
||
|
Остаток аминокислоты |
Группа лиганда |
Длина водородной связи, Å |
||
|
1с |
-5,22 |
Arg 166 |
-COOH |
1,981 |
|
2с |
-6,55 |
Asn 267 |
-COOH |
2,135 |
|
Tyr 268 |
-COOH |
1,916 |
||
|
3с |
-6,75 |
Tyr 268 |
-COOH |
1,910 |
|
4с |
-7,56 |
Trp 1 |
-СОNH- |
1,984 |
|
5с |
-5,67 |
Thr 301 |
-COOH |
2,126 |
|
Arg 166 |
-COOH |
2,169 |
||
|
6с |
-6,75 |
Tyr 268 |
-COOH |
2,013 |
|
7с |
-7,29 |
Gly 266 |
-COOH |
2,053 |
|
8с |
-8,82 |
— |
— |
— |
|
9с |
-6,52 |
— |
— |
— |
|
10с |
-6,86 |
Gln 269 |
-COOH |
1,803 |
|
11с |
-5,30 |
Tyr 268 |
-COOH |
1,859 |
|
12с |
-7,15 |
Thr 301 |
-COOH |
2,007 |
|
13с |
-8,11 |
Arg 166 |
-COOH |
1,856 |
|
Arg 166 |
COOH |
2,041 |
||
|
Телапривир |
-7,52 |
Arg 166 |
-N= |
2,113 |
Однако структура 13с, содержащая остаток β-аланина, предположительно обладает соизмеримой энергией взаимодействия в сравнении с соединениями-лидерами (-8,11 ккал/моль, 13с). Экспериментальные исследования in vitro производные 6,7-диметоксихиназолин-4(3H)-она показали достаточно высокую способность ингибировать протеазы SARS-Cov-2 (Таблица 3). Наиболее выраженная активность наблюдается у вещества 8с, содержащего остаток глицилтриптофана. Причем согласно литературным данным, соединения-лидеры превосходят по активности известный препарат эбселен, для которого концентрация полумаксимального ингибирования для главной протезы приводится равная 0,67±0,09 мкМ/мл, а для папаиноподобной — 0,52±0,06 мкМ/мл [20].
ВЛИЯНИЕ ИССЛЕДУЕМЫХ СОЕДИНЕНИЙ
НА ИНГИБИРОВАНИЕ MPRO И PLPRO SARS-COV-2
Таблица 3
|
Соединение |
IC50, мкМ/мл |
Соединение |
IC50, мкМ/мл |
|
Ингибирование главной протеазы SARS-Cov-2 |
|||
|
1c |
0,09±0,008 |
8c |
0,012±0,0048 |
|
2c |
0,09±0,008 |
9c |
0,098±0,0076 |
|
3c |
0,1±0,007 |
10c |
0,062±0,0004 |
|
4c |
0,08±0,009 |
11c |
0,046±0,001 |
|
5c |
0,062±0,0015 |
12c |
0,09±0,004 |
|
6c |
0,081±0,0046 |
13c |
0,1±0,007 |
|
7c |
0,032±0,0087 |
эбселен |
0,67±0,09 [20] |
Бюллетень науки и практики / Bulletin of Science and Practice Т. 9. №8. 2023
|
Соединение |
IC50, мкМ/мл |
Соединение |
IC50, мкМ/мл |
|
Ингибирование папаино |
подобной протеазы SARS-Cov-2 |
||
|
1c |
0,08±0,002 |
8c |
0,013±0,0093 |
|
2c |
0,07±0,004 |
9c |
0,041±0,001 |
|
3c |
0,07±0,004 |
10c |
0,074±0,0038 |
|
4c |
0,05±0,006 |
11c |
0,023±0,002 |
|
5c |
0,016±0,0006 |
12c |
0,07±0,007 |
|
6c |
0,025±0,0072 |
13c |
0,1±0,003 |
|
7c |
0,018±0,001 |
эбселен |
0,52±0,06 [20] |
В результате проведенного исследования осуществлено молекулярное конструирование и дальнейший целенаправленный синтез производных 6,7-диметоксихиназолин-4(3H)-она с остатками нейроактивных аминокислот и дипептидов обладающих антикоронавирусным действием. Полученные вещества следует рассматривать в качестве перспективных кандидатов для разработки новых противовирусных лекарственных средств. Предложенный алгоритм поиска инновационных противовирусных препаратов in silico достаточно хорошо согласуется с результатами эксперимента in vitro .
Финансирование: Исследование выполнено при финансовой поддержке РФФИ в рамках научного проекта №20-315-90060.
Financing . The reported study was funded by RFBR, project no. 20-315-90060.
Список литературы Моделирование и изучение антикоронавирусной активности производных 6,7-диметоксихиназолин-4(3H)-она в отношении SARS-CoV-2 методами in silico и in vitro
- Wu D., Wu T., Liu Q., Yang Z. The SARS-CoV-2 outbreak: what we know // International journal of infectious diseases. 2020. V. 94. P. 44-48. https://doi.org/10.1016/j.ijid.2020.03.004
- Pozzi C., Vanet A., Francesconi V., Tagliazucchi L., Tassone G., Venturelli A., Tonelli M. Antitarget, Anti-SARS-CoV-2 Leads, Drugs, and the Drug Discovery–Genetics Alliance Perspective // Journal of Medicinal Chemistry. 2023. V. 66. №6. P. 3664-3702. https://doi.org/10.1021/acs.jmedchem.2c01229
- Золотых Д. С., Дайронас Ж. В., Поздняков Д. И. Противовирусная активность соединений, содержащих структурный фрагмент бензопиран-2-она // Вопросы биологической, медицинской и фармацевтической химии. 2022. Т. 25. №6. С. 22-27. https://doi.org/10.29296/25877313-2022-06-03
- Чиряпкин А. С. Обзор производных пиримидина как фармакологически активных соединений // Juvenis Scientia. 2022. Т. 8. №5. С. 16-30. https://doi.org/10.32415/jscientia_2022_8_5_16-30
- Verma V. A., Saundane A. R., Meti R. S., Vennapu D. R. Synthesis of novel indolo [3, 2-c] isoquinoline derivatives bearing pyrimidine, piperazine rings and their biological evaluation and docking studies against COVID-19 virus main protease // Journal of Molecular Structure. 2021. V. 1229. P. 129829. https://doi.org/10.1016/j.molstruc.2020.129829
- Abu-Zaied M. A., Elgemeie G. H., Mahmoud N. M. Anti-covid-19 drug analogues: synthesis of novel pyrimidine thioglycosides as antiviral agents against SARS-COV-2 and avian influenza H5N1 viruses // ACS omega. 2021. Vol. 6. №26. P. 16890-16904. https://doi.org/10.1021/acsomega.1c01501
- Alamshany Z. M., Khattab R. R., Hassan N. A., El-Sayed A. A., Tantawy M. A., Mostafa A., Hassan A.A. Synthesis and Molecular Docking Study of Novel Pyrimidine Derivatives against COVID-19 // Molecules. 2023. Vol. 28. №2. P. 739. https://doi.org/10.3390/molecules28020739
- Khan S., Kale M., Siddiqui F., Nema N. Novel pyrimidine-benzimidazole hybrids with antibacterial and antifungal properties and potential inhibition of SARS-CoV-2 main protease and spike glycoprotein // Digital Chinese Medicine. 2021. V. 4. №2. P. 102-119. https://doi.org/10.1016/j.dcmed.2021.06.004
- Algethami F. K., Cherif M., Jlizi S., Ben Hamadi N., Romdhane A., Elamin M. R., ... & Ben Jannet H. Design, Microwave-Assisted Synthesis and In Silico Prediction Study of Novel Isoxazole Linked Pyranopyrimidinone Conjugates as New Targets for Searching Potential Anti- SARS-CoV-2 Agents // Molecules. 2021. V. 26. №20. P. 6103. https://doi.org/10.3390/molecules26206103
- Кодониди И. П., Чиряпкин А. С., Творовский Д. Е. Молекулярное конструирование N-ацильных производных 2-(2-оксопиролидин-1-ил)-ацетамида, обладающих ГАМК-ергической и глутаматергической активностями // Фармация и фармакология. 2021. Т. 9. №1. С. 84-97. https://doi.org/10.19163/2307-9266-2021-9-1-84-97
- Chiriapkin A. S., Kodonidi I. P., Pozdnyakov D. I. Synthesis and evaluation of cerebroprotective activity of novel 6,7-dimethoxyquinazolin-4(3H)-one derivatives containing residues of amino acids and dipeptides // Chimica Techno Acta. 2022. Vol. 9. №2. P. 20229212. https://doi.org/10.15826/chimtech.2022.9.2.12
- Morris G. M., Huey R., Lindstrom W., Sanner M. F., Belew R. K., Goodsell D. S., Olson A. J. Autodock4 and AutodockTools4: Automated Docking with Selective Receptor Flexibility // J. Comput. Chem. 2009. V. 30. P. 2785-2791. https://doi.org/10.1002/jcc.21256
- Berman H. M., Westbrook J., Feng Z., Gilliland G., Bhat T. N., Weissig H., Bourne P. E. The Protein Data Bank // Nucleic Acids Res. 2000. V. 28. №1. P. 235-242. https://doi.org/10.1093/nar/28.1.235
- Brian J. T. Hyperchem, release 2: molecular modeling for the personal computer // J. Chem. Inf. Comput. Sci. 1992. V. 32. P. 757–759. https://doi.org/10.1021/ci00010a025
- O'Boyle N. M. Banck M., James C. A., Morley C., Vandermeersch T., Hutchison G. R. Open Babel: An open chemical toolbox // J. Cheminform. 2011. V. 3. №1. P. 33. https://doi.org/10.1186/1758-2946-3-33
- Andi B., Kumaran D., Kreitler D. F., Soares A. S., Keereetaweep J., Jakoncic J., .McSweeney S. Hepatitis C virus NS3/4A inhibitors and other drug-like compounds as covalent binders of SARS-CoV-2 main protease // Sci. Rep. 2022. V. 12. P. 12197. https://doi.org/10.1038/s41598-022-15930-z
- Shen Z., Ratia K., Cooper L., Kong D., Lee H., Kwon Y., Xiong R. Design of SARSCoV- 2 PLpro Inhibitors for COVID-19 Antiviral Therapy Leveraging Binding Cooperativity // J. Med. Chem. 2022. V. 65. №4. P. 2940-2955. https://doi.org/10.1021/acs.jmedchem.1c01307
- Yang H., Xie W., Xue X., Yang K., Ma J., Liang W., Rao Z. Design of wide-spectrum inhibitors targeting coronavirus main proteases // PLoS Biol. 2005. V. 3. №11. P. e428. https://doi.org/10.1371/journal.pbio.0030428
- Pitsillou E., Liang J., Ververis K., Lim K. W., Hung A., Karagiannis T. C. Identification of small molecule inhibitors of the deubiquitinating activity of the SARS-CoV-2 papain-like protease: in silico molecular docking studies and in vitro enzymatic activity assay // Front. Chem. 2020. V. 8. P. 623971. https://doi.org/10.3389/fchem.2020.623971
- Amporndanai K., Meng X., Shang W., Jin Z., Rogers M., Zhao Y., Samar Hasnain S. Inhibition mechanism of SARS-CoV-2 main protease by ebselen and its derivatives // Nat. Commun. 2021. V. 12. №1. P. 3061. https://doi.org/10.1038/s41467-021-23313-7


