Молекулярно-генетический анализ начальных этапов каллусогенеза in vitro у Bryophyllum daigremontianum
Автор: Геращенков Григорий Алексеевич, Абрамов Сергей Николаевич, Горбунова Валентина Юрьевна, Рожнова Наталья Анатольевна, Гильмаева Алина Владиславовна
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Биотехнология
Статья в выпуске: 3-5 т.15, 2013 года.
Бесплатный доступ
В данной работе рассмотрены особенности начальных этапов развития каллусов в культуре листовых эксплантов каланхоэ. Выполнен молекулярно-генетический анализ каллусогенеза на основе ключевых локусов клеточного цикла.
Каланхоэ, морфогенез, каллусогенез, днк-маркеры
Короткий адрес: https://sciup.org/148202049
IDR: 148202049 | УДК: 62-762.001.5
Текст научной статьи Молекулярно-генетический анализ начальных этапов каллусогенеза in vitro у Bryophyllum daigremontianum
Вивипария – это способ воспроизведения потомства, при котором из полового зародыша в семени либо из выводковой почки образуются проростки (пропагулы) на материнском организме без периода покоя [1]. Проблема вивипарии напрямую связана с феноменом функционирования стволовых клеток растений, молекулярные механизмы этого процесса далеки от понимания. Цель нашего исследования – создание коллекции молекулярногенетических маркеров на начальных этапах морфогенеза при формировании пропагулы Bryophyllum daigremontianum .
РНК выделяли из меристематических и паренхимных клеток фенольно - детергентным методом. Пробойником диаметром 1-1,5 мм брали ткань как из меристематической зоны (М2 - стадия глобулярного зародыша), так и окружающей паренхимы (П2 - стадия глобулярного зародыша) и каллуса (К) [2]. В работе использовали амплификационный полиморфизм специфичных последовательностей, или АПСП-ПЦР (SSAP, Sequence-Specific Amplification Polymorphisms) [3]. Вкратце метод можно описать следующим образом. Геномная ДНК или кДНК разрезается на мелкие фрагменты эндонуклеазой рестрикции, которая преимущественно расщепляет ДНК за пределами якорной последовательности. Таким образом, происходит упрощение генома. ДНК фрагменты затем лигируются (пришиваются) к адапторам, и осуществляется ПЦР амплификация при использовании праймеров, соответствующих адаптеру и оконечности специфической якорной последовательности. Этот метод создает набор АПСП-ПЦР фрагментов, содержащих участки якорной последовательности вплоть до ближайшего рестрикционного сайта [3].
Предположение, лежащее в основе метода, заключается в том, что каждый специфический якорный элемент расположен на статистически различном расстоянии от сайта рестрикции и, как ожидается, создает уникальный размер АПСП-ПЦР фрагмента. Затем фрагменты фракционируют в агарозных или полиакриламидных гелях для получения АПСП-ПЦР профилей, которые могут быть использованы в оценке генетического полиморфизма. Очевидно АПСП-ПЦР является модификацией метода AFLP (полиморфизма длины амплификаци-онных фрагментов).
Формирование адаптеров V20/dv12 для предсе-лективной амплификации осуществляли на основе олигонуклеотидов, представленных в таблице 1.
Таблица 1. Праймеры на основе адаптеров для SSAP анализа
|
№№ |
5`5`-3` последовательность |
|
1. |
V V20 ctcgaacacaccgtctcctc |
|
2. |
D dv12 tagaggagacga |
|
3. |
V V23 ctcgaacacaccgtctcctctac |
|
4. |
V Vgcc ctcgaacacaccgtctcctctac-gcc |
|
5. |
V Vtac gaacacaccgtctcctctac-tac |
В отличие от классического варианта AFLP, где используются анонимные праймеры, метод АПСП-ПЦР предполагает применение одного праймера на основе адаптера и второго локус специфичного праймера, комплементарного к изучаемому геномному локусу. Использованные праймеры на основе адаптера содержали по 3 селективных нуклеотида на 3` конце для улучшения специфичности АПСП – ПЦР, а именно праймеры Vgcc и Vtac (табл. 1).
В качестве локус специфичных олигонуклеотидов использовали праймеры, комплементарные локусам, некоторые из которых вовлечены в работу клеточного цикла (табл. 2). Использованы 12 вариантов таких праймеров (табл. 2). Результаты АПСП–ПЦР представлены на рис. 1-4. Как следует из представленных рисунков, в большинстве случаев нам удалось детектировать полиморфизм экс-прессионных спектров, демонстрирующих принципиальную возможность участия этих локусов в каллусогенезе у B. daigremontianum .
Таблица 2 . Олигонуклеотиды для SSAP анализа
|
№ |
5`----3` последовательность |
Фрагмент гена, аминокислотная последовательность |
|
1 |
Grig2 tgc-cgc-tac-ccc-tac-ga |
Протеинкиназа сdc25, IID-CRYPYE |
|
2 |
Grig3 ggc-gaa-ggc-aca-tac-gg |
Протеинкиназа cdc2, GEGTYG |
|
3 |
Grig4 gcg-aga-tgg-tat-gaa-tgg |
Протеинкиназа cdc5, CKAR-WYEW |
|
4 |
Grig5 gac-ata-cac-ggg-caa-t |
Щелочная фосфатаза PP1, СGDIHGQY |
|
5 |
Grig6 act-gtg-tgt-ggc-gat-gt |
Щелочная фосфатаза PPP1/2, V(I)TVCGDV |
|
6 |
Grig7 gat-ggg-gac-gga-atg-gt |
Ген вивипарии VP14, LFDGDGMV |
|
7 |
CDKB2,1_F ATGGACGAGGGAGTTATAGCAGTTTC |
CyclinB2 |
|
8 |
CDKB2,1_R TCAAAAACCAGGTACAGTACAGTTTTGCC |
CyclinB2 |
|
9 |
CYCD4,2_F TCAGAATCCAAGCCCTTGGT |
CyclinD4 |
|
10 |
CYCD4,2_R ACGGTGTCACTGCCCGTAAC |
CyclinD4 |
|
11 |
TUB4_F CTCTGTGCATCAGCTTGTCGAAAACG |
Tubulin |
|
12 |
TUB4_R CCGAGGGAGCCATTGACAACATCTT |
Tubulin |
На рис. 1 видно, что спектры амплификации кДНК каллуса отличаются от спектров кДНК М2 и Р2 для локусов протеинкиназ cdc2 и cdc5, вовлеченных в функционирование клеточного цикла. В дорожке № 6 детектируется маркерные фрагменты около 250 п.о. и 350 п.о., а в дорожке № 9 - ампликоны около 280 п.о. и 320 п.о.
Как следует из рис. 2, спектры амплификации кДНК каллуса отличаются от спектров кДНК М2 и Р2 для локуса щелочной фосфатазы РР1. В треке № 3 детектируется маркерный фрагмент около 500 п.о. (комбинация праймеров Grig5+Vtac).
Рис. 3 демонстрирует, что спектры амплификации кДНК каллуса отличаются от спектров кДНК М2 и Р2 для генов циклинов B и D , вовлеченных в функционирование клеточного цикла. Так, в дорожке №3 можно отметить ампликон около 850 п.о. и в дорожке № 6 ампликон около 650 п.о. Оба маркера получены для гена циклина В. В дорожке № 9 виден фрагмент около 700 п.о., ассоциированный с возможной экспрессионной активностью гены цик-лина D4.
На рис. 4 показано, что спектры амплификации кДНК каллуса отличаются от спектров кДНК М2 и Р2 для генов циклина D и тубулина. Так, в треке № 3 виден фрагмент около 650 п.о. характеризующий активность гена циклина D. В дорожке № 6 можно отметить маркерный фрагмент около 300 п.о., а в дорожке № 9 - маркерный фрагмент около 350 п.о., характеризующие активность гена тубулина.
Можно предполагать, что полученные нами молекулярные АПСП-ПЦР маркеры ассоциированы с процессами морфогенеза, а именно каллусогенеза, в культуре in vitro у B.daigremontianum , и потому представляют интерес огромный научный интерес для понимания проблем специализации растительных клеток и тканей.
Выявленные маркеры предстоит фракционировать в полиакриламидном геле, клонировать и сек-венировать.
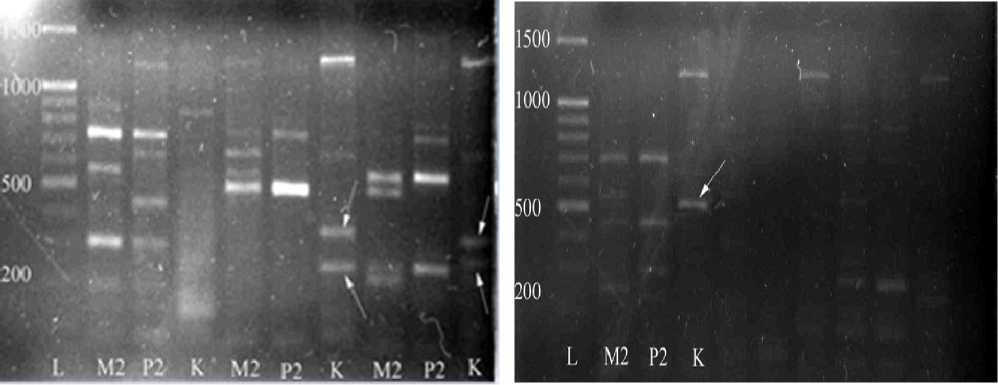
Рис. 2. Амплификационный полиморфизм специфичных последовательностей, или АПСП-ПЦР. Праймеры Grig5+Vtac. Маркерный фрагмент 500 п.о.
Рис. 1. Амплификационный полиморфизм специфичных последовательностей, или АПСП-ПЦР. (1) Праймеры Grig3+Vtac. Маркерные фрагменты около 250 и 350 п.о. (2) Праймеры Grig4+Vtac. Маркерные фрагменты около 280 и 320 п.о.
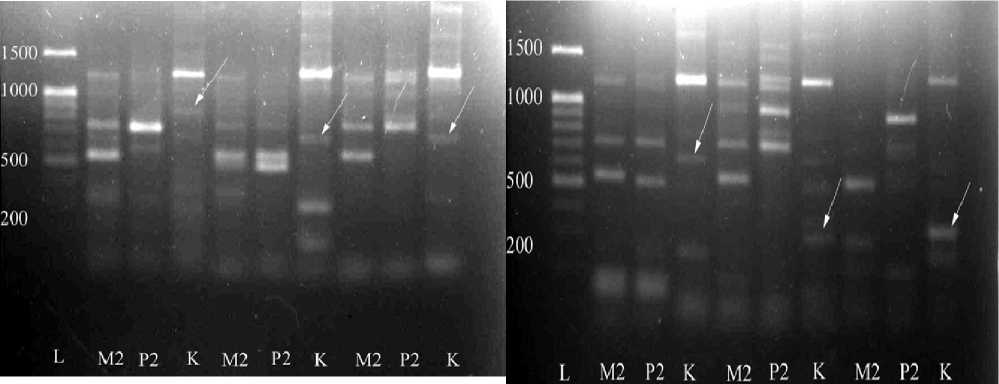
Рис. 3. Амплификационный полиморфизм специфичных последовательностей, или АПСП-ПЦР. (1) CYCD_R+Vtac Маркерный фрагмент около 850 п.о. (2) Праймеры CYCD_R+Vtac. Маркерный фрагмент около 650 п.о. (3) Праймеры CYCD_F+Vtac Маркерный фрагмент 700 п.о.
Рис. 4. Амплификационный полиморфизм специфичных последовательностей, или АПСП-ПЦР. (1) Праймеры CYCD_+Vtac Маркерный фрагмент около 650 п.о. (2) Праймеры TUB4_F+Vtac Маркерный фрагмент 300 п.о. (3) Праймеры TUB4_R+Vtac. Маркерный фрагмент около 350 п. о.
ВЫВОДЫ
У B. daigremontianum обнаружены специфичные молекулярные маркеры на начальных этапах морфогенеза при формировании каллуса:
фрагменты около 250 п.о. и 350 п.о. на основе локуса протеинкиназы Cdc2;
фрагменты около 320 п.о. и 280 п.о. на основе локуса протеинкиназы Cdc5;
фрагмент около 500 п.о. на основе локуса щелочной фосфатазы РР1;
фрагменты около 850 п.о. на основе локуса цик-лина В2.1;
фрагмент около 700 п.о. на основе локуса цик-лина D4;
фрагмент около 300 п.о. на основе локуса тубу- лина.
Очевидно, морфогенез in vitro и метод ампли-фикационного полиморфизма специфичных последовательностей (АПСП-ПЦР) могут быть использован в качестве биотехнологии нового поколения для снижения техногенной нагрузки на экосистемы при нетрадиционных системах размножения растений.
Работа выполнена при частичной поддержке Российского фонда фундаментальных исследований (гранты № 11_04_97039 р_поволжье_а, № 13_04_01873 и № 13_04_01404).
-
1. Батыгина Т.Б., Брагина Е.А. Вивипария // Эмбриология цветковых растений. Терминология и концепции. Т. 3 / Под ред. Т.Б. Батыгиной. СПб.: Мир и семья, 2000. С.3942.
-
2. Геращенков Г.А., Постригань Б.Н., Бакирова А.В., Абрамов С.Н., Горбунова В.Ю. Скрининг молекулярных маркеров при вивипарии у Bryophyllum daigremontianum
(Berger) // Мат. междунар. научно-практ. конф. «Роль классических университетов в формировании инновационной среды регионов». Уфа: БашГУ, 2009.
-
3. Waugh R., McLean K., Flavell A.J. et. al. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (SSAP). // Mol. Gen. Genet. 1997. V. 253. P. 687-694.
MOLECULAR-GENETIC ANALYSIS OF BRYOPHYLLUM DAIGREMONTIANUM AT THE INITIAL STAGES OF CALLUSOGENESIS IN VITRO
A.V. Gilmaeva1
-
1Bashkir State Pedagogical University named M. Akmullah, Ufa
-
2Institute of Biochemistry and Genetics, Ufa Sci. Center of RAS, Ufa
Список литературы Молекулярно-генетический анализ начальных этапов каллусогенеза in vitro у Bryophyllum daigremontianum
- Батыгина Т.Б., Брагина Е.А. Вивипария//Эмбриология цветковых растений. Терминология и концепции. Т. 3/Под ред. Т.Б. Батыгиной. СПб.: Мир и семья, 2000. С.39-42.
- Геращенков Г.А., Постригань Б.Н., Бакирова А.В., Абрамов С.Н., Горбунова В.Ю. Скрининг молекулярных маркеров при вивипарии у Bryophyllum daigremontianum (Berger)//Мат. междунар. научно-практ. конф. «Роль классических университетов в формировании инновационной среды регионов». Уфа: БашГУ, 2009.
- Waugh R., McLean K., Flavell A.J. et. al. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (SSAP).//Mol. Gen. Genet. 1997. V. 253. P. 687-694.


