Молекулярно-генетическое типирование генов, контролирующих скорость оперения крыла у кур (Gallus gallus L.), в связи с разделением по полу
Автор: Алексеев Я.И., Бородин А.М., Никулин А.В., Емануйлова Ж.В., Ефимов Д.Н., Фисинин В.И.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Птицеводство: наука и технологии
Статья в выпуске: 2 т.52, 2017 года.
Бесплатный доступ
Традиционная селекция животных длительна по времени и требует больших материальных затрат. Применение лабораторных молекулярно-генетических методов значительно ускоряет и удешевляет процесс создания пород и кроссов с нужными свойствами, отбор желательных генотипов. Молекулярное типирование по аллелям, сцепленным с полом, важно для получения исходных линий птицы, дающих аутосексное потомство. Разделение по полу при выращивании бройлеров - известный технологический прием, который обеспечивает более эффективное использование корма, лучшую выживаемость, сортность, повышенное сходство по массе. Нами разработан тест, позволяющий, используя технологию количественной ПЦР в реальном времени (ПЦР-РВ), различать у 1-суточных цыплят гомо- и гетерозиготное состояние сцепленных с полом аллелей К и k, отвечающих за скорость роста перьев крыла. Применение количественной ПЦР-РВ для анализа генотипов сводится к дискриминации содержания одной копии гена в геноме от двух. Для получения надежных результатов при проведении анализа необходимо придерживаться определенных правил: во-первых, эффективность ПЦР должна приближаться к максимуму, во-вторых, без надлежащей статистической обработки данных возможно получение ложных результатов. Для преодоления ограничений количественной ПЦР-РВ предложен новый алгоритм тестирования: все пробы подвергаются двум последовательным независимым анализам одновременно с референсными образцами (известные генотипы), а с пробами, давшими противоположные результаты, проводится повторный анализ без учета предыдущих данных. Такой подход исключает возможные ошибки. Предложена новая система из трех (вместо четырех) праймеров для амплификации двух генов и зонда, позволяющая эффективно дискриминировать генотипы КК, Кk и kk. Данные количественной ПЦР-РВ анализировали по методу ΔΔCt и обрабатывали при помощи пакета программ статистики SPSS с применением ROC-анализа. Использовав разработанный тест, мы определили процентное соотношение генотипов КК, Кk и kk среди 145 петухов исходных линий Б5, Б6, Б7 и Б9 отечественного мясного кросса Смена 8. Показано, что все протестированные 19 петухов линии Б5 и все 15 петухов линии Б6 имели генотип kk. Из 46 петухов линии Б7 ни у одного не обнаружили генотип kk, у 17 особей (37 %) выявили генотип Кk, у 29 особей (63 %) - генотип КК. Из 65 петухов линии Б9 ни один не был гомозиготным по аллелю k (генотип kk ), 17 особей (26 %) оказались гетерозиготами с генотипом Кk и 48 особей (74 %) имели генотип КК. Для исключения влияния возможной вариабельности нуклеотидных последовательностей ДНК на результаты анализа провели секвенирование и установили отсутствие каких-либо нуклеотидных замен в участках отжига праймеров и зондов. Полученные результаты определения пола у 1-суточных цыплят с помощью количественной ПЦР-РВ (на основании выявления аллелей К и k, отвечающих за скорость роста перьев крыла) позволяют ускорить селекцию кур с целью создания нового отечественного мясного кросса. Дальнейшая селекционная работа предполагает оценку птицы традиционными и молекулярно-генетическими методами.
Полимеразная цепная реакция в реальном времени, генотип, копийность генов, аутосексные куры, селекция птицы, мясной кросс
Короткий адрес: https://sciup.org/142214173
IDR: 142214173 | УДК: 636.52/.58:636.082.2:577.21 | DOI: 10.15389/agrobiology.2017.2.367rus
Текст научной статьи Молекулярно-генетическое типирование генов, контролирующих скорость оперения крыла у кур (Gallus gallus L.), в связи с разделением по полу
Разделение цыплят по полу (сексирование), используя простые фенотипические приемы (например, дифференциация по скорости роста перьев крыла, по цвету перьев), дает целый ряд преимуществ по сравнению с традиционным методом, при котором определяют наличие половых бугорков. Решение этой актуальной задачи позволяет значительно снизить требования к персоналу, уменьшить процент ошибок и увеличить скорость сортировки цыплят. Для сексирования у бройлеров используется разная скорость оперяемости крыла у цыплят противоположного пола в 1-су-
При проведении исследования использовано научное оборудование центра коллективного пользования «Биотехнология» Всероссийского НИИ сельскохозяйственной биотехнологии. Работа выполнена при финансовой поддержке ФАНО России, дополнительное государственное задание ФГБУ СГЦ Смена на IV квартал 2016 года ¹ 007 от 5 августа 2016 года, в рамках выполнения Федеральной научно-технической программы развития сельского хозяйства на 2017-2025 годы (Подпрограмма 2 «Создание отечественного конкурентоспособного кросса мясной птицы»).
точном возрасте (1-3). Генетика этого явления хорошо изучена (4-6). Аллель медленной оперяемости К расположен на Z хромосоме и сцеплен с полом. У петухов в геноме присутствуют две мужские половые хромосомы ZZ, у куриц — одна мужская Z и одна женская W. Определить пол у цыплят в 1-суточном возрасте можно по степени развития оперения крыла, которая наследуется через сцепленные с полом аллели — доминантный К и рецессивный к (быстрая оперяемость) (7). У 1-суточных цыплят, имеющих генотип КК , Кk или Кw , формирование перьевого покрова крыла замедлено (слабо развиты маховые перья, они либо одинаковые по длине, либо кроющие перья длиннее маховых), у особей с генотипами кк или кw маховые перья крыла хорошо развиты, а кроющие короче маховых. Использование в скрещивании петухов, гомозиготных по гену быстрой опе-ряемости kk , и кур Кw — носителей гена медленной оперяемости приводит к получению потомства, среди которого все цыплята с быстрым оперением крыла — курочки kw , с медленным — петушки Кk .
Детальное исследование гена К , расположенного в области интеграции эндогенного ретровируса ev21, позволило установить связь между удвоением тандема частично делетированных генов жгутикового белка сперматозоидов dSpef2 и рецептора пролактина dPrlr с генотипом K (8). Изучение механизмов, определяющих медленное оперение, показало важность экспрессии продуктов генов dSpef2 и dPrlr в кожном покрове (9, 10). Рецептор пролактина, лигандом которого служит гормон гипофиза пролактин, относится к семейству рецепторов цитокинов и экспрессируется во всех тканях позвоночных. Пролактину приписывается около 300 различных функций, в том числе поддержание водно-солевого баланса, участие в процессах роста и развития, обмена веществ, в деятельности гормональной системы и мозга, в контроле поведения, в регуляции и работе иммунной системы (11), в воспроизводстве, продукции яиц у кур (12), индукции линьки (13).
Особенность отечественного кросса Смена 8 — его универсальность. При стабильно высокой скорости роста молодняка, что позволяет сократить сроки выращивания, птица имеет устойчивую репродукцию, хорошо адаптирована к российским условиям (в отличие от зарубежных кроссов), может использоваться при клеточной и напольной технологиях содержания. Родительское стадо имеет высокий пик яйцекладки (85 %). Для кросса также характерна хорошая жизнеспособность, однородность стада, высокий выход грудных и ножных мышц, эффективная конверсия корма .
Получение аутосексных цыплят с разной оперяемостью в 1-суточном возрасте (на примере скрещивания петухов исходной линии Б7 и кур исходной линии Б9 мясного кросса Смена 8) иллюстрирует схема:
Линия Б7 ( d) х Б9 ( $)
Генотип kk Кw
Признак Быстрое оперение крыла Медленное оперение крыла Потомство Б79
Генотип Кк ( d) kw ( $)
Признак Медленное оперение крыла Быстрое оперение крыла
Сексирование необходимо для раздельного выращивания бройлеров. Этот технологический прием обеспечивает более эффективное использование корма, лучшую выживаемость, сортность, повышенное сходство по массе, то есть способствует улучшению выходных показателей бройлеров при меньших экономических издержках.
Нами предложена новая тест система, позволяющая определять ко-пийность гена, ответственного за скорость оперения кур, а также алгоритм анализа копийности генов при помощи количественной ПЦР в реальном 368
времени. Это дает возможность эффективно и с высокой пропускной способностью генотипировать аллели К и k кур для ускоренного получения исходных линий мясного кросса Смена 8, дающих аутосексное потомство.
Цель исследования заключалась в разработке теста для молекулярного типирования генов быстрой и медленной оперяемости у кур с использованием ПЦР в реальном времени (ПЦР-РВ).
Методика . Испытание тест-системы и алгоритма генотипирования проводили на 145 образцах ДНК петухов линий Б5, Б6, Б7 и Б9 отечественного мясного кросса Смена 8.
ДНК выделяли из отобранных перьев кур с использованием набора М-Сорб (ЗАО «Синтол», Россия). Предварительно проводили пробоподго-товку: 0,5 см кончика очина пера помещали в пробирку объемом 1,5 мл (ООО «Техоснастка», Россия). Добавляли 400 мкл лизирующего раствора и инкубировали при 37 ° С в течение 20 мин с перемешиванием, лизат осаждали в высокоскоростной микроцентрифуге Циклотемп-902 (ЗАО «Циклотемп», Россия) в течение 3 мин при 13 тыс. об/мин. Супернатант переносили в пробирку объемом 1,5 мл и продолжали выделение в соответствии со стандартным протоколом к набору М-Сорб. Концентрацию ДНК определяли на спектрофотометре NanoPhotometer («Implene», Германия). В реакцию брали около 3 мкг ДНК (А260/280 = 1,8-2,0). Праймеры и зонды для ПЦР-РВ и секвенирования, представленные ниже, разработаны на основе известных последовательностей ДНК (9, 10, 14) и синтезированы в ЗАО «Синтол»; использованные флуоресцентные метки — карбоксифлуоресцеин (FAM) и 6-карбоксиродамин (R6G) с 3´- концевой модификацией фосфатом (р):
LFBF1
LFBR1
LFBV1 (зонд) NFR
NFP (зонд) PROSF PROSR SPEFR
5' ^ 3': CTCACTGAAACCATCCCTGGA
5' ^ 3': CTAACCTGAGACAAGTGTCGGA
5' ^ 3': (6FAM)-CCCCTTAAATGCC(dT-BHQ1)TGCTTTTCCATC-p
5' ^ 3': CTGTGGTTTGCTCGGTTTTGGA
5' ^ 3': (5R6G)-CTCCATCTCTCC(dT-BHQ1)TGCTTTTCCATC-p
5' ^ 3': GTTTGCCTATCACAGCATTGTAGA
5' ^ 3': GCTGGTTCCTCATCCTGTCTGA
5' ^ 3': GTGCAATTTAGCAGTGCATGTGA
Мультиплексную ПЦР-РВ (каналы детекции — FAM и R6G) проводили на приборе АНК-32М («Институт аналитического приборостроения РАН», Россия) по следующей температурно-временной циклограмме (40 циклов): денатурация при 93 ° С — 15 с, отжиг при 60 ° С — 30 с. Использовали реакционную смесь (20 мкл) для ПЦР-РВ (ЗАО «Синтол», Россия). Концентрация праймеров в реакционной смеси составляла 450 нМ, концентрация зондов — 250 нМ. Специфичность ПЦР-РВ подтверждали секвенированием продуктов амплификации, полученных при помощи праймеров PROSF, PROSR и SPEFR, на генетическом анализаторе Нанофор 05 («Институт аналитического приборостроения РАН», Россия) в соответствии с протоколом изготовителя.
Результаты . Система праймеров LFBF1, LFBR1 и зонда LFBV1 позволяла обнаружить ген dSpef2 dPrlr , система праймеров LFBF1, NFR и зонда NFP выявляла референсный ген рецептора пролактина кур Prlr . Праймеры PROSF, PROSR и SPEFR использовались для секвенирования фрагментов генов Spef2 и Prlr .
Определение генотипов КК и Кк при помощи ПЦР-РВ сводится к задаче дискриминации двух копий гена в геноме от одной (8, 15, 16). Она может решаться при помощи количественной ПЦР (8, 17), а также методом гибридизации нуклеиновых кислот (15, 18, 19). Каждый из перечисленных подходов имеет свои преимущества и недостатки. Гибридизация по Саузерну трудоемка, занимает много времени и требует разнообразного оборудования. При использовании количественной ПЦР (ПЦР-РВ) для решения поставленной задачи результаты могут оказаться неадекватными, если не соблюдаются необходимые условия при выполнении теста и последующей статистической обработки данных (15, 18, 19). Однако при соответствующем исполнении этот метод высокотехнологичен и позволяет за короткое время накопить большой массив информации. Для надежного тестирования необходимо, чтобы за один цикл ПЦР количество ДНК удваивалось и эффективность амплификации приближалась к максимальной (20, 21). Также нужно тщательно отслеживать ее вероятные флуктуации, которые могут приводить к неправильным результатам.
Подобранные праймеры и зонды анализировали в мультиплексной ПЦР-РВ для двух образцов (¹ 64 и ¹ 65) в сериях из пяти 2-кратных разведений ДНК в двух повторностиях. Эффективность амплификации гена dSpef2 dPrlr составила 92 % (R2 = 0,9945), референсного гена пролактина — 94 % (R2 = 0,9991). Применение одного общего праймера LFBF1 для амплификации двух генов делает предложенную систему более устойчивой за счет уменьшения числа компонентов и сглаживает возможные различия в работе праймеров.
В предварительных экспериментах по определению генотипов кур образцы исследовали в 2-кратных повторностях. До 10 % проб в одном из экспериментов дали результаты, не только лежащие в так называемой «серой» области, но и достоверно противоположные в повторе одной пробы. Для повышения достоверности теста мы разработали новый алгоритм, который отличается от описанного ранее (8). Для каждой партии исследуемых образцов обязательно использование не менее четырех референсных проб для каждого из тех генотипов, относительно которых делается анализ. Затем проводится повторное независимое определение с аналогичным набором проб. Подсчитываются результаты в каждом из двух тестов, и при обнаружении расхождений выполняется новое определение с референсными пробами в двух независимых повторах. Такой алгоритм позволяет свести вероятность ошибки к минимуму.
Использовав новый алгоритм, проанализировали 19 образцов ДНК петухов из исходной линии Б5 и 15 образцов — из исходной линии Б6 мясного кросса Смена 8. В 100 % случаев был подтвержден генотип кк , что полностью коррелировало с фенотипом для этих двух исходных линий.
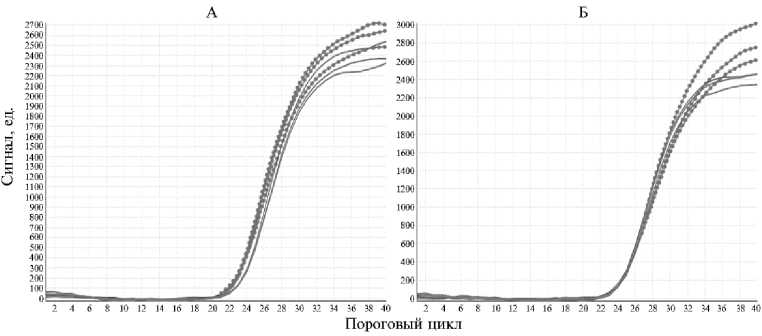
Результат генотипирования петухов линии Б7 кросса Смена 8 с помощью ПЦР в реальном времени: А — образец ¹ 64 ( Δ Сt = 0,8) для генотипа КК , Б — образец ¹ 65 ( Δ Сt = 0,02) для генотипа Кк ; « -- » — канал детекции FAM (ген dSpef2 dPrlr ), « — » — канал детекции R6G (ген Prlr ).
Для исследования петухов линий Б7 и Б9 использовали четыре референсных петуха с генотипом КК и четыре — с генотипом Кk . Примеры кинетических кривых ПЦР-амплификации, полученных для образцов ¹ 64 и ¹ 65 исходной линии Б7 приведены на рисунке. Для каждого проанализированного образца проводили расчет разницы пороговых циклов реакций по тандему генов dSpef2 dPrlr (канал детекции FAM) и гену пролактина (канал детекции R6G) по формуле Δ Сt = Ct (FAM) - Ct (R6G). Как видно из рисунка, пороговые циклы амплификации фрагмента референсного гена пролактина кур (канал детекции R6G) больше, чем пороговые циклы амплификации фрагмента гена dSpef2 dPrlr (канал детекции FAM) (см. рис., А), либо равны им (см. рис., 2). Разница объясняется тем, что в образцах ДНК особей, имеющих генотип КК , содержится в 2 раза больше копий гена К , чем у петухов с генотипом Кk . Генотипы образцов анализировали с использованием метода ΔΔ Ct (22) в соответствии с описанием (8), данные обрабатывали при помощи пакета программ статистики SPSS (23), применяя ROC-анализ (24). Использование других бинарных классификаторов (25) и методов статистики (18) для увеличения надежности результатов может быть перспективным при подобного рода исследованиях.
Результаты изучения образцов от 46 петухов исходной линии Б7 показали, что ни у одного не выявлен генотип kk , 17 (37 %) имели генотип Кk и 29 (63 %) — генотип КК . По данным анализа образцов от 65 петухов исходной линии Б9, ни у одного не обнаружили генотип kk , у 17 (26 %) — генотип Кk и у 48 (74 %) — генотип КК . Чтобы исключить возможное влияние вариабельности исследуемых участков генов, провели секвенирование продуктов амплификации ДНК, полученных при помощи пар праймеров PROSF и PROSR (ген Prlr ), а также PROSF и SPEFR (ген dSpef2 dPrlr ). Было установлено, что последовательности в анализируемых областях в целом инвариантны и не содержат нуклеотидных замен в участках отжига праймеров и зондов.
В дальнейшем предполагается оценить поголовье традиционными и молекулярно-генетическими методами. По результатам петухи линии Б7 с генотипами КК и Кk , а также петухи линии Б9 с генотипами Кk и kk и их потомки будут исключены из селекции, что позволит получить линии с заданным генотипом.
Таким образом, разработан тест и предложен алгоритм выявления у 1-суточных цыплят гомо- и гетерозиготного состояния сцепленных с полом аллелей К и k , отвечающих за скорость роста перьев крыла, с использованием количественной ПЦР в реальном времени. С помощью предложенного теста определено процентное соотношение генотипов КК , Кk и kk среди 145 петухов исходных линий Б5, Б6, Б7 и Б9 отечественного мясного кросса Смена 8. Дальнейшая селекционная работа предполагает оценку птицы традиционными и молекулярно-генетическими методами с целью исключить из селекции петухов линии Б7 с генотипами КК и Кk , петухов линии Б9 с генотипами Кk и kk , а также их потомков как не соответствующих целевым параметрам.
Список литературы Молекулярно-генетическое типирование генов, контролирующих скорость оперения крыла у кур (Gallus gallus L.), в связи с разделением по полу
- Serebrovsky A.S. Crossing-over involving three sex-linked genes in chickens. Amer. Nat., 1922, 56: 571-572 ( ) DOI: 10.1086/279898
- Warren D.C. Inheritance of rate of feathering in poultry. J. Hered., 1925, 16(1): 13-18 ( ) DOI: 10.1093/oxfordjournals.jhered.a102502
- Somes R.G. Jr. Delayed feathering, a third allele at the K locus in the domestic fowl. J. Hered., 1969, 60(5): 281-288 ( ) DOI: 10.1093/oxfordjournals.jhered.a107994
- Jones D.G., Hutt F.B. Multiple alleles affecting feathering in the fowl. J. Hered., 1946, 37(7): 197-205 ( ) DOI: 10.1093/oxfordjournals.jhered.a105617
- Warren D.C. Retarded feathering in the fowl. A new factor affecting manner of feathering. J. Hered., 1933, 24(11): 431-434 ( ) DOI: 10.1093/oxfordjournals.jhered.a103691
- Warren D.C. Developing early-feathering strains in heavy breeds of poultry. Agricultural Experiment Station, Kansas State College of Agriculture and Applied Science, 1944.
- McGibbon W.H. A sex-linked mutation affecting rate of feathering in chickens. Poult. Sci., 1977, 56(3): 872-875 ( ) DOI: 10.3382/ps.0560872
- Elferink M.G., Vallee A.A.A., Jungerius A.P., Crooijmans R.P.M.A., Groenen M.A.M. Partial duplication of the PRLR and SPEF2 genes at the late feathering locus in chicken. BMC Genomics, 2008, 9: 391 ( ) DOI: 10.1186/1471-2164-9-391
- Zhao J., Yao J., Li F., Yang Z., Sun Z., Qu L., Wang K., Su Y., Zhang A., Montgomery S.A., Geng T., Cui H. Identification of candidate genes for chicken early-and late-feathering. Poult. Sci., 2016, 95(7): 1498-1503 ( ) DOI: 10.3382/ps/pew131
- Bu G., Huang G., Fu H., Li J., Huang S., Wang Y. Characterization of the novel duplicated PRLR gene at the late-feathering K locus in Lohmann chickens. J. Mol. Endocrinol., 2013, 51: 261-276 ( ) DOI: 10.1530/jme-13-0068
- Bole-Feysot C., Goffin V., Edery M., Binart N., Kelly P.A. Prolactin (PRL) and its receptor: actions, signal transduction pathways and phenotypes observed in PRL receptor knockout mice. Endocrine Reviews, 1998, 19(3): 225-268 ( ) DOI: 10.1210/edrv.19.3.0334
- Cui J.-X., Du H-L., Liang Y., Deng X.-M., Li N., Zhang X.-Q. Association of polymorphisms in the promoter region of chicken prolactin with egg production. Poult. Sci., 2006, 85(1): 26-31 ( ) DOI: 10.1093/ps/85.1.26
- Juhn M., Harris P.C. Molt of capon feathering with prolactin. Exp. Biol. Med., 1958, 98(3): 669-672 ( ) DOI: 10.3181/00379727-98-24146
- International Chicken Genome Sequencing Consortium. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution. Nature, 2004, 432: 695-717 ( ) DOI: 10.1038/nature03154
- Bubner B., Baldwin I.T. Use of real-time PCR for determining copy number and zygosity in transgenic plants. Plant. Cell Rep., 2004, 23: 263-271 ( ) DOI: 10.1007/s00299-004-0859-y
- Mieog J.C., Howitt C.A., Ral J.P. Fast-tracking development of homozygous transgenic cereal lines using a simple and highly flexible real-time PCR assay. BMC Plant Biol., 2013, 13: 71 ( ) DOI: 10.1186/1471-2229-13-71
- Stefano B., Patrizia B., Matteo C., Massimo G. Inverse PCR and quantitative pcr as alternative methods to Southern blotting analysis to assess transgene copy number and characterize the integration site in transgenic woody plants. Biochem. Genet., 2016, 54(3): 291-305 ( ) DOI: 10.1007/s10528-016-9719-z
- Yuan J.S., Burris J., Stewart N.R., Mentewab A., Stewart C.N. Jr. Statistical tools for transgene copy number estimation based on real-time PCR. BMC Bioinformatics, 2007, 8(Suppl 7): S6 ( ) DOI: 10.1186/1471-2105-8-S7-S6
- Huang Y., Yin X., Zhu C., Wang W., Grierson D., Xu C., Chen K. Standard addition quantitative real-time PCR (SAQPCR): a novel approach for determination of transgene copy number avoiding PCR efficiency estimation. PLoS One, 2013, 8(1): e53489 ( ) DOI: 10.1371/journal.pone.0053489
- Sochivko D.G., Fedorov A.A., Varlamov D.A., Kurochkin V.E., Petrov R.V. Simulation of the PCR amplification as two-type-particle branching process. Dokl. Biochem. Biophys., 2010, 434: 239-241 ( ) DOI: 10.1134/S1607672910050054
- Sochivko D.G., Fedorov A.A., Lavrov V.V., Varlamov D.A., Kurochkin V.E., Petrov R.V. Stochastic modeling of polymerase chain reaction kinetic curves. Dokl. Biochem. Biophys., 2011, 439: 188-191 ( ) DOI: 10.1134/S1607672911040119
- Livak K.J., Schmittgen T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 2001, 25(4): 402-408 ( ) DOI: 10.1006/meth.2001.1262
- Bryman A., Cramer D. Quantitative data analysis with IBM SPSS 17, 18 and 19: A guide for social scientists. Routledge, NY, 2011 (ISBN 978-0-415-57918-6).
- Fawcett T. An introduction to ROC analysis. Pattern Recognition Letters, 2006, 27(8): 861-874 ( ) DOI: 10.1016/j.patrec.2005.10.010
- Saito T., Rehmsmeier M. The precision-recall plot is more informative than the ROC plot when evaluating binary classifiers on imbalanced datasets. PLoS ONE, 2015, 10(3): e0118432 ( ) DOI: 10.1371/journal.pone.0118432


