Мультиплексная система микросателлитного анализа для исследования интрогрессии фрагментов a-, b-, c-геномов у видов рода Brassica L. при отдаленной гибридизации
Автор: Анискина Ю.В., Родионова Д.А., Зубко О.Н., Монахос С.Г., Велишаева Н.С., Колобова О.С., Шилов И.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярные технологии
Статья в выпуске: 3 т.55, 2020 года.
Бесплатный доступ
Род Brassica L. - источник масличных, овощных, пряных, кормовых и декоративных культур, широко выращиваемых по всему миру. Большинство культурных растений относятся к трем диплоидным видам B. rapa L. (2 n = 20, геном AA), B. nigra (L.) K. Koch (2 n = 16, BB) и B. oleracea L. (2 n = 18, CC) и трем аллотетраплоидным видам B. juncea (L.) Czern. (2 n = 36, AABB), B. napus L. (2 n = 38, AACC) и B. carinata A. Braun (2 n = 34, BBCC), которые возникли в результате естественной гибридизации диплоидов. Эффективным способом увеличения генетического разнообразия и улучшения агрономических признаков культур Brassica служит отдаленная гибридизация. Для контроля интрогрессии геномного материала при отдаленной гибридизации необходимо применение методов генетического маркирования. В настоящей работе предложен эффективный подход для контроля интрогрессии геномов A, B и C Brassica у растений, полученных в результате межвидовой гибридизации. Впервые выявлены специфичные для A-, B- и C-геномов Brassica микросателлитные маркеры, длина которых определена в системе высокоразрешающего электрофореза с точностью до одного нуклеотида, а также изучена возможность применения этих маркеров для анализа селекционных образцов, полученных в результате межвидовой гибридизации. Целью исследования была разработка высокопроизводительной технологии контроля интрогрессии геномов A, B и C у представителей рода Brassica при отдаленной гибридизации на основе мультиплексного микросателлитного ПЦР-анализа геном-специфичных микросателлитных маркеров. В качестве контрольных образцов исследовали растения, полученные из Центра генетических ресурсов CGN (Centre for Genetic Resources, Нидерланды) и Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР, г. Санкт-Петербург). Растительный материал для анализа интрогрессий был предоставлен сотрудниками ООО Селекционная станция им. Н.Н. Тимофеева (г. Москва). Геномную ДНК получали методом адсорбции на сорбенте. ДНК амплифицировали в ПЦР с праймерами, специфичными для локусов Na10-D09, Na12-A02, Na12-F12, Ni2-B02, Ni2-F02, Ni3-G04B, Ol12-A04, Ra2-E12, BRMS-043, BN6A2. Анализ флуоресцентно меченных ПЦР-фрагментов осуществляли методом высокоразрешающего электрофореза (генетический анализатор Нанофор-05, ООО НРФ «Синтол»-ФГБНУ Институт аналитического приборостроения РАН, Россия). Длину ПЦР-фрагментов устанавливали с помощью программного обеспечения ДНК Фрагментный анализ (ФГБНУ ИАП РАН, Россия). В результате проведенного исследования разработана система для мультиплексного ПЦР-анализа на основе шести микросателлитных локусов (Na12-A02, BRMS-043, Na10-D09, Ol12-A04, Ni2-F02, BN6A2), позволяющая в одну стадию определить наличие маркеров геномов A, B и C у видов рода Brassica . Маркеры, специфичные для геномов A, B и C, были установлены в процессе мультиплексного ПЦР-анализа контрольных образцов Brassica с известной видовой принадлежностью и геномным составом: B. rapa (AA), B. nigra (BB), B. oleracea (CC), B. napus (AACC), B. juncea (AABB), B. carinata (BBCC). Длину маркерных фрагментов определили в системе высокоразрешающего электрофореза на генетическом анализаторе с точностью до одного нуклеотида. Маркеры, специфичные для генома A, выявлены в локусах Na12-A02 (178, 180 и 182 п.н.), BRMS-043 (303, 307 и 313 п.н.), Na10-D09 (283, 285, 291, 293 и 299 п.н.), для генома B - в локусах Na12-A02 (196, 198, 200, 202, 204, 212, 214 и 216 п.н.), Ol12-A04 (125, 127 и 129 п.н.), Ni2-F02 (198, 200, 202, 204 и 208 п.н.), BN6A2 (222 п.н.), для генома C - в локусах Na12-A02 (164, 168 и 170 п.н.), Ni2-F02 (164, 166, 168 и 186 п.н.). С использованием разработанной мультиплексной системы в генетических профилях образцов (Эт2 ½ КК)2 ½ Цв9, (Эт2 ½ КК)1, Грин ½ FBLM(1), JR ½ Агр2ки, БК ½ ЗМ1-1(6), БК ½ ЗМ1-1(8), БК и КБ, полученных в результате отдаленной гибридизации, выявлена интрогрессия фрагментов геномов A, B и/или C . Таким образом, у образцов с известной селекционной историей подтверждено наличие соответствующих геномов. Благодаря автоматизации всех этапов анализа в формате 96-луночного планшета эта технология позволяет осуществлять широкомасштабный скрининг селекционных образцов и может быть внедрена в практику как инструмент контроля интрогрессии A-, B- и C-геномного материала, а также контроля его наследования в последующих поколениях.
Род brassica, эволюционная модель u, геномы brassica, отдаленная гибридизация, интрогрессия, микросателлитные маркеры, геном-специфичные маркеры
Короткий адрес: https://sciup.org/142226313
IDR: 142226313 | УДК: 633:631.522/.524:575:57.06 | DOI: 10.15389/agrobiology.2020.3.510rus
Текст научной статьи Мультиплексная система микросателлитного анализа для исследования интрогрессии фрагментов a-, b-, c-геномов у видов рода Brassica L. при отдаленной гибридизации
Среди растений семейства Brassicaceae наибольшую экономическую ценность представляет род Brassica L. как источник масличных, овощных, пряных, кормовых и декоративных культур, широко возделываемых по всему миру. Род Brassica включает 39 видов (1). Большинство культурных растений относятся к видам B. napus L. (масличный рапс, брюква), B. rapa L. (азиатские листовые и кочанные капусты, репа и турнепс), B. oleracea L. (кочанная, брюссельская и цветная капуста, брокколи, кольраби), B. juncea Czern. (горчица сарептская) B. carinata A. Braun (горчица эфиопская) и B. nigra (L.) K. Koch (горчица черная).
Широко исследовались морфологические, цитогенетические и молекулярные взаимосвязи между культурными видами Brassica . Установлены шесть цитогенетических групп Brassica (2). Согласно эволюционной модели N. U (3), в результате естественной гибридизации трех диплоидных видов B. rapa (2 n = 20, геном AA), B. nigra (2 n = 16, BB) и B. oleracea (2 n = 18, CC) возникли амфидиплоидные виды B. juncea (2 n = 36, AABB), B. napus (2 n = 38, AACC) и B. carinata (2 n = 34, BBCC). Сравнительный анализ трех геномов Brassica обнаруживает значительную консервативность, которая свидетельствует о том, что все три генома полностью наследовались от одного общего предка, но значительно перестроились (4). Различие в числе хромосом предположительно возникло в результате слияния/фрагментации хромосом в процессе дивергенции видов (5). На основе анализа хлоропластных, митохондриальных и ядерных геномов были установлены два направления эволюции видов Brassica : B. nigra (В-геном) и B. rapa / B. oleracea (A/C-геномы) (6). Согласно данным генетического картирования, геномы A и C, несмотря на различие в числе хромосом, высококолинеарны (7). Предположительно дивергенция B. nigra и B. rapa / B. oleracea произошла 7,9 млн лет назад. Виды B. rapa и B. oleracea дивергировали около 4 млн лет назад. Гибридизация видов с образованием B. napus , по-видимому, произошла 10 тыс. лет назад (6). Также были выявлены некоторые различия в структуре субгеномов аллотетраплоидных видов по сравнению с соответствующими геномами диплоидных видов (8, 9). Дифференциация среди различных субгеномов в процессе межвидовой гибридизации и длительного одомашнивания полиплоидных видов Brassica могла произойти из-за транслокации, инверсии, делеции, дупликации, а также гомеологичной рекомбинации (8) .
Отдаленная гибридизация — эффективный способ улучшения агрономических признаков культур Brassica , таких как высокая урожайность, устойчивость к болезням и неблагоприятным условиям среды (10). В скрещиваниях видов Brassica между собой и с другими родственными представителями семейства Brassicaceae барьер межвидовой или межродовой несовместимости был успешно преодолен методами спасения зародышей или соматической гибридизации (11, 12).
Источником потенциально полезных агрономических признаков для улучшения культур могут быть дикорастущие представители семейства Bras-sicaceae (12, 13), например Brassica maurorum Durieu и Eruca versicaria ssp. sativa (Mill.) Thell. (устойчивость к белой ржавчине, возбудитель Albugo candida Pers.) (14), Raphanus sativus L. (устойчивость к нематоде Heterodera schachtii Schmidt) (15) , Sinapis alba L. (устойчивость к нематоде H. schachtii и черной ножке, возбудитель Leptosphaeria maculans (Sowerby) P. Karst.) (16), Sinapis arvensis L. (устойчивость к черной ножке, L. maculans ) (17), Sinapis incana L. и Diplotaxis L. (источник цитоплазматической мужской стерильности) (18).
У культурных видов Brassica также обнаружены полезные агрономические признаки, например устойчивость к мучнистой росе (возбудитель Hy-aloperonospora parasitica (Pers.) Constant) у B. oleracea (12), к киле (Plasmodiophora brassicae Woronin) — у B. rapa, B. oleracea и B. napus (19), к сосудистому бактериозу (Xanthomonas campestris (Pammel) Dowson) — у B. carinata (20), к тяжелым металлам — у B. juncea (21). Виды Brassica, содержащие геном B, служат источником устойчивости к черной ножке и неблагоприятным условиям среды (22, 23).
Межвидовая гибридизация — естественное природное явление, результатом которого становится образование новых видов или интрогрессия полезных адаптивных признаков. Ресинтез аллотетраплоидов Brassica из диплоидных родительских видов способствует увеличению их генетического разнообразия. С целью расширения генетической основы были повторно получены аллотетраплоиды B. juncea , B. napus и B. carinata (24-26) . Геном Raphanus был использован для получения синтетических аллотетраплоидов, устойчивых к нематоде (12). Аллогексаплоиды Brassica (2 n = 54, AABBCC) созданы посредством скрещивания B. napus ½ B. nigra и B. carinata ½ B. rapa (27). В результате гибридизации B. carinata и B. rapa получены мейотически стабильные аллогексаплоиды (28-31). Из-за мейотических аберраций в результате неконтролируемого спаривания между тремя геномами долгое время не удавалось создать стабильный аллогексаплоид с ожидаемым числом хромосом (32). Синтетические полиплоиды используются в скрещиваниях как источники новых агрономических признаков.
Понимание взаимосвязей между геномами A, B и C имеет важное практическое значение. Степень колинеарности между геномами B. nigra и широко культивируемыми B. oleracea и B. rapa может влиять на возможность переноса признаков из B. nigra и родственных представителей родов Sinapis и Raphanus в важные культуры Brassica . Знание гомологичных участков геномов повысит возможность переноса генетических детерминант признаков посредством гомеологичной рекомбинации в ресинтезированные гибриды и селекции форм с желаемыми гибридными хромосомами с помощью технологии генетических маркеров.
С развитием современных технологий селекции, позволяющих осуществлять отдаленную гибридизацию, появилась необходимость введения системы эффективного контроля селекционного процесса на основе методов генетического анализа. Наряду с цитогенетическими методами — флуоресцентной гибридизацией in situ (fluorescence in situ hybridization, FISH) (31) и геномной гибридизацией in situ (genomic in situ hybridization, GISH) (32, 33) для исследования генетических взаимосвязей между видами Brassica , оценки эволюционных изменений геномов и контроля наследования хромосом при создании дигеномных и тригеномных гибридов широко используются ДНК маркеры: RFLP (restriction fragment length polymorphism), RAPD (random amplified polymorphic DNA), AFLP (amplified fragment length polymorphism) (34), SSR (simple sequence repeats) (35-37) и SNP (single nucleotide polymorphism) (38).
Эффективным инструментом для исследования интрогрессии генетического материала у межвидовых гибридов служат микросателлитные маркеры. Они равномерно распределены в геноме растений, кодоминантно наследуются и высокополиморфны, что позволяет получать информацию о гомозиготном или гетерозиготном состоянии локусов. Поскольку геномы родственных видов Brassica высококолинеарны, для них характерна гомология фланкирующих последовательностей микросателлитных локусов. Благодаря консервативной природе фланкирующих последовательностей, локусы, выявленные у одного вида, в большинстве случаев могут быть использованы для исследования родственных видов.
Микросателлитные локусы растений рода Brassica разработаны неза- висимо несколькими исследовательскими коллективами (39-41). Для многих из таких локусов установлены группы сцепления и расположение на генетической карте. В ряде работ показана перспективность использования микросателлитных локусов для исследования межвидового и внутривидового разнообразия Brassica (42, 43), оценки отличимости, однородности и стабильности сортов (44, 45), а также в качестве маркеров генов устойчивости к болезням, например к киле (46, 47) и сосудистому бактериозу (48).
В настоящей работе впервые выявлены эффективные геном-специ-фичные микросателлитные маркеры A-, B- и C-геномов Brassica , длина которых определена в системе высокоразрешающего электрофореза с точностью до одного нуклеотида, а также исследована возможность применения этих маркеров для анализа селекционных образцов, полученных в результате межвидовой гибридизации.
Нашей целью была разработка эффективной технологии контроля интрогрессии геномов A, B и C у представителей рода Brassica при отдаленной гибридизации на основе мультиплексного микросателлитного ПЦР-анализа геном-специфичных микросателлитных маркеров.
Методика. В качестве контрольных образцов использовали растения, полученные из Центра генетических ресурсов CGN (Centre for Genetic Resources, Нидерланды) и Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР, г. Санкт-Петербург). Растительный материал для исследования интрогрессии геномного материала был предоставлен сотрудниками ООО Селекционная станция им. Н.Н. Тимофеева (г. Москва).
Геномную ДНК получали методом адсорбции на сорбенте в соответствии с инструкцией к набору Фитосорб (ООО НПФ «Синтол», Россия). В исследованиях использовали растительный материал 5 растений каждого наименования. Микрообъемы растворов дозировали автоматически (дозаторы фирмы «Ленпипет», Россия). Осаждение проводили на микроцентрифуге Centrifuge 5415D («Eppendorf», Германия). Лизис растительных образцов осуществляли в термостате Термит (ООО НПО «ДНК-Технология», Россия). Для перемешивания и осаждения ДНК-образцов использовали мини-центрифугу-вортекс Microspin FV-2400 («SIA Biosan», Латвия).
Растительную ДНК амплифицировали методом ПЦР с праймерами, специфичными для локусов Na10-D09, Na12-A02, Na12-F12, Ni2-B02, Ni2-F02, Ni3-G04B, Ol12-A04, Ra2-E12 (39), BRMS-043 (40), BN6A2 (41). ПЦР проводили в реакционной смеси объемом 25 мкл следующего состава: 67 мМ Tris-HCl, pH 8,8; 16,6 мM (NH 4 ) 2 SO 4 ; 2,5 мМ MgCl 2 ; 5 ед/мкл Тaq-ДНК-полимеразы (ООО НПО «ДНК-Технология», Россия), 25 мМ dNTP (ООО «Медиген», Россия), 5-20 пмоль каждого праймера в зависимости от степени флуоресценции (ООО НПФ «Синтол», Россия), 10 нг ДНК. Амплификацию осуществляли в термоциклере CFX-96 («Bio-Rad», США) по программе: 5 мин при 95 ° C; 30 с при 94 ° C, 30 с при 48 ° C, 30 с при 72 ° C (30 циклов); 5 мин при 72 ° C. Наличие продуктов амплификации подтверждали в 2 % агарозном геле с окрашиванием бромистым этидием.
Анализ флуоресцентно меченных ПЦР-фрагментов осуществляли методом капиллярного электрофореза в денатурирующих условиях в генетическом анализаторе Нанофор-05 (ООО НПФ «Синтол»—ФГБНУ Институт аналитического приборостроения РАН, Россия) согласно инструкции к прибору (ЦКП «Биотехнология» ФГБНУ Всероссийский НИИ сельскохозяйственной биотехнологии). Для проведения фрагментного анализа 1 мкл ПЦР-продукта смешивали с 1 мкл маркера молекулярной массы S-450 (ООО НПФ «Синтол», Россия) и 8 мкл формамида Super DI («MCLab»,
США), затем денатурировали 5 мин при 95 ° С.
Длину ПЦР-фрагментов устанавливали с помощью программного обеспечения ДНК Фрагментный анализ (ФГБНУ ИАП РАН, Россия).
Результаты. Ранее при изучении полиморфизма микросателлитных локусов в 8 % полиакриламидном геле мы отобрали наиболее полиморфные локусы, позволяющие различать растения видов B. rapa , B. nigra , B. oleracea , B. napus , B. juncea и B. carinata (49-51). Поскольку большинство этих локусов имеют консервативные фланкирующие последовательности в геномах A, B и C, их амплификация наблюдается у всех шести видов Brassica , что позволяет провести сравнительный анализ и выявить геном-специ-фичные маркеры.
На основании полученных данных для исследования были выбраны 10 микросателлитных локусов. Полиморфизм длин микросателлитных фрагментов изучали у контрольных образцов шести видов: B rapa (AA), B. nigra (BB), B. oleracea (CC), B. napus (AACC), B. juncea (AABB), B. carinata (BBCC), для которых известны видовая принадлежность и геномный состав (табл. 1).
1. Представители шести видов рода Brassica L. и межвидовые гибриды, у которых был изучен полиморфизм длин микросателлитных фрагментов
Обозначение Описание
Контрольные образцы
1-02 (ВИР), Урал (ВИР), ВикРос (ВИР) CGN06832, CGN06818, 114 (ВИР), 107 (ВИР) CGN06619, CGN006634, CGN02656
CGN03950, CGN03952
CGN15778, CGN06998, CGN07004, CGN07022 CGN04588, CGN04594, CGN015193
B. napus L. (2 n = 38, геном AACC)
B. rapa L. (2 n = 20, геном AA)
B. nigra L. (2 n = 16, геном BB)
B. carinata A. Braun (2 n = 34, геном BBCC)
B. oleracea L. (2 n = 18, геном CC)
B. juncea (L.) Czern. (2 n = 36, геном AABB)
¹ 63, ¹ 69, ¹ 70, ¹ 97, ¹ 98, ¹ 106
Селекционные образцы
(Эт2 ½ КК)2 ½ Цв9
Цв9
КК
(Эт2 ½ КК)1
Грин ½ FBLM(1)
FBLM
JR
JR ½ Агр2ки
ПР3
ЗМ тетр
БК ½ ЗМ1-1(6), БК ½ ЗМ1-1(8)
B. carinata 1
БК
B. carinata 2
КБ
Растения-регенеранты, полученные в культуре изолированных микроспор от растений ВС 2 при межвидовой гибридизации B. oleracea и B. carinata :
{[( B. oleracea ½ B. carinata ) ½ B. oleracea ] ½ B. oleracea } Потомство ВС 1 от межвидовой гибридизации
( B. oleracea ½ B. rapa ) ½ B. oleracea
Инбредная линия капусты белокочанной B. oleracea
Инбредная линия капусты пекинской B. rapa Межвидовой гибрид F 1 ( B. oleracea ½ B. rapa ) Межвидовой гибрид F 1 ( B. oleracea ½ B. juncea ) Инбредная линия горчицы сарептской B. juncea Инбредная линия репы B. rapa
Межвидовой гибрид F 1 ( B. rapa ½ B. oleracea ) Инбредная линия капусты белокочанной B. oleracea Тетраплоидная линия капусты белокочанной B. oleracea Растения ВС 1 от межвидовой гибридизации B. oleracea и B. carinata : [( B. oleracea ½ B. carinata ) ½ B. oleracea )]
Инбредная линия горчицы эфиопской B. carinata Межвидовой гибрид F 1 ( B. oleracea ½ B. carinata ) Инбредная линия горчицы эфиопской B. carinata Межвидовой гибрид F 1 ( B. carinata ½ B. oleracea )
П р и м еч а ни е. CGN — Centre for Genetic Resources (Нидерланды), ВИР — Всероссийский института генетических ресурсов растений им. Н.И. Вавилова (г. Санкт-Петербург). Селекционные образцы выведены на ООО Селекционная станция им. Н.Н. Тимофеева (г. Москва).
Фрагменты определенной длины, выявленные у растений вида B. rapa (AA) и отсутствующие у контрольных образцов B. nigra (BB) и B. oleracea (CC), были приняты за маркеры присутствия материала генома A (А-геном-специфичные). Фрагменты, выявленные у B. nigra (BB) и отсутствующие у B. rapa (AA) и B. oleracea (CC), стали маркерами генома B (B-геном-специфичные). Фрагменты, выявленные у B. oleracea (CC) и отсутствующие у B. rapa (AA) и B. nigra (BB), были приняты за маркеры генома C (C-геном-специфичные).
В результате ПЦР-анализа локуса Na12-A02 у растений видов B. rapa
(AA), B. nigra (BB), B. oleracea (CC) были выявлены А-, В-, С-геном-спе-цифичные фрагменты. Также установлено кодоминантное сочетание соответствующих фрагментов у амфидиплоидных видов B. napus (AACC), B. juncea (AABB), B. carinata (BBCC). Локус BRMS-043 амплифицировался только у растений с геномом A — B. rapa (AA), B. juncea (AABB), B. napus (AACC), что позволяет отличить их от растений с В- и С-геномами. В локусе Na10-D09 были обнаружены А-геном-специфичные фрагменты. ПЦР-фрагменты синтезировались у представителей всех шести видов. Однако только фрагменты, выявленные у растений B. rapa (AA), значительно отличались по длине от фрагментов у растений B. nigra (BB), B. oleracea (CC). В локусах Ol12-A04 и BN6A2 установили В-геном-специфичные фрагменты. В результате анализа локуса Ni2-F02 выявили фрагменты, специфичные для В- и С-геномов. В локусах NI2-B02, RA2-E12, NA12-F12 обнаружили сходные по длине фрагменты у контрольных образцов B. rapa , B. nigra и B. oleracea . То есть эти локусы оказались непригодными для изучения интрогрессии A-, B- и C-геномов и были исключены из исследования.
2. Структура мультиплексной системы, разработанной на основе микросателлит-ных локусов для анализа интрогрессии геномов A, B и C видов рода Brassica L.
|
Локус |
Геном-специфичность |
Краситель |
Длина, п.н. |
|
Na12-A02 |
ABC |
FAM |
164-216 |
|
BRMS-043 |
A |
FAM |
303-313 |
|
Na10-D09 |
A |
R6G |
155-299 |
|
Ol12-A04 |
B |
ROX |
125-153 |
|
Ni2-F02 |
ВC |
ROX |
164-200 |
|
BN6A2 |
B |
Sy630 |
108-222 |
В результате изучения контрольных образцов отобрали шесть мик-росателлитных локусов: Na12-A02, BRMS-043, Na10-D09, Ol12-A04, Ni2-F02 и BN6A2. На их основе мы разработали систему мультиплексного ПЦР-анализа, позволяющую оценивать образцы по всем локусам одновременно (табл. 2). Для амплификации микросателлитных локусов в одной ПЦР-пробе была подобрана единая оптимальная температура отжига нескольких пар праймеров (Т отж = 48 ° C). В этой мультиплексной системе амплификация каждого микросателлитного локуса осуществлялась с парой специфичных праймеров, один из которых был помечен определенным флуоресцентным красителем (FAM, R6G, ROX и Sy630), что позволяло анализировать получаемые по каждому локусу ПЦР-фрагменты отдельно по соответствующему каналу детекции (см. табл. 2). Локусы были подобраны по длине таким образом, чтобы при анализе по одному каналу диапазоны длин их фрагментов не перекрывались. Длина полученных ПЦР-фрагментов определялась с точностью до одного нуклеотида в результате разделения электрофорезом высокого разрешения в капиллярах на автоматическом генетическом анализаторе Нанофор-05, с использованием технологии флуоресцентной детекции.
С помощью разработанной мультиплексной системы мы получили оцифрованные генетические профили образцов Brassica, на которых каждый пик соответствовал ПЦР-фрагменту определенной длины (рис.). В результате микросателлитного анализа контрольных растений были установлены фрагменты, специфичные для геномов А, В и С. Так, в генетическом профиле контрольного образца CGN06832 (B. rapa) присутствовали фрагменты, характерные для генома А, — 180 п.н. (локус Na12-A02), 303 п.н. (локус BRMS-043) и 293 п.н. (локус Na10-D09) (см. рис., А). В профиле образца CGN006634 (B. nigra) были выявлены отличительные маркеры генома В: 198 п.н. и 211 п.н. (локус Na12-A02), 125 п.н. и 129 п.н. (локус OL12-A04), 200 п.н. (локус NI2-F02), 222 п.н. (локус BN6A2) (см. рис., Б). У образца CGN15778 (B. oleracea) обнаружили фрагменты, специфичные для генома С, — 170 п.н. (локус Na12-A02) и 186 п.н. (локус Ni2-F02) (см. рис., В). В генетических профилях аллотетраплоидных видов наблюдалось кодоминантное сочетание фрагментов геномов соответствующих диплоидных видов (см. рис., Г-Е).
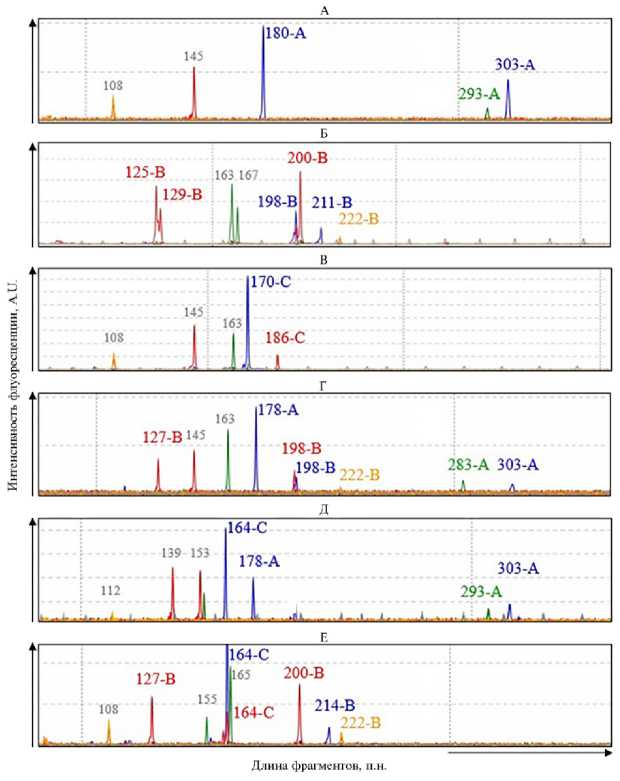
Генетические профили контрольных образцов растений шести видов рода Brassica L., полученные в результате мультиплексного ПЦР-анализа микросателлитных локусов: А — CGN06832 ( В. rapa , геном АА), Б — CGN006634 ( B. nigra , ВВ), В — CGN15778 ( B. oleracea , CC), Г — CGN015193 ( B. juncea , ААВВ), Д — ВИР ВикРос ( B. napus , ААСС), Е — CGN03950 ( B. carinata , ВВСС). Цвет пика (фрагмента) соответствует каналу детекции на приборе Нанофор-05 (ООО НПФ «Синтол»—ФГБНУ Институт аналитического приборостроения РАН, Россия) и определяет принадлежность фрагмента к определенному локусу: Na12-A02 и BRMS-043 — FAM (синий), NA10-D09 — R6G (зеленый), Ol12-A04 и Ni2-F02 — ROX (красный), BN6A2 — SY630 (оранжевый). Серым цветом обозначены недискриминирующие фрагменты, которые амплифицируются одновременно у двух или трех геномов. Описание образцов см. в таблице 1.
На основе полученных генетических характеристик определили состав аллелей каждого микросателлитного локуса, среди которых были выявлены маркеры, специфичные для геномов A, B и C (табл. 3).
-
3. А-, В- и С-геном-специфичные маркеры, выявленные у представителей шести видов рода Brassica L. с использованием мультиплексной системы ПЦР-анализа микросателлитных локусов
-
4. Геном-специфичные маркеры (п.н.) , выявленные у линий и межвидовых гибридов рода Brassica L. с использованием разработанной системы мультиплексного ПЦР-анализа микросателлитных локусов
|
Локус |
Геном |
Длина выявленных микросателлитных фрагментов, п.н. |
||
|
A-специфичные |
B-специфичные |
C-специфичные |
||
|
Na12-A02 |
ABC |
178, 180, 182 |
196, 198, 200, 202, 204, 212, 214, 216 |
164,168,170 |
|
BRMS-043 |
A |
303, 307, 313 |
||
|
Na10-D09 |
A |
283, 285, 287, 291, 293, 299 |
||
|
Ol12-A04 |
B |
125, 127, 129 |
||
|
Ni2-F02 |
BC |
198, 200, 202, 204, 208 |
164, 166, 168, 186 |
|
|
BN6A2 |
B |
222 |
||
П р и м е ч а н и е. Прочерки означают отсутствие в локусе соответствующего геном-специфичного маркера.
|
Образец |
Локус |
Геном |
|||||
|
Na12-A02 |
1BRMS-043 Na10-D09 1 Ol12-A04т |
Ni2-F02 |
1 BN6A2 |
||||
|
КК |
180А |
303А |
293А |
145 |
108 |
AA |
|
|
JR |
180А |
311А |
285А 341А |
143 |
114 |
AA |
|
|
¹ 63 |
164С |
155 |
166С |
108 |
CC |
||
|
¹ 69 |
164С |
155 |
166С |
108 |
CC |
||
|
¹ 70 |
164С |
155 |
166С |
108 |
CC |
||
|
¹ 97 |
164С |
155 |
166С |
108 |
CC |
||
|
¹ 98 |
164С |
155 |
166С |
108 |
CC |
||
|
¹ 106 |
164С |
155 |
166С |
108 |
CC |
||
|
Цв9 |
164С |
155 |
166С |
108 |
CC |
||
|
ПР3 |
164С |
166С 186С |
108 |
CC |
|||
|
ЗМ тетр |
164С |
166С 186С |
108 |
CC |
|||
|
БК ½ ЗМ1-1(6) |
164С 168С |
167 |
108 |
CC |
|||
|
БК ½ ЗМ1-1(8) |
164С 168С |
155 |
166С |
108 |
CC |
||
|
(Эт2 ½ КК)2 ½ Цв9 |
170С 180А |
303А |
155 293А |
145 |
166С |
108 |
AACC |
|
(Эт2 ½ КК)1 |
170С 180А |
303А |
293 А |
145 |
166С |
108 |
AACC |
|
JR ½ Агр2ки |
164С 180А |
311А |
287А |
145 |
166С |
108 114 |
AACC |
|
FBLM |
178А 198В |
313А |
163 285А |
127В 145 |
198В |
222 В |
AABB |
|
Грин ½ FBLM(1) |
164С 178А 198В |
313А |
163 287А |
127В 145 |
166С 198В |
108 222В |
AABBCC |
|
B. carinata 1 |
16 |
155 167 |
127В |
164С 200В |
108 |
BBCC |
|
|
БК |
164С 214В |
155 167 |
127В |
166С 200В |
108 222В |
BBCC |
|
|
B. carinata 2 |
164С 214В |
155 167 |
127В |
166С 200В |
108 222В |
BBCC |
|
|
КБ |
164С 168С 214В |
155 167 |
127В |
166С 200В |
108 222В |
BBCC |
|
П р и м е ч а н и е. Верхние индексы (А, В, С) указывают геномную специфичность маркерного фрагмента. Прочерки означают отсутствие у образца соответствующего геном-специфичного маркера по указанному локусу. Описание образцов см. в таблице 1.
Разработанную систему ПЦР-анализа использовали для выявления А-, В- и С-геном-специфичных фрагментов у селекционных образцов (см. табл. 1). В результате получили их генетические профили и обнаружили ге-ном-специфичные фрагменты, определяющие геномную принадлежность образцов (табл. 4). Следовательно, используемая мультиплексная система позволяет достоверно выявлять интрогрессию фрагментов геномов A, B и
C у видов рода Brassica . Кодоминантное сочетание фрагментов геномов в генетических профилях селекционных образцов (Эт2 ½ КК)2 ½ Цв9, полученных в результате гибридизации ( B. oleracea ½ B. rapa ) ½ B. oleracea , (Эт2 ½ КК)1 (F 1 B. oleracea ½ B. rapa ), Грин ½ FBLM (1) (F 1 B. oleracea ½ B. jun-cea ), JR ½ Агр2ки (F 1 B. rapa ½ B. oleracea ), БК (F 1 B. oleracea ½ B. carinata ) и КБ (F 1 B. carinata ½ B. oleracea ) свидетельствовало об интрогрессии генетического материала в результате межвидовой гибридизации. Данные, полученные в результате микросателлитного анализа, соответствовали селекционной истории этих образцов.
Система мультиплексного микросателлитного анализа на основе шести микросателлитных локусов, специфичных для геномов A, B и C у представителей рода Brassica , позволяет надежно различить растения шести видов треугольника U, а также получить информацию о генетическом разнообразии, поскольку каждый геномный маркер характеризуется несколькими аллельными вариантами. Каждый геном определяется минимум двумя маркерами, которые служат друг для друга внутренними контролями.
При использовании мультиплексной системы можно проследить интрогрессию определенных участков генома, поскольку для входящих в ее состав локусов были установлены группы сцепления . Например, показано, что локус BRMS-043 связан с устойчивостью к сосудистому бактериозу B. rapa (48).
Эффективность применения ДНК маркеров для генотипирования новых форм и контроля передачи геномного материала при их создании продемонстрирована в ряде работ. С использованием 162 комбинаций A-, B- и C-геном-специфичных SSR-праймеров был проведен анализ потенциальных генетических изменений у 25 синтезированных аллогексаплоидов (H1 B. rapa ½ B. carinata , AABBCC, 2 n = 54) (31). Для оценки генетической изменчивости новой формы B. napus , полученной в результате скрещивания B. rapa с гексаплоидом ( B. napus ½ B. oleracea , AACCCC), использовали 153 комбинации праймеров для микросателлитным локусам (32). С помощью 34 SSR-маркеров была обнаружена гомеологическая рекомбинация хромосом у потомства тригеномных гибридов B. napus ½ B. carinata (ABCC), полученного из микроспор (37).
Для идентификации видов Brassica и оценки генетического разнообразия разработан массив SNP маркеров для аллотетраплоида B. napus — Illumina Infinium B. napus 60K SNP («Illumina, Inc.», США) (52). Также для быстрой идентификации видов Brassica, входящих в треугольник N. U, предложен мультиплексный ПЦР-анализ (MPCR) специфичных последовательностей геномов A, B и C с использованием пяти комбинаций праймеров (35). Достоверность результатов MPCR была проверена на 120 генетически охарактеризованных образцах Brassica . Благодаря прямой детекции специфичных фрагментов в 2 % агарозном геле MPCR анализ полезен как доступный и быстрый диагностический подход, который может быть легко реализован в обычной лаборатории. Однако, как отмечают сами авторы, этот метод имеет достаточно низкую пропускную способность, и его следует адаптировать для высокопроизводительного скрининга в режиме реального времени.
В отличие от дорогостоящих и более сложных в исполнении методов, требующих специальной обработки результатов (например, при применении ДНК-чипа Illumina Infinium B. napus 60K SNP), предлагаемая нами технология доступнее и удобнее в использовании. Электрофоретический анализ осуществляется в автоматическом режиме на высокоточном оборудовании. Это значительно повышает надежность интерпретации полученных данных (в отличие от эмпирической оценки в геле без точного определения длин анализируемых фрагментов ДНК) (35, 45, 48). Благодаря автоматизации всех этапов анализа в формате 96-луночного планшета предложенный подход позволяет осуществлять широкомасштабный скрининг селекционных образцов. Рассматриваемая мультиплексная система может применяться для оценки интрогрессии геномов Brassica при межвидовой гибридизации и контроля наследования геномного материала в последующих поколениях.
Таким образом, нами разработана система мультиплексного ПЦР-анализа шести микросателлитных локусов Brassica (Na12-A02, BRMS-043, Na10-D09, Ol12-A04, Ni2-F02, BN6A2) для детекции фрагментов геномов А, В и С. Достоверность системы подтверждена на контрольных образцах с известным геномным составом и видовой принадлежностью. С использованием разработанных маркеров геномов А, В и С выявлены геном-специ-фичные фрагменты и определен геномный состав ряда селекционных образцов. Результаты исследования могут применяться для обнаружения последовательностей, специфичных для геномов А, В, С, у представителей рода Brassica и контроля наследования генетического материала при отдаленной гибридизации.
Список литературы Мультиплексная система микросателлитного анализа для исследования интрогрессии фрагментов a-, b-, c-геномов у видов рода Brassica L. при отдаленной гибридизации
- Warwick S.I., Francis A., Al-Shehbaz I.A. Brassicaceae: Species checklist and database on CD-ROM. Plant Systematics and Evolution, 2006, 259(2-4): 249-258 ( ). DOI: 10.1007/s00606-006-0422-0
- Morinaga T. Interspecific hybridization in Brassica. IV. The cytology of F1 hybrid of B. juncea and B. nigra. Cytologia, 1934, 6: 62-67 ( ). DOI: 10.1508/cytologia.6.62
- U N. Genome analysis in Brassica with special reference to the experimental formation of B. napus and peculiar mode of fertilization. Japanese Journal of Botany, 1935, 7: 389-452.
- Lagercrantz U. Comparative mapping between Arabidopsis thaliana and Brassica nigra indicates that Brassica genomes have evolved through extensive genome replication accompanied by chromosome fusions and frequent rearrangements. Genetics, 1998, 150(3): 1217-1228.
- Kowalski S.P., Lan T.H., Feldmann K.A., Paterson A.H. Comparative mapping of Arabidopsis thaliana and Brassica oleracea chromosomes reveals islands of conserved organization. Genetics, 1994, 138(2): 499-510.
- Rana D., van den Boogaart T., O'Neill C.M., Hynes L., Bent E., Macpherson L., Park J.Y., Lim Y.P., Bancroft I. Conservation of the microstructure of genome segments in Brassica napus and its diploid relatives. Plant Journal, 2004, 40(5): 725-733 ( ).
- DOI: 10.1111/j.1365-313X.2004.02244.x
- Lagercrantz U., Lydiate D. Comparative genome mapping in Brassica. Genetics, 1996, 144(4): 1903-1910.
- Zou J., Hu D., Liu P., Raman H., Liu Z., Liu X., Parkin I.A.P., Chalhoub B., Meng J. Co-linearity and divergence of the A subgenome of Brassica juncea compared with other Brassica species carrying different A subgenomes. BMC Genomics, 2016, 17: 18 ( ).
- DOI: 10.1186/s12864-015-2343-1
- Jiang C., Ramchiary N., Ma Y., Jin M., Feng J., Li R., Wang H., Long Y., Choi S.R., Zhang C., Cowling W.A., Park B.S., Lim Y.P., Meng J. Structural and functional comparative mapping between the Brassica A genomes in allotetraploid Brassica napus and diploid Brassica rapa. Theoretical and Applied Genetics, 2011, 123(4): 927-941 ( ).
- DOI: 10.1007/s00122-011-1637-1
- Mason A.S., Batley J. Creating new interspecific hybrid and polyploid crops. Trends in Biotechnology, 2015, 33(8): 436-441 ( ).
- DOI: 10.1016/j.tibtech.2015.06.004
- Snowdon R.J. Cytogenetics and genome analysis in Brassica crops. Chromosome Research, 2007, 15(1): 85-95 ( ).
- DOI: 10.1007/s10577-006-1105-y
- Katche E., Quezada-Martinez D., Katche E.I., Vasquez-Teuber P., Mason A.S. Interspecific hybridization for Brassica crop improvement. Crop Breeding, Genetics and Genomics, 2019, 1: e190007 ( ).
- DOI: 10.20900/cbgg20190007
- Siemens J. Interspecific hybridisation between wild relatives and Brassica napus to introduce new resistance traits into the oilseed rape gene pool. Czech Journal of Genetics and Plant Breeding, 2002, 38(3-4): 155-157 ( ).
- DOI: 10.17221/6258-CJGPB
- Chrungu B., Verma N., Mohanty A., Pradhan A., Shivanna K.R. Production and characterization of interspecific hybrids between Brassica maurorum and crop Brassicas. Theoretical and Applied Genetics, 1999, 98(3-4): 608-613 ( ).
- DOI: 10.1007/s001220051111
- Lelivelt C.L.C., Krens F.A. Transfer of resistance to the beet cyst nematode (Heterodera schachtii Schm.) into the Brassica napus L. gene pool through intergeneric somatic hybridization with Raphanus sativus L. Theoretical and Applied Genetics, 1992, 83(6-7): 887-894 ( ).
- DOI: 10.1007/BF00226712
- Lelivelt C.L.C., Leunissen E.H.M., Frederiks H.J., Helsper J.P.F.G., Krens F.A. Transfer of resistance to the beet cyst nematode (Heterodera schachtii Schm.) from Sinapis alba L. (white mustard) to the Brassica napus L. gene pool by means of sexual and somatic hybridization. Theoretical and Applied Genetics, 1993, 85(6-7): 688-696 ( ).
- DOI: 10.1007/BF00225006
- Snowdon R.J., Winter H., Diestel A., Sacristán M.D. Development and characterisation of Brassica napus - Sinapis arvensis addition lines exhibiting resistance to Leptosphaeria maculans. Theoretical and Applied Genetics, 2000, 101(7): 1008-1014 ( ).
- DOI: 10.1007/s001220051574
- Yamagishi H., Bhat S.R. Cytoplasmic male sterility in Brassicaceae crops. Breeding Science, 2014, 64(1): 38-47 ( ).
- DOI: 10.1270/jsbbs.64.38
- Piao Z., Ramchiary N., Lim Y.P. Genetics of clubroot resistance in Brassica species. Journal of Plant Growth Regulation, 2009, 28(3): 252-264 ( ).
- DOI: 10.1007/s00344-009-9093-8
- Sharma B.B., Kalia P., Singh D., Sharma T.R. Introgression of black rot resistance from Brassica carinata to cauliflower (Brassica oleracea botrytis group) through embryo rescue. Frontiers in Plant Science, 2017, 8: 1255 ( ).
- DOI: 10.3389/fpls.2017.01255
- Belimov A.A., Safronova V.I., Demchinskaya S.V., Dzyuba O.O. Intraspecific variability of cadmium tolerance in hydroponically grown Indian mustard (Brassica juncea (L.) Czern.) seedlings. Acta Physiologiae Plantarum, 2007, 29(5): 473-478 ( ).
- DOI: 10.1007/s11738-007-0057-y
- Plieske J., Struss D., Röbbelen G. Inheritance of resistance derived from the B-genome of Brassica against Phoma lingam in rapeseed and the development of molecular markers. Theoretical and Applied Genetics, 1998, 97: 929-936 ( ).
- DOI: 10.1007/s001220050973
- Navabi Z.K., Parkin I.A.P., Pires J.C., Xiong Z., Thiagarajah M.R., Good A.G., Rahman M.H. Introgression of B-genome chromosomes in a doubled haploid population of Brassica napus ½ B. carinata. genome, 2010, 53(8): 619-629 ( ).
- DOI: 10.1139/g10-039
- Abel S., Möllers C., Becker H. Development of synthetic Brassica napus lines for the analysis of "fixed heterosis" in allopolyploid plants. Euphytica, 2005, 46(1-2): 157-163 ( ).
- DOI: 10.1007/s10681-005-3364-7
- Srivastava A., Mukhopadhyay A., Arumugam N., Gupta V., Verma J.K., Pental D., Pradhan A.K. Resynthesis of Brassica juncea through interspecific crosses between B. rapa and B. nigra. Plant Breeding, 2004, 123(2): 204-206 ( ).
- DOI: 10.1046/j.1439-0523.2003.00933.x
- Narasimhulu S.B., Kirti P.B., Prakash S., Chopra V.L. Resynthesis of Brassica carinata by protoplast fusion and recovery of a novel cytoplasmic hybrid. Plant Cell Reports, 1992, 11(8): 428-432 ( ).
- DOI: 10.1007/BF00234376
- Chen S., Nelson M.N., Chèvre A.M., Jenczewski E., Li Z., Mason A.S. Trigenomic bridges for Brassica improvement. Critical Reviews in Plant Sciences, 2011, 30(6): 524-547 ( ).
- DOI: 10.1080/07352689.2011.615700
- Монахос Г.Ф., Игнатов А.Н., Джалилов Ф.С. Синтез аллогексаплоида с геномной формулой ААВВСС рода Brassica L. как донора устойчивости к киле и сосудистому бактериозу крестоцветных. Известия ТСХА, 2001, 4: 56-68.
- Malek M.A., Rahman L., Das M.L., Hassan L., Rafii M.Y., Ismail M.R. Development of hexaploid Brassica (AABBCC) from hybrids (ABC) of Brassica carinata (BBCC) ½ B. rapa (AA). Ausralian Journal of Crop Science, 2013, 7(9): 1375-1382.
- Zhou J., Tan C., Cui C., Ge X., Li Z. Distinct subgenome stabilities in synthesized Brassica allohexaploids. Theoretical and Applied Genetics, 2016, 129(7): 1257-1271 ( ).
- DOI: 10.1007/s00122-016-2701-7
- Gupta M., Atri C., Agarwal N., Banga S.S. Development and molecular-genetic characterization of a stable Brassica allohexaploid. Theoretical and Applied Genetics, 2016, 129(11): 2085-2100 ( ).
- DOI: 10.1007/s00122-016-2759-2
- Li Q., Mei J., Zhang Y., Li J., Ge X., Li Z., Qian W. A large-scale introgression of genomic components of Brassica rapa into B. napus by the bridge of hexaploid derived from hybridization between B. napus and B. oleracea. Theoretical and Applied Genetics, 2013, 126(8): 2073-2080 ( ).
- DOI: 10.1007/s00122-013-2119-4
- Ma N., Li Z.-Y., Cartagena J. A., Fukui K. GISH and AFLP analyses of novel Brassica Napus lines derived from one hybrid between B. napus and Orychophragmus Violaceus. Plant Cell Reports, 2006, 25(10): 1089-1093 ( ).
- DOI: 10.1007/s00299-006-0171-0
- Монахос С.Г. Интеграция современных биотехнологических методов в селекции овощных культур. Докт. дис. М., 2015.
- Koh J.C.O., Barbulescu D.M., Norton S., Redden B., Salisbury P.A., Kaur S., Cogan N., Slater A.T. A multiplex PCR for rapid identification of Brassica species in the triangle of U. Plant Methods, 2017, 13(1): 49 ( ).
- DOI: 10.1186/s13007-017-0200-8
- Thakur A.K., Singh K.H., Singh L., Nanjundan J., Khan Y.J., Singh D. SSR marker variations in Brassica species provide insight into the origin and evolution of Brassica amphidiploids. Hereditas, 2017, 155: 6 ( ).
- DOI: 10.1186/s41065-017-0041-5
- Mason A.S., Nelson M.N., Castello M.C., Yan G., Cowling W.A. Genotypic effects on the frequency of homoeologous and homologous recombination in Brassica napus ½ B. carinata hybrids. Theoretical and Applied Genetics, 2011, 122(3): 543-553 ( ).
- DOI: 10.1007/s00122-010-1468-5
- Clarke W.E., Higgins E.E., Plieske J. et al. A high-density SNP genotyping array for Brassica napus and its ancestral diploid species based on optimised selection of single-locus markers in the allotetraploid genome. Theoretical and Applied Genetics, 2016, 129(10): 1887-1899 ( ).
- DOI: 10.1007/s00122-016-2746-7
- Lowe A.J., Moule C., Trick M., Edwards K.J. Efficient large-scale development of microsatellites for marker and mapping applications in Brassica crop species. Theoretical and Applied Genetics, 2004, 108(6): 1103-1112 ( ).
- DOI: 10.1007/s00122-003-1522-7
- Suwabe K., Iketani H., Nunome T., Kage T., Hirai M. Isolation and characterization of microsatellites in Brassica rapa L. Theoretical and Applied Genetics, 2002, 104(6-7): 1092-1098 ( ).
- DOI: 10.1007/s00122-002-0875-7
- Kresovich S., Szewc-McFadden A.K., Bliek S.M., McFerson J.R. Abundance and characterisation of simple-sequence repeats (SSRs) isolated from a size-fractionated genomic library of Brassica napus L. (rapeseed). Theoretical and Applied Genetics, 1995, 91(2): 206-211 ( ).
- DOI: 10.1007/BF00220879
- Plieske J., Struss D. Microsatellite markers for genome analysis in Brassica. I. Development in Brassica napus and abundance in Brassicaceae species. Theoretical and Applied Genetics, 2001, 102(5): 689-694 ( ).
- DOI: 10.1007/s001220051698
- Hasan M., Seyis F., Badani A.G., Pons-Kühnemann J., Friedt W., Lühs W., Snowdon R.J. Analysis of genetic diversity in the Brassica napus L. gene pool using SSR markers. Genetic Resources and Crop Evolution, 2006, 53(4): 793-802 ( ).
- DOI: 10.1007/s10722-004-5541-2
- Tommasini L., Batley J., Arnold G.M., Cooke R.J., Donini P., Lee D., Law J.R., Lowe C., Moule C., Trick M., Edwards K.J. The development of multiplex simple sequence repeat (SSR) markers to complement distinctness, uniformity and stability testing of rape (Brassica napus L.) varieties. Theoretical and Applied Genetics, 2003, 106(6): 1091-1101 ( ).
- DOI: 10.1007/s00122-002-1125-8
- Дубина Е.В., Королева С.В., Гаркуша С.В., Юрченко С.А., Есаулова Л.В. Разработка методической схемы оценки гибридности семян F1 Brаssiса olеrасеа L., основанной на полиморфизме микросателлитных ДНК-маркеров. Достижения науки и техники ΑΠΚ, 2016, 30(8): 49-51.
- Suwabe K., Tsukada H., Iketani H., Hatakeyama K., Fujimura M., Nunome T., Fukuoka H., Matsumoto S., Hirai M. Identification of two loci for resistance to clubroot (Plasmodiophora brassicae Wornin) in Brassica rapa L. Theoretical and Applied Genetics, 2003, 107(6): 997-1002 ( ).
- DOI: 10.1007/s00122-003-1309-x
- Kato T., Hatakeyama K., Fukino N., Matsumoto S. Fine mapping of the clubroot resistance gene CRb and development of a useful selectable marker in Brassica rapa. Breeding Science, 2013, 63(1): 116-124 ( ).
- DOI: 10.1270/jsbbs.63.116
- Артемьева А.М., Игнатов А.Н., Волкова А.И., Кочерина Н.В., Коноплева М.Н., Чесноков Ю.В. Физиолого-генетические компоненты устойчивости к сосудистому бактериозу у линий удвоенных гаплоидов Brassica rapa L. Сельскохозяйственная биология, 2018, 53(1): 157-169 ( ).
- DOI: 10.15389/agrobiology.2018.1.157rus
- Шилов И.А. Применение технологии микросателлитного анализа ДНК в растениеводстве. В сб.: Проблемы агробиотехнологии. М., 2012: 140-162.
- Анискина Ю.В. Технология генотипирования культурных и дикорастущих форм Brassica на основе анализа полиморфизма микросателлитов. Автореф. канд. дис. М., 2006.
- Анискина Ю.В., Велишаева Н.С., Шилов И.А., Хавкин Э.Е. Генотипирование пасленовых и крестоцветных растений методом микросателлитного анализа. Метод. реком. М., 2005.
- Mason A.S., Zhang J., Tollenaere R., Vasquez Teuber P., Dalton-Morgan J., Hu L., Edwards D., Redden R., Batley J. High-throughput genotyping for species identification and diversity assessment in germplasm collections. Molecular Ecology Resources, 2015, 15(5): 1091-1101 ( ).
- DOI: 10.1111/1755-0998.12379


