Некоторые аспекты построения генетических карт
Автор: Чесноков Ю.В.
Журнал: Овощи России @vegetables
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 2 (40), 2018 года.
Бесплатный доступ
Построение генетических карт имеет принципиальное значение при практическом использовании установленных ассоциаций «маркер-признак». Последовательное развитие генетических исследований привело кустановлению групп сцепления и локализации генов, а цитогенетическое изучение дало возможность идентифицировать группы сцепления с определенными хромосомами у целого ряда организмов. Широкое использование молекулярных ДНК-маркеров, а также применение методологии идентификации и локализации на хромосомах QTL (quantitative trait loci) даже в тех случаях, когда они не способствовали появлению новых областей науки, значительно расширили границы биологических исследований, в том числе, увеличили точность и масштабируемость в построении генетических карт сцепления. На сегодня исследование сцепления генов - важнейший подход, используемый как для кэр™рования генома, так и в генетико-селекционной деятельности. Насыщение генетических карт дает возможность компилировать на карте большое число сегрегирующих локусов одной популяции практически до полного «насыщения» генома. В то же время, эффекты воздействия «генотип-среда» могут вызывать вариабельность, которая является основной причиной того, почему различные популяции некоторых видов не обязательно проявляют идентичную длину карт, а разница при этом может достигать 20%. Гены, локусы или даже целые районы хромосом, картированные у организма одного таксона, с достаточно высокой долей достоверности могут быть оценены и идентифицированы с помощью консервативных молекулярных маркеров, картированных у другого организма, как правило, из родственного таксона. Кроме того, если проведены тщательные межвидовые сравнения, то предсказания по расположению и картированию генов или QTL (quantitative trait loci), сделанные для одного вида, могут быть применены и к другому родственному ему виду.
Генетическая карта, длина карты, сравнительное картирование, организация генома
Короткий адрес: https://sciup.org/140223762
IDR: 140223762 | УДК: 575.116.4
Текст научной статьи Некоторые аспекты построения генетических карт
Г енетическая карта сцепления – одномерная схема взаиморасположения локусов хромосом, генов и/или генетических маркеров в группах сцепления данного организма с указанием расстояний между ними в парах нуклеотидов, определенных с помощью секвенирования, или в сантиморганидах, установленных по частоте кроссинговера [1].
Генетические карты сцепления правильно отражают порядок расположения генов и/или генетических маркеров на хромосомах, однако полученные при этом значения расстояний между ними не соответствуют реальным физическим расстояниям. Обычно данный факт связывают с тем, что эффективность рекомбинации между хроматидами на отдельных участках хромосом может сильно различаться. В частности, она подавлена в гетерохроматиновых участках хромосом [2]. С другой стороны, в хромосомах часто встречаются «горячие точки» рекомбинации. Использование частот рекомбинации для построения физических генетических карт без учета этих факторов будет приводить к искажениям реальных расстояний между генами и/или генетическими маркерами.
Таким образом, генетические карты сцепления являются наименее точными из всех имеющихся типов генетических карт, и их можно рассматривать только в качестве первого приближения к реальным физическим картам. Тем не менее, на практике именно они позволяют локализовать на группах сцепления детерминанты, определяющие сложные хозяйственно ценные признаки, и дают возможность их дальнейшего изучения. Необходимо помнить, что в отсутствие кроссинго-вера все гены, находящиеся на индивидуальной хромосоме, передавались бы от родителей потомству вместе, поскольку они физически сцеплены друг с другом. Поэтому индивидуальные хромосомы образуют группы сцепления генов, и одной из первых задач построения генетических карт сцепления является отнесение исследуемого гена или определенной последовательности нуклеотидов (маркеров) к конкретной группе сцепления.
Для установления принадлежности гена к той или иной группе сцепления необходимо произвести скрещивания, на основании которых определяется характер наследования этого гена по отношению к другим генам, принадлежность которых к той или иной группе сцепления определена ранее. Например, чтобы у кукурузы определить принадлежность гена к одной из десяти групп сцепления, необходимо растения с изучаемым геном скрестить поочередно с десятью растениями, каждое из которых несет рецессивные гены, принадлежащие к разным группам сцепления. Ввиду того, что изучаемый ген может принадлежать только, к одной из десяти групп сцепления, с генами девяти остальных групп он будет проявлять независимое наследование и расщепление в F2 9:3:3:1.
На сегодняшний день исследование сцепления генов – важнейший подход, используемый как для картирования генома, так и в генетико-селекционной деятельности. Последовательное развитие генетических исследований привело к установлению групп сцепления и локализации генов, а цитогенетическое изучение дало возможность идентифицировать группы сцепления с определенными хромосомами у целого ряда организмов. Существенный вклад в исследование сцепления генов внесло открытие молекулярных маркеров [3]. Так начатое в 80-х – 90-х годах прошлого столетия широкое использование молекулярных ДНК-маркеров, а также применение методологии идентификации и локализации на хромосомах QTL (quantitative trait loci) даже в тех случаях, когда они не способствовали появлению новых областей науки, значительно расширили границы биологических исследований, в том числе, увеличили точность и масштабируемость в построении генетических карт сцепления. Именно благодаря им удалось достичь так называемого насыщения генетических карт, что дает возможность компилировать на карте большое число сегрегирующих локусов одной популяции, причем практически до полного «насыщения» генома, т.е. до тех пор, пока каждая точка генома не будет генетически связана (сцеплена), как минимум с одним маркером. На таких картах количество групп сцепления равно числу хромосом.
Насыщение карт
Как указывалось выше, насыщение генетической карты наступает тогда, когда каждая точка на карте хромосом генетически сцеплена как минимум с одним маркером. Каким же образом можно достигнуть этой цели? В случае если карта не насыщена, в ней, как правило, существует много небольших групп сцепления и несцеп-ленных маркеров. Увеличение числа маркеров обычно приводит к сцеплению независимых групп. Насыщение маркерами имеет смысл проводить до
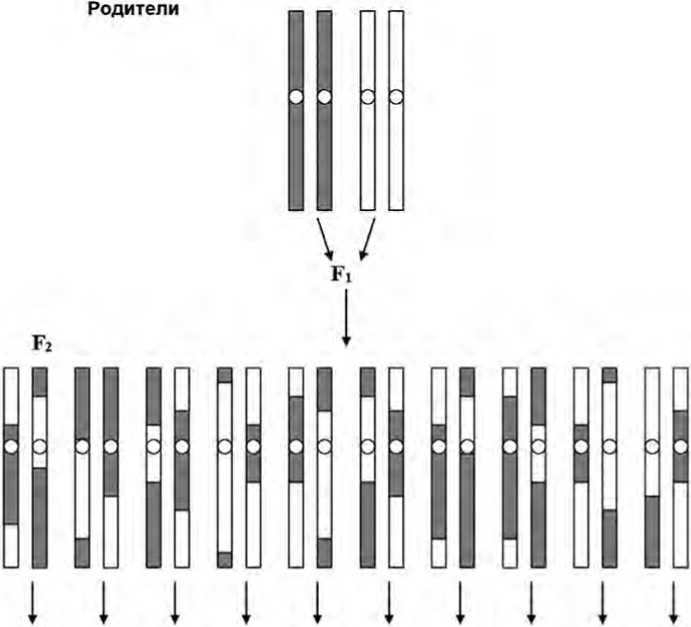
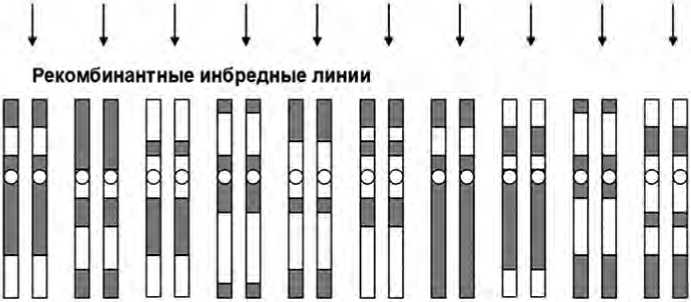
Рисунок. Схема получения рекомбинантных инбредных линий. Каждую особь F2, как и последующие поколения, самоопыляют, отбирая только по одной особи в качестве родителя очередного поколения. Каждая пара хромосом представляет геномную структуру семьи рекомбинантных инбредных линий полученных от поколения особей F2. Белые и затемненые районы обозначают соответствующие «вклады» родительских линий. Несмотря на преемственность поколений, геном, как бы «порван» на различные фрагменты, поскольку в каждом мейозе число кроссоверных обменов ограничено (по [8]).
Fig. Scheme of obtaining of recombinant inbred lines. Eash individual F2 as well as the next generations self-pollinate separate only one individual as a parent the next generation. Each chromosome pair are the genome structure of recombinant inbred lines family obtained from F2 individual generation. White and blackened regions mean respective «contribution» parent lines. In spite of generation genome as if “tear” on different fragments, as far as in each meiosis the number of crossovers are limited (base on [8])
тех пор, пока групп сцепления не станет столько же сколько хромосом у вида, и при этом не останется несцеп-ленных маркеров. Поскольку маркеры выбирают случайным образом, то иногда для заполнения всех брешей на карте необходимо тестированием перепробовать большое число маркеров. Более того, некоторые районы генома могут оказаться бедны марке- рами, поскольку они имеют высокий уровень рекомбинации. Для таких «неподдающихся» районов лучше использовать совокупный сегрегационный анализ (bulk segregant analysis, BSA) и маркеры, фланкирующие этот район, нежели случайным образом заполнять геном большим числом маркеров [3].
Но нахождение всех групп сцепле- ния, соответствующих числу хромосом, не гарантирует полного насыщения, которое может быть продемонстрировано двумя способами. Непрямой метод состоит в накоплении маркеров. Когда генетическая длина карты больше не увеличивается можно предположить, что насыщение достигнуто. Однако существует проблема, заключающаяся в том, что при увеличении числа маркеров, увеличивается число ошибок генотипирования, что в свою очередь приводит к ошибке в вычислении длины карты. Контроль ошибок должен быть одним из приоритетов в генетическом картировании (программное обеспечение, такое, например, как Mapmaker 3.0, имеет функцию поиска ошибок, основанную на определении рекомбинационных событий, которые «слишком» близки). Прямой метод, в отличие от непрямого, заключается в использовании маркерных проб или праймеров специфичных к субтеломерным районам, расположенным на концах хромосом, и которые, как правило, состоят из повторяющихся последовательностей. Если такие маркеры генетически сцеплены с рядом дистальных маркеров хромосом, то это значит, что карта насыщена. Такой подход был продемонстрирован для нескольких хромосом кукурузы [4]. Если подобного рода пробы недоступны, в скорейшем нахождении маркеров, прилежащих к наиболее дистальным маркерам может помочь метод BSA. И, наконец, если карта еще не насыщена, исходя из числа расположенных на ней маркеров и типа карти- рующей популяции, для достижения полной генетической длины, могут быть использованы теоретические методологии [5, 6]. Иллюстрацию такого подхода можно найти в [7].
В таблице приведены генетические длины геномов наиболее значимых видов растений, для которых были построены достаточно плотно насыщенные карты. Значения указанных длин варьируют от 630 сМ для генома арабидопсиса и до 3500 сМ для генома пшеницы. В отношении гаплоидного числа хромосом показано, что средняя генетическая длина хромосомы составляет примерно 100 сМ (1 морганиду). Это означает, что в каждом мейозе в среднем существует один кроссоверный обмен на хромосому с учетом больших флуктуаций по хромосомам и в соответствии с видом растений. Таким образом, мейоз может быть представлен как процесс, который разрывает геном на большие фрагменты. Рисунок иллюстрирует пример, когда даже в рекомбинантных инбредных линиях достаточно грубо «перетасовываются» два родительских генома, при этом целостность длинных участков сохраняется.
Исходя из значений таблицы, порядок увеличения числа маркеров, которые должны быть картированы, может быть подсчитан как функция степени желаемого насыщения. Для генома в 1000 сМ, 50 маркеров, расположенных на расстоянии 20 сМ друг от друга, в принципе, достаточно. На практике необходимо картировать больше маркеров, поскольку по геному они располагаются друг относи- тельно друга неравномерно. Одна из первых базовых карт кукурузы, опубликованная в 1993 году, содержала всего 97 маркеров с максимальной дистанцией между ними 30 сМ и покрывала практически все 1860 сМ генома данного вида растения [9]. Безусловно, на сегодняшний день, в геноме кукурузы картировано гораздо больше маркеров, и, кроме того, на картах они организованы в так называемые «бины» (Bins (англ.) – в данном случае интервалы между двумя фиксированными реперными маркерными локусами).
Относительность длины карты
Генетические дистанции не абсолютны. Как известно, уровни рекомбинации могут изменяться под воздействием различных факторов.
Прежде всего, на уровни рекомбинации влияет пол, хотя общих правил в данном случае нет. При сравнении карт, полученных при реципрокных скрещиваниях у томата [10], показано, что для этой культуры рекомбинация выше у растений, которые использовали в качестве материнской формы (карта у них на 20% длиннее). Такая же тенденция отмечалась у ячменя [11], но у кукурузы и арабидопсиса [12] наблюдался обратный эффект. Это происходит потому, что различия между полами локально высоко вариабельны.
Эволюционные генетические дистанции между родителями также играют роль. Ранние исследования показывают, что популяции, полученные в результате межвидовых скре-
Таблица. Взаимоотношения между физическими и генетическими дистанциями у некоторых видов растений, для которых построены насыщенные генетические карты групп сцепления
Table 1. Interaction between physical and genetic distances in some plant species for which constructed saturated genetic maps of linkage groups
|
Виды |
Общая длина генома* (п.о. х 109) |
Общая генетическая длина генома (сМ) |
Длина ДНК/сМ (т.п.о.) |
Число хромосом |
|
Arabidopsis |
0,15 |
630 |
140 |
5 |
|
Рис |
0,43 |
1575 |
280 |
12 |
|
Соя |
1,2 |
2700 |
440 |
20 |
|
Томат |
0,95 |
1267 |
750 |
12 |
|
Бобы |
0,65 |
830 |
780 |
11 |
|
Рапс |
1,2 |
1016 |
1200 |
19 |
|
Кукуруза |
2,5 |
1860 |
1400 |
10 |
|
Пшеница |
16 |
3500 |
4600 |
21 |
|
Сосна |
24 |
1800 |
13000 |
12 |
* - по [21].
щиваний, приводят к получению таких межлокусных генетических дистанций, которые меньше тех, что получают при внутривидовых скрещиваниях [13]. Карты молекулярных маркеров достаточно ясно показали и подтвердили это наблюдение. Так, у картофеля генетическая карта, построенная на основе межвидового скрещивания, имела размер на 35% меньше, чем карта, построенная при проведении внутривидового скрещивания [14, 15].
Подобного же рода наблюдения были сделаны для томата [16] и риса [17]. Объяснение этому явлению заключается в том, что уменьшение идентичности между гомологичными нитями ДНК в межвидовом скрещивании приводит к уменьшению частоты кроссоверных обменов [18].
И, наконец, существует вариабельность уровней рекомбинации, которая контролируется генетически. Вместе с возможными эффектами воздействия окружающей среды, такая вариабельность, несомненно, является основной причиной того, почему различные популяции некоторых видов не обязательно проявляют идентичную длину карт, а разница при этом может достигать 20% [19, 20].
Отношения между генетическими и физическими дистанциями
Низкая вариация длины генетических карт у различных видов контрастирует со значительными изменениями количества ДНК на клетку. У покрытосеменных количество ДНК, по крайней мере, для видов, изученных по этим параметрам, варьирует в 660 раз (при сравнении крайних значений). Так, например, арабидопсис ( Brassicaceae ) содержит всего лишь 0,3 пикограмм ДНК на диплоидную клетку (или на 2С), в то время как растение рябчик ( Liliaceae ) – 255 пг/2С [22]. В среднем значения 2С у покрытосеменных растений варьирует между 1 и 10 пг. У голосеменных средние значения содержания ДНК на диплоидную клетку преимущественно высоки и варьируют, как правило, между 10 и 60 пг/2С. Поскольку число экспрессируемых генов у высших растений довольно велико, то (благодаря высоко достоверному варьированию количества нетранскрибируемых геномных повторов) и выявленные различия в количестве ДНК у различных видов растений также значительны. Если оставить в стороне идущие до сих пор дебаты о предполагаемой адаптивной значимости такого варьирования, то важным остается тот факт, что подобного рода варьирование не отражается на общей генетической длине геномов. Другими словами, ясно, что частота кроссоверных обменов на единицу физической длины уменьшается, если размер геномов увеличивается. Это может быть подтверждено обменами между хромосомами отдельно взятого вида: у дрожжей наименьшая хромосома имеет в среднем в два раза больше рекомбинаций на одну т.п.о., чем наибольшая [23].
Такие результаты согласуются с наблюдениями, указывающими на то, что районы гетерохроматина, содержащие высоко повторяющиеся последовательности, соответствуют зонам с низкой рекомбинацией [24]. И, наконец, исследования, также проведенные на дрожжах, показали соответствие между рекомбинацией и зонами транскрипции [25].
Среднее число пар оснований (п.о.) на сантиморганиду зависит от рассматриваемого вида. Таблица показывает, что от арабидопсиса до сосны эти значения варьируют в сотни раз, от 140 т.п.о. до 13000 т.п.о. Подобного рода информация особенно важна для позиционного клонирования. Например, для того, чтобы найти маркер на расстоянии 1 сМ от нужного гена у видов с маленьким размером генома, вполне достаточен метод «прогулки по хромосоме» или метод «посадки на хромосому», но такой подход вряд ли подойдет для кукурузы или пшеницы. Однако, это заключение нельзя назвать безусловным, поскольку величины чисел п.о./сМ усреднены для всего генома, а внутри генома могут существовать большие локальные вариации уровней рекомбинации. Так, например, было установлено [26], что одна сантиморганида по соседству от локуса bronze кукурузы соответствует 14 т.п.о., что в 100 раз меньше, чем средняя величина целого генома. И, наоборот, по соседству с центромерным районом, где есть ограничения рекомбинации, число т.п.о. на сантиморганиду может быть значительно выше, чем на весь оставшийся геном. Так у арабидопсиса при сравнении относительных дистанций физической (YAC) и генетической (RFLP) карт хромосомы 4, показано, что между ними локально могут наблюдаться достоверные различия [27].
Анализ организации генома
При анализе геномов таких видов, как кукуруза, рапс или рожь, значительный процент использованных в исследовании проб выявил более чем один локус. Для данных растений определенный порядок расположения маркеров на одной группе сцепления (или какой-то ее части) соответствовал порядку расположения тех же маркеров на другой группе сцепления, а проведенное картирование отобранных зондов помогло выявить гомологии внутри генома. Подобного рода гомологии могут быть интерпретированы в том смысле, что геномы рассматриваемых видов прошли частичную или полную дупликацию в процессе эволюции [28-30]. Этот феномен наблюдается и у видов с малым размером геномов, таких, например, как рис [31] и арабидопсис [32, 33].
Сравнительное картирование
Идея сравнения генетических карт различных видов живых организмов отнюдь не нова, особенно в отношении видов животных. Первые сравне- ния сцеплений между геном альбинизма и геном розовой окраски глаз у крысы и мыши проведены в 1920 году (цит. по: [34]). С тех пор осуществлен целый ряд сравнительных анализов отдельных генов, которые, судя по картам млекопитающих, являются гомологичными. В дополнение к этому установлено [35], что несколько групп сцепления консервативны у представителей практически всего отряда позвоночных – от рыб к человеку.
Возможность использования гетерологичных проб, при построении генетических карт у растений, позволило провести многочисленные сравнения порядка расположения генов у видов, принадлежащих к одному ботаническому семейству. Так, например, у Solanaceae показано [14, 36], что геномы томата и картофеля практически полностью коллинеарны, за исключением пяти парацентромер-ных инверсий. Это очень хороший пример эволюционной консервативности синтении . (О синтении можно говорить тогда, когда у организмов, относящихся к разным таксонам, локусы сцепленые, либо несцепленые генетически, схожим образом расположены в идентичных группах сцепления. При асинтении похожие генетические локусы расположены в разных хромосомах или группах сцепления.) У томата и перца, которые филогенетически не так близки, перестроения геномов более многочисленны (около 20), но локально порядок генов остается консервативным [37-39]. А у Fabaceae сравнение карт чечевицы и гороха выявило 8 высоко консервативных районов, которые составляют 40% генома [40]. Что же говорить о представителях Poaceae , которые всегда являлись объектом более интенсивного изучения. Так, в трибе Triticae порядок маркеров довольно консервативен у пшеницы, ячменя и ржи [41-43]. И это несмотря на то, что геном ржи кажется весьма отличным от такового у пшеницы за счет существования многих транслокаций. В трибе Andropogonae схожая консервативность групп сцепления и близкий порядок расположения маркеров отмечались у кукурузы и сорго [44, 45]. К этой группе, благодаря своей плоидности, по-видимому, принадлежит и геном сахарного тростника [46]. Группы сцепления, найденные у сорго и сахарного тростника, имеют два геномных района, гомологичных таковым у кукурузы. Можно предположить, что в данном случае, эти районы явились результатом единой эволюционной дупликации. Однако, у кукурузы, по-видимому, произошло больше эволюционных перестроек, нежели у сорго и сахарного тростника, поскольку эти три вида, в конце концов, дивергировали. Также возможно проведение сравнения между трибами внутри Poaceae . В этой связи, установлено, что рис, пшеница и кукуруза содержат несколько коллинеарных геномных районов [47-49].
С фундаментальной точки зрения, полученные и примененные к ряду видов результаты по сравнительному картированию, могут быть использованы для анализа путей эволюции структуры геномов семейства Poaceae, что является наглядным примером реализации на практике закона гомологических рядов в наследственной изменчивости, сформулированного Н.И. Вавиловым еще в 1920 году [50]. Другими словами, гены, локусы или даже целые районы хромосом, картированные у организма одного таксона, с достаточно высокой долей достоверности могут быть оценены и идентифицированы с помощью консервативных молекулярных маркеров, картированных у другого организма, как правило, из родственного таксона. Кроме того, если проведены тщательные межвидовые сравнения, то предсказания по расположению и картированию генов или QTL, сделанные для одного вида, могут быть применены и к другому родственному ему виду.
Даже если учесть, что пробы, наиболее часто используемые в экспериментах, в основном анонимны, идея, заключающаяся в том, что позиционная гомология генов ассоциирована с функциональной гомологией, получила подтверждение. Примером тому могут служить классические случаи картирования мутаций, детерминирующих потерю лигулы, у кукурузы и риса [47], или основного гена, определяющего устойчивость к заморозкам и необходимости яровизации, найденного у ржи, пшеницы и ячменя [51]. Сравнительное картирование является важным и зачастую основным методическим подходом для научных программ, в которых проводятся работы по интегрированному изучению аналогичных линий различных видов растений. Эта точка зрения была поддержана рядом ученых [52, 53], которые предположили, что злаки представляют собой «единую генетическую систему» и активно пропагандировали свою идею «единого генетического начала». Однако, независимо от этого и в дополнение к вышесказанному, следует отметить, что при сравнительном картировании:
-
• Литература
-
1. Чесноков Ю.В. Картирование локусов количественных признаков у растений. СПб., ВИР. 2009. – 100 с.
-
2. Жученко А.А., Король А.Б. Рекомбинация в эволюции и селекции. М.: Наука. 1985. – 400 с.
-
3. Чесноков Ю.В. Молекулярно-генетические маркеры и их использование в предселекцион-ных исследованиях. СПб: АФИ. 2013. – 116 с.
-
4. Burr B., Burr F.A., Matx E.C., Romero-Severson J. Pinning down loose ends: mapping telomeres and factors affecting their length // The Plant Cell. – 1992. – Vol. 4. – P. 953-960
-
5. Chakravarti A., Lasher L.A., Reefer J.E. A maximum likelihood method for estimating genome length using linkage data // Genetics. – 1991. – Vol. 128. – P. 175-182
-
6. Hulbert S.H., Hott T.W., Legg E.J., Lincoln S.E., Lander E.S., Michelmore R.W. Genetic analysis of the fungus, Bremia lactucae, using restriction length polymorphism // Genetics. - 1988. – Vol. 120. - P. 947-958
-
7. Gerber S., Rodolphe F. An estimation of the genome length of maritime pine (Pinus pinaster Ait.) // Theor. Appl. Genet. - 1994b. - Vol.88. – P. 289-292
-
8. Burr B., Burr F.A. Recombinant inbreds for molecular mapping in maize // Trends Genet. - 1991. – Vol. 7. - P. 55-60
-
9. Gardiner J.M., Coe E.H., Melia-Hancock S., Hoisington D.A., Chao S. Development of a core RFLP map using an immortalized F2 population // Genetics. - 1993. - Vol. 134. – P. 917-930
-
10. De Vicente M.C., Tanksley S.D. Genome-wide reduction in recombination of backcross progeny derived from male versus female gametes in an interspecific cross of tomato // Theor. Appl. Genet. - 1991. - Vol. 83. – P. 173-178
-
11. Graner A., Jahorr A., Schondelmaier J., Siedler H., Pileen K., Fishbeck G., Wenzel G., Herrmann R.G. Construction of an RFLP map of barley // Theor. Appl. Genet. - 1991. – Vol. 83. – P. 250-256 12. Vizir I., Korol A.B. Sex difference in recombination frequency in Arabidopsis // Heredity. - 1990. – Vol. 65. – P. 379-383
-
-
13. Rick C.M. Controlled introgression of chromosomes of Solanum penneli into Lycopersicon escu-lentum: segregation and recombination // Genetics. - 1969. – Vol. 62. - P. 753-768
-
14. Bonierbale M.W., Plaisted R.L., Tanksley S.D. RFLP maps based on a common set of clones reveal modes of chromosomal evolution in potato and tomato // Genetics. - 1988. – Vol. 120. – P. 1095-1103
-
15. Gebhardt C., Ritter E., Barone A., Debener T., Walkemeier B., Schachtschabel U., Kaufmann H., Thompson R.D., Bonierbale M.W., Ganal M.W., Tanksley S.D., Salamini F. RFLP maps of potato and their alignment with the homologous tomato genome // Theor. Appl. Genet. - 1991. - Vol. 83. – P. 49-57
-
16. Paterson A.H., Lander E.S., Hewitt J.D., Peterson S., Loncoln S.E. Tanksley S.D. Resolution of quantitative traits into Mendelian factors by using a complete linkage map of restriction fragment length polymorphisms // Nature. - 1988. – Vol. 335. – P. 721-726
-
17. Causse M.A., Fulton T.M., Cho Y.G., Ahn S.N., Chunwongse J., Wu K., Xiao J., Yu Z., Ronald P.C., Herrington S.E., Second G., McCouch S.R., Tanksley S.D. Saturated molecular map of the rice genome based on an interspecific backcross population // Genetics. - 1994. – Vol. 138. – P. 12511274
-
18. Borts R.H., Haber J.E. Meiotic recombination in yeast: alteration by multiple heterozygosities // Science. - 1987. – Vol. 237. - P. 1459-1465
-
19. Beavis W.D., Grant D. A linkage map based on information from F2 populations of maize (Zea mays L.) // Theor. Appl. Genet. - 1991. – Vol. 82. – P. 636-644
-
20. Burr B., Burr F.A., Thompson K.H., Albertsen M.C., Stuber C.W. Gene mapping with recombinant inbreeds in maize // Genetics. - 1988. – Vol. 118. – P. 519-526
-
21. Arumuganathan K., Earle E.D. Nuclear DNA content of some important plant species // Plant Mol. Biol. Rep. - 1991. – Vol. 9. P. 208-218
-
22. Bennett M.D., Smith J.B. Nuclear DNA amount in angiosperms // Proc. R. Soc. Lond. B. - 1991. – Vol. 334. – P. 309-345
-
23. Kaback D.B., Guacci V., Barber D., Mahon J.W. Chromosome size dependent control of meiot-ic recombination // Science. - 1992. – Vol. 256. - P. 228-232
-
24. Flavell R.B. Chromosome architecture: the distribution of recombination sites, the structure of ribosomal DNA loci and the multiplicity of sequences containing inverted repeats. In: Molecular Form and Function of the Plant Genome. Van Vloten-Dtin L., Groot G.S.P., Hall I.C. (eds.). NATO ASI, 83. Plenum Press. New York. - 1985. - P. 1-14
-
25. Oliver S.G., Van der Aart Q.J., Agostoni-Garbone M.L., Aigle M., Alberghina L., Alexandraki D., Antoine G. et al. The complete DNA sequence of yeast chromosome III // Nature. - 1992. – Vol. 357. – P. 38-46
-
26. Donner H.K. Genetic fine structure of the bronze locus in maze // Genetics. - 1986. – Vol. 113. P. 1021-1036
-
27. Schmidt R., West J., Love K., Lenehan Z., Lister C., Thompson H., Bouchez D., Dean C. Physical map and organization of Arabidopsis thaliana chromosome 4 // Science. - 1995. – Vol. 270. – P. 480-483
-
28. Rocher J.P., Prioul J.L., Lecharny A., Reyss A., Joussaume M. Genetic variability in carbon fixation, sucrose-P-synthase and ADP glucose pyrophosphorylase in maize plants of differing growth rate // Plant Physiol. - 1989. – Vol. 89. – P. 416-420
-
29. Slocum M.K., Figdore S.S., Kennard W.C., Suzuki J.Y., Osborn T.C. Linkage arrangement of restriction fragment length polymorphism loci in Brassica oleracea // Theor. Appl. Genet. - 1990. – Vol. 80. – P. 57-64
-
30. Song K.M., Suzuki J.Y., Slocum M.K., Williams P.H., Osborn T.C. A linkage map of Brassica rapa (syn. campestris) based on restriction length polymorphism loci // Theor. Appl. Genet. - 1991. – Vol. 82. – P. 296-304
-
31. Kishimoto N., Higo H., Abe K., Arai S., Siato A., Higo G. Identification of the duplicated segments in rice chromosomes 1 and 5 by linkage analysis of cDNA markers of known functions // Theor. Appl. Genet. - 1994. – Vol. 88. – P. 722-726
-
32. Blanc G., Barakat A., Guyot R., Cooke R., Denseny M. Extensive duplication and reshuffling in the Arabidopsis genome // Plant Cell. - 2000. – Vol. 12. – P. 1093-1101
-
33. Grant D., Cregan P., Schoemaker R.C. Genome organization in dicots: genome duplication in Arabidopsis and synteny between soybean and Arabidopsis // Proc. Natl. Acad. Sci. USA. - 2000. – Vol. 97. – P. 4168-4173
-
34. Lyon M.F. Dunn and mouse genetic mapping // Genetics. - 1990. – Vol. 125. – P. 231-236
-
35. Morizot D.C. Use fish gene maps to predict ancestral vertebrate genome organization. In: Isozymes: Structure, Function and Use in Biology and medicine. Ogita Z.I., Marker C.L. (eds.). Liss, Wiley, New York. - 1990. - P. 207-234
-
36. Tanksley S.D., Ganal M.W., Prince J.P., De Vicente M.C., Bonierbale M.W., Broun P., Fulton T.M., Giovannoni J.J., Grandillo S., Martin G.B., Messeguer R., Miller L., Paterson A.H., Pinedo O., Roder M.S., Wing R.A., Wu W., Young N.D. High density molecular linkage maps of the tomato and potato genomes // Genetics. - 1992. – Vol. 132. – P. 1141-1160
-
37. Lefebvre V., Palloux A., Caranta C., Pochard E. Construction of an intraspecific integrated linkage map of pepper using molecular markers and double haploid progenies // Genome. - 1995. – Vol. 38. P. 112-121
-
38. Livingstone K.D., Lackney V.K., Blauth J.R., Van Wijk R., Jahn M.K. Genome mapping in Capsicum and the evolution of genome structure in the Solanaceae // Genetics. - 1999. – Vol. 152. – P. 1183-1202
-
39. Tanksley S.D., Bernatzky R., Lapitan N.L., Prince J.P. Conservation of gene repertoire but not gene order in paper and tomato // Proc. Natl. Acad. Sci. USA. - 1988. - Vol. 85. P. 6419-6423
-
40. Weeden N.F., Muehlbauer F.J., Ladizinsky G. Extensive conservation of linkage relationships between pea and lentil genetic maps // J. Hered. - 1992. – Vol. 83. – P. 123-129
-
41. Devos K.M., Atkinsin M.D., Chinoy C.N., Liu C.J., Gale M.D. RFLP-based genetic map of the homoeologous group 3 chromosomes of wheat and rye // Theor. Appl. Genet. - 1992. – Vol. 83. – P. 931-939
-
42. Sharp P.J., Kreis M., Shewry P.R., Gale M.D. Location of β -amilase sequences in wheat and its relatives // Theor. Appl. Genet. - 1988. – Vol. 75. – P. 286-290
-
43. Wang M.L., Atkinson M.D., Chinoy C.N., Devos K.M., Gale M.D. Comparative RFLP-based genetic maps of barley chromosome 5 1H and rye chromosome 1R // Theor. Appl. Genet. - 1992. – Vol. 84. – P. 339-344
-
44. Melake-Berhan A., Hulbert S.H., Bultmer L.G., Bennetzen J.L. Structure and evolution of the genomes of sorghum bicolor and Zea mays // Theor. Appl. Genet. - 1993. – Vol. 86. – P. 598-604
-
45. Whitkus R., Doebley J., Lee M. Comparative genome mapping of sorghum and maize // Genetics. - 1992. – Vol. 132. – P. 1119-1130
-
46. Grivet L., DґHont A., Dufour P., Hamon P., Roques D., Glaszmann J.C. Comparative genome mapping of sugar cane with other species within the Andropogoneae tribe // Heredity. - 1994. – Vol. 73. – P. 500-508
-
47. Ahn S., Anderson J.A., Sorrells M.E., Tanksley S.D. Homoeologous relationships of rice, wheat and maize chromosomes // Mol. Gen. Genet. - 1993. – Vol. 241. – P. 483-490
-
48. Ahn S., Tanksley S.D. Comparative linkage maps of the rice and maize genomes // Proc. Natl. Acad. Sci. USA. - 1993. – Vol. 90. – P. 7980-7984
-
49. Kurata N., Moore G., Nagamura Y., Foote T., Yano M., Minobe Y., Gale M. Conservation of genome structure between rice and wheat // BioTechnology. - 1994. – Vol. 12. – P. 276-278
-
50. Вавилов Н.И. Закон гомологических рядов в наследственной изменчивости. В: Теоретические основы селекции растений. М., Л. - 1935. - Т.1. - C.75-128
-
51. Plaschke J., Boerner A., Xie D.X., Koebner R.M.D., Schlegel R., Gale M.D. RFLP mapping of genes affecting plant height and growth habit in rye // Theor. Appl. Genet. - 1993. – Vol. 85. – P. 1049-1054
-
52. Bennetzen J.L., Freeling M. Grasses as a single genetic system: genome composition, collinearity and compatibility // Trends in Genet. - 1993. – Vol. 9. – P. 259-261
-
53. Freeling M. Grasses as a single genetic system: reassessment // Plant Physiol. - 2001. – Vol. 125. – P. 1191-1197
-
54. Asnaghi C., Paulet F., Kaye C., Grivet L., Deu M., Glaszmann J.C., DґHont A. Application of syn-teny across Poaceae to determine the map location of a sugar cane rust resistance gene // Theor. Appl. Genet. - 2000. - Vol. 101. – P. 962-969
-
55. Robert L.S., Robson F., Sharpe A., Lyndiate D., Coupland G. Conserved structure and function of the Arabidopsis flowering time gene CONSTANS in Brassica napus // Plant Mol. Biol. - 1998. – Vol. 37. – P. 763-772
-
• известные маркеры видов могут быть использованы для насыщения данного района у родственных видов. Этот метод может оказаться особенно полезным у видов с высоко плоид-ным уровнем, где картирование целого генома практически не осуществимо [54].
-
• ген гомологичный (ортологичный) по отношению к искомому гену в родственных модельных видах с малым геномом для которых существуют YAC и BAC библиотеки, может быть клонирован, а информация о его первичной нуклеотидной последовательности (сиквенс) использована для выделения гена из изучаемого вида [55].
Заключение
Таким образом, с появлением все большего и большего количества насыщенных генетическими маркерами молекулярных карт для наиболее распространенных видов растений, а также с доступностью таких карт для все более широкого круга исследователей и сфер их деятельности, генетики, а в перспективе и селекционеры, будут нуждаться только в скрининге родителей известных картированных популяций для установления вариаций в экспрессии представляющих интерес признаков, что позволит методом экстраполяции оценить соответствующую картированную популяцию по изучаемому признаку. Разумеется, что использование, напри- мер, QTL анализа для тестирования генов-кандидатов будет требовать наличия картированных генов в уже изученной популяции. Однако, техники и методологии картирования становятся все легче и быстрее, и нет принципиальных ограничений (пожалуй, только за исключением финансовых возможностей) для обеспечения исследователей современным лабораторным оборудованием, необходимым для проведения молекулярно-биологических работ с целью картирования геномов живых организмов.
-
• References
-
1. Chesnokov Yu.V. Quantitative trait loci mapping in plants. SPb.,: VIR. 2009. - 100 p.
-
2. Zhuchenko A.A., Korol A.B. Recombination in evolution and breeding. М.: Nauka. 1985. - 400 p.
-
3. Chesnokov Yu.V. Molecular and genetic markers and its using in prebreeding investigations. SPb: AFI. 2013. - 116 p.
-
4. Burr B., Burr F.A., Matx E.C., Romero-Severson J. Pinning down loose ends: mapping telomeres and factors affecting their length // The Plant Cell. - 1992. – Vol. 4. – P. 953-960
-
5. Chakravarti A., Lasher L.A., Reefer J.E. A maximum likelihood method for estimating genome length using linkage data // Genetics. - 1991. - Vol. 128. – P. 175-182
-
6. Hulbert S.H., Hott T.W., Legg E.J., Lincoln S.E., Lander E.S., Michelmore R.W. Genetic analysis of the fungus, Bremia lactucae, using restriction length polymorphism // Genetics. - 1988. – Vol. 120. - P. 947-958
-
7. Gerber S., Rodolphe F. An estimation of the genome length of maritime pine (Pinus pinaster Ait.) // Theor. Appl. Genet. - 1994b. - Vol.88. – P. 289-292
-
8. Burr B., Burr F.A. Recombinant inbreds for molecular mapping in maize // Trends Genet. -1991. – Vol. 7. - P. 55-60
-
9. Gardiner J.M., Coe E.H., Melia-Hancock S., Hoisington D.A., Chao S. Development of a core RFLP map using an immortalized F2 population // Genetics. - 1993. - Vol. 134. – P. 917-930
-
10. De Vicente M.C., Tanksley S.D. Genome-wide reduction in recombination of backcross progeny derived from male versus female gametes in an interspecific cross of tomato // Theor. Appl. Genet. - 1991. - Vol. 83. – P. 173-178
-
11. Graner A., Jahorr A., Schondelmaier J., Siedler H., Pileen K., Fishbeck G., Wenzel G., Herrmann R.G. Construction of an RFLP map of barley // Theor. Appl. Genet. - 1991. – Vol. 83. – P. 250-256
-
12. Vizir I., Korol A.B. Sex difference in recombination frequency in Arabidopsis // Heredity. - 1990. – Vol. 65. – P. 379-383
-
13. Rick C.M. Controlled introgression of chromosomes of Solanum penneli into Lycopersicon esculentum: segregation and recombination // Genetics. - 1969. – Vol. 62. - P. 753-768
-
14. Bonierbale M.W., Plaisted R.L., Tanksley S.D. RFLP maps based on a common set of clones reveal modes of chromosomal evolution in potato and tomato // Genetics. - 1988. – Vol. 120. – P. 1095-1103
-
15. Gebhardt C., Ritter E., Barone A., Debener T., Walkemeier B., Schachtschabel U., Kaufmann H., Thompson R.D., Bonierbale M.W., Ganal M.W., Tanksley S.D., Salamini F. RFLP maps of potato and their alignment with the homologous tomato genome // Theor. Appl. Genet. - 1991. - Vol. 83. – P. 49-57
-
16. Paterson A.H., Lander E.S., Hewitt J.D., Peterson S., Loncoln S.E. Tanksley S.D. Resolution of quantitative traits into Mendelian factors by using a complete linkage map of restriction fragment length polymorphisms // Nature. - 1988. – Vol. 335. – P. 721-726
-
17. Causse M.A., Fulton T.M., Cho Y.G., Ahn S.N., Chunwongse J., Wu K., Xiao J., Yu Z., Ronald P.C., Herrington S.E., Second G., McCouch S.R., Tanksley S.D. Saturated molecular map of the rice genome based on an interspecific backcross population // Genetics. - 1994. – Vol. 138. – P. 1251-1274
-
18. Borts R.H., Haber J.E. Meiotic recombination in yeast: alteration by multiple heterozygosities // Science. - 1987. – Vol. 237. - P. 1459-1465
-
19. Beavis W.D., Grant D. A linkage map based on information from F2 populations of maize (Zea mays L.) // Theor. Appl. Genet. - 1991. – Vol. 82. – P. 636-644
-
20. Burr B., Burr F.A., Thompson K.H., Albertsen M.C., Stuber C.W. Gene mapping with recombinant inbreeds in maize // Genetics. - 1988. – Vol. 118. – P. 519-526
-
21. Arumuganathan K., Earle E.D. Nuclear DNA content of some important plant species // Plant Mol. Biol. Rep. - 1991. – Vol. 9. P. 208-218
-
22. Bennett M.D., Smith J.B. Nuclear DNA amount in angiosperms // Proc. R. Soc. Lond. B. -1991. – Vol. 334. – P. 309-345
-
23. Kaback D.B., Guacci V., Barber D., Mahon J.W. Chromosome size dependent control of mei-otic recombination // Science. - 1992. – Vol. 256. - P. 228-232
-
24. Flavell R.B. Chromosome architecture: the distribution of recombination sites, the structure of ribosomal DNA loci and the multiplicity of sequences containing inverted repeats. In: Molecular Form and Function of the Plant Genome. Van Vloten-Dtin L., Groot G.S.P., Hall I.C. (eds.). NATO ASI, 83. Plenum Press. New York. - 1985. - P. 1-14
-
25. Oliver S.G., Van der Aart Q.J., Agostoni-Garbone M.L., Aigle M., Alberghina L., Alexandraki D., Antoine G. et al. The complete DNA sequence of yeast chromosome III // Nature. - 1992. – Vol. 357. – P. 38-46
-
26. Donner H.K. Genetic fine structure of the bronze locus in maze // Genetics. - 1986. – Vol. 113. P. 1021-1036
-
27. Schmidt R., West J., Love K., Lenehan Z., Lister C., Thompson H., Bouchez D., Dean C. Physical map and organization of Arabidopsis thaliana chromosome 4 // Science. - 1995. – Vol. 270. – P. 480-483
-
28. Rocher J.P., Prioul J.L., Lecharny A., Reyss A., Joussaume M. Genetic variability in carbon fixation, sucrose-P-synthase and ADP glucose pyrophosphorylase in maize plants of differing growth
rate // Plant Physiol. - 1989. – Vol. 89. – P. 416-420
-
-
29. Slocum M.K., Figdore S.S., Kennard W.C., Suzuki J.Y., Osborn T.C. Linkage arrangement of restriction fragment length polymorphism loci in Brassica oleracea // Theor. Appl. Genet. - 1990. – Vol. 80. – P. 57-64
-
30. Song K.M., Suzuki J.Y., Slocum M.K., Williams P.H., Osborn T.C. A linkage map of Brassica rapa (syn. campestris) based on restriction length polymorphism loci // Theor. Appl. Genet. - 1991. – Vol. 82. – P. 296-304
-
31. Kishimoto N., Higo H., Abe K., Arai S., Siato A., Higo G. Identification of the duplicated segments in rice chromosomes 1 and 5 by linkage analysis of cDNA markers of known functions // Theor. Appl. Genet. - 1994. – Vol. 88. – P. 722-726
-
32. Blanc G., Barakat A., Guyot R., Cooke R., Denseny M. Extensive duplication and reshuffling in the Arabidopsis genome // Plant Cell. - 2000. – Vol. 12. – P. 1093-1101
-
33. Grant D., Cregan P., Schoemaker R.C. Genome organization in dicots: genome duplication in Arabidopsis and synteny between soybean and Arabidopsis // Proc. Natl. Acad. Sci. USA. - 2000. – Vol. 97. – P. 4168-4173
-
34. Lyon M.F. Dunn and mouse genetic mapping // Genetics. - 1990. – Vol. 125. – P. 231-236
-
35. Morizot D.C. Use fish gene maps to predict ancestral vertebrate genome organization. In: Isozymes: Structure, Function and Use in Biology and medicine. Ogita Z.I., Marker C.L. (eds.). Liss, Wiley, New York. - 1990. - P. 207-234
-
36. Tanksley S.D., Ganal M.W., Prince J.P., De Vicente M.C., Bonierbale M.W., Broun P., Fulton T.M., Giovannoni J.J., Grandillo S., Martin G.B., Messeguer R., Miller L., Paterson A.H., Pinedo O., Roder M.S., Wing R.A., Wu W., Young N.D. High density molecular linkage maps of the tomato and potato genomes // Genetics. - 1992. – Vol. 132. – P. 1141-1160
-
37. Lefebvre V., Palloux A., Caranta C., Pochard E. Construction of an intraspecific integrated linkage map of pepper using molecular markers and double haploid progenies // Genome. - 1995. – Vol. 38. P. 112-121
-
38. Livingstone K.D., Lackney V.K., Blauth J.R., Van Wijk R., Jahn M.K. Genome mapping in Capsicum and the evolution of genome structure in the Solanaceae // Genetics. - 1999. – Vol. 152. – P. 1183-1202
-
39. Tanksley S.D., Bernatzky R., Lapitan N.L., Prince J.P. Conservation of gene repertoire but not gene order in paper and tomato // Proc. Natl. Acad. Sci. USA. - 1988. - Vol. 85. P. 6419-6423
-
40. Weeden N.F., Muehlbauer F.J., Ladizinsky G. Extensive conservation of linkage relationships between pea and lentil genetic maps // J. Hered. - 1992. – Vol. 83. – P. 123-129
-
41. Devos K.M., Atkinsin M.D., Chinoy C.N., Liu C.J., Gale M.D. RFLP-based genetic map of the homoeologous group 3 chromosomes of wheat and rye // Theor. Appl. Genet. - 1992. – Vol. 83. – P. 931-939
-
42. Sharp P.J., Kreis M., Shewry P.R., Gale M.D. Location of β -amilase sequences in wheat and its relatives // Theor. Appl. Genet. - 1988. – Vol. 75. – P. 286-290
-
43. Wang M.L., Atkinson M.D., Chinoy C.N., Devos K.M., Gale M.D. Comparative RFLP-based genetic maps of barley chromosome 5 1H and rye chromosome 1R // Theor. Appl. Genet. - 1992. – Vol. 84. – P. 339-344
-
44. Melake-Berhan A., Hulbert S.H., Bultmer L.G., Bennetzen J.L. Structure and evolution of the genomes of sorghum bicolor and Zea mays // Theor. Appl. Genet. - 1993. – Vol. 86. – P. 598-604
-
45. Whitkus R., Doebley J., Lee M. Comparative genome mapping of sorghum and maize // Genetics. - 1992. – Vol. 132. – P. 1119-1130
-
46. Grivet L., DґHont A., Dufour P., Hamon P., Roques D., Glaszmann J.C. Comparative genome mapping of sugar cane with other species within the Andropogoneae tribe // Heredity. - 1994. – Vol. 73. – P. 500-508
-
47. Ahn S., Anderson J.A., Sorrells M.E., Tanksley S.D. Homoeologous relationships of rice, wheat and maize chromosomes // Mol. Gen. Genet. - 1993. – Vol. 241. – P. 483-490
-
48. Ahn S., Tanksley S.D. Comparative linkage maps of the rice and maize genomes // Proc. Natl. Acad. Sci. USA. - 1993. – Vol. 90. – P. 7980-7984
-
49. Kurata N., Moore G., Nagamura Y., Foote T., Yano M., Minobe Y., Gale M. Conservation of genome structure between rice and wheat // BioTechnology. - 1994. – Vol. 12. – P. 276-278
-
50. Vavilov N.I. The law of homological rows in hereditable variation. In: Theoretical basis of plant breeding. М., L. - 1935. - V.1. - P.75-128
-
51. Plaschke J., Boerner A., Xie D.X., Koebner R.M.D., Schlegel R., Gale M.D. RFLP mapping of genes affecting plant height and growth habit in rye // Theor. Appl. Genet. - 1993. – Vol. 85. – P. 1049-1054
-
52. Bennetzen J.L., Freeling M. Grasses as a single genetic system: genome composition, collinearity and compatibility // Trends in Genet. - 1993. – Vol. 9. – P. 259-261
-
53. Freeling M. Grasses as a single genetic system: reassessment // Plant Physiol. - 2001. – Vol. 125. – P. 1191-1197
-
54. Asnaghi C., Paulet F., Kaye C., Grivet L., Deu M., Glaszmann J.C., DґHont A. Application of synteny across Poaceae to determine the map location of a sugar cane rust resistance gene // Theor. Appl. Genet. - 2000. - Vol. 101. – P. 962-969.
-
55. Robert L.S., Robson F., Sharpe A., Lyndiate D., Coupland G. Conserved structure and function of the Arabidopsis flowering time gene CONSTANS in Brassica napus // Plant Mol. Biol. -1998. – Vol. 37. – P. 763-772.
Список литературы Некоторые аспекты построения генетических карт
- Чесноков Ю.В. Картирование локусов количественных признаков у растений. СПб., ВИР. 2009. -100 с.
- Жученко А.А., Король А.Б. Рекомбинация в эволюции и селекции. М.: Наука. 1985. -400 с.
- Чесноков Ю.В. Молекулярно-генетические маркеры и их использование в предселекционных исследованиях. СПб: АФИ. 2013. -116 с.
- Burr B., Burr F.A., Matx E.C., Romero-Severson J. Pinning down loose ends: mapping telomeres and factors affecting their length//The Plant Cell. -1992. -Vol. 4. -P. 953-960
- Chakravarti A., Lasher L.A., Reefer J.E. A maximum likelihood method for estimating genome length using linkage data//Genetics. -1991. -Vol. 128. -P. 175-182
- Hulbert S.H., Hott T.W., Legg E.J., Lincoln S.E., Lander E.S., Michelmore R.W. Genetic analysis of the fungus, Bremia lactucae, using restriction length polymorphism//Genetics. -1988. -Vol. 120. -P. 947-958
- Gerber S., Rodolphe F. An estimation of the genome length of maritime pine (Pinus pinaster Ait.)//Theor. Appl. Genet. -1994b. -Vol.88. -P. 289-292
- Burr B., Burr F.A. Recombinant inbreds for molecular mapping in maize//Trends Genet. -1991. -Vol. 7. -P. 55-60
- Gardiner J.M., Coe E.H., Melia-Hancock S., Hoisington D.A., Chao S. Development of a core RFLP map using an immortalized F2 population//Genetics. -1993. -Vol. 134. -P. 917-930
- De Vicente M.C., Tanksley S.D. Genome-wide reduction in recombination of backcross progeny derived from male versus female gametes in an interspecific cross of tomato//Theor. Appl. Genet. -1991. -Vol. 83. -P. 173-178
- Graner A., Jahorr A., Schondelmaier J., Siedler H., Pileen K., Fishbeck G., Wenzel G., Herrmann R.G. Construction of an RFLP map of barley//Theor. Appl. Genet. -1991. -Vol. 83. -P. 250-256
- Vizir I., Korol A.B. Sex difference in recombination frequency in Arabidopsis//Heredity. -1990. -Vol. 65. -P. 379-383
- Rick C.M. Controlled introgression of chromosomes of Solanum penneli into Lycopersicon esculentum: segregation and recombination//Genetics. -1969. -Vol. 62. -P. 753-768
- Bonierbale M.W., Plaisted R.L., Tanksley S.D. RFLP maps based on a common set of clones reveal modes of chromosomal evolution in potato and tomato//Genetics. -1988. -Vol. 120. -P. 1095-1103
- Gebhardt C., Ritter E., Barone A., Debener T., Walkemeier B., Schachtschabel U., Kaufmann H., Thompson R.D., Bonierbale M.W., Ganal M.W., Tanksley S.D., Salamini F. RFLP maps of potato and their alignment with the homologous tomato genome//Theor. Appl. Genet. -1991. -Vol. 83. -P. 49-57
- Paterson A.H., Lander E.S., Hewitt J.D., Peterson S., Loncoln S.E. Tanksley S.D. Resolution of quantitative traits into Mendelian factors by using a complete linkage map of restriction fragment length polymorphisms//Nature. -1988. -Vol. 335. -P. 721-726
- Causse M.A., Fulton T.M., Cho Y.G., Ahn S.N., Chunwongse J., Wu K., Xiao J., Yu Z., Ronald P.C., Herrington S.E., Second G., McCouch S.R., Tanksley S.D. Saturated molecular map of the rice genome based on an interspecific backcross population//Genetics. -1994. -Vol. 138. -P. 12511274
- Borts R.H., Haber J.E. Meiotic recombination in yeast: alteration by multiple heterozygosities//Science. -1987. -Vol. 237. -P. 1459-1465
- Beavis W.D., Grant D. A linkage map based on information from F2 populations of maize (Zea mays L.)//Theor. Appl. Genet. -1991. -Vol. 82. -P. 636-644
- Burr B., Burr F.A., Thompson K.H., Albertsen M.C., Stuber C.W. Gene mapping with recombinant inbreeds in maize//Genetics. -1988. -Vol. 118. -P. 519-526
- Arumuganathan K., Earle E.D. Nuclear DNA content of some important plant species//Plant Mol. Biol. Rep. -1991. -Vol. 9. P. 208-218
- Bennett M.D., Smith J.B. Nuclear DNA amount in angiosperms//Proc. R. Soc. Lond. B. -1991. -Vol. 334. -P. 309-345
- Kaback D.B., Guacci V., Barber D., Mahon J.W. Chromosome size dependent control of meioic recombination//Science. -1992. -Vol. 256. -P. 228-232
- Flavell R.B. Chromosome architecture: the distribution of recombination sites, the structure of ribosomal DNA loci and the multiplicity of sequences containing inverted repeats. In: Molecular Form and Function of the Plant Genome. Van Vloten-Dtin L., Groot G.S.P., Hall I.C. (eds.). NATO ASI, 83. Plenum Press. New York. -1985. -P. 1-14
- Oliver S.G., Van der Aart Q.J., Agostoni-Garbone M.L., Aigle M., Alberghina L., Alexandraki D., Antoine G. et al. The complete DNA sequence of yeast chromosome III//Nature. -1992. -Vol. 357. -P. 38-46
- Donner H.K. Genetic fine structure of the bronze locus in maze//Genetics. -1986. -Vol. 113. P. 1021-1036
- Schmidt R., West J., Love K., Lenehan Z., Lister C., Thompson H., Bouchez D., Dean C. Physical map and organization of Arabidopsis thaliana chromosome 4//Science. -1995. -Vol. 270. -P. 480-483
- Rocher J.P., Prioul J.L., Lecharny A., Reyss A., Joussaume M. Genetic variability in carbon fixation, sucrose-P-synthase and ADP glucose pyrophosphorylase in maize plants of differing growth rate//Plant Physiol. -1989. -Vol. 89. -P. 416-420
- Slocum M.K., Figdore S.S., Kennard W.C., Suzuki J.Y., Osborn T.C. Linkage arrangement of restriction fragment length polymorphism loci in Brassica oleracea//Theor. Appl. Genet. -1990. -Vol. 80. -P. 57-64
- Song K.M., Suzuki J.Y., Slocum M.K., Williams P.H., Osborn T.C. A linkage map of Brassica rapa (syn. campestris) based on restriction length polymorphism loci//Theor. Appl. Genet. -1991. -Vol.82. -P. 296-304
- Kishimoto N., Higo H., Abe K., Arai S., Siato A., Higo G. Identification of the duplicated segments in rice chromosomes 1 and 5 by linkage analysis of cDNA markers of known functions//Theor. Appl. Genet. -1994. -Vol. 88. -P. 722-726
- Blanc G., Barakat A., Guyot R., Cooke R., Denseny M. Extensive duplication and reshuffling in the Arabidopsis genome//Plant Cell. -2000. -Vol. 12. -P. 1093-1101
- Grant D., Cregan P., Schoemaker R.C. Genome organization in dicots: genome duplication in Arabidopsis and synteny between soybean and Arabidopsis//Proc. Natl. Acad. Sci. USA. -2000. -Vol. 97. -P. 4168-4173
- Lyon M.F. Dunn and mouse genetic mapping//Genetics. -1990. -Vol. 125. -P. 231-236
- Morizot D.C. Use fish gene maps to predict ancestral vertebrate genome organization. In: Isozymes: Structure, Function and Use in Biology and medicine. Ogita Z.I., Marker C.L. (eds.). Liss, Wiley, New York. -1990. -P. 207-234
- Tanksley S.D., Ganal M.W., Prince J.P., De Vicente M.C., Bonierbale M.W., Broun P., Fulton T.M., Giovannoni J.J., Grandillo S., Martin G.B., Messeguer R., Miller L., Paterson A.H., Pinedo O., Roder M.S., Wing R.A., Wu W., Young N.D. High density molecular linkage maps of the tomato and potato genomes//Genetics. -1992. -Vol. 132. -P. 1141-1160
- Lefebvre V., Palloux A., Caranta C., Pochard E. Construction of an intraspecific integrated linkage map of pepper using molecular markers and double haploid progenies//Genome. -1995. -Vol. 38. P. 112-121
- Livingstone K.D., Lackney V.K., Blauth J.R., Van Wijk R., Jahn M.K. Genome mapping in Capsicum and the evolution of genome structure in the Solanaceae//Genetics. -1999. -Vol. 152. -P. 1183-1202
- Tanksley S.D., Bernatzky R., Lapitan N.L., Prince J.P. Conservation of gene repertoire but not gene order in paper and tomato//Proc. Natl. Acad. Sci. USA. -1988. -Vol. 85. P. 6419-6423
- Weeden N.F., Muehlbauer F.J., Ladizinsky G. Extensive conservation of linkage relationships between pea and lentil genetic maps//J. Hered. -1992. -Vol. 83. -P. 123-129
- Devos K.M., Atkinsin M.D., Chinoy C.N., Liu C.J., Gale M.D. RFLP-based genetic map of the homoeologous group 3 chromosomes of wheat and rye//Theor. Appl. Genet. -1992. -Vol. 83. -P. 931-939
- Sharp P.J., Kreis M., Shewry P.R., Gale M.D. Location of p-amilase sequences in wheat and its relatives//Theor. Appl. Genet. -1988. -Vol. 75. -P. 286-290
- Wang M.L., Atkinson M.D., Chinoy C.N., Devos K.M., Gale M.D. Comparative RFLP-based genetic maps of barley chromosome 5 1H and rye chromosome 1R//Theor. Appl. Genet. -1992. -Vol. 84. -P. 339-344
- Melake-Berhan A., Hulbert S.H., Bultmer L.G., Bennetzen J.L. Structure and evolution of the genomes of sorghum bicolor and Zea mays//Theor. Appl. Genet. -1993. -Vol. 86. -P. 598-604
- Whitkus R., Doebley J., Lee M. Comparative genome mapping of sorghum and maize//Genetics. -1992. -Vol. 132. -P. 1119-1130
- Grivet L., DrHont A., Dufour P., Hamon P., Roques D., Glaszmann J.C. Comparative genome mapping of sugar cane with other species within the Andropogoneae tribe//Heredity. -1994. -Vol. 73. -P. 500-508
- Ahn S., Anderson J.A., Sorrells M.E., Tanksley S.D. Homoeologous relationships of rice, wheat and maize chromosomes//Mol. Gen. Genet. -1993. -Vol. 241. -P. 483-490
- Ahn S., Tanksley S.D. Comparative linkage maps of the rice and maize genomes//Proc. Natl. Acad. Sci. USA. -1993. -Vol. 90. -P. 7980-7984
- Kurata N., Moore G., Nagamura Y., Foote T., Yano M., Minobe Y., Gale M. Conservation of genome structure between rice and wheat//BioTechnology. -1994. -Vol. 12. -P. 276-278
- Вавилов Н.И. Закон гомологических рядов в наследственной изменчивости. В: Теоретические основы селекции растений. М., Л. -1935. -Т.1. -C.75-128
- Plaschke J., Boerner A., Xie D.X., Koebner R.M.D., Schlegel R., Gale M.D. RFLP mapping of genes affecting plant height and growth habit in rye//Theor. Appl. Genet. -1993. -Vol. 85. -P. 1049-1054
- Bennetzen J.L., Freeling M. Grasses as a single genetic system: genome composition, collinearity and compatibility//Trends in Genet. -1993. -Vol. 9. -P. 259-261
- Freeling M. Grasses as a single genetic system: reassessment//Plant Physiol. -2001. -Vol. 125. -P. 1191-1197
- Asnaghi C., Paulet F., Kaye C., Grivet L., Deu M., Glaszmann J.C., Dr Hont A. Application of synteny across Poaceae to determine the map location of a sugar cane rust resistance gene//Theor. Appl. Genet. -2000. -Vol. 101. -P. 962-969
- Robert L.S., Robson F., Sharpe A., Lyndiate D., Coupland G. Conserved structure and function of the Arabidopsis flowering time gene CONSTANS in Brassica napus//Plant Mol. Biol. -1998. -Vol. 37. -P. 763-772


