Некоторые проблемы идентификации подвидов медоносной пчелы и их решение на примере изучения Apis mellifera в Сибири
Автор: Островерхова Н.В., Конусова О.Л.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Структура генома, геномные технологии
Статья в выпуске: 2 т.57, 2022 года.
Бесплатный доступ
При изучении медоносной пчелы Apis mellifera L. применяются как классический морфометрический, так и молекулярно-генетические методы, включая полногеномное секвенирование. С использованием морфометрического анализа тридцать подвидов A. mellifera были распределены по четырем эволюционным линиям, которые соответствовали их географическому происхождению. Исследование полиморфизма мтДНК, например локуса COI-COII, позволило выделить три основные эволюционные линии - А, М и С. Вместе с тем метод имеет ограничение, связанное с материнским наследованием митохондриального генома. Поскольку у медоносной пчелы нет половых хромосом, информацию о наследовании по линии трутней (а также по линии матки) можно получить только на основании анализа аутосомных локусов, таких как SNP (single nucleotide poly-morphism) и SSR (simple sequence repeats) маркеры. В настоящей работе впервые проведена оценка информативности морфометрического и молекулярно-генетического методов для идентификации подвидов A. mellifera , обитающих на пасеках Сибири, и показано, что анализ изменчивости основных параметров крыла (кубитальный индекс, гантельный индекс, дискоидальное смещение) и локуса COI-COII мтДНК позволяет точно определить происхождение пчелиной семьи. Впервые изучено генетическое разнообразие среднерусской породы сибирских популяций с использованием микросателлитных локусов. Выявлены породо- и экоспецифичные диагностические аллели, позволяющие дифференцировать среднерусскую породу и ее экотипы, а также подвиды южного происхождения (карпатскую породу, карнику). Цель работы - оценить информативность морфометрического и молекулярно-генетических методов для идентификации подвидов A. mellifera при изучении медоносной пчелы в Сибири. Объектом исследования были медоносные пчелы, полученные с 92 пасек 69 населенных пунктов, расположенных в пяти регионах Сибири: в Томской и Кемеровской областях, в Красноярском крае, Алтайском крае и Республике Алтай. Чтобы изучить породный состав медоносных пчел на пасеках Сибири, первоначально рабочие особи из 414 пчелиных семей были исследованы с использованием морфометрического метода и анализа вариабельности локуса COI-COII мтДНК. На втором этапе выявленные пчелиные семьи среднерусской породы A. m. mellifera были изучены с использованием комплекса SSR-маркеров. Исследована ва-риабельность 31 микросателлитного локуса. С целью поиска уникальных или специфичных для разных подвидов медоносной пчелы SSR-маркеров также изучено генетическое разнообразие пчел двух подвидов A. m. carpathica и A. m. carnica , имеющих южное происхождение (группа сравнения). Расчет популяционно-генетических параметров (частота аллелей, показатели наблюдаемой и ожидаемой гетерозиготности) проводили с использованием программы GenAlEx 6.5 (https://biology-assets.anu.edu.au/GenAlEx/). Оценка степени интрогрессии генов эволюционной линии С в линию М была выполнена на основе данных полиморфизма микросателлитных локусов с использованием программы STRUCTURE 2.3.4 (https://web.stanford.edu/group/pritchardlab/home.html). Выявлено, что три показателя крыла, а именно кубитальный индекс, гантельный индекс и дискоидальное смещение, совместно с данными по полиморфизму мтДНК необходимы и достаточны для дифференциации подвидов A. mellifera , причем показатель «дискоидальное смещение» одним из первых отклоняется от стандартных значений породы в процессе гибридизации пчел. В результате микросателлитного анализа выявлены локусы, которые дифференцируют как подвиды разных эволюционных линий (линии М и С), так и различные экотипы среднерусской пчелы. В качестве диагностических ДНК-маркеров могут быть рассмотрены локусы А043, Аp081, Аp049, AT139, A113, mrjp3 , состав и частота преобладающих аллелей которых (породоспецифичные аллели) различались у подвидов A. m. mellifera (линия М) и двух подвидов южного происхождения (карпатская порода A. m. carpathica и карника A. m. carnica , линия С). У пчел среднерусской породы A. m. mellifera аллели размером 128 п.н. локуса А043, 124 п.н. локуса Ap081, 127 п.н. локуса Ap049, 190 п.н. локуса AT139, 218 п.н. локуса A113 и 529 п.н. - mrjp3 регистрировали с высокой частотой (0,54-0,99), тогда как у пчел южного происхождения A. m. carpathica и A. m. carnica эти аллели оказались более редкими (0,01-0,27). Микросателлитный локус А008 рассматривается как наиболее перспективный молекулярный маркер для дифференциации различных экотипов темной лесной пчелы A. m. mellifera , обитающих на территории Сибири, Урала и Европы (выявлены экоспецифичные аллели). На основании изучения генетического разнообразия медоносных пчел, обитающих на территории Сибири, с использованием микросателлитных локусов разработана диагностическая панель молекулярных маркеров для дифференциации подвидов и экотипов медоносной пчелы, принадлежащих к эволюционным линиям М и С ( A. m. mellifera , A. m. carpathica и A. m. carnica ).
Apis mellifera, медоносная пчела, морфометрические признаки, мтднк, микросателлитные локусы, coi-coii, днк-маркеры, сибирь
Короткий адрес: https://sciup.org/142235672
IDR: 142235672 | УДК: 638.123:577.2 | DOI: 10.15389/agrobiology.2022.2.283rus
Текст научной статьи Некоторые проблемы идентификации подвидов медоносной пчелы и их решение на примере изучения Apis mellifera в Сибири
В настоящее время при исследовании медоносной пчелы Apis mellifera L. применяются как классический морфометрический (1, 2), так и молекулярно-генетические методы (3-5), включая полногеномное секвенирование (6). Вместе с тем морфометрия и анализ полиморфизма митохондриальной ДНК (мтДНК) наиболее часто используются для идентификации подвидов медоносной пчелы (1, 7, 8).
Морфометрический метод предполагает анализ около 40 качественных (окраска тергитов, форма задней границы воскового зеркальца, характеристики волосяной каймы на брюшке рабочих пчел и т.д.) и количественных (тарзальный индекс, кубитальный индекс, площадь воскового зеркальца и т.д.) признаков медоносной пчелы (9, 10). С использованием морфометрического анализа 30 подвидов A. mellifera были распределены по четырем эволюционным линиям, которые соответствовали их географическому происхождению: подвиды Африканского континента — линия А (Африканская), подвиды западной части Средиземноморья и северо-западной Европы — линия М (Мellifera), подвиды юго-восточной Европы и восточного Средиземноморья — линия С (Carnica), подвиды Ближнего Востока и западной части Азии — линия О (Oriental) (10).
Исследование полиморфизма мтДНК, например локуса COI-COII (последовательность между генами цитохромоксидазы I и цитохромокси-дазы II), позволило выделить три основные эволюционные линии — А, М и С (7, 11, 12). При этом варианты мтДНК не всегда согласуются с систематикой, основанной на морфологии. Например, подвиды морфологических линий С и О характеризуются одним и тем же вариантом мтДНК (12) . Согласно анализу мтДНК, у подвидов линии С выявлена самая короткая последовательность локуса COI-COII, или вариант Q. Наоборот, у подвидов линий М и А более длинная последовательность, которая включает минимум один элемент Q, а также элемент P (варианты PQ, PQQ, PQQQ, PQQQQ или PQQQQ) (13, 14).
Вместе с тем метод имеет ограничение, связанное с материнским наследованием митохондриального генома. Поскольку у медоносной пчелы нет половых хромосом, информацию о наследовании по линии трутней (а также по линии матки) можно получить только на основании анализа аутосомных локусов, таких как SNP (single nucleotide polymorphism, однонуклеотидный полиморфизм) и SSR (simple sequence repeats, микросателлитные локусы) маркеры. Так, микросателлитные локусы широко применяются в исследованиях медоносной пчелы, например для характеристики популяционно-генетической структуры (15-17), выявления гибридизации между разными подвидами (5, 18, 19), дифференциации подвидов (20, 21) и оценки адаптационного потенциала пчел различного происхождения и эволюционных линий (22-24). Однако генетическое разнообразие разных подвидов медоносной пчелы по микросателлитным локусам и другим ядерным маркерам изучено недостаточно, референсные материалы не разработаны. При этом на основании данных полиморфизма SNP-маркеров у пчел 14 подвидов выделены группы, в значительной степени соответствующие морфологическим эволюционным линиям. Также были установлены дополнительные линии африканского происхождения (например, линия Y — Yem-enitica, Эфиопия) (7, 25).
В России в связи с разнообразием природно-климатических условий к разведению рекомендованы три подвида пчел (23): темная лесная пчела, или среднерусская порода Apis mellifera mellifera L., кавказская порода A. m. cauca-sica Gorb. и карпатская порода A. m. carpathica (производная A. m. carnica Poll.). Также разводят карнику A. m. carnica, итальянскую пчелу A. m. ligus-tica Spin. и дальневосточнаую пчелу (продукт бессистемного скрещивания украинских степных A. m. sossimai Engel и среднерусских пчел при участии кавказских и итальянских пчел). В Сибири первоначально культивировалась среднерусская пчела A. m. mellifera , которую завезли 230 лет назад в Томскую губернию . Пчелы среднерусской породы хорошо адаптировались к местным суровым климатическим условиям и растительности, но зимовку пчелиных семей контролирует человек. С конца XX века на территории Сибири наблюдается активный завоз пчел южного происхождения, что привело к массовой гибридизации (26).
В настоящей работе впервые проведена оценка информативности морфометрического и молекулярно-генетического методов для идентификации подвидов A. mellifera , обитающих на пасеках Сибири, и показано, что анализ изменчивости основных параметров крыла (кубитальный индекс, гантельный индекс, дискоидальное смещение) и локуса COI-COII мтДНК позволяет точно определить происхождение пчелиной семьи. Впервые изучено генетическое разнообразие среднерусской породы сибирских популяций с использованием микросателлитных локусов. Выявлены породо- и экоспецифичные диагностические аллели, позволяющие дифференцировать среднерусскую породу и ее экотипы, а также подвиды южного происхождения (карпатскую породу, карнику).
Цель работы — оценить информативность морфометрического и молекулярно-генетических методов для идентификации подвидов Apis mellifera при изучении медоносной пчелы в Сибири.
Ìåòîäèêà . Исследования проводили в 2008-2018 годах. Объектом были медоносные пчелы, полученные с 92 пасек 69 населенных пунктов, расположенных в пяти регионах Сибири: Томской области (52 пункта/71 пасека/340 пчелиных семей), Кемеровской области (4/5/16), Красноярском крае (6/6/25), Алтайском крае (6/9/31) и Республике Алтай (1/1/2). Чтобы изучить породный состав медоносных пчел на пасеках Сибири, первоначально исследовали рабочих особей из 414 пчелиных семей с использованием морфометрического метода и анализа мтДНК.
На втором этапе выявленные пчелиные семьи среднерусской породы A. m. mellifera были изучены с использованием комплекса SSR-маркеров. Исследовали вариабельность 31 микросателлитного локуса. С целью поиска уникальных или специфичных для разных подвидов медоносной пчелы SSR-маркеров также было изучено генетическое разнообразие пчел двух подвидов — A. m. carpathica и A. m. carnica , имеющих южное происхождение (группа сравнения).
В процессе морфометрического анализа изучали основные породоопределяющие показатели крыла пчел (дискоидальное смещение, кубитальный и гантельный индексы), а также окраску тергитов. От каждой семьи брали 25-30 рабочих особей (27). Полученные данные морфометрического анализа сравнивали с показателями морфологических признаков согласно стандарту породы (28).
Для молекулярно-генетического анализа выделяли образцы ДНК из грудных мышц рабочих пчел с помощью набора ДНК-Экстран-2 (ЗАО «Синтол», Россия) и анализировали методом полимеразной цепной реакции (ПЦР) с использованием набора реагентов БиоМастер HS-Taq ПЦР-Color
(ООО «Биолабмикс», Россия) на амплификаторе BioRad Т100 («Bio-Rad Laboratories, Inc.», США).
Анализ вариабельности локуса COI-COII мтДНК выполняли с использованием следующих последовательностей праймеров: F-5 ′ -САСАТТТ-AGAAATTCCATTA-3', R-5 ‘ -AТAAAТAТGAAТСAТGТGGA-3' (29). Условия ПЦР: 5 мин при 95 °С (первичная денатурация); 1 мин при 95 °С (денатурация), 2 мин при 57 °С (отжиг праймеров), 2 мин при 72 °С (элонгация цепи) (35 циклов); 7 мин при 72 °С (финальная элонгация). ПЦР-продукты разделяли в 1,5 % агарозном геле и анализировали в программе Image LabTM Software («Bio-Rad Laboratories, Inc.», США). У среднерусской породы медоносной пчелы локус COI-COII был представлен аллелями PQQ и PQQQ, имеющими размеры соответственно 600 и 800 п.н., тогда как у пород южного происхождения (карпатская порода, карника и др.) присутствовал аллель Q размером 350 п.н. Анализировали 5 рабочих особей от пчелиной семьи.
Для SSR-анализа использовали предварительно описанные последовательности праймеров и условия амплификации (30-32). Была изучена вариабельность микросателлитных локусов А008, Ap049, А043, AC117, Ap243, H110, A024, A113, SV185, Ap066, Ap081, A088, A007, A028, 6339, SV220, K0457B, K1168, Ap033, K0820, Ap007, Ap068, K0405, K1615, K0711, SV167, Ap249, Ap226, A056, AT139, mrjp3. Один праймер из каждой пары имел флуоресцентную метку. Генотипирование образцов проводили на генетическом анализаторе ABI Prism 3730 («Applied Biosystems, Inc.», США; Центр коллективного пользования НИИ медицинской генетики Томского НИМЦ РАН «Медицинская геномика»). Использовали стандарт длины молекул ДНК GeneScan500-ROX в условиях, рекомендуемых производителем. Размер фрагментов анализировали с помощью программного обеспечения GeneMapper Software («Applied Biosystems, Inc.», США). Для каждой выборки пчел по разным микросателлитным локусам было исследовано минимум 27 и максимум 534 рабочих особи. В выборке пчел алтайской популяции микросател-литный анализ не проводили для локусов A028, A088, A056, K0711, Ap068, K1615 и Ap249, у пчел томской популяции — для локусов Ap033 и SV167.
Расчет популяционно-генетических параметров (частота аллелей, показатели наблюдаемой и ожидаемой гетерозиготности — Ho и He) проводили в программе GenAlEx 6.5 (33). Статистически значимые различия между наблюдаемой и ожидаемой гетерозиготностью выявляли с помощью t-критерия Стьюдента. Степень интрогрессии генов эволюционной линии С (карпатская порода A. m. car-pathica и карника A. m. carnica) в ядерном геноме среднерусских пчел (эволюционная линия М) разных популяций Сибири оценивали по данным полиморфизма микросателлитных локусов с использованием программы STRUCTURE 2.3.4 на основе байесовского анализа (Bayesian analysis). Применяли метод кластеризации Монте-Карло с цепями Маркова (Monte Carlo Markov Chain, MCMC) при заданном числе кластеров K = 2, K = 3 и K = 5 с использованием модели смешивания (admixture model) и повторности MCMC (iteration) 500000 (34). Подготовительную обработку данных проводили в приложении Microsoft Excel 2010, статистические расчеты выполняли в приложении StatSoft STATISTICA 8.0 for Windows («StatSoft, Inc.», США). В таблицах представлены средние значения показателей (M) и стандартные ошибки средних (±SEM).
Результаты . Согласно данным, полученным при исследовании морфометрических показателей и митохондриального генома медоносных пчел, большинство пчелиных семей, обитающих на территории Сибири, было 286
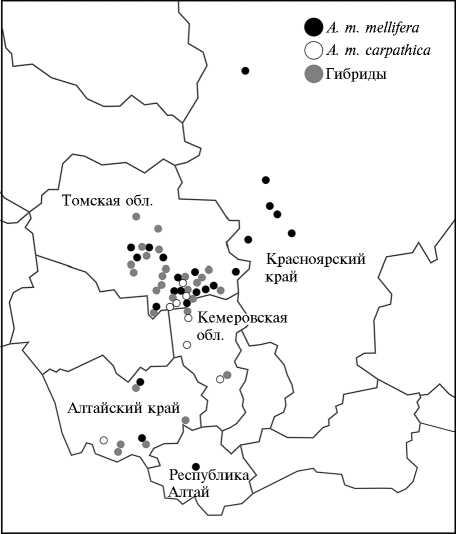
Рис. 1. Карта распределения подвидов Apis mellifera mel-lifera и A. m. carpathica , а также гибридов между этими подвидами на пасеках Сибирского региона Российской Федерации по данным морфометрического исследования и анализа мтДНК (2008-2018 годы) .
представлено гибридами среднерусской и карпатской пород (рис. 1). При этом более 60 % семей по материнской линии произошли от среднерусской пчелы, согласно данным анализа мтДНК (35).
Выраженная гетерогенная картина наблюдалась в регионах с хорошо развитым пчеловодством (Кемеровская область, южные районы Томской области, Алтайский край), куда активно завозятся пчелы южного происхождения (преимущественно карпатская порода, карника) и гибриды. Например, в Кемеровской области на большинстве пасек были представлены гибриды среднерусской пчелы и пород южного происхождения, но также имелись промышленные пасеки, где культивировались породы южного происхождения ( A. m. carpathica , A. m. carnica ). Пасек, где разводили чистопородную среднерусскую пчелу, мы не обнаружили.
В более отдаленных регионах Сибири (северные районы Томской области, Красноярский край, Республика Алтай) были обнаружены территории с однородным по породному составу массивом пчел, происходящих от среднерусской пчелы A. m. mellifera . Значительный интерес представляли популяции среднерусской породы на территории Красноярского края (енисейская популяция), на севере Томской области (обская популяция) и в горных районах Республики Алтай (алтайская популяция), а также отдельные промышленные пасеки в некоторых районах Алтайского края (Змеиногорский, Чарышский районы), где культивировалась A. m. mellifera (рис. 1).
В таблице 1 приведены основные параметры крыла и вариант локуса COI-COII мтДНК у пчел 34 чистопородных и гибридных семей, обитающих в Сибири. Для некоторых семей среднерусской породы, полученных с изолированных пасек (например, семья 3 с пасеки с. Остяцкое и семья ¹ 1 с пасеки в окрестностях с. Туруханск Красноярского края; семья ¹ 1 с пасеки в окрестностях с. Онгудай Республики Алтай), показано отклонение от стандарта породы по кубитальному индексу, что может свидетельствовать в пользу генетического дрейфа как результата длительной изоляции семей. В то же время отклонение некоторых морфометрических показателей от стандарта породы, например в семьях с пасеки п. Заречный (наличие в семье 27 % особей с нейтральным дискоидальным смещением), могло указывать на влияние подвидов южного происхождения (см. табл. 1). Пасека в п. Заречный находится в южном районе Томской области, где на близко расположенных пасеках разводятся пчелы разного происхождения.
-
1. Морфометрические показатели и вариант локуса COI-COII мтДНК у пчел ( Apis mellifera L.) некоторых чистопородных и гибридных семей, обитающих на территории Сибири (2008-2018 годы)
Населенный пункт
Вариант локуса
Кубитальный индекс, усл. ед.
Гантельный индекс, усл. ед.
Дискоидальное смещение, %
¹ семьи
COI-COII мтДНК
min-max
M ±SEM
min-max
M ±SEM
-
\ 0 1
+
Среднерус
ская порода
(Томская популяция)
п. Чалково
1
PQQ
1,30-2,00
1,75±0,04
0,720-0,871
0,811±0,010
100,0
0,0
0,0
2
PQQ
1,32-2,00
1,57±0,04
0,712-0,851
0,789±0,010
100,0
0,0
0,0
с. Тегульдет
1
PQQQ
1,44-2,10
1,75±0,03
0,692-1,000
0,854±0,011
100,0
0,0
0,0
2
PQQQ
1,26-2,22
1,74±0,04
0,701-0,914
0,825±0,010
100,0
0,0
0,0
с. Могочино
1
PQQ
1,36-2,00
1,73±0,02
0,693-0,923
0,821±0,006
100,0
0,0
0,0
2
PQQ
1,57-2,82
1,88±0,04
0,770-1,000
0,855±0,015
92,0
8,0
0,0
п. Заречный
1
PQQQ
1,39-2,33
1,65±0,04
0,712-0,932
0,825±0,009
73,5
26,5
0,0
2
PQQQ
1,19-2,05
1,64±0,06
0,679-1,000
0,865±0,012
73,1
26,9
0,0
Среднерусск
ая порода (Красноярская
популяция)
с. Остяцкое
1
PQQ
1,24-2,00
1,61±0,04
0,675-0,892
0,795±0,011
100,0
0,0
0,0
2
PQQ
1,25-1,76
1,60±0,04
0,722-0,849
0,812±0,011
100,0
0,0
0,0
3
PQQ
1,26-1,79
1,51±0,04
0,743-0,910
0,849±0,012
97,0
3,0
0,0
с. Колмогорово
1
PQQ
1,32-2,10
1,60±0,05
0,724-0,900
0,820±0,009
97,0
3,0
0,0
2
PQQ
1,28-1,76
1,58±0,05
0,716-0,919
0,820±0,012
93,0
7,0
0,0
3
PQQ
1,28-1,86
1,56±0,04
0,746-0,985
0,810±0,011
97,0
3,0
0,0
с. Озерное
1
PQQ
1,02-2,00
1,62±0,04
0,746-1,000
0,845±0,011
100,0
0,0
0,0
2
PQQ
1,45-1,95
1,65±0,04
0,768-1,000
0,867±0,010
93,3
6,7
0,0
3
PQQ
1,35-2,05
1,65±0,04
0,716-0,951
0,806±0,010
100,0
0,0
0,0
п. Якша
1
PQQ
1,43-1,82
1,65±0,04
0,803-0,915
0,859±0,011
100,0
0,0
0,0
с. Ярцево
1
PQQ
1,31-1,85
1,59±0,02
0,711-0,846
0,775±0,008
100,0
0,0
0,0
с. Туруханск
1
PQQ
1,15-1,89
1,52±0,04
0,779-0,919
0,849±0,011
100,0
0,0
0,0
Среднерусская порода
(Алтайская популяция)
с. Барановка
1
PQQ
1,42-2,10
1,75±0,04
0,732-0,883
0,833±0,012
100,0
0,0
0,0
Окрестности с. Онгудай
1
PQQ
1,00-1,67
1,44±0,04
0,740-0,919
0,840±0,012
100,0
0,0
0,0
-
2. Параметры генетического разнообразия 25 микросателлитных локусов у медоносных пчел ( Apis mellifera L.) различного происхождения, обитающих на территории Сибири (2008-2018 годы)
Подвид медоносной пчелы A. mellifera
mellifera
carpathica
1 carnica
Параметр
эволюционная линия M
эволюционная линия C
(PQQ, PQQQ
локуса COI-COII мтДНК)
(Q локуса COI-COII мтДНК)
Томская
Красноярский
Алтай
семьи, завезенные
область
край
в Сибирь из
питомников
Локус Ap066
Число:
исследованных пчел
206
323
36
196
97
аллелей
6
7
5
6
3
Частота преобладающих аллелей:
90 п.н.
0,325±0,023а
0,130±0,013
0,306±0,054а
0,933±0,018а
0,921±0,014а
96 п.н.
0,187±0,019
0,370±0,019а
0,417±0,058а
0,046±0,015
0,041±0,010
98 п.н.
0,347±0,024а
0,252±0,017
0,222±0,049
0
0
H o
0,782±0,029
0,625±0,027***
0,694±0,077
0,148±0,025
0,113±0,032
H e
0,726±0,010
0,728±0,007
0,682±0,026
0,150±0,024
0,127±0,032
Локус A024
Число:
исследованных пчел
307
534
34
183
106
аллелей
6
8
4
6
9
Частота преобладающих аллелей:
92 п.н.
0,660±0,019а
0,473±0,015а
0,779±0,050а
0
0
96 п.н.
0,007±0,003
0,156±0,011
0
0
0
98 п.н.
0,011±0,004
0,028±0,005
0,029±0,021
0,219±0,022
0,099±0,021
100 п.н.
0,186±0,016
0,126±0,010
0,162±0,045
0,301±0,024а
0,448±0,034а
102 п.н.
0,007±0,003
0,029±0,005
0
0,156±0,019
0,297±0,031
104 п.н.
0
0,059±0,007
0
0,169±0,020
0
106 п.н.
0,130±0,014
0,085±0,009
0
0,101±0,016
0,057±0,016
H o
0,505±0,029
0,468±0,022***
0,265±0,076
0,563±0,037***
0,566±0,048*
H e
0,513±0,020
0,722±0,012
0,365±0,065
0,798±0,009
0,693±0,021
Локус Ap081
Число:
исследованных пчел
150
372
33
193
137
аллелей
5
4
3
6
4
Частота преобладающих аллелей:
124 п.н.
0,820±0,022а
0,973±0,006а
0,955±0,026а
0,204±0,024
0,042±0,010
130 п.н.
0,037±0,011
0,009±0,004
0,030±0,021
0,762±0,022а
0
132 п.н.
0
0
0
0,101±0,015
0,708±0,028а
H o
0,120±0,027***
0,032±0,009
0,091±0,050
0,337±0,034
0,489±0,043
H e
0,318±0,034
0,053±0,011
0,088±0,047
0,404±0,030
0,452±0,031
Локус A043
Число:
исследованных пчел
305
418
33
177
106
аллелей
4
4
3
4
3
Частота преобладающих аллелей:
128 п.н.
0,831±0,015а
0,974±0,006а
0,864±0,042а
0,071±0,014
0,222±0,029
132 п.н.
0,012±0,004
0,006±0,003
0,121±0,040
0,020±0,007
0
Продолжение таблицы 2
138 п.н.
0
0
0
0,105±0,016
0,009±0,007
140 п.н.
0,156±0,015
0,014±0,004
0,015±0,015
0,805±0,021а
0,769±0,029а
H o
0,279±0,026
0,022±0,007*
0,030±0,030**
0,266±0,033
0,443±0,048
H e
0,285±0,021
0,052±0,011
Локус
0,239±0,063
A007
0,336±0,030
0,360±0,032
Число
исследованных пчел
206
381
36
193
114
аллелей 3
Частота преобладающих аллелей:
6
3
9
8
104 п.н.
0,063±0,012
0,154±0,013
0,444±0,059а
0,005±0,004
0,081±0,019
108 п.н.
0,820±0,019а
0,810±0,014а
0,542±0,059а
0,357±0,025а
0,257±0,030
112 п.н.
0,117±0,016
0,013±0,004
0
0,378±0,025а
0,114±0,022
114 п.н.
0
0,008±0,003
0,014±0,014
0,114±0,016
0,252±0,030
131 п.н.
0
0
0
0,082±0,014
0,200±0,028
H o
0,199±0,028**
0,297±0,023
0,806±0,066
0,609±0,036*
0,495±0,049***
H e
0,309±0,027
0,320±0,020
Локус
0,509±0,020
A008
0,708±0,013
0,808±0,011
Число:
исследованных пчел
306
451
34
193
135
аллелей 6
Частота преобладающих аллелей:
6
3
9
7
162 п.н.
0,868±0,014а
0,938±0,008а
0,868±0,041а
0,106±0,016
0,315±0,028а
170 п.н.
0
0
0,118±0,039
0,065±0,013
0,067±0,015
174 п.н.
0,023±0,006
0,009±0,003
0,015±0,015
0,443±0,025а
0,415±0,030а
178 п.н.
0
0
0
0,153±0,018
0,096±0,018
H o
0,226±0,024
0,093±0,014
0,265±0,076
0,580±0,036***
0,607±0,042*
H e
0,243±0,023
0,119±0,015 Локус
0,233±0,062
AC117
0,751±0,018
0,710±0,017
Число:
исследованных пчел
301
497
34
185
137
аллелей 4
Частота преобладающих аллелей:
4
3
4
4
177 п.н.
0,098±0,012
0,139±0,011
0,029±0,021
0,014±0,006
0,157±0,022
181 п.н.
0,292±0,019
0,163±0,012
0,044±0,025
0,203±0,021
0,190±0,024
185 п.н.
0,517±0,020а
0,687±0,015а
0,927±0,032а
0,778±0,022а
0,650±0,029а
H o
0,389±0,028***
0,302±0,021*** 0,147±0,061
0,297±0,034
0,219±0,035***
H e
0,629±0,014
0,482±0,016 Локус
0,139±0,055
AT139
0,353±0,026
0,517±0,028
Число:
исследованных пчел
38
162
34
129
135
аллелей 3
Частота преобладающих аллелей:
3
2
7
5
177 п.н.
0,211±0,047
0,080±0,015
0,015±0,015
0,124±0,021
0,656±0,029а
179 п.н.
0
0
0
0,349±0,030а
0
182 п.н.
0
0
0
0,349±0,030а
0,230±0,026
186 п.н.
0,118±0,037
0
0
0,012±0,007
0
190 п.н.
0,671±0,054а
0,880±0,018а
0,985±0,015а
0,008±0,006
0,022±0,009
H o
0,500±0,081
0,154±0,028
0,029±0,029
0,674±0,041
0,430±0,043
H e
0,491±0,053
0,218±0,029
Локус
0,029±0,028
Ap243
0,718±0,013
0,510±0,028
Число:
исследованных пчел
212
316
32
198
107
аллелей 8
Частота преобладающих аллелей:
8
8
4
7
252 п.н.
0,014±0,006
0,137±0,014
0,141±0,044
0,970±0,009а
0,869±0,023а
255 п.н.
0,427±0,024а
0,242±0,017
0,031±0,022
0
0,037±0,013
257 п.н.
0
0,008±0,004
0,563±0,062а
0
0
263 п.н.
0,330±0,023а
0,390±0,020а
0,125±0,041
0,005±0,004
0,033±0,012
H o
0,439±0,034***
0,468±0,028*
** 0,500±0,088
0,020±0,010*
0,140±0,034
H e
0,694±0,014
0,753±0,010 Локус
0,642±0,058
SV185
0,059±0,016
0,241±0,039
Число:
исследованных пчел
243
278
36
57
30
аллелей 5
Частота преобладающих аллелей:
5
3
4
5
263 п.н.
0,288±0,021
0,158±0,016
0,417±0,058а
0,149±0,033
0,100±0,039
266 п.н.
0,117±0,015
0,385±0,021а
0,097±0,035
0,149±0,033
0,300±0,059а
269 п.н.
0,578±0,022а
0,421±0,021а
0,486±0,059а
0,386±0,046а
0,233±0,055
272 п.н.
0,010±0,005
0,002±0,002
0
0,316±0,044а
0,350±0,062а
H o
0,527±0,032
0,550±0,030*
* 0,583±0,082
0,439±0,066***
0,867±0,062*
H e
0,569±0,017
0,649±0,009
0,581±0,027
0,707±0,019
0,723±0,023
Продолжение таблицы 1
Карпатская порода
|
Разные регионы Сибири |
1 |
Q |
2,20-3,40 |
2,80±0,10 |
0,919-1,208 |
0,972±0,009 |
0,0 |
23,3 |
76,7 |
|
2 |
Q |
2,00-3,50 |
2,93±0,07 |
0,967-1,230 |
1,118±0,015 |
0,0 |
0,0 |
100,0 |
|
|
3 |
Q |
2,11-4,10 |
3,12±0,10 |
0,905-1,205 |
1,048±0,015 |
3,4 |
10,4 |
86,2 |
|
|
4 |
Q |
1,68-3,64 |
2,51±0,06 |
0,867-1,210 |
1,050±0,010 |
4,0 |
20,0 |
76,0 |
|
|
5 |
Q |
2,00-5,56 |
3,41±0,02 |
0,905-1,310 |
1,125±0,016 |
0,0 |
3,0 |
97,0 |
|
|
Гибридные |
семьи (пасеки Томской |
области) |
|||||||
|
п. Курлек |
1 |
PQQQ |
1,74-3,29 |
2,14±0,07 |
0,857-1,053 |
0,937±0,100 |
32,14 |
57,14 |
10,72 |
|
2 |
Q |
1,30-2,29 |
1,66±0,04 |
0,735-0,965 |
0,878±0,011 |
72,40 |
27,60 |
0,00 |
|
|
с. Кривошеино |
1 |
PQQ |
1,00-2,67 |
1,79±0,07 |
0,707-1,000 |
0,884±0,018 |
27,27 |
18,18 |
54,55 |
|
п. Синий Утес |
1 |
Q |
1,83-2,87 |
2,37±0,06 |
0,815-1,053 |
0,931±0,012 |
6,70 |
76,70 |
16,60 |
|
с. Подгорное |
1 |
Q |
1,35-2,87 |
2,19±0,10 |
0,797-1,100 |
0,910±0,010 |
34,30 |
45,70 |
20,00 |
|
2 |
Q |
1,72-3,01 |
2,53±0,06 |
0,852-1,178 |
0,977±0,011 |
51,40 |
14,30 |
34,30 |
|
|
Окрестности г. Томска |
1 |
Q |
1,64-2,69 |
2,12±0,05 |
0,722-0,946 |
0,895±0,011 |
90,00 |
6,70 |
3,30 |
|
Стандарты |
породы |
||||||||
|
A. m. mellifera а |
1,30-2,10 |
1,70 |
0,600-0,923 |
- |
- |
- |
- |
||
|
A. m. mellifera б |
1,30-1,90 |
1,5-1,7 |
0,600-0,923 |
- |
91-100 |
5-10 |
0 |
||
|
A. m. carpathica а |
Q |
2,30-3,00 |
2,65 |
- |
0,925 |
0-5 |
0-20 |
80-100 |
|
Примечание. а — Европейский стандарт подвидов пчел, разработанный на основе значений кубитального и гантельного индексов (28), б — стандарт породы пчел, принятый в России. Методом морфометрического анализа исследовано минимум 25 особей от семьи; анализ мтДНК (вариабельность локуса COI-COII) проведен для 5 особей от пчелиной семьи. Прочерки означают отсутствие данных.
Таким образом, сибирские популяции медоносной пчелы характеризуются сложной и мозаичной генетической структурой. Нами выявлены как чистопородные пчелиные семьи различного происхождения, так и гибриды. Пчелиные семьи A. m. mellifera можно рассматривать как уникальный материал для сохранения и восстановления генофонда среднерусской пчелы на территории России.
Генетическое разнообразие медоносных пчел среднерусской породы изучали по 31 микросателлитному локусу. В качестве группы сравнения использовали медоносных пчел от семей южных подвидов A. m. carpathica и A. m. carnica . Среди изученных локусов были отобраны 22 полиморфных SSR-маркера, наиболее информативных для дифференциации подвидов и экотипов медоносной пчелы (табл. 2). Для остальных 9 микросателлитных локусов отмечали сходный состав и частоту аллелей, включая преобладающие у пчел разного происхождения (в таблице 2 в качестве примера таких локусов приведены AC117, SV185 и H110).
Локус Ap049
|
Число: |
|||||
|
исследованных пчел |
309 |
442 |
36 |
183 |
85 |
|
аллелей |
5 |
7 |
4 |
9 |
5 |
|
Частота преобладающих аллелей: |
|||||
|
120 п.н. |
0,123±0,013 |
0,002±0,002 |
0 |
0,227±0,022 |
0,194±0,030 |
|
127 п.н. |
0,673±0,019а |
0,761±0,014а |
0,542±0,059а |
0,030±0,009 |
0,035±0,014 |
|
130 п.н. |
0,175±0,015 |
0,154±0,012 |
0,250±0,051 |
0,160±0,019 |
0,235±0,033 |
|
139 п.н. |
0,023±0,006 |
0,042±0,007 |
0 |
0,511±0,026а |
0,453±0,038а |
|
152 п.н. |
0 |
0,008±0,003 |
0,181±0,045 |
0,055±0,012 |
0,082±0,021 |
|
H o |
0,447±0,028 |
0,378±0,023 |
0,389±0,081* |
0,564±0,037* |
0,506±0,054** |
|
H e |
0,501±0,020 |
0,394±0,019 |
0,611±0,040 |
0,658±0,019 |
0,694±0,022 |
|
Локус |
A113 |
||||
|
Число: |
|||||
|
исследованных пчел |
290 |
509 |
33 |
194 |
136 |
|
аллелей |
11 |
4 |
2 |
9 |
6 |
|
Частота преобладающих аллелей: |
|||||
|
212 п.н. |
0,107±0,013 |
0,048±0,007 |
0 |
0,874±0,017а |
0,599±0,030а |
|
218 п.н. |
0,571±0,021а |
0,818±0,012а |
0,879±0,040а |
0,034±0,009 |
0,272±0,027 |
|
220 п.н. |
0,255±0,018 |
0,129±0,011 |
0,121±0,040 |
0,039±0,010 |
0,015±0,007 |
|
H o |
0,521±0,029* |
0,236±0,019** 0,000±0,000*** |
0,180±0,027 |
0,456±0,043* |
|
|
H e |
0,597±0,017 |
0,312±0,017 |
0,213±0,060 |
0,233±0,029 |
0,562±0,025 |
|
Локус |
H110 |
||||
|
Число: |
|||||
|
исследованных пчел |
282 |
439 |
36 |
195 |
77 |
|
аллелей |
3 |
5 |
5 |
5 |
6 |
|
Частота преобладающих аллелей: |
|||||
|
154 п.н. |
0 |
0 |
0 |
0,005±0,004 |
0,110±0,025 |
|
158 п.н. |
0 |
0,040±0,007 |
0,014±0,014 |
0,126±0,017 |
0,013±0,009 |
|
162 п.н. |
0,789±0,017а |
0,452±0,017а |
0,111±0,037 |
0,385±0,025а |
0,097±0,024 |
|
166 п.н. |
0,027±0,007 |
0,126±0,011 |
0,722±0,053а |
0,413±0,025а |
0,630±0,039а |
|
170 п.н. |
0,184±0,016 |
0,342±0,016а |
0,028±0,019 |
0,072±0,013 |
0,143±0,028 |
|
H o |
0,333±0,028 |
0,421±0,024* |
* 0,333±0,079 |
0,631±0,035 |
0,584±0,056 |
|
H e |
0,343±0,022 |
0,660±0,009 |
0,450±0,065 |
0,661±0,012 |
0,561±0,040 |
|
Локус |
A028 |
||||
|
Число: |
|||||
|
исследованных пчел |
126 |
343 |
0 |
170 |
109 |
|
аллелей |
3 |
4 |
Нет данных |
5 |
2 |
|
Частота преобладающих аллелей: |
|||||
|
126 п.н. |
0,770±0,027а |
0,848±0,014а |
0,071±0,014 |
0,243±0,029 |
|
|
132 п.н. |
0,167±0,024 |
0,054±0,009 |
0,829±0,020а |
0,757±0,029а |
|
|
H o |
0,397±0,044 |
0,274±0,024 |
0,341±0,036 |
0,431±0,047 |
|
|
H e |
0,376±0,034 |
0,268±0,021 |
0,301±0,031 |
0,368±0,030 |
|
|
Локус |
Ap226 |
||||
|
Число: |
|||||
|
исследованных пчел |
120 |
345 |
55 |
167 |
100 |
|
аллелей |
5 |
3 |
2 |
5 |
5 |
|
Частота преобладающих аллелей: |
|||||
|
227 п.н. |
0,033±0,012 |
0,197±0,015 |
0,200±0,127 |
0,087±0,015 |
0,065±0,017 |
|
233 п.н. |
0,871±0,022а |
0,801±0,015а |
0,800±0,127а |
0,051±0,012 |
0,260±0,031 |
|
235 п.н. |
0,004±0,004 |
0 |
0 |
0,210±0,022 |
0,090±0,020 |
|
237 п.н. |
0,017±0,008 |
0 |
0 |
0,608±0,027а |
0,400±0,035а |
|
247 п.н. |
0 |
0 |
0 |
0,045±0,011 |
0,185±0,028 |
|
H o |
0,225±0,038 |
0,035±0,010* |
* 0,000±0,000* |
0,473±0,039 |
0,620±0,049 |
|
H e |
0,235±0,035 |
0,319±0,018 |
0,320±0,143 |
0,575±0,025 |
0,726±0,017 |
|
Локус |
A088 |
||||
|
Число: |
|||||
|
исследованных пчел |
82 |
236 |
0 |
82 |
77 |
|
аллелей |
2 |
2 |
Нет данных |
2 |
3 |
|
Частота преобладающих аллелей: |
|||||
|
141 п.н. |
0,927±0,020а |
0,998±0,002а |
0,098±0,023 |
0,234±0,034 |
|
|
150 п.н. |
0 |
0 |
0,902±0,023а |
0,753±0,035а |
|
|
H o |
0,122±0,036 |
0,004±0,004 |
0,024±0,017* |
0,260±0,050 |
|
|
H e |
0,136±0,035 |
0,004±0,004 |
0,176±0,037 |
0,378±0,037 |
|
|
Локус |
A056 |
||||
|
Число: |
|||||
|
исследованных пчел |
39 |
41 |
0 |
146 |
110 |
|
аллелей |
3 |
2 |
Нет данных |
5 |
4 |
|
Частота преобладающих аллелей: |
|||||
|
280 п.н. |
0,026±0,018 |
0 |
0,188±0,023 |
0,336±0,032а |
|
|
282 п.н. |
0,667±0,053а |
0,720±0,050а |
0,089±0,017 |
0,127±0,023 |
|
|
284 п.н. |
0,256±0,049 |
0,281±0,050 |
0,630±0,028а |
0,473±0,034а |
|
|
H o |
0,013±0,013 |
0,463±0,078 |
0,473±0,041 |
0,536±0,048* |
|
|
H e |
0,039±0,022 |
0,404±0,044 |
0,555±0,029 |
0,643±0,019 |
|
Локус K0711
Число:
|
исследованных пчел |
82 |
170 |
0 |
127 |
53 |
|
аллелей |
3 |
3 |
Нет данных |
5 |
3 |
|
Частота преобладающих аллелей: |
|||||
|
212 п.н. |
0,976±0,012а |
0,918±0,015а |
0,020±0,009 |
0 |
|
|
219 п.н. |
0,018±0,011 |
0,071±0,014 |
0,815±0,024а |
0,868±0,033а |
|
|
222 п.н. |
0 |
0 |
0,130±0,021 |
0,113±0,031 |
|
|
H o |
0,049±0,024 |
0,094±0,022 |
0,299±0,041 |
0,264±0,061 |
|
|
H e |
0,048±0,023 |
0,153±0,025 |
0,318±0,035 |
0,234±0,050 |
|
|
Локус |
Ap068 |
||||
|
Число: |
|||||
|
исследованных пчел |
125 |
242 |
0 |
157 |
55 |
|
аллелей |
10 |
5 |
Нет данных |
7 |
9 |
|
Частота преобладающих аллелей: |
|||||
|
146 п.н. |
0,020±0,009 |
0,074±0,012 |
0,255±0,025 |
0,118±0,031 |
|
|
150 п.н. |
0,008±0,006 |
0 |
0,242±0,024 |
0,073±0,025 |
|
|
152 п.н. |
0,092±0,018 |
0 |
0,303±0,026а |
0,209±0,039 |
|
|
154 п.н. |
0,036±0,012 |
0,008±0,004 |
0,045±0,012 |
0,227±0,040 |
|
|
158 п.н. |
0,392±0,031а |
0,552±0,023а |
0,061±0,014 |
0,173±0,036 |
|
|
162 п.н. |
0,212±0,026 |
0,337±0,022а |
0,083±0,016 |
0,073±0,025 |
|
|
164 п.н. |
0,128±0,021 |
0 |
0 |
0 |
|
|
H o |
0,816±0,035 |
0,488±0,032* |
0,605±0,039*** |
0,782±0,056 |
|
|
H e |
0,769±0,018 |
0,576±0,015 |
0,772±0,010 |
0,841±0,013 |
|
|
Локус |
Ap033 |
||||
|
Число: |
|||||
|
исследованных пчел |
0 |
274 |
35 |
90 |
84 |
|
аллелей |
Нет данных |
6 |
6 |
11 |
9 |
|
Частота преобладающих аллелей: |
|||||
|
221 п.н. |
0,113±0,014 |
0,300±0,055а |
0,006±0,006 |
0 |
|
|
225 п.н. |
0,007±0,004 |
0,286±0,054 |
0,017±0,010 |
0,131±0,026 |
|
|
227 п.н. |
0 |
0 |
0,233±0,032 |
0,262±0,034 |
|
|
229 п.н. |
0 |
0 |
0 |
0,113±0,024 |
|
|
231 п.н. |
0 |
0 |
0,356±0,036а |
0,107±0,024 |
|
|
233 п.н. |
0,016±0,005 |
0,100±0,036 |
0,200±0,030 |
0,030±0,013 |
|
|
235 п.н. |
0,312±0,020а |
0,229±0,050 |
0,061±0,018 |
0,262±0,034 |
|
|
239 п.н. |
0,146±0,015 |
0,043±0,024 |
0 |
0 |
|
|
241 п.н. |
0,405±0,021а |
0,043±0,024 |
0 |
0 |
|
|
H o |
0,631±0,029* |
0,857±0,059 |
0,489±0,053*** |
0,607±0,053*** |
|
|
H e |
0,704±0,010 |
0,762±0,021 |
0,770±0,017 |
0,816±0,014 |
|
|
Локус |
SV167 |
||||
|
Число: |
|||||
|
исследованных пчел |
0 |
251 |
36 |
80 |
82 |
|
аллелей |
Нет данных |
4 |
3 |
8 |
8 |
|
Частота преобладающих аллелей: |
|||||
|
198 п.н. |
0,492±0,022а |
0,458±0,059а |
0 |
0 |
|
|
201 п.н. |
0,400±0,022а |
0,444±0,059а |
0,475±0,040а |
0,482±0,039а |
|
|
204 п.н. |
0 |
0 |
0 |
0,177±0,030 |
|
|
207 п.н. |
0,104±0,014 |
0,097±0,035 |
0,188±0,031 |
0,037±0,015 |
|
|
H o |
0,558±0,031 |
0,444±0,083 |
0,475±0,056*** |
0,512±0,055** |
|
|
H e |
0,587±0,011 |
0,583±0,026 |
0,716±0,030 |
0,711±0,030 |
|
|
Локус |
SV220 |
||||
|
Число: |
|||||
|
исследованных пчел |
81 |
362 |
36 |
82 |
81 |
|
аллелей |
7 |
7 |
5 |
7 |
6 |
|
Частота преобладающих аллелей: |
|||||
|
176 п.н. |
0,062±0,019 |
0,003±0,002 |
0 |
0,117±0,025 |
0,277±0,037 |
|
179 п.н. |
0,031±0,014 |
0 |
0,028±0,019 |
0,599±0,039а |
0,453±0,041а |
|
182 п.н. |
0 |
0,354±0,018а |
0,097±0,035 |
0,031±0,014 |
0,014±0,010 |
|
185 п.н. |
0,617±0,038а |
0,403±0,018а |
0,681±0,055а |
0,037±0,015 |
0,047±0,017 |
|
188 п.н. |
0,173±0,030 |
0,079±0,010 |
0,167±0,044 |
0,043±0,016 |
0,101±0,025 |
|
191 п.н. |
0,090±0,023 |
0,006±0,003 |
0 |
0,173±0,030 |
0,095±0,024 |
|
H o |
0,432±0,055* |
0,503±0,026*** 0,528±0,083 |
0,500±0,058** |
0,519±0,056 |
|
|
H e |
0,574±0,039 |
0,693±0,010 |
0,498±0,061 |
0,697±0,026 |
0,594±0,037 |
|
Локус |
K1615 |
||||
|
Число: |
|||||
|
исследованных пчел |
42 |
282 |
0 |
49 |
40 |
|
аллелей |
5 |
4 |
Нет данных |
5 |
5 |
|
Частота преобладающих аллелей: |
|||||
|
208 п.н. |
0,512±0,055а |
0,910±0,012а |
0,010±0,010 |
0,025±0,018 |
|
|
210 п.н. |
0 |
0 |
0 |
0,175±0,043 |
|
|
212 п.н. |
0 |
0 |
0,796±0,041а |
0,700±0,051а |
|
|
214 п.н. |
0,429±0,054а |
0,016±0,005 |
0,102±0,031 |
0 |
|
*, **, *** Различия между наблюдаемой от ожидаемой гетерозиготностью статистически значимы соответственно при p < 0,05; p < 0,01; p < 0,001.
В результате сравнительного анализа вариабельности микросател-литных локусов у среднерусских, карпатских пчел и пчел карника для некоторых SSR-маркеров были обнаружены различия в составе и/или частоте аллелей между подвидами. Так, для локусов Ap081, А008, А043, A139, А113, Ар243, Ар049, A024, A088, Ap226, K0711, SV220, K1615 и mrjp3 выявлены преобладающие аллели (частота регистрации более 0,40), состав которых различался у пчел среднерусской породы (линия М) и южных подвидов (линия С). Для локуса А043 преобладающим аллелем у A. m. mellifera (линия М) был аллель размером 128 п.н. (частота регистрации более 0,83), тогда как у A. m. carpathica и A. m. carnica (линия С) — аллель размером 140 п.н. (частота более 0,76). Для локуса А113 преобладающий аллель у A. m. carpathica и A. m. carnica имел размер 212 п.н., у A. m. mellifera — 218 п.н. (частота более 0,57). Для ряда локусов (например, Ap066, A007) выявлены общие преобладающие аллели, но с разной частотой встречаемости у подвидов линий М и С (см. табл. 2).
Характеристика генетического разнообразия на основе показателей гетерозиготности выявила сходные результаты для большинства изученных локусов у разных подвидов пчел: показаны более низкие значения наблюдаемой гетерозиготности (H o ) по сравнению с ожидаемой (H e ). Для некоторых SSR маркеров, например локуса Ap066 у пчел среднерусской породы и локусов A043, SV185 у пчел карника, значение H o было выше H e . Среди групп среднерусских пчел эта ситуация была наиболее характерна для алтайской популяции, что может быть обусловлено небольшим числом особей в выборке.
Статистически значимые различия между значениями H o и H e мы выявили для разных выборок по большинству локусов, кроме AT139, A028 и K0711. Так, статистически значимые различия между H o и H e были обнаружены у пчел среднерусской породы (енисейская популяция) для локусов Ap066, A024, AC117, Ap243, H110, Ap226 и SV220 ( t > 3,69, p < 0,001), SV185
и A113 ( t ≥ 2,98, p < 0,01), A043, Ap033 и Ap068 ( t ≥ 2,30, p < 0,05); у пчел томской популяции — для Ap081, AC117, Ap243, mrjp3 ( t ≥ 4,55, p < 0,001), A007 ( t = 2,83, p < 0,01), A113 и SV220 ( t ≥ 2,26, p < 0,05); у пчел алтайской популяции — для A113 ( t = 3,55, p < 0,001), A043 ( t = 3,00, p < 0,01), Ap049 и Ap226 ( t ≥ 2,46, p < 0,05); у A. m. carpathica — для A024, A008, SV185, SV167, Ap068, Ap033 ( t ≥ 3,90, p < 0,001), SV220 и Ap249 ( t = 3,10, p < 0,01), А007, Аp243, Аp049, A088 ( t ≥ 2,07, p < 0,05); у пчел A. m. carnica — для A007, AC117, Ap033, mrjp3 ( t ≥ 4,30, p < 0,001), Аp049 и SV167 ( t ≥ 3,18, p < 0,01), A113, A008, A024, SV185, A056 и K1615 ( t ≥ 2,13, p < 0,05) (см. табл. 2).
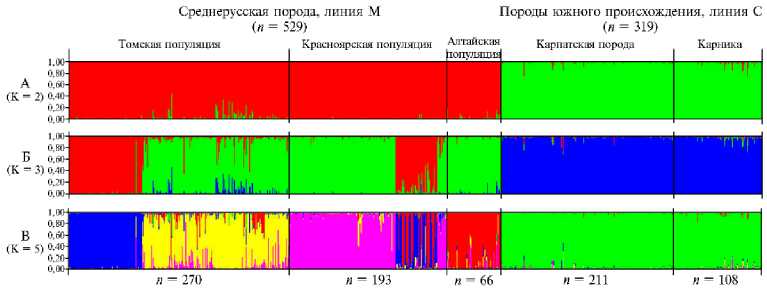
Рис. 2. Гистограмма, построенная в программе STRUCTURE 2.3.4 на основании данных по вариабельности 24 микросателлитных локусов у медоносных пчел ( Apis mellifera L.) среднерусской породы и пород южного происхождения, обитающих на территории Сибири (2008-2018 годы) . Вертикальные линии представляют долю членства особи в обозначенном цветом кластере.
А (K = 2): зеленый цвет отражает представленность ядерных генов, характерных для пород южного происхождения (карпатской породы A. m. carpathica и карники A. m. carnica ), красный цвет — генов среднерусской породы A. m. mellifera .
Б (K = 3): синий цвет отражает представленность ядерных генов, характерных для пород южного происхождения, остальные цвета — генов среднерусской породы.
В (K = 5): зеленый цвет отражает представленность ядерных генов, характерных для пород южного происхождения, остальные цвета — генов среднерусской породы.
Для оценки интрогрессии генов пород южного происхождения (карпатская порода, карника, эволюционная линия С) в линию M (среднерусская порода), а также выявления границ популяций A. m. mellifera мы провели сравнительное исследование генетического разнообразия среднерусской породы, карпатской породы и карники по комплексу маркеров ядер-ного генома (рис. 2). Гистограмма, построенная на основании данных по вариабельности 24 микросателлитных локусов, с одной стороны, наглядно показывает низкую степень интрогрессии генов линии С в линию М, то есть чистопородность среднерусских пчел сибирских популяций. С другой стороны, важной характеристикой сибирских популяций среднерусской породы был генетический полиморфизм, причем наибольшее разнообразие выявили для пчел томской популяции (пасеки северных и южных районов), а более однородными оказались пчелы алтайской популяции (преимущественно из репродуктора среднерусской породы, а также изолированной пасеки Республики Алтай). Вместе с тем наблюдалось перекрывание генетических вариантов у среднерусских пчел разных популяций, что, вероятно, связано с перемещением пчелиных семей, которое пчеловоды проводят на территории Сибири (например, с изолированных пасек Красноярского края или Республики Алтай на территорию Томской области). Наконец, показано высокое генетическое родство для подвидов A. m. carnica и A. m. car-pathica (производная карники), несмотря на различные условия обитания
(карпатская порода длительное время культивировалась в условиях Карпат).
При изучении медоносных пчел, обитающих на пасеках Сибири, мы столкнулись с некоторыми проблемами при идентификации подвидов пчел и выявлении гибридов, что привело к необходимости разработки алгоритма исследования и выбора наиболее информативных маркеров, позволяющих дифференцировать разные породы, прежде всего среднерусскую пчелу.
Морфометрический метод, с одной стороны, прост и достаточно экономичен, но с другой, — это трудоемкий подход, который предполагает анализ большого числа морфологических признаков, что значительно усложняет и удлиняет процедуру исследования пчелиных семей (6). Чтобы упростить морфометрический анализ, используют геометрическую морфометрию (анализ формы крыла вместо измерения углов и расстояний ячеек) (6, 36, 37), специальные программы для измерений (38, 39), а также подходы, предусматривающие уменьшение числа морфометрических параметров при анализе пчел (27) или использование отдельной ячейки крыла (40). В России для идентификации пород (подвидов) пчел широко применяется морфометрический метод, часто с применением компьютерных программ (41, 42), однако для исключения ошибок в интерпретации результатов требуется верификация полученных данных.
При использовании морфометрического метода также важно учитывать и тот факт, что факторы окружающей среды могут оказывать влияние на изменчивость некоторых морфологических признаков, таких как длина хоботка (9). Наконец, морфометрический метод недостаточно информативен при оценке гибридных семей (43), и необходимо исследование дополнительных маркеров, например локуса мтДНК (27). Следовательно, актуален вопрос как выбора наиболее информативных морфометрических маркеров, так и алгоритма исследования пчел в целом.
В настоящей работе мы показали, что среди морфометрических параметров три показателя крыла (дискоидальное смещение, кубитальный и гантельный индексы) высокоинформативны и достаточны (минимально необходимы) для идентификации подвидов A. mellifera (26, 27). Вместе с тем установлено, что отдельные пчелиные семьи по некоторым параметрам не соответствуют морфометрическим стандартам подвидов медоносной пчелы. Причем такая ситуация наблюдается не только в зонах гибридизации пчел, но и на изолированных пасеках (см. табл. 1). Кроме того, некоторые пчелиные семьи, соответствующие стандарту породы по морфологическим показателям (например, стандарту среднерусской породы), имели происхождение по материнской линии от пород южного происхождения, согласно данным анализа мтДНК (выявлен вариант Q локуса COI-COII), то есть представляют собой «семьи-перевертыши».
Следовательно, использование локуса COI-COII мтДНК в качестве дополнительного маркера позволило нам уточнить данные морфометрического анализа, несмотря на то, что при анализе митохондриального генома оценивается генетический вклад только по линии матки. Алгоритм исследования, примененный при изучении породного состава медоносных пчел, обитающих в Сибири, был следующим: анализ мтДНК (выявление изменчивости локуса COI-COII) для установления происхождения семьи по материнской линии; оценка морфометрических показателей крыла (кубитальный индекс, гантельный индекс, дискоидальное смещение); выявление соответствия данных морфометрии и анализа мтДНК стандарту пород. Таким образом, комплексный подход с применением морфометрического метода и анализа мтДНК позволил нам значительно упростить морфометрический анализ пчел и провести точную идентификацию подвидов и гибридов медоносной пчелы, обитающих на территории Сибири.
На основании данных по изменчивости 31 SSR-маркера была создана база данных по микросателлитным локусам (стандартная аллельная лестница) для среднерусских пчел сибирских популяций, а также проведен поиск молекулярных маркеров, информативных для дифференциации подвидов пчел эволюционных линий М и С. Наиболее перспективными мик-росателлитными локусами, которые могут быть включены в диагностическую ДНК-панель для дифференциации подвида A. m. mellifera (линия М) и двух подвидов южного происхождения A. m. carpathica и A. m. carnica (линия С), оказались А043, Аp081, Аp049, AT139, A024, A113, А088, А028, А008 и mrjp3. В целом, среди изученных микросателлитных локусов можно выделить три класса маркеров, различающихся по информативности для диагностики пород и экотипов медоносной пчелы.
К первому классу относятся локусы, для которых были обнаружены породо-специфичные аллели (например, А043, Аp081, Аp049, AT139, A024, A113, A088, А028, А008 и mrjp3 ). Для этих локусов зарегистрирован преобладающий аллель (аллели) у пчел среднерусской породы A. m. mellifera , тогда как у A. m. carpathica и A. m. carnica он не выявлен или встречался с низкой частотой (см. табл. 2). Например, для локуса А043 аллель размером 128 п.н. преобладал у пчел A. m. mellifera разных популяций Сибири (частота регистрации более 0,83), а также бурзянской популяции (Башкортостан) и большинства популяций Европы (частота встречаемости от 0,68 до 0,90) (11, 12, 21), тогда как для южных подвидов был характерен аллель размером 140 п.н. (частота встречаемости более 0,76). Для mrjp3 также были выявлены разные преобладающие аллели у пчел эволюционных линий М и С: аллель размером 529 п.н. зарегистрирован с высокой частотой (более 0,57) у среднерусских пчел, тогда как у карпатских пчел и пчел подвида карника встречался редко (частота регистрации менее 0,05). Наоборот, аллель размером 406 п.н. преобладал у пчел A. m. carpathica и A. m. carnica (частота регистрации соответственно 0,49 и 0,40) и не был выявлен у среднерусских пчел сибирских популяций . Аналогичная ситуация наблюдалась для локусов Ap081, Ap049, AT139, A024, A113 (см. табл. 2). Кроме того, для некоторых локусов (Ap081, AT139 и др.) выявили различия в составе и частоте доминирующих аллелей у A. m. carpathica и A. m. carnica , что может быть информативным для дифференциации двух южных подвидов.
Во второй класс вошли локусы, для которых были обнаружены эко-специфичные аллели, то есть зарегистрированы различные состав и частота аллелей у пчел A. m. mellifera из разных популяций России и Европы. Значительный интерес представляет микросателлитный локус А008. У среднерусских пчел сибирских популяций аллель размером 162 п.н. встречался с высокой частотой (более чем 0,86). У пчел подвида карника этот аллель также зарегистрирован, но с более низкой частотой (менее 0,32). В связи с этим локус А008 интересен для дифференциации не столько подвидов, сколько экотипов A. m. mellifera : выявлены различия по составу аллелей между пчелами A. m. mellifera разных популяций России и Европы. Так, если для среднерусских пчел Сибири преобладающим был аллель размером 162 п.н., то у A. m. mellifera уральской и европейских популяций доминирующими оказались более короткие аллели размером соответственно 154 и 148 п.н. (11, 12, 21). При этом аллель размером 148 п.н. у среднерусских пчел сибирских популяций вообще не был обнаружен. Поскольку у пчел
-
A. m. mellifera из разных популяций России и Европы выявлен географический градиент размера доминирующего аллеля (148 п.н.—154 п.н.—162 п.н.) в направлении запад-восток, можно предположить, что локус А008 ассоциирован с географическими/экологическими условиями обитания пчел. Проблема генетической специфичности разных подвидов/экотипов пчел к локальным условиям окружающей среды активно обсуждается в научных публикациях (7, 44-46).
Неспецифические локусы (например, AC117, H110, SV185), относящиеся к третьему классу, — это маркеры, для которых был показан сходный состав и близкие частоты аллелей у пчел разного происхождения и/или географической локализации.
В целом, среди микросателлитных локусов, вариабельность которых изучена у пчел сибирских популяций, ряд маркеров может быть использован для установления происхождения подвидов и/или экотипов медоносной пчелы. Несмотря на существующие ограничения при применении ДНК-маркеров ядерного генома (отсутствие или недоступность базы данных и референс-материалов по вариабельности SSR-маркеров у пчел разных популяций/экотипов/подвидов), в некоторых случаях микросателлит-ные локусы высокоинформативны и широко используются при оценке интрогрессии генов одной эволюционной линии в другую и выявлении следов гибридизации (5, 19-21).
Таким образом, в настоящее время не существует универсального метода или диагностического маркера (морфометрического, молекулярного) для идентификации подвидов A. mellifera , но при комплексном применении разных методов морфометрические и ДНК-маркеры дополняют друг друга.
Важность комплексного подхода отмечается во многих работах по систематике и филогении животных. Так, при описании новых видов наездников ( Hymenoptera ) показано, что наряду с морфологическим подходом имеют значение молекулярно-генетические, цитогенетические и другие методы (47). Для идентификации гибридов и оценки интрогрессии или потока генов наиболее информативен комплексный анализ ядерных и митохондриальных маркеров (3, 21).
Вместе с тем выбор надежного» (адекватного) ДНК-маркера для молекулярно-генетических исследований, как и поиск морфологического признака, относится к трудным и слабо разработанным вопросам (48, 49). Хороший молекулярный маркер предполагает наличие достаточного количества информативных сайтов, обладает низкой степенью гомоплазии и относительно равномерной скоростью эволюции в рамках анализируемой группы организмов (50). Если для анализа крупных таксонов, характеризующихся значительной дивергенцией (трибы, семейства), необходим ДНК-маркер с невысокой степенью изменчивости, то при изучении групп низкого таксономического уровня (виды) желательно использовать быстро эволюционирующий маркер с изменчивостью, не достигшей предела насыщения (49). Наконец, перед использованием нового ДНК-локуса в качестве филогенетического маркера необходимо выявить степень его изменчивости и информативности, например на группе организмов с хорошо изученной систематикой и эволюционной историей (50).
Следует подчеркнуть, что различные подходы, такие как морфологический и молекулярно-генетический методы, используемые в систематике и филогении организмов, дополняют друг друга и не являются конкурирующими или взаимоисключающими, тем более молекулярно-генетические методы не заменят морфологическую систему определения видов
-
(51). Оптимальным и наиболее информативным для выделения видов и других таксонов, а также установления границы между видами и описания видового разнообразия служит комплексный подход с привлечением широкого арсенала методов и учетом данных различных дисциплин (сравнительной анатомии, экологии, этологии, популяционной генетики, филогеографии) (52). Особого доверия заслуживают исследования, в которых филогенетическая гипотеза обосновывается анализом нескольких независимо эволюционирующих молекулярных маркеров (52, 53) или предварительно изучена вариабельность молекулярного маркера и показана его информативность и надежность для разрешения филогенетических связей на данном таксономическом уровне (48, 50, 54).
Итак, при исследовании медоносных пчел на территории Сибири нами был использован комплексный подход, включающий анализ морфометрических признаков, маркеров митохондриального и ядерного геномов, и проведена оценка информативности разных методов. Оптимальным алгоритмом исследования, учитывающим различную разрешающую способность, информативность и экономичность используемых методов, можно считать следующий: анализ происхождения семьи по материнской линии с использованием маркеров митохондриального генома; изучение морфометрических признаков и оценка их соответствия данным анализа мтДНК; микросателлитный анализ с целью уточнения породной принадлежности семей, происхождения гибридов, а также выявления генетического разнообразия пчел разных эволюционных линий. Такой подход позволил нам идентифицировать подвиды медоносной пчелы, выявить популяции среднерусской породы Apis mellifera mellifera и охарактеризовать их генетическое разнообразие, оценить генотипический состав пчелиных семей, определить зоны гибридизации пчел. Результаты, полученные в настоящей работе, представляют собой научную основу для генетической паспортизации пчел и селекционно-племенной работы по отбору чистопородных семей с необходимыми биологическими и хозяйственно значимыми признаками, что служит важным условием сохранения и рационального использования аборигенных пород/экотипов пчел.
Список литературы Некоторые проблемы идентификации подвидов медоносной пчелы и их решение на примере изучения Apis mellifera в Сибири
- Bouga M., Alaux C., Bienkowska M., Büchler R., Carreck N.L., Cauia E., Chlebo R., Dahle B., Dall'Olio R., De la Rúa P., Gregorc A., Ivanova E., Kence A., Kence M., Kezic N., Kiprijanovska H., Kozmus P., Kryger P., Le Conte Y., Lodesani M., Murilhas A.M., Siceanu A., Soland G., Uzunov A., Wilde J. A review of methods for discrimination of honey bee populations as applied to European beekeeping. Journal of Apicultural Research,2011, 50(1): 51-84 (doi: 10.3896/IBRA.1.50.1.06).
- Nawrocka A., Kandemir İ., Fuchs S., Tofilski A. Computer software for identification of honey bee subspecies and evolutionary lineages. Apidologie, 2018, 49: 172-184 (doi: 10.1007/s13592-017-0538-y).
- Pinto M.A., Henriques D., Chávez-Galarza J., Kryger P., Garnery L., van der Zee R., Dahle B., Soland-Reckeweg G., De la Rúa P., Dall'Olio R., Carreck N.L., Johnston J.S. Genetic integrity of the Dark European honey bee (Apis mellifera mellifera)from protected populations: a genome-wide assessment using SNPs and mtDNA sequence data. Journal of Apicultural Research,2014, 53(2): 269-278 (doi: 10.3896/IBRA.1.53.2.08).
- Muñoz I., Henriques D., Johnston J.S., Chávez-Galarza J., Kryger P., Pinto M.A. Reduced SNP panels for genetic identification and introgression analysis in the dark honey bee (Apis mellifera mellifera). PLoS ONE, 2015, 10(4): e0124365 (doi: 10.1371/journal.pone.0124365).
- Parejo M., Henriques D., Pinto M.A., Soland-Reckeweg G., Neuditschko M. Empirical comparison of microsatellite and SNP markers to estimate introgression in Apis mellifera mellifera. Journal of Apicultural Research, 2018, 57(4): 504-506 (doi: 10.1080/00218839.2018.1494894).
- Henriques D., Chávez-Galarza J.C., Quaresma A., Neves C.J., Lopes A.R., Costa C., Costa F.O., Rufino J., Pinto M.A. F rom the popular tRNAleu-COX2 intergenic region to the mitogenome: insights from diverse honey bee populations of Europe and North Africa. Apidologie, 2019, 50(2): 215-229 (doi: 10.1007/s13592-019-00632-9).
- Meixner M.D., Pinto M.A., Bouga M., Kryger P., Ivanova E., Fuchs S. Standard methods for characterising subspecies and ecotypes of Apis mellifera. Journal of Apicultural Research, 2013, 52(4): 1-28 (doi: 10.3896/IBRA.1.52.4.05).
- Porrini L.P., Quintana S., Brasesco C., Porrini M.P., Garrido P.M., Eguaras M.J., Müller F., Iriarte P.F. Southern limit of Africanized honey bees in Argentina inferred by mtDNA and wing geometric morphometric analysis. Journal of Apicultural Research, 2020, 59(4): 648-657 (doi: 10.1080/00218839.2019.1681116).
- Алпатов В.В. Породы медоносной пчелы. М., 1948.
- Ruttner F. Biogeography and taxonomy of honey bees. Berlin, Germany, 1988.
- Franck P., Garnery L., Solignac M., Cornuet J.-M. Molecular confirmation of a fourth lineage in honeybees from the Near East. Apidologie, 2000, 31(2): 167-180 (doi: 10.1051/apido:2000114).
- Garnery L., Cornuet J.M., Solignac M.Evolutionary history of the honey bee Apis mellifera inferred from mitochondrial DNA analysis. Molecular Ecology, 1992, 1(3): 145-154 (doi: 10.1111/j.1365-294x.1992.tb00170.x).
- Cornuet J.M., Garnery L., Solignac M. Putative origin and function of the intergenic region between COI and COII of Apis mellifera L. mitochondrial DNA. Genetics, 1991, 128(2): 393-403 (doi: 10.1093/genetics/128.2.393).
- Rortais A., Arnold G., Alburaki M., Legout H., Garnery L. Review of the DraI COI-COII test for the conservation of the black honeybee (Apis mellifera mellifera). Conservation Genetics Resources, 2011, 3(2): 383-391 (doi: 10.1007/s12686-010-9351-x).
- Dall’Olio R., Marino A., Lodesani M., Moritz R.F.A. Genetic characterization of Italian honeybees, Apis mellifera ligustica, based on microsatellite DNA polymorphisms. Apidologie, 2007, 38(2): 207-217 (doi: 10.1051/apido:2006073).
- Cánovas F., de la Rúa P., Serrano J., Galián J.Microsatellite variability reveals beekeeping influences on Iberian honeybee populations. Apidologie, 2011, 42(3): 235-251 (doi: 10.1007/s13592-011-0020-1).
- Ostroverkhova N.V., Kucher A.N., Konusova O.L., Kireeva T.N., Sharakhov I.V. Genetic diversity of honeybees in different geographical regions of Siberia. International Journal of Environmental Studies, 2017, 74(5): 771-781 (doi: 10.1080/00207233.2017.1283945).
- Soland-Reckeweg G., Heckel G., Neumann P., Fluri P., Excoffier L. Gene flow in admixed populations and implications for the conservation of the Western honeybee, Apis mellifera. Journal of Insect Conservation, 2009, 13: 317-328 (doi: 10.1007/s10841-008-9175-0).
- Oleksa A., Chybicki I., Tofilski A., Burczyk J. Nuclear and mitochondrial patterns of introgression into native dark bees (Apis mellifera mellifera) in Poland. Journal of Apicultural Research, 2011, 50(2): 116-129 (doi: 10.3896/IBRA.1.50.2.03).
- Nikolova S. Genetic variability of local Bulgarian honey bees Apis mellifera macedonica (rodopica) based on microsatellite DNA analysis. Journal of Apicultural Science, 2011, 55(2): 117-129.
- Ильясов Р.А., Поскряков А.В., Петухов А.В., Николенко А.Г. Молекулярно-генетический анализ пяти сохранившихся резерватов темной лесной пчелы Apis mellifera mellifera Урала и Поволжья. Генетика, 2016, 52(8): 931-942 (doi: 10.7868/S0016675816060059).
- Зиновьева Н.А., Кривцов Н.И., Форнара М.С., Гладырь Е.А., Бородачев А.В., Березин А.С., Лебедев В.И. Микросателлиты как инструмент для оценки динамики аллелофонда при создании приокского типа среднерусской породы медоносной пчелы Apismellifera. Сельскохозяйственная биология, 2011, 6: 75-79.
- Кривцов Н.И., Зиновьева Н.А., Бородачев А.В., Лебедев В.И., Форнара М.С.Дифференциация основных пород пчел с использованием микросателлитов. Вестник Рязанского государственного агротехнологического университета имени П.А. Костычева, 2011, 4(12): 23-27.
- Hassett J., Browne K.A., McCormack G.P., Moore E., Native Irish Honey Bee Society, Soland G., Geary M. A significant pure population of the dark European honey bee (Apis mellifera mellifera) remains in Ireland. Journal of Apicultural Research, 2018, 57(3): 337-350 (doi: 10.1080/00218839.2018.1433949).
- Whitfield C.W., Behura S.K., Berlocher S.H., Clark A.G., Johnston J.S., Sheppard W.S., Smith D.R., Suarez A.V., Weaver D., Tsutsui N.D. Thrice out of Africa: ancient and recent expansions of the honey bee, Apis mellifera. Science, 2006, 314(5799): 642-645 (doi: 10.1126/science.1132772).
- Островерхова Н.В., Россейкина С.А., Конусова О.Л., Кучер А.Н., Киреева Т.Н. Разнообразие медоносной пчелы Apismellifera L. в Томской области по морфометрическим и молекулярно-генетическим маркерам. Вестник Томского государственного университета. Биология, 2019, 47: 142-173 (doi: 10.17223/19988591/47/8).
- Конусова О.Л., Островерхова Н.В., Кучер А.Н., Курбатский Д.В., Киреева Т.Н. Характеристика морфометрической изменчивости медоносных пчел Apismellifera L., отличающихся вариантами локуса COI-COII мтДНК. Вестник Томского государственного университета. Биология, 2016, 1(33): 62-81 (doi: 10.17223/19988591/33/5).
- Căuia E., Usurelu D., Magdalena L.M., Cimponeriu D., Apostol P., Siceanu A., Holban A., Gavrilă L. Preliminary researches regarding the genetic and morphometric characterization of honeybee (A. mellifera L.) from Romania. Scientific Papers Animal Science and Biotechnologies, 2008, 41(2): 278-286.
- Никоноров Ю.М., Беньковская Г.В., Поскряков А.В., Николенко А.Г., Вахитов В.А. Использование метода ПЦР для контроля чистопородности пчелосемей Apis mellifera mellifera L. в условиях Южного Урала. Генетика, 1998, 34(11): 1574-1577.
- Solignac M., Vautrin D., Loiseau A., Mougel F., Baudry E., Estoup A., Garnery L., Haberl M., Cornuet J.-M. Five hundred and fifty microsatellite markers for the study of the honeybee (Apis mellifera L.) genome. Molecular Ecology Notes, 2003, 3(2): 307-311 (doi: 10.1046/j.1471-8286.2003.00436.x).
- Baitala T.V., Faquinello P., de Toledo V.d.A.A., Mangolin C.A., Martins E.N., Ruvolo-Takasusuki M.C.C. Potential use of major royal jelly proteins (MRJPs) as molecular markers for royal jelly production in Africanized honeybee colonies. Apidologie, 2010, 41: 160-168 (doi: 10.1051/apido/2009069).
- Albert S., Klaudiny J., Šimúth J. Molecular characterization of MRJP3, highly polymorphic protein of honeybee (Apis mellifera) royal jelly. Insect Biochemistry and Molecular Biology, 1999, 29(5): 427-434 (doi: 10.1016/s0965-1748(99)00019-3).
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research — an update. Bioinformatics, 2012, 28(19): 2537-2539 (doi: 10.1093/bioinformatics/bts460).
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945-959 (doi: 10.1093/genetics/155.2.945).
- Островерхова Н.В., Конусова О.Л., Кучер А.Н., Киреева Т.Н., Воротов А.А., Белых Е.А. Генетическое разнообразие локуса COI-COII мтДНК медоносной пчелы Apis mellifera L. в Томской области. Генетика, 2015, 51(1): 89-100 (doi: 10.7868/S0016675815010105).
- Kandemir İ., Özkan A., Fuchs S. Reevaluation of honeybee (Apis mellifera) microtaxonomy: a geometric morphometric approach. Apidologie, 2011, 42(5): 618-627 (doi: 10.1007/s13592-011-0063-3).
- Özkan A.K., Kandemir İ. Comparison of two morphometric methods for discriminating honey bee (Apis mellifera L.) populations in Turkey. Turkish Journal of Zoology, 2013, 37(2): 205-210 (doi: 10.3906/zoo-1104-10).
- Klingenberg C.P. MorphoJ: an integrated software package for geometric morphometrics. Molecular Ecology Resources, 2011, 11(2): 353-357 (doi: 10.1111/j.1755-0998.2010.02924.x).
- Charistos L., Hatjina F., Bouga M., Mladenovic M., Maistros A.D. Morphological discrimination of Greek honey bee populations based on geometric morphometrics analysis of wing shape. Journal of Apicultural Science, 2014, 58(1): 75-84 (doi: 10.2478/JAS-2014-0007).
- Francoy T.M., Prado P.R.R., Gonçalves L.S., Costa L.F., De Jong D. Morphometric differences in a single wing cell can discriminate Apis mellifera racial types. Apidologie, 2006, 37(1): 91-97 (doi: 10.1051/apido:2005062).
- Люто А.А., Иванова О.В., Толстопятов Л.П. Морфологические показатели крыльев пчел Манского и Енисейского районов Красноярского края. Пчеловодство, 2015, 9: 21-22.
- Брандорф А.З., Ивойлова М.М. Популяционная структура медоносных пчел Кировской области. Биомика, 2016, 8(2): 73-75.
- Guzmín-Novoa E., Page R.E.Jr., Fondrk M.K. Morphometric techniques do not detect intermediate and low levels of Africanization in honey bee (Hymenoptera: Apidae) colonies. Annals of the Entomological Society of America, 1994, 87(5): 507-515 (doi: 10.1093/aesa/87.5.507).
- De la Rúa P., Jaffé R., Dall’Olio R., Muñoz I., Serrano J. Biodiversity, conservation and current threats to European honeybees. Apidologie, 2009, 40: 263-284 (doi: 10.1051/apido/2009027).
- Meixner M.D., Büchler R., Costa C., Francis R.M., Hatjina F., Kryger P., Uzunov A., Carreck N.L. Honey bee genotypes and the environment. Journal of Apicultural Research, 2014, 53(2): 183-187 (doi: 10.3896/IBRA.1.53.2.01).
- Hatjina F., Costa C., Büchler R., Uzunov A., Drazic M., Filipi J., Charistos L., Ruottinen L., Andonov S., Meixner M.D., Bienkowska M., Dariusz G., Panasiuk B., Le Conte Y., Wilde J., Berg S., Bouga M., Dyrba W., Kiprijanovska H., Korpela S., Kryger P., Lodesani M., Pechhacker H., Petrov P., Kezic N. Population dynamics of European honey bee genotypes under different environmental conditions. Journal of Apicultural Research, 2014, 53(2): 233-247 (doi: 10.3896/IBRA.1.53.2.05).
- Гохман В.Е. Объем и границы вида у паразитических перепончатокрылых (Hymenoptera): смена парадигмы? Журналобщейбиологии, 2017, 78(5): 37-45.
- Банникова А.А. Молекулярные маркеры и современная филогенетика млекопитающих. Журнал общей биологии, 2004, 65(4): 278-305.
- Абрамсон Н.И. Молекулярные маркеры, филогеография и поиск критерия разграничения видов. Труды Зоологического института РАН,2009, 1: 185-198.
- Тарасов О.В., Журавлева Г.А., Абрамсон Н.И. Оценка возможности применения гена, кодирующего фактор терминации трансляции eRF3, в качестве филогенетического маркера. Молекулярная биология, 2008, 42(6): 937-946.
- Винарский М.В. Судьба категории подвида в зоологической систематике. 2. Современность. Журнал общей биологии, 2015, 76(2): 99-110.
- Синёв С.Ю. О соотношении классических и новейших методов исследования в систематике насекомых. Энтомологическое обозрение, 2011, XC(4): 821-832.
- Лухтанов В.А, Шаповал Н.А. Выявление симпатрично обитающих видов-двойников бабочек из комплекса Agrodiaetus kendevani (Lepidoptera, Lycaenidae) с помощью популяционного анализа несцепленных генетических маркеров. Доклады Академии наук, 2008, 423(3): 421-426.
- Wiens J.J., Kuczynski C.A., Townsend T., Reeder T.W., Mulcahy D.G., Sites J.W.Jr. Combining phylogenomics and fossils in higher-level squamate reptile phylogeny: molecular data change the placement of fossil taxa. Systematic Biology, 2010, 59(6): 674-688 (doi: 10.1093/sysbio/syq048).


