Образование биологически активных пептидов в мясном сырье под влиянием лизатов стартовых бактериальных культур
Автор: Чернуха И.М., Машенцева Н.Г., Вострикова Н.Л., Ковалев Л.И., Ковалева М.А., Афанасьев Д.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Функциональность пищевых продуктов
Статья в выпуске: 6 т.55, 2020 года.
Бесплатный доступ
В настоящее время препараты, основанные на лизатах бактерий, используются главным образом в медицине. В пищевых производствах, в том числе в мясной промышленности при получении функциональных продуктов, бактериальные лизаты пока что мало распространены. Их перспективность для получения функциональных продуктов предсказуема, но эффективность и специфичность действия, которые зависят от особенностей штамма и метода дезинтеграции клеток, требует изучения. У стартовых культур идентифицирован набор пептидаз, в частности эндопептидазы, аминопептидазы, дипептидазы, трипептидазы и пролинспецифические пептидазы, что стимулирует интерес к применению лизатов этих микроорганизмов в пищевых биотехнологиях. В настоящей работе мы показали, что лизаты штаммов Pediococcus pentosaceus 28, Staphylococcus carnosus 108, Lactobacillus curvatus 1, P. acidilactici 38, L. sakei 103, L. sakei 105, L. curvatus 2 и L. acidophilus AT-41, полученные при физическом разрушении клеток, обладают наиболее широкими спектрами ферментов и биологически активных веществ. Нашей целью было определение биохимического состава и ферментативной активности лизатов стартовых бактериальных культур и их роли в образовании биологически активных пептидов в мясном сырье. Рассмотрены два способа получения лизатов из клеток стартовых культур - ферментативный и физический. Первый способ заключается в ферментативной обработке суспензии бактериальных клеток ферментом лизоцимом с последующим отделением экстракта от клеточного дебриса путем центрифугирования, второй - в ультразвуковой обработке. По результатам исследований было выявлено, что наиболее эффективным является физический способ лизиса. Проведена комплексная оценка полученных лизатов - определены протеолитическая, липолитическая и коллагеназная активности, содержание органических кислот, концентрация белка и количество свободных аминокислот. С применением тест-систем API®ZYM определены ферментативные активности лизатов, характерные для использованных видов микроорганизмов. Наиболее широкий спектр внутриклеточных ферментов, а именно лейциновая и валиновая ариламидаза, кислая фосфатаза, нафтол-AS-BI-фосфогидролаза и b-галактозидаза, представлен в лизатах штаммов L. curvatus 2, L. acidophilus AT-41, P. acidilactici 38 и S. carnosus 108. Наибольшую протеолитическую активность выявили в лизатах штаммов S. carnosus 108 (115,94 ед. ПС/мг белка), L. acidophilus AT-41 (66,7 ед. ПС/мг белка), L. curvatus 1 (91,03 ед. ПС/мг белка) и L. curvatus 2 (72,20 ед. ПС/мг белка). Содержание яблочной, молочной и янтарной кислот в лизатах варьировало в пределах соответственно 0,002-0,02; 0,02-0,06 и 0,2-0,9 мг/100 г образца. Наиболее широкий спектр свободных аминокислот представлен в образце лизата из P . acidilactici 38 (13 из 17 исследуемых аминокислот), узкий спектр - в образце лизата из L . sakei 105 (7 из 17 исследуемых аминокислот). Сравнительный анализ полученных двумерных электрофореграмм ферментированного мясного сырья показал как общее действие (снижение количества белкового материала), так и специфичность влияния лизатов на ряд белков в зависимости от используемого лизата стартовой культуры (белки-мишени, в частности актин скелетной мышцы, образование белковых конъюгатов). Полученные данные позволяют рассматривать лизаты стартовых культур как источник ряда ферментов для практического применения в пищевой промышленности (например, для повышения функционально-технологических и биокоррегирующих характеристик мясных продуктов).
Лизаты, стартовые культуры, ферментолиз, биологически активные пептиды, двумерный электрофорез, ief-page, maldi-tof, масс-спектрометрическая идентификация
Короткий адрес: https://sciup.org/142229449
IDR: 142229449 | УДК: 637.03:573.6.086.83.001.26 | DOI: 10.15389/agrobiology.2020.6.1182rus
Текст научной статьи Образование биологически активных пептидов в мясном сырье под влиянием лизатов стартовых бактериальных культур
В современный рацион человека должны входить биологически активные ингредиенты с известными физико-химическими характеристиками, для которых выявлены и научно обоснованы полезные для сохранения и улучшения здоровья свойства, установлена суточная физиологическая потребность. Ежедневное потребление функциональных продуктов питания
∗ При проведении протеомного исследования использовалось оборудование ЦКП ФИЦ Биотехнологии РАН (идентификатор RFMEFI62114Х0002). Исследование профинансировано за счет гранта РНФ (проект ¹ 16-16-10073П).
рассматривается как способ снижения риска заболеваний (1). Многочисленные исследования сосредоточены на производстве биологически активных пептидов в качестве нутрицевтиков и функциональных пищевых ингредиентов из-за их пользы для здоровья. Эти короткие пептиды, проявляющие антигипертензивную, антиоксидантную, минералсвязывающую, иммуномодулирующую и антимикробную активности, латентны в составе первичных последовательностей пищевых белков и высвобождаются в процессе ферментативного протеолиза (2).
При микробной ферментации бактерии синтезируют витамины и минеральные соединения, с участием протеиназ и пептидаз образуют биологически активные пептиды и удаляют некоторые непитательные вещества. Микробную ферментацию рассматривают как один из основных и экономически наиболее пригодных процессов получения биологически активных пептидов (3). Молочнокислые бактерии, обладающие сложными протеолитическими системами, успешно используются в качестве стартовых культур при производстве разнообразных ферментированных мясных продуктов. Более глубокое понимание функциональности протеолитической системы стартовых культур открывает будущие возможности для получения новых пищевых соединений с потенциальными оздоровительными свойствами (4). Ферментированные пищевые продукты сочетают набор полезных для здоровья свойств благодаря антиоксидантной, антимикробной, антимикотической, противовоспалительной, антидиабетической и антиате-росклеротической активности (5).
В последнее время в связи с задачей получения функциональных препаратов и продуктов растет интерес к лизатам клеток микроорганизмов, подвергнутых механическому, химическому или ферментативному разрушению. Клетки состоят из воды, неорганических ионов и углеродсодержащих (органических) молекул. Вода — самая распространенная молекула в клетках, на ее долю приходится от 70 % общей массы клеток. Неорганические ионы клетки, включая натрий (Na+), калий (K+), магний (Mg2+), кальций (Ca2+), фосфат (HPO 4 2 - ), хлорид (Cl - ) и бикарбонат (HCO 3 - ), составляют до 1 % массы клетки. Эти ионы вовлечены в клеточный метаболизм и играют важную роль в функционировании клеток. Однако уникальность живой клетки определяют органические молекулы, большинство которых относятся к одному из четырех классов соединений — к углеводам, липидам, белкам и нуклеиновым кислотам. Белки, нуклеиновые кислоты и большинство углеводов (полисахариды) представляют собой макромолекулы, образующиеся в результате полимеризации низкомолекулярных предшественников — аминокислот, нуклеотидов или простых сахаров. Такие макромолекулы составляют 80-90 % сухой массы большинства клеток. Липиды также входят в число основных компонентов клетки. Остальная часть клеточной массы состоит из множества мелких органических молекул, включая макромолекулярные предшественники (6). Клеточная стенка грампо-ложительных бактерий представляет собой сложную структуру, образованную гликополимерами и белками. Она состоит из окружающих цитоплазматическую мембрану слоев пептидогликана (муреиновый мешок), прошитого в поперечном направлении тейхоевыми кислотами, и содержит полисахариды и белки (7).
Бактерии служат источником различных внутри- и внеклеточных ферментов. Стартовые культуры синтезируют протеазы и пептидазы, гликозидазы, полисахаридные ферменты, малолактические ферменты, эстеразы, уреазы, фенолоксидазы и липазы (8). Протеолитическая система молочнокислых бактерий, используемых в качестве стартовых культур, состоит из протеиназ (расщепляют белки до пептидов), пептидаз (расщепляют образующиеся пептиды на более мелкие пептиды и аминокислоты) и транспортных систем, которые участвуют в клеточном поглощении мелких пептидов и аминокислот. У молочнокислых бактерий идентифицирован широкий спектр пептидаз, в частности эндопептидазы, аминопептидазы, дипептидазы, трипептидазы и пролинспецифические пептидазы (9, 10). Протеолитические системы лактококков и лактобацилл удивительно схожи по компонентам и способу действия, их протеолитическая система состоит из внеклеточной сериновой протеиназы, транспортных систем, специфичных для ди-трипептидов и олигопептидов (> 3 остатков), а также множества внутриклеточных пептидаз (11). Кроме того, важность протеолитических и пептидолитических ферментов молочнокислых бактерий заключается в том, что ряд штаммов, например, Lactobacillus helveticus CP790, L. rhamnosus GG, L. bulgaricus SS1 и L. lactis subsp. cremoris FT4 участвуют в высвобождение биоактивных пептидов (12).
Бактерии могут вырабатывать как промежуточные, так и конечные продукты бактериального метаболизма — молочную кислоту, перекись водорода и бактериоцины, а также метаболиты малых молекул (гистамин, витамины, короткоцепочечные жирные кислоты, полиненасыщенные жирные кислоты, серпины, лактоцепины, секретируемые белки). При этом метаболический потенциал микробов сильно разнится между видами и даже у штаммов одного и того же вида (13).
Таким образом, с химической точки зрения клеточные лизаты — это смесь короткоцепочных пептидов, свободных аминокислот, органических кислот, полисахаридов, витаминов групп В, С, РР, фолиевой кислоты, летучих жирных кислот, пептидогликана клеточных стенок, а также различных ферментов.
Изучение внутриклеточных ферментных систем бактерий требует дезинтеграции клеток (14, 15). Сравнение некоторых общепринятых методов (ультразвуковая обработка, механическое измельчение с помощью стеклянных шариков, замораживание-оттаивание, химический лизис растворами толуола в ацетоне или этаноле) показало, что ультразвуковая обработка и измельчение клеток Bacillus subtilis , Pseudomonas putrefaciens и Streptococcus durans позволили получить больше белка в бесклеточных экстрактах, чем другие методы (16).
В настоящее время препараты, основанные на лизатах бактерий, используются главным образом в медицине. Клинические исследования показали, что бактериальные лизаты при пероральном приеме снижают необходимость в антибиотиках и риск рецидивов респираторных инфекций у детей и взрослых (12). Описано применение этих препаратов для лечения бронхиальной астмы (17, 18). Сообщалось о регенеративном действии бактериальных лизатов резидентной непатогенной микрофлоры родниковой воды Комано (Comano Terme, Trento, Италия) в культуре фибробластов кожи человека in vitro. Бактериальные штаммы, выделенные из этой воды, были охарактеризованы методом геномного секвенирования. Коллекция включала 182 изолята, бактериальные лизаты получали при автоклавировании (121 °С, 20 мин) (19). На модели линии кератиноцитов человека HaCaT in vitro показана реэпитализация поврежденных тканей при использовании растворимой фракции из лизата семи различных пробиотических штаммов, принадлежащих к родам Streptococcus, Lactobacillus и Bifidobacterium (20). В исследовании in vitro выявлено увеличение содержания гиалуроновой кислоты в клетках HaCaT, обработанных лизатами L. plantarum K8 (21). При использовании микрофлюидизированных лизатов Lactobacillus rhamnosus в реконструированном эпидермисе человека Keraskinтм улучшалась барьерная функции кожи (22). Лизаты кишечной палочки и других патогенов успешно применяются при лечении инфекций мочевыводящих путей в случаях, когда наблюдается высокая резистентность бактерий к противомикробным препаратам (23). В интервенционных исследованиях с использованием пробиотиков, пребиотиков и их гидролизованной формы и бактериальных лизатов установлено снижение пищевой сенсибилизации и положительный эффект при аллергических заболеваниях, включая атопический дерматит (24, 25). Лизаты молочнокислых бактерий используются для лечения болезней желудочно-кишечного тракта (26).
Бесклеточные системы экспрессии генов становятся важной платформой для решения широкого спектра задач в синтетической биологии и биотехнологии, включая производство надежных биосенсоров (27).
В пищевых производствах, в частности в мясной промышленности, бактериальные лизаты пока что мало распространены, хотя их перспективность предсказуема, что стимулирует интерес к подобного рода исследованиям. Например, показано, что под воздействием культуры штамма L. plantarum CRL 681 (первоначально выделен из мясных продуктов) в сочетании с его клеточным лизатом происходил протеолиз как саркоплазматических, так и миофибриллярных белков с образованием различных пептидов гидрофобной природы (28). При усилении ферментации субстрат обогащается биологически активными соединениями, которые продуцируют бактерии, ответственные за ферментацию (конъюгированные линолевые кислоты понижают кровяное давление, экзополисахариды проявляют пребиотические свойства, бактериоцины — антимикробные эффекты, сфинголипиды обладают антиканцерогенными и антимикробными свойствами) (29), в дополнение к биоактивным пептидам (БАП), проявляющим антиоксидантное, антимикробное, опиоидное антагонистическое, антиаллергенное и понижающее кровяное давление действие (30).
Ранее мы рассмотрели влияние стартовых культур на образование БАП в мясе и мясных продуктах (31, 32). В развитие этих исследований в настоящей работе мы показали, что лизаты изученных стартовых культур обладают набором ферментативных активностей, в том числе проявляют высокую общую и специфическую протеолитическую активность (наличие белков-мишеней, формирование белковых конъюгатов) с образованием спектров низкомолекулярных пептидов и могут найти практическое применение для повышения функционально-технологических и биокоррегирую-щих характеристик мясных продуктов.
Цель работы заключалась в определении биохимического состава и ферментативной активности лизатов стартовых бактериальных культур и их роли в образовании биологически активных пептидов в мясном сырье.
Ìåòîäèêà . Использовали штаммы Pediococcus pentosaceus 28, Staphylococcus carnosus 108, Lactobacillus curvatus 1, P. acidilactici 38, L. sakei 103, L. sakei 105, L. curvatus 2, L. acidophilus AT-41 (коллекция ФГБОУ ВО Московского государственного университета пищевых производств).
Для получения бактериальной биомассы каждого штамма суспензию клеток (109 КОЕ/мл) вносили в жидкую питательную среду de Man, Rogosa и Sharpe (MRS) из расчета 1 мл суспензии на 10 мл среды. Культивировали в течение 24 ч при 37 ° С (33). Культуральную жидкость помещали в две центрифужные пробирки объемом 40 мл. Клетки осаждали (4000 об/мин, 4 ° С, 15 мин), надосадочную жидкость сливали. Осадок ресуспендировали в 10 мл 100 мМ фосфатного буфера (pH 7), образцы объединяли, центрифугировали (4000 об/мин, 4 ° С, 15 мин), повторно ресуспендировали в 10 мл
100 мМ фосфатного буфера (pH 7) и дополнительно центрифугировали при тех же условиях.
Лизаты получали двумя способами — обработкой клеток лизоцимом и ультразвуковой дезинтеграцией.
При использовании лизоцима после удаления супернатанта (второе центрифугирование) к биомассе приливали 100 мМ фосфатный буфер (pH = 7) с добавлением лизоцима (2,5 мг/мл) и сахарозы (20 мг/мл) до плотности клеточной суспензии 109 КОЕ/мл (по стандарту мутности МакФарланда). Биомассу тщательно ресуспендировали. В стеклянную пробирку объемом 40 мл отбирали 20 мл лизируемой суспензии и помещали в термостат на качалку (для равномерной обработки лизоцимом) на 1 ч при 30 ° С. Затем образцы центрифугировали (4000 об/мин, 4 ° С, 15 мин) для отделения клеточного дебриса от протопластов. Надосадочную жидкость сливали, осадок ресуспендировали в 10 мл 100 мМ фосфатного буфера (pH 7) с сахарозой (5 мг/мл) для создания условий гипотонической среды, приводящей к физическому разрыву протопластов. Образцы тщательно перемешивали в течение 5 мин, центрифугировали (15000 об/мин, 8 мин). Осадок отделяли от супернатанта (лизата).
Для ультразвуковой дезинтеграции клеток после удаления супернатанта (второе центрифугирование) к биомассе добавляли 100 мМ фосфатный буфер (pH 7) до концентрации клеток в суспензии 109 КОЕ/мл (по стандарту мутности Мак-Фарланда). В центрифужную пробирку объемом 40 мл отбирали 20 мл лизируемой суспензии. Пробирку помещали на ледяную баню и далее обрабатывали на ультразвуковом дезинтеграторе Soniprep 150 («MSE», Великобритания) в следующем режиме: обработка — 30 с, охлаждение — 30 с (шесть циклов с перемешиванием каждые два цикла для равномерной дезинтеграции; рабочая длина волны 9 мкм). По завершении ультразвуковой дезинтеграции смесь тщательно перемешивали в течение 5 мин, центрифугировали (15000 об/мин, 8 мин), осадок отделяли от супернатанта (лизата).
Полноту лизиса контролировали методом просвечивающей электронной микроскопии (ПЭМ) (JEM-1400, «Jeol», Япония; рабочее напряжение 80 кВ, длина волны 500 нм). Для этого осадок, полученный после центрифугирования лизируемой суспензии и отделения супернатанта (лизата) ресуспендировали в 0,5 мл 100 мМ фосфатного буфера (pH 7), из аликвот каждой суспензии, разведенных в 100 раз 100 мМ фосфатным буфером, готовили препараты для ПЭМ. Образцы наносили на опорную медную сетку, покрытую формваровой пленкой и укрепленную углеродом, высушивали в течение 15 мин на воздухе, просматривали и фотодокументировали; негативы сканировались и обрабатывались в графическом редакторе.
Концентрацию белка в лизате измеряли по методу Брэдфорд на спектрофотометре BioSpectrometer basic («Eppendorf», Австрия) на основе реакции с Coomassie Brilliant Blue R-250 ( X = 595 нм). В качестве стандарта для построения калибровочной кривой использовали бычий сывороточный альбумин (БСА) (34).
Протеолитическую активность лизатов определяли с помощью модифицированного метода Ансона по ГОСТ 20264.2-88 «Препараты ферментные. Методы определения протеолитической активности (с Изменением ¹ 1)» по количеству тирозина, образующегося при гидролизе субстрата — казеината натрия ( X = 670 нм). При пересчете значений OD использовали калибровочную кривую для серии стандартных растворов с известной концентрацией тирозина. Значения протеолитической активности выражали в единицах протеолитической способности в 1 мг белка (ед.
ПС/мг белка).
Липолитическую активность лизатов измеряли модифицированным методом Ото и Ямады (35) с использованием щелочи для титрования жирных кислот, образовавшихся под действием липазы, и оливкового масла в качестве субстрата. Значения липолитической активности выражали в единицах липолитической способность в 1 мг белка (ед. ЛС/мг белка).
Коллагеназную активность лизатов (36) оценивали методом, основанным на определении содержания оксипролина в смеси образовавшегося в результате гидролиза нативного коллагена, с построением калибровочной кривой для зависимости между концентрацией оксипролина в буферном растворе (в диапазоне 2-20 ммоль/мл; OD 555 ). Значения коллагеназной активности выражали в процентах растворенного коллагена (%рк).
Для измерения ферментативных активностей лизатов использовали тест-системы API®ZYM («BioM é rieux», Франция), позволяющие определять алкалиновую фосфатазу, эстеразу, эстеразную липазу, липазу, лейциновую, валиновую и цистеиновую ариламидазы, трипсин, а -химотрипсин, кислую фосфотазу, нафтол-AS-BI-фосфогидролазу, а -галактозидазу, р -галактози-дазу, p -глюкуронидазу, а -глюкозидазу, р -глюкозидазу, N-ацетил- р -глюкоз-аминидазу, а -маннозидазу и а -фукозидазу.
Аминокислотный состав лизатов исследовали методом, основанным на осаждении белков трихлоруксусной кислотой (ТХУ) с последующим извлечением свободных (несвязанных) аминокислот. Выделенные аминокислоты подвергали дериватизации о-фталевым альдегидом (OPA) и 9-флуоро-метил-хлорформиатом (FMOC). ОРА применяли для определения первичных аминокислот, FMOC — вторичных с последующим анализом методом ВЭЖХ-ДМ и детекцией при длинах волн соответственно 338 и 262 нм (колонка С18 PA, 3,5 µ m½150 mm). Использовали следующие реагенты: деионизированная вода, полученная с помощью системы очистки воды Milli-Q («Merck Millipore», США), ацетонитрил для ВЭЖХ ( > 99,9 %) («Panreac», Франция), метанол для ВЭЖХ («Merck», США), соляная кислота ( > 37 %), ТХУ ( > 99,0 %), FMOC (9-флюоренилметилхлорформиат, 10 мг/мл) («Sigma-Aldrich», США), ОРА (ортофталевый альдегид, 10 мг/мл) («Sigma-Aldrich», США). В качестве стандартов использовали смесь D,L-аминокислот («Merck», США). Состав растворов для градиентного элюирования: элюент А — ацетонитрил: метанол: вода (45:45:10), элюент В — боратный буфер (10 мМ Na 2 HPO 4 , 10 мМ Na 2 B 4 O 7 , pH 8,2). Аминокислоты анализировали по стандартным протоколам на жидкостном хроматографе Agilent 1260 Infinity LC с диодноматричным детектором («Agilent Technologies», США), температура колонки в термостате 40 ° С, рабочее давление 1,6 МПа, скорость потока элюента 1,5 мл/мин, время анализа 25 мин.
Органические кислоты в лизатах определяли методом, основанным на экстракции органических кислот водным раствором ТХУ. Белки из экстрактов, мешающие определению органических кислот, осаждали центрифугированием, пробы анализировали методом ВЭЖХ на Agilent 1260 Infinity LC с УФ-детектором («Agilent Technologies», США) по стандартным протоколам. Использовали анионообменную колонку для ВЭЖХ длиной 50150 мм и диаметром 2,1-4,6 мм, размер частиц 1,8-5,0 мкм. Стандартами служили растворы органических кислот с содержанием основного вещества не менее 99,0 % («Merck», США). Кислоты идентифицировали по абсолютному времени удерживания, массовую долю определяли по площади хроматографического пика анализируемого образца, сопоставляя с пиком образца сравнения с заведомо известной концентрацией. Используемые для определения реагенты и условия проведения анализа аналогичны реагентам и условиям, установленным для исследования аминокислотного состава.
Белковые профили мясного сырья после обработки определяли в мышце longissimus dorsi Bos taurus . Лизат инъецировали в образец мышечной ткани (5 мл на 50 г сырья, соотношение 1:10). Образцы выдерживали в вакуумных контейнерах в течение 48 ч при температуре +4± 1 ° С и до анализа хранили при - 30 ° С в течение 5 сут. Для протеомного исследования обработанного мясного сырья 100 мг измельченного образца гомогенизировали в 2 мл в системе тефлон-стекло в лизирующем растворе (9 М мочевина, 5 % меркаптоэтанола, 2 % Triton Х-100, 2 % амфолинов, рH 3,5-10). Полученный гомогенат осветляли центрифугированием при 800 об/мин в течение 5 мин, надосадочную фракцию, содержащую солюбилизированные белки (экстракт), использовали для фракционирования.
Для протеомного анализа белки разделяли методом двумерного электрофореза (2D) по О’Фарреллу с изоэлектрофокусированием в амфо-линовом полиакриламидном геле (IEF-PAGE), как описано ранее (37, 38). Для визуализации белки на 2D электрофореграммах последовательно окрашивали Coomassie Brilliant Blue R-250 и азотнокислым серебром (39). Молекулярные массы белковых фракций определяли с использованием набора высокоочищенных рекомбинантных белков с молекулярными массами 10-170 кДа PageRuler™ Prestained Protein Ladder (#SM0671 — 10 белков, «Fermentas», США).
При компьютерной денситометрии использовали двумерные электрофореграммы, находившиеся во влажном состоянии. Их полные цифровые изображения и/или изображения отдельных фрагментов получали сканированием (Expression 1680, «Epson», США) (40) (разрешение 300 dpi, 48 bit Color, сохранение результатов в формате * .tiff). Полученные цифровые изображения обрабатывали в графическом редакторе и рассчитывали содержание белков с помощью пакета программ ImageMaster 2D Platinum версий 7 («GE Healthcare», Швейцария). При определении количества белка использовалось не менее трех электрофореграмм с равным нанесением. Разброс значений оптической плотности составлял не более ±1,5 %.
Для идентификации белков вырезанные фрагменты 2D геля измельчали и проводили их трипсинолиз, как описано ранее (41). Наборы пептидов изучали методами MALDI-TOF MS и MS/MS масс-спектрометрии на MALDI-времяпролетном масс-спектрометре Ultraflex («Bruker», Германия) с УФ-лазером (X = 336 нм) в режиме положительных ионов в диапазоне масс 500-8000 Да с калибровкой по известным пикам автолиза трипсина. Для расшифровки масс-спектров (пептидные фингерпринты) применяли традиционные биоинформатические технологии. Масс-спектры триптических пептидов анализировали с помощью программы Mascot, опция Peptide Fingerprint («Matrix Science», США) (точность определения массы МН+ 0,01 %) с использованием базы данных Protein NCBI . При сравнительном анализе протеомных профилей представленных образцов использовали информационные модули Белки скелетной мышцы коров (Bos taurus) базы данных Протеомика мышечных органов .
Статистическую обработку количественных данных проводили с использованием пакета программ STATISTICA 14.0 («StatSoft, Inc.», США). Все измерения выполняли в 3 повторностях. Результаты представлены в виде взвешенного среднего (WAM, weighted arithmetic mean) со стандартным отклонением (±SD). Статистическую достоверность рассчитывали с применением непараметрических U-критерия Манна-Уитни (Mann-Whitney U-test) и H-критерия Краскела-Уоллиса (Kruskal–Wallis H-test). Критический уровень значимости нулевой статистической гипотезы (p) принимали равным 0,05.
Ðåçóëüòàòû . Пептиды, образуемые стартовыми культурами, включают рибосомально синтезируемые бактериоцины и продукты гидролиза белков — биоактивные пептиды, которые могут выполнять функции соответственно природных консервантов и нутрицевтиков. Биоактивные пептиды образуются из субстратных белков под действием внутримембранных протеаз и представляют собой внеклеточные белковые остатки, которые не используются протеолитической системой стартовых культур для ассимиляции азота и выделяются из клетки (11). В некоторых случаях необходим лизис клеток и высвобождение ферментов, участвующих в протеолизе и генерации биоактивных пептидов (42).
Мы лизировали клеточную биомассу стартовых культур двумя способами — ферментативным (обработка лизоцимом) и физическим (ультразвуковая дезинтеграция клеток в суспензии). В обоих случаях клеточный дебрис отделяли центрифугированием. Полученный супернатант представлял собой продукт лизиса клеток стартовых культур.
Э лектронная микро скопия. Микроскопия показала, что лизис клеток лизоцимом менее эффективен, чем ультразвуковая дезинтеграция (рис. 1). При этом клеточная стенка лишь незначительно подвергается разрушению: и у штамма Pediococcus pentosaceus 28, и у штамма Lactobacillus sakei 103 можно наблюдать нарушение целостности поверхностных слоев клеточной стенки (появление шероховатости), а также повышение ее пористости. Обработка клеток ультразвуком дала аналогичный результат, однако заметно, что разрушение происходит равномерно по поверхности клетки, в то время как при обработке клеток лизоцимом — локально.
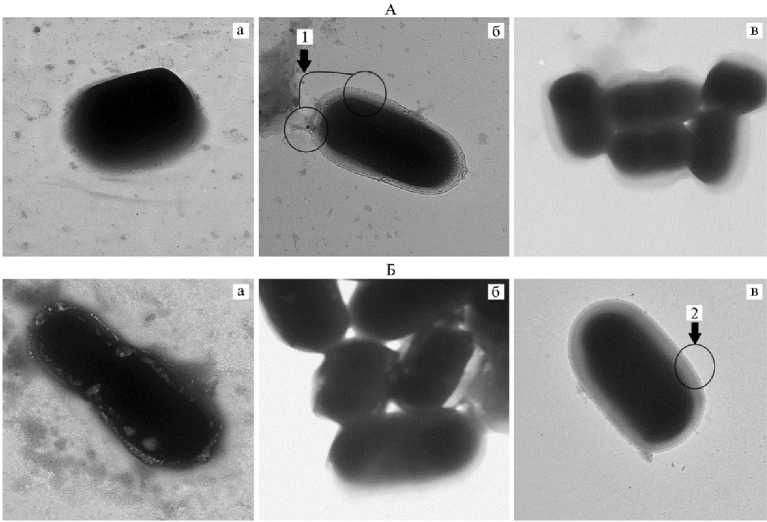
Рис. 1. Контроль полноты лизиса клеток Pediococcus pentosaceus 28 (А) и Lactobacillus sakei 103 при разных способах получения лизатов (½30000) : a — контрольный образец (клетки P . pento-saceus 28 и L . sakei 103 без обработки); б — опытный образец (обработка лизоцимом клеток P . pentosaceus 28 и L . sakei 103); в — опытный образец (ультразвуковая дезинтеграция клеток P . pentosaceus 28 и L . sakei 103); 1 — локальное нарушение целостности поверхностных слоев клеточной стенки (появление шероховатости), 2 — разрушение происходит равномерно по поверхности клетки (просвечивающая электронная микроскопия, JEM-1400, «Jeol», Япония).
Ультразвуковая обработка образцов для ПЭМ сначала проводилась в четыре цикла при рабочей длине волны 4 мкм. По результатам микроскопии было принято решение увеличить число циклов обработки до шести при длине волны 9 мкм. Полученные данные согласуются с результатами исследования F. Tabatabaie и A. Mortazavi (43), где была проведена ультразвуковая дезинтеграция клеток пробиотических бактерий ( Lactobacillus acidophilus , Lactobacillus casei , Lactococcus lactis subsp. cremoris и Lactococus lactis subsp. lactis ). Полученные ими результаты ПЭМ подтвердили разрушение клеточной стенки бактерий после обработки. Авторы отмечали, что характер повреждения зависит от продолжительности обработки. При минимальном времени воздействия на поверхности клетки образуются микротрещины и микропустоты, при более длительном — разрывы клеточной стенки, повышается ее пористость. Дальнейшее увеличение продолжительности дезинтеграции приводит к разрыву клетки и выходу внутриклеточного содержимого в рабочую среду (43).
Содержание белка и спектры ферментативной активности лизатов. По итогам сравнения двух методов получения лизатов в дальнейшем для этих целей мы использовали ультразвуковую обработку. Результаты определения концентрации белка и протеолитической активности образцов представлены в таблице 1.
1. Концентрация белка (мкг/мл) и протеолитическая активность (ед. ПС/мг белка) в лизатах стартовых культур при разных методах обработки клеток ( WAM ±SD)
|
Штамм |
Концентрация белка |
Протеолитическая активность, (ультразвук) |
|
|
лизоцим* 1 |
ультразвук* |
||
|
Staphylococcus carnosus 108 |
0,24±0,012 |
0,99±0,006 |
114,88±2,162 |
|
Lactobacillus acidophilus AT-41 |
0,81±0,015a |
1,94±0,050e |
67,46±0,661 |
|
Lactobacillus curvatus 2 |
0,80±0,020c |
1,79±0,026 |
72,19±0,717 |
|
Lactobacillus curvatus 1 |
0,34±0,006 |
1,28±0,038 |
91,07±0,905 |
|
Lactobacillus sakei 105 |
0,19±0,006b |
1,00±0,041 |
55,77±1,137 |
|
Pediococcus acidilactici 38 |
0,47±0,021 |
1,27±0,035 |
42,17±1,478 |
|
Pediococcus pentosaceus 28 |
0,15±0,015b, d |
0,80±0,036f |
45,01±0,705 |
|
Lactobacillus sakei 103 |
0,23±0,021 |
0,86±0,017f |
51,24±1,372 |
Примечание. ПС — протеолитическая способность. Для анализа использовали объединенную пробу (три повторных определения).
a-b, c-d, e-f Различия между лизатами для варианта обработки (в столбце) статистически значимы при р < 0,05. * Различия между вариантами обработки статистически значимы при р < 0,05.
Содержание белка в образцах биомассы стартовых культур до лизиса было низкое (до 0,05 мкг/мкл), но после обработки лизоцимом составило от 0,17 до 0,82 мкг/мкл, ультразвуком — от 0,79 до 1,95 мкг/мкл. Следовательно, лизис биомассы при ультразвуковой обработке проходит интенсивнее, чем при ферментативной, что соответствует результатам электронной микроскопии. Полученные данные согласуются с результатами исследования I. Mehmeti с соавт. (15), которые также проводили ультразвуковую дезинтеграцию клеток стартовых культур. В их работе концентрации белка в лизатах клеток Lactococcus lactis NIZO 0900 в среднем составляла 1,25±0,02 мкг/мкл, Pediococcus pentosaceus OZF — 1,32±0,01 мкг/мкл.
Наибольшую протеолитическую активность проявляли штаммы Staphylococcus carnosus 108, Lactobacillus acidophilus AT-41, L. curvatus 1 и L. cur-vatus 2. Молочнокислые бактерии, используемые в качестве стартовых культур, считаются слабо протеолитическими. Тем не менее протеолиз — одна из основных ферментативных реакций, происходящих в мясных продуктах под действием микроорганизмов. Молочнокислые бактерии обладают сложной протеолитической системой, которая состоит из трех компонентов: протеаз, связанных с клеточной стенкой, которые инициируют распад белка на олигопептиды, переносчиков пептидов и внутриклеточных пептидаз, которые разлагают пептиды на более короткие пептиды и свободные аминокислоты
(44). Как правило, опубликованные результаты исследований описывают протеолитическую активность лактобактерий непосредственно в среде культивирования (среда MRS, молоко) (45). В работе O.N. Donkor с соавт. (44) трипептидазная активность лактобактерий в среде MRS находилась в диапазоне 200,0-3020,0 Ед/мг белка, дипептидазная — 50,0-1100,0 Ед/мг белка. L. Parra с соавт. (46) оценивали способность целых клеток, бесклеточных экстрактов и клеточных лизатов ускорять протеолиз в творожных суспензиях. Полученные результаты позволили предположить, что разработанная модельная система ускоренного созревания на основе клеточных лизатов индикативна и может применяться для экспресс-оценки вклада штаммов в протеолиз при созревании сыра.
Pediococcus pentosaceus 28


Lactobacillus curvatus 1

К
+ +
± + ‒ +
±
Pediococcus acidilactici 38
Lactobacillus sakei 103

К ‒ ‒ ± ‒ ± ‒ ‒ ‒ ‒ ± + ‒ + ‒ ‒ ‒ ± ‒ ‒
Lactobacillus curvatus

1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
К ± ‒ ‒ ‒ + + ‒ ‒ ‒ + + ‒ + ‒ ‒ + ‒ ‒ ‒
Lactobacillus acidophilus AT-41

123456789 10 11 12 13 14 15 16 17 18 19 20
К ± ‒ ‒ ± + + ‒ ± ‒ + + ‒ + ‒ ‒ ‒ ‒ ‒ ‒
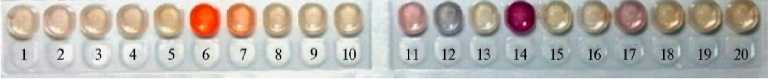
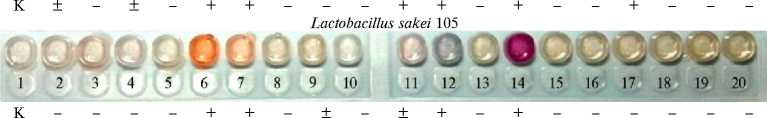
Рис. 2. Ферментативная активность лизатов стартовых культур при ультразвуковой дезинтеграции клеток : 1 — контрольная проба, 2 — алкалиновая фосфатаза, 3 — эстераза, 4 — эстеразная липаза, 5 — липаза, 6 — лейциновая ариламидаза, 7 — валиновая ариламидаза, 8 — цистеиновая ариламидаза, 9 — трипсин, 10 — α -химотрипсин, 11 — кислая фосфотаза, 12 — нафтол-AS-BI-фосфогидролаза, 13 — α -галактозидаза, 14 — β -галактозидаза, 15 — β -глюкуронидаза, 16 — α -глюкозидаза, 17 — β -глюкозидаза, 18 — N-ацетил- β -глюкозаминидаза, 19 — α -манно-зидаза, 20 — α -фукозидаза; «+», « - », «±» — соответственно наличие, отсутствие и слабое проявление активности (скрининг методом стрипов API ® ZYM, «BioM e rieux», Франция).
Способность штаммов молочнокислых бактерий продуцировать биоактивные пептиды основана на особенностях гидролитических реакций участвующих в них белков. Различия, обнаруженные в протеиназах молочнокислых бактерий, объясняют разнообразие образующихся биоактивных пептидов, наблюдаемое даже при действии на один и тот же белковый матрикс. Состав биоактивных пептидов также зависит от субстрата, в котором протекала гидролитическая ферментативная реакция (42).
Для определения ферментативных активностей в лизатах использовались тест-системы API®ZYM с визуальной оценкой наличия активности по цветной реакции (рис. 2) (47). У большинства продуцентов мы обнаружили ферменты нафтол-AS-BI-фосфогидролазу, лейциновую ариламидазу и характерную для многих молочнокислых микроорганизмов β -галактози-дазу (см. рис. 2). Наши данные согласуются с результатами других исследований: известно наличие у молочнокислых бактерий β -глюкозидазы, β -га-лактозидазы (48, 49) и лейциновой ариламидазы (50), что делает их полезными стартовыми культурами в пищевых биотехнологиях.
Скрининг с API®ZYM показал, что у изученных нами штаммов липолитические ферменты либо отсутствуют, либо содержатся в незначительных количествах. Использовав модифицированный метод Ото и Ямады, мы не выявили в лизатах липолитическую активность. У молочнокислых бактерий липолитическая активность обнаруживается редко. Тем не менее показано, что она встречается у Lactobacillus plantarum (51, 52). Эта культура — один из наиболее ценных продуцентов многих ферментов, среди которых выделяют липазу и различные эстеразы (53). Также встречаются упоминания о липазной активности у культуры Pediococcus acidilactici , но у этого вида липаза синтезируется в ограниченных количествах и при определенных способствующих синтезу условиях (54). Есть сообщения о липолитической активности у штаммов видов L. helveticus , L. delbrueckii , L. bulgaricus , L. casei , L. plantarum и L. acidophilus (55).
В лизатах не было обнаружено коллагеназной активности, определяемой по оксипролину, выделившемуся в результате гидролиза нативного коллагена. Возможно, это следствие антиколлагеназной активности штаммов: в литературе имеются подтверждения того, что молочнокислые микроорганизмы обладают такой активностью, в частности среди их метаболитов можно выделить липотейхоевую кислоту, ингибирующую гидролиз коллагена и активирующую его синтез (56-58).
Анализ биохимического состава лизатов. Согласно данным таблицы 2, содержание свободных аминокислот в лизатах представлено следующими диапазонами (мг/100 г образца): аспарагиновая кислота — 0,07-0,23 (исключение — 1,5 мг/100 г у L . sakei 105); серин — 0,16-0,31 (исключение — 8,95 мг/100 г у L . sakei 105); гистидин — 0,8-2,2; аргинин — 0,9-3,5; аланин — 1,7-6,6; валин — 0,18-0,70; фенилаланин — 0,26-0,39; изолейцин — 0,44-0,95; лейцин — 1,00-1,51; лизин — 1,60-2,07; пролин — 0,801,16. Цистин обнаружен только у представителей рода Pediococcus — P . pentosaceus 28 и P . acidilactici 38 (соответственно 0,010 и 0,424 мг/100 г). Наличие метионина также зафиксировано в образцах лизатов P . pentosaceus 28 и P . acidilactici 38 (0,293 и 0,782 мг/100 г), а также у штамма L . sakei 103 (0,072 мг/100 г). Глутаминовая кислота, глицин, треонин и тирозин не детектировались ни в одном образце. Наиболее широкий спектр свободных аминокислот представлен в образце лизата из P . acidilactici 38, наименее – в образце лизата из L . sakei 105. Эти данные согласуются с представленными А.А. Шайхиевым (59) результатами аминокислотного анализа суммарных белков у трех штаммов молочнокислых бактерий в культуральной среде.
-
2. Содержание свободных аминокислот (мг/100 г лизата) у штаммов Lactobacillus , Pediococcus и Staphylococcus при ультразвуковой дезинтеграции клеток ( WAM ±SD)
3. Содержание органических кислот
(мг/100 г образца)
в лизатах штаммов стартовых культур при ультразвуковой дезинтеграции клеток
(
WAM
±SD)
|
Аминокислота |
1 L. acidophilus AT-41г |
P. pentosaceus 28 |
L. curvatus 1 |
P. acidilactici 38 |
^ L. curvatus 2 |
L. sakei 103 1 |
S. carnosus 108 |
1 L. sakei 105 |
|
Аспарагиновая кислота |
0,219±0,004 |
0,214±0,011 |
0,040±0,002 |
0,234±0,012 |
0,228±0,011 |
0,075±0,004 |
0,153±0,008 |
1,500±0,075 |
|
Глутаминовая кислота |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
|
Серин |
0,167±0,008 |
‒ |
‒ |
0,302±0,006 |
0,204±0,010 |
0,165±0,003 |
‒ |
8,950±0,447 |
|
Гистидин |
0,898±0,045 |
1,093±0,055 |
1,298±0,065 |
2,166±0,108 |
1,074±0,054 |
1,106±0,055 |
0,874±0,044 |
‒ |
|
Глицин |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
|
Треонин |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
|
Аргинин |
2,058±0,103 |
2,508±0,125 |
0,966±0,048 |
3,449±0,172 |
1,184±0,024 |
2,516±0,126 |
2,190±0,109 |
‒ |
|
Аланин |
3,260±0,163 |
3,821±0,191 |
1,799±0,090 |
5,139±0,257 |
6,598±0,330 |
3,882±0,194 |
3,235±0,162 |
‒ |
|
Тирозин |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
‒ |
|
Цистин |
‒ |
0,010±0,001 |
‒ |
0,424±0,021 |
‒ |
‒ |
‒ |
‒ |
|
Валин |
0,470±0,024 |
0,460±0,023 |
0,692±0,035 |
0,600±0,030 |
0,527±0,026 |
0,447±0,022 |
0,442±0,022 |
0,185±0,009 |
|
Метионин |
‒ |
0,293±0,015 |
‒ |
0,782±0,039 |
‒ |
0,072±0,004 |
‒ |
‒ |
|
Фенилаланин |
0,336±0,017 |
0,272±0,014 |
0,471±0,024 |
0,385±0,019 |
0,364±0,018 |
0,313±0,016 |
0,279±0,014 |
0,265±0,013 |
|
Изолейцин |
0,452±0,023 |
0,448±0,022 |
0,943±0,047 |
0,930±0,047 |
0,515±0,026 |
0,565±0,028 |
0,575±0,029 |
0,795±0,040 |
|
Лейцин |
1,153±0,058 |
1,143±0,057 |
1,506±0,075 |
1,305±0,065 |
1,300±0,065 |
1,148±0,057 |
1,109±0,055 |
1,014±0,051 |
|
Лизин |
1,505±0,075 |
1,465±0,073 |
0,646±0,032 |
1,659±0,083 |
2,033±0,102 |
2,040±0,102 |
1,652±0,083 |
0,710±0,036 |
|
Пролин |
1,112±0,056 |
0,897±0,045 |
1,160±0,058 |
0,887±0,044 |
0,986±0,049 |
0,966±0,048 |
0,994±0,050 |
‒ |
|
Общее содержание |
11,630±0,582 |
12,624±0,631 |
9,521±0,476 |
18,262±0,913 |
15,013±0,751 |
13,295±0,665 |
11,503±0,575 |
13,419±0,671 |
Примечание. Для анализа использовали объединенную пробу (три повторных определения). Прочерки означают, что указанная аминокислота в лизате не детектируется. * Различия между вариантами обработки статистически значимы при р < 0,05.
У изученных А.А. Шайхиевым штаммов (59) качественный аминокислотный состав оказался идентичен (всего 18 аминокислот). При этом по содержанию лейцина, треонина, фенилаланина, изолейцина, метионина, триптофана, аргинина, глутаминовой и аспарагиновой кислот, а также пролина в культурах эти штаммы молочнокислых бактерий практически не различались.
|
Штамм Яблочная Молочная |
Янтарная Общее содержание |
|
Lactobacillus acidophilus AT-41 0,0038±0,0002 0,0333±0,0017 Pediococcus pentosaceus 28 0,0170±0,0009 0,0436±0,0022 Lactobacillus curvatus 1 0,0048±0,0002 0,0474±0,0024 Pediococcus acidilactici 38 0,0029±0,0001 0,0575±0,0029 Lactobacillus curvatus 2 0,1780±0,0089 0,0345±0,0017 Lactobacillus sakei 103 0,0194±0,0010 0,0375±0,0019 Staphylococcus carnosus 108 0,0076±0,0004 0,0439±0,0022 Lactobacillus sakei 105 0,0640±0,0032 0,0248±0,0012 |
0,6915±0,0346 0,7286±0,0340 0,5417±0,0271 0,6023±0,0301 0,0370±0,0019 0,0892±0,0045 0,2195±0,0110 0,2799±0,0140 0,5251±0,0263 0,7376±0,0370 0,8692±0,0435 0,9261±0,0463 0,8223±0,0412 0,8738±0,0437 0,0089±0,0004 0,0977±0,0049 |
Примечание. Для анализа использовали объединенную пробу (три повторных определения). * Различия между вариантами обработки статистически значимы при р < 0,05.
В среднем содержание яблочной кислоты в лизатах (табл. 3) варьировало в пределах 0,002-0,02; молочной кислоты — 0,02-0,06 и янтарной кислоты — 0,2-0,9 мг/100 г образца. Исключением по содержанию яблочной кислоты стал лизат L. curvatus 2, где содержание этой кислоты было на несколько порядков больше, чем в остальных образцах, — 0,178 мг/100 г. В лизатах L. curvatus 1 и L. sakei 105 содержание янтарной кислоты, напротив, оказалось на несколько порядков меньше, чем в других образцах — соответственно 0,037 и 0,0089 мг/100 г. В литературе имеются данные об исследовании способности молочнокислых бактерий образовывать органические кислоты. Так, после 5 сут жидкого брожения методом ВЭЖХ в культурах выявили ацетат, цитрат, формиат, лактат и сукцинат, при этом лактат и ацетат доминируют среди продуктов брожения (44, 60).
Результаты протеомного исследования. В исследуемых лизатах стартовых культур был выявлен ряд эффектов и специфичность действия на мышечную ткань Bos taurus по сравнению с контролем (рис. 3).
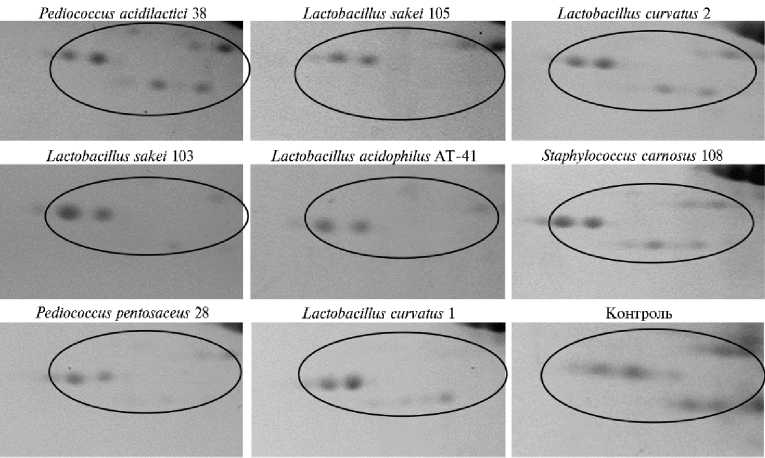
Рис. 3. Фрагменты 2D электрофореграмм (IEF-PAGE) белков мышечной ткани longissimus dorsi
Bos taurus после обработки лизатами стартовых культур бактерий. В овалы заключены зоны фракций медленных скелетномышечных тропонинов Т. Окрашивание Coomassie Brilliant Blue R-250 (линейное связывание красителя с белком).
Сравнительный анализ полученных 2D электрофореграмм показал, что при обработке мясного сырья изученными лизатами стартовых культур количество белка во фракциях уменьшалось с определенной избирательностью. В частности, такой эффект отмечен для некоторых фракций транскрипционных вариантов медленных скелетномышечных тропонинов Т (см. рис. 3) — продуктов гена TNNT1. Положение этих фракций и результаты их идентификации представлены в информационном модуле Белки скелетной мышцы коров (Bos taurus) базы данных Протеомика мышечных органов (табл. 4).
-
4. Результаты масс-спектрометрической идентификации (MALDI-TOF MS и MS/MS) белковых фракций мышечной ткани longissimus dorsi Bos taurus , в которых проявились изменения после обработки лизатами стартовых культур
Белок (ген)
Номер в
Protein NCBI
S/M/C
Мм/pI
эксп.
1 расчет.
Агрегат скелетномышечной регуляторной легкой цепи миозина 2 ( MYL2 )
NP_001069115.1
157/12/82
240,0/5,10
19,0/4,91
Конъюгированная смесь пептидов титина ( TTN )в(1)
DAA32835.1
59/8/< 1
160,0/6,60
3 713 421,0/6,07
Конъюгированные пептиды мышечной креатинфосфокиназы ( CPK-M )а (1)
NP_777198.2
216/7/23
200,0/7,40
42,9/6,63
Конъюгат миоглобина ( MB )а (1)
NP_776306.1
125/11/69
160,0/7,80
17,0/6,90
Фрагмент L-лактатдегидрогеназы А ( LDHA )а (1)+ Acetyl (Protein N-term)
NP_776524.1
76/24/66
27,0/7,50
36,0/8,12
Фрагмент L-лактатдегидрогеназы А ( LDHA )а (1)+ Acetyl (Protein N-term)
XP_005900750.1
127/9/30
26,5/7,50
36,0/8,12
Фрагмент легкой цепи миозина 6В ( MYL6B )
NP_001069181.1
234/26/82
22,0/5,30
23,3/5,40
Скелетномышечный α -актин ( ACTA1 )
NP_001091.1
154/11/38
43,0/5,40
42,0/5,23
Фрагмент а.п. 21-286 скелетномышечного α -актина ( ACTA1 )а (3) + Methyl (75H)
NP_776650.1
413/27/50
28,0/4,90
42,1/5,31
Фрагмент а.п. 21-256 скелетномышечного α -актина ( ACTA1 )а (3) + Methyl (75H)
NP_776650.1
413/37/57
26,0/5,10
42,1/5,31
Фрагмент а.п. 241-375 скелетномышечного α -актина ( ACTA1 )а (3)
NP_776650.1
189/18/
17,0/5,00
42,1/5,31
Примечание. а.п. — аминокислотная последовательность.
S/M/C: Score
— показатель соответствия,
или «счет очков» (Protein scores greater than 68 are significant, p < 0,05); Match peptides — число совпавших пептидов; Coverage — процент покрытия полной аминокислотной последовательности белка выявленными пептидами. Мм/pI (эксп.) — молекулярная масса/изоэлектрическая точка по результатам определения 2D электрофоретической подвижности, Мм/pI (расчет.) — молекулярная масса/изоэлектрическая точка, рассчитанная на основании данных об аминокислотной последовательности с учетом удаления сигнального пептида с помощью программы ExPASy Compute pI/Mw tool. а — msms (указание на подтверждающую идентификацию с помощью тандемной масс-спектрометрии, в скобках указано число секвенированных триптических пептидов). MALDI-времяпролетном масс-спектрометре Ultraflex («Bruker», Германия) с УФ-лазером, режим положительных ионов в диапазоне масс 500-8000 Да.
В исходном мясном сырье обычно присутствуют пять фракций, различающихся по pI и молекулярной массе. Более высокомолекулярный набор содержит три электрофоретические изоформы, а низкомолекулярный включает две изоформы. Обработка лизатами штаммов Lactobacillus sakei 105, L. acidophilus AT-41 и Pediococcus pentosaceus 28 привела к полному исчезновению низкомолекулярных форм, а лизатами L. sakei 103 и L. curva-tus 1 — к явному уменьшению их количества, то есть исследуемые лизаты стартовых культур проявили избирательность действия на актомиозиновый комплект мышечной ткани.
Специфичных изменений в образцах, обработанных лизатами Pedi-ococcus acidilactici 38, L. sakei 103, L. acidophilus AT-41 и Staphylococcus carno-sus 108, мы не выявили. Действие лизата L. curvatus 2 привело к образованию нетипичной фракции с молекулярной массой ∼ 240 кДа (рис. 4, см. табл. 4), идентифицированной как агрегат скелетномышечной регуляторной легкой цепи миозина 2, которая в мономерном виде имеет молекулярную массу 19 кДа. Эта фракция на 2D электрофореграмме присутствует в большом количестве и образует зону с характерной подвижностью, но часть этих молекул под воздействием лизата бактерий перешла в олигомерную форму, которая состоит примерно из 12 субъединиц. В результате обработки лизатом L. curvatus 2 появился конъюгат (∼ 160 кДа), образованный фрагментами белка титина (см. рис. 4, табл. 4). Сам титин имеет молекулярную массу около 4000 кДа и в силу своего размера не входит в пластину PAGE. Триптические пептиды, идентифицированные во фракции 160 кДа, находятся в разных областях молекулы этого белка. Следовательно, фракция 160 кДа не может быть ничем другим как образовавшимся конъюгатом пептидов титина. В целом можно предположить, что в лизате штамма L. curva-tus 2 обнаруживаются специфичные белки и протеазы, влияющие на протеолиз и изменение фолдинга белков.
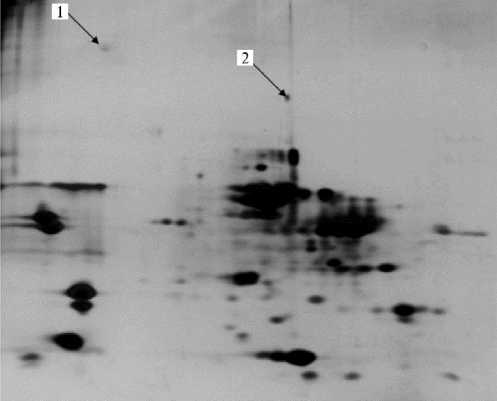
Рис. 4. 2D электрофореграмма (IEF-PAGE) белков мышечной ткани longissimus dorsi Bos taurus после обработки лизатом штамма Lactobacillus curvatus 2: 1 — агрегат скелетномышечной регуляторной легкой цепи миозина 2 ( MYL2 ), 2 — конъюгированная смесь пептидов титина (TTN)в(1). Окрашивание азотнокислым серебром.
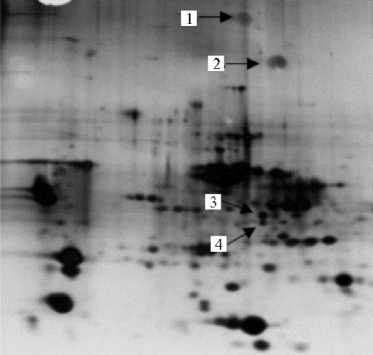
При обработке лизатом L. sakei 105 образовались конъюгаты мышечной креатинфосфокиназы и миоглобина при сохранности мономеров, а также появились фрагменты субъединицы А L-лактатдегидрогеназы, ацетилированные по N-концу (рис. 5, см. табл. 4). Действие лизата L. curvatus 1 привело к появлению крупного фрагмента легкой цепи миозина 6В (см. рис. 5, табл. 4), деаминированного по нескольким остаткам глутамина (Q), что, очевидно, послужило защитным механизмом (судя по тому, что при использовании других лизатов эта фракция полностью разрушалась).
Выраженные изменения в структуре актина мясного сырья наблюдали при использовании лизатов Staphylococcus carnosus 108 и Pediococcus pentosaceus 28 (рис. 6, см. табл. 4). При воздействии ферментного комплекса из Staphylococcus carnosus 108 фракция актина полностью исчезла при сохранении тропомиозинов и легких цепей миозина, а обработка лизатом Pediococcus pentosaceus 28 привела к образованию трех крупных фрагментов актина, два из которых представляли N-концевую часть молекул разной длины, метилированной по аминокислотному остатку 75H. Более низкомолекулярный компонент содержал фрагмент только С-концевой части молекулы, что свидетельствует о наличии у этого вида микроорганизмов специ- ализированного фермента рестрикции, узнающего уникальный участок аминокислотной последовательности актина.
Lactobacillus sakei 105
Lactobacillus curvatus 1

Рис. 5. 2D электрофореграмма (IEF-PAGE) белков мышечной ткани longissimus dorsi Bos taurus после обработки лизатами штаммов Lactobacillus sakei 105 и Lactobacillus curvatus 1:
1 — конъюгированные пептиды мышечной креатинфосфокиназы, 2 — конъюгат миоглобина, 3 и 4 — фрагмент L-лактатдегидрогеназы А, 5 — фрагмент легкой цепи миозина 6В (появившиеся высокомолекулярные олигомеры/конъ-югаты белков и фрагменты L-лактатдегидрогеназы А обозначены стрелками). Окрашивание азотнокислым серебром.
Staphylococcus carnosus 108 Pediococcus pentosaceus 28 Контроль
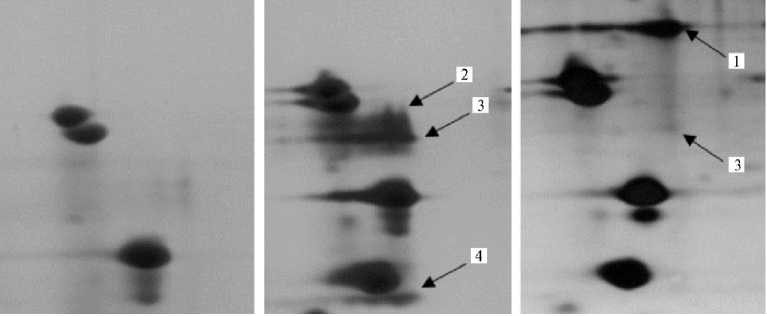
Рис. 6. Фрагменты 2D электрофореграмм (IEF-PAGE) зон актина, тропомиозина и легких цепей миозина, иллюстрирующие действие лизатов на фракцию актина: 1 — скелетномышечный α -актин, 2 — фрагмент аминокислотной последовательности (а.п.) 21-286 скелетномышечного α -актина, 3 — фрагмент а.п. 21-256 скелетномышечного α -актина, 4 — фрагмент а.п. 241-375 скелетномышечного α -актина. Окрашивание азотнокислым серебром.
В целом можно заключить, что нами выявлены специфические белки — мишени ферментных препаратов из разных стартовых культур.
Ранее мы изучили действие цельных стартовых культур на мышечную ткань крупного рогатого скота и мясную продукцию и обнаружили ряд изменений в белковом составе (28), в частности образование агрегатов миоглобина и тропонина I, особенно при применении культуры Pediococcus pentosaceus 31. Отметим, что в раннем исследовании мы анализировали изменения, происходящие под действием живых стартовых культур, постоянно продуцирующих наборы специфичных ферментов. В представленном исследовании использовались ферменты, высвободившиеся из клеток при лизисе с применением ультразвуковой дезинтеграции. Очевидно, что в препаратах клеточных лизатов эти ферменты имеют определенное время жизни и явно меньшую концентрацию. В таком случае эффект, естественно, менее выражен, что и наблюдалось в нашем эксперименте, но общие механизмы подтвердились.
В настоящей работе под воздействием лизатов стартовых культур происходили выраженные изменения белков мясного сырья, в том числе с продукцией низкомолекулярных пептидов, которые могут обладать биологической активность. При ферментации белков животного происхождения протеолитическими ферментами, секретируемыми Lactobacillus helveticus , L. lactis subsp. ñremoris FT4 и L. delbrueckii subsp. bulgaricus SS1, образуются биологически активные пептиды, включая гипотензивные пептиды, которые ингибируют ангиотензин I-превращающий фермент (АПФ), опиоидные агонисты и антагонисты пептидов, а также минералосвязывающие, иммуномодулирующие, антибактериальные и антитромботические пептиды. Ангиотензин I-преобразующий фермент регулирует кровяное давление через синтез вазопрессора ангиотензина II из ангиотензина I (44). Появление биоактивных пептидов зависит от ферментативной активности культуры, факторы, влияющие на их продукцию, специфичны для штамма. На молекулярном уровне на проявление активности протеолитической системы влияет наличие белков, аминокислот и углерода (61, 62). Кроме того, ферментативная активность зависит от фазы роста микроорганизмов; активность сохраняется в течение экспоненциальной фазы и в начальный период стационарной фазы, но уменьшается по мере прогрессирования стационарной фазы (63); изменения ферментативной активности также связаны с целостностью бактериальных клеток.
На сегодняшний день потенциал получения биоактивных пептидов с участием стартовых культур можно предсказать двумя способами: с помощью геномного анализа компонентов протеолитической системы стартовых культур для выявления различных ферментативных активностей или субэлементов и их стратегических особенностей (64) и на основе подхода in silico с использованием методов оценки количественного соотношения структура—активность (QSAR) для анализа белкового матрикса, который может высвобождать биоактивные пептиды (65). Для прогноза по большой выборке штаммов стартовых культур наиболее эффективно объединение биоинформатических инструментов с экспериментальными данными (42).
Таким образом, штаммы Pediococcus pentosaceus 28, Staphylococcus car-nosus 108, Lactobacillus curvatus 1, P. acidilactici 38, L. sakei 103, L. sakei 105, L. curvatus 2 и L. acidophilus AT-41, выбраненные нами для получения лизатов, — это активные продуценты многих ферментов, они широко применяются в пищевой промышленности как стартовые культуры. Штаммы подобраны таким образом, чтобы результаты по лизатам можно было сравнить с полученными ранее по действию соответствующих живых культур на мясное сырье и готовые мясные продукты. Для этих штаммов разработана полная схема получения бактериальных лизатов методом ультразвуковой дезинтеграции.
Итак, самым широким набором внутриклеточных ферментов, включая лейциновую и валиновую ариламидазу, кислую фосфотазу, нафтол-AS-BI-фосфогидролазу и β-галактозидазу, обладают лизаты Lactobacillus curva-tus 2, L. acidophilus AT-41, Pediococcus acidilactici 38 и Staphylococcus carnosus 108. Для лизатов L. acidophilusAT-41, L. curvatus 1 и L. curvatus 2 характерно наличие алкалиновой фосфорилазы. В лизате L. acidophilus AT-41 обнаруживаются следы липазы, а также трипсин, в лизатах Staphylococcus carnosus 108 и Pediococcus acidilactici 38 — следы эстеразной липазы. Во всех образцах, кроме лизата L. acidophilus AT-41, присутствует β-глюкозидаза. Наибольшую протеолитическую активность имели лизаты Staphylococcus carnosus 108 (115,94 ед. ПС/мг белка), L. acidophilus AT-41 (66,7 ед. ПС/мг белка), L. curva-tus 1 (91,03 ед. ПС/мг белка) и L. curvatus 2 (72,20 ед. ПС/мг белка). При ферментации мясного сырья лизатами стартовых культур выявлено образование нестандартных белковых конъюгатов, для некоторых культур определены специфические белки-мишени, в частности актин скелетной мышцы. Полученные данные будут использованы в технологиях обработки мышечной ткани животных для повышения функциональности продуктов. Продолжением и дополнением экспериментальных исследований должен быть геномный анализ компонентов протеолитической системы молочнокислых бактерий для выявления различных ферментативных активностей.
Список литературы Образование биологически активных пептидов в мясном сырье под влиянием лизатов стартовых бактериальных культур
- Чернуха И.М., Котенкова Е.А., Василевская Е.Р., Иванкин А.Н., Лисицын А.Б., Федулова Л.В. Изучение биологических эффектов ягод годжи разного географического происхождения на крысах с алиментарной гиперхолестеринемией. Вопросы питания, 2020, 89(1): 37-45 (doi: 10.24411/0042-8833-2020-10004).
- Vostrikova N.L., Chernukha I.M. Identification of tissue-specific proteins and peptides forming innovative meat products corrective properties to confirm authenticity of meat raw materials. Foods and Raw Materials, 2018, 6(1): 201-209 (doi: 10.21603/2308-4057-2018-1-201-209).
- Marco M.L., Heeney D., Binda S., Cifelli C.J., Cotter P.D., Foligne B., Ganzle M., Kort R., Pasin G., Pihlanto A., Smid E.J., Hutkins R. Health benefits of fermented foods: microbiota and beyond. Current Opinion in Biotechnology, 2017, 44: 94-102 (doi: 10.1016/j.copbio.2016.11.010).
- Al Alwan I., Khadora M., Amir L., Nasrat G., Omair A., Brown L., Al Dubayee M., Badri M. Turner syndrome genotype and phenotype and their effect on presenting features and timing of diagnosis. International Journal of Health Sciences, 2014, 8(2): 195-202 (doi: 10.12816/0006086).
- §anlier N., Gokcen B.B., Sezgin A.C. Health benefits of fermented foods. Review. Critical Reviews in Food Science and Nutrition, 2019, 59(3): 506-527 (doi: 10.1080/10408398.2017.1383355).
- Cooper G.M. The cell: a molecular approach. Sinauer Associates, Sunderland, Massachusetts, 2000.
- Vinogradov E., Sadovskaya I., Grard T., Murphy J., Mahony J., Chapot-Chartier M.-P., van Sinderen D. Structural studies of the cell wall polysaccharide from Lactococcus lactis UC509.9. Carbohydrate Research, 2018, 461: 25-31 (doi: 10.1016/j.carres.2018.03.011).
- Matthews A., Grimaldi A., Walker M., Bartowsky E., Grbin P.R., Jiranek V. Lactic acid bacteria as a potential source of enzymes for use in vinification. Applied and Environmental Microbiology, 2004, 70(10): 5715-5731 (doi: 10.1128/AEM.70.10.5715-5731.2004).
- Kunji E.R., Mierau I., Hagting A., Poolman B., Konings W.N. The proteolytic systems of lactic acid bacteria. Review. Antonie Van Leeuwenhoek, 1996, 70(2-4): 187-221 (doi: 10.1007/bf00395933).
- Law J., Haandrikman A. Proteolytic enzymes of lactic acid bacteria. International Dairy Journal, 1997, 7(1): 1-11 (doi: 10.1016/0958-6946(95)00073-9).
- Kunji E.R., Mierau I., Poolman B., Konings W.N., Venema G., Kok J. Fate of peptides in peptidase mutants of Lactococcus lactis. Molecular Microbiology, 1995, 21(1): 123-131 (doi: 10.1046/j.1365-2958.1996.6231339.x).
- Savijoki K., Ingmer H., Varmanen P. Proteolytic systems of lactic acid bacteria. Review. Applied Microbiology and Biotechnology, 2006, 71(4): 394-406 (doi: 10.1007/s00253-006-0427-1).
- Jurkiewicz D., Zielnik-Jurkiewicz B. Bacterial lysates in the prevention of respiratory tract infections. Review. Polish Journal of Otolaryngology, 2018, 72(5): 1-8 (doi: 10.5604/01.3001.0012.7216).
- Hugo W.B. The mode of action of antibacterial agents. The Journal of Applied Bacteriology, 1967, 30(1): 17-50 (doi: 10.1111/j.1365-2672.1967.tb00273.x).
- Mehmeti I., Kiran F., Osmanagaoglu O. Comparison of three methods for determination of protein concentration in lactic acid bacteria for proteomics studies. African Journal of Biotechnology, 2011, 10(11): 2178-2185 (doi: 10.5897/AJB10.1881).
- Vadehra D.V., Wallace D.L., Harmon L.G. Comparison of methods of extracting intracellular proteases from bacteria. Applied Microbiology, 1965, 13(6): 1010-1013.
- Kearney S.C., Dziekiewicz M., Feleszko W. Immunoregulatory and immunostimulatory responses of bacterial lysates in respiratory infections and asthma. Review. Ann. Allergy Asthma Immunol., 2015, 114(5): 364-369 (doi: 10.1016/j.anai.2015.02.008).
- Lei Y., Kuang S.-J., Liao C.-S. Effects of bacterial lysates and all trans-retinoic acid on airway inflammation in asthmatic mice. Chinese Journal of Contemporary Pediatrics, 2018, 20(6): 514518 (doi: 10.7499/j.issn.1008-8830.2018.06.016) (Chinese).
- Nicoletti G., Saler M., Tresoldi M.M., Faga A., Benedet M., Cristofolini M. Regenerative effects of spring water-derived bacterial lysates on human skin fibroblast in in vitro culture: preliminary results. Journal of International Medical Research, 2019, 47(11): 5777-5786 (doi: 10.1177/0300060519880371).
- Lombardi F., Palumbo P., Mattei A., Augello F.R., Cifone M.G., Giuliani M., Cinque B. Soluble fraction from lysates of selected probiotic strains differently influences re-epithelialization of HaCaT scratched monolayer through a mechanism involving nitric oxide synthase 2. Biomol-ecules, 2019, 9(12): 756 (doi: 10.3390/biom9120756).
- Kim H., Kim H.R., Jeong B.J., Lee S.S., Kim T.-R., Jeong J.H., Lee M., Lee S., Lee J.S., Chung D.K. Effects of oral intake of kimchi-derived lactobacillus plantarum k8 lysates on skin moisturizing. Journal of Microbiology and Biotechnology, 2015, 25(1): 74-80 (doi: 10.4014/jmb.1407.07078).
- Jung Y.-O., Jeong H., Cho Y., Lee E.-O., Jang H.-W., Kim J., Nam K., Lim K.-M. Lysates of a probiotic, Lactobacillus rhamnosus, can improve skin barrier function in a reconstructed human epidermis model. International Journal of Molecular Science, 2019, 20(17): 4289 (doi: 10.3390/ijms20174289).
- Ahumada-Cota R.E., Hernandez-Chicas U., Milián-Suazo F., Chávez-Berrocal M.E., Navarro-Ocaca A., Martínez-Gómez D., Patiño-López G., Salazar-Jiménez E.P., Eslava C.A. Effect and analysis of bacterial lysates for the treatment of recurrent urinary tract infections in adults. Pathogens, 2020, 9(2): E102 (doi: 10.3390/pathogens9020102).
- Pfefferle P.I., Prescott S.L., Kopp M. Microbial influence on tolerance and opportunities for intervention with prebiotics/probiotics and bacterial lysates. Review. The Journal of Allergy and Clinical Immunology, 2013, 131(6): 1453-1463 (doi: 10.1016/j.jaci.2013.03.020).
- Lau S. Bacterial lysates in food allergy prevention. Review. Current Opinion in Allergy and Clinical Immunology, 2013, 13(3): 293-295 (doi: 10.1097/ACI.0b013e328360ede9).
- Roberts J.D., Suckling C.A., Peedle G.Y, Murphy J.A., Dawkins T.G., Roberts M.G. An exploratory investigation of endotoxin levels in novice long distance triathletes, and the effects of a multi-strain probiotic/prebiotic, antioxidant intervention. Nutrients, 2016, 8(11): 733 (doi: 10.3390/nu8110733).
- Didovyk A., Tonooka T., Tsimring L., Hasty J. Rapid and scalable preparation of bacterial lysates for cell-free gene expression. ACS Synth. Biol, 2017, 6(12): 2198-2208 (doi: 10.1021/acssyn-bio.7b00253).
- Fadda S., Sanz Y., Vignolo G., Aristoy M.C., Oliver G., Toldra F. Characterization of muscle sarcoplasmic and myofibrillar protein hydrolysis caused by Lactobacillus plantarum. Applied and Environmental Microbiology, 1999, 65(8): 3540-3546.
- Przybylski R., Firdaous L., Chataigné G., Dhulster P., Nedjar N. Production of an antimicrobial peptide derived from slaughterhouse byproduct and its potential application on meat as preservative. Food Chemistry, 2016, 211: 306-313 (doi: 10.1016/j.foodchem.2016.05.074).
- Чернуха И.М., Машенцева Н.Г., Афанасьев Д.А., Вострикова Н.Л. Биологически активные пептиды мяса и мясных продуктов: обзор. Часть 2. Функциональность мясных биоактивных пептидов. Теория и практика переработки мяса, 2020, 5(12): 12-19.
- Афанасьев Д.А., Машенцева Н.Г., Чернуха И.М. Биологически активные пептиды как продукт микробной ферментации мясного сырья и готовых мясных продуктов. Пищевая промышленность, 2019, 4: 20-22 (doi: 10.24411/0235-2486-2019-10009).
- Афанасьев Д.А., Клабукова Д.Л., Машенцева Н.Г., Ахремко А.Г., Куликовский А.В., Чернуха И.М. Влияние стартовых культур на технологические и биохимические показатели сырокопченых колбас. Мясная индустрия, 2016, 12: 18-22.
- de Man J.C., Rogosa M., Sharpe M.E. A medium for the cultivation of Lactobacillus. Journal of Applied Bacteriology, 1960, 23: 130-135 (doi: 10.1111/j.1365-2672.1960.tb00188.x).
- M®hre H.K., Dalheim L., Edvinsen G.K., Elvevoll E.O., Jensen I.-J. Protein determination — method matters. Foods, 2018, 7(1): 5 (doi: 10.3390/foods7010005).
- Полыгалина Г.В., Чередниченко B.C., Римарева Л.В. Определение активности ферментов. М., 2003. Ryhanen L., Rantala-Ryhanen S., Tan E.M.L., Uitto J. Assay of collagenase activity by a rapid, sensitive, and specific method. Collagen and Related Research, 1982, 2(2): 117-130 (doi: 10.1016/S0174- 173X(82)80028-9).
- Чернуха И.М., Машенцева Н.Г., Вострикова Н.Л., Ковалев Л.И., Ковалева М.А., Афанасьев Д.А., Бажаев А.А. Образование биологически активных пептидов в мясном сырье под влиянием протеаз различного происхождения. Сельскохозяйственная биология, 2018, 53(6): 1247-1261 (doi: 10.15389/agrobiology.2018.6.1247rus).
- Ковалев Л.И., Шишкин С.С., Ковалева М.А., Иванов А.В., Вострикова Н.Л., Чернуха И.М. Протеомное изучение белков в образцах свинины и выработанных из нее мясных продуктах. Всё о мясе, 2013, 3: 32-34.
- Kovalyov L.I., Kovalyova M.A., Kovalyov P.L., Serebryakova M.V., Moshkovskii S.A., Shish-kin S.S. Polymorphism of delta 3,5-delta2,4-dienoyl-coenzyme A isomerase (the ECH1 gene product protein) in human striated muscle tissue. Biochemistry, 2006, 71(4): 448-453 (doi: 10.1134/s0006297906040146).
- Shishkin S.S., Kovalev L.I., Kovaleva M.A., Krakhmaleva I.N., Lisitskaya K.V., Eremina L.S., Ivanov A.V., Gerasimov E.V., Sadykhov E.G., Ulasova N.Yu., Sokolova O.S., Toropygin I.Yu., Popov V.O. Database. Proteomics of prostate cancer. Acta Naturae, 2010, 2(4): 95-104 (doi: 10.32607/20758251-2010-2-4-95-104).
- Zvereva E.A., Kovalev L.I., Ivanov A.V., Kovaleva M.A., Zherdev A.V., Shishkin S.S., Lisitsyn A.B., Chernukha I.M., Dzantiev B.B. Enzyme immunoassay and proteomic characterization of troponin I as a marker of mammalian muscle compounds in raw meat and some meat products. Meat Science, 2015, 105: 46-52 (doi: 10.1016/j.meatsci.2015.03.001).
- Venegas-Ortega M.G., Flores-Gallegos A.C., Martínez-Hernández J.L., Aguilar C.N., Nevárez-Moorillón G.V. Production of bioactive peptides from lactic acid bacteria: a sustainable approach for healthier foods. Comprehensive Reviews in Food Science and Food Safety, 2019, 18(4): 10391051 (doi: 10.1111/1541-4337.12455).
- Tabatabaie F., Mortazavi A. Studying the effects of ultrasound shock on cell wall permeability and survival of some LAB in milk. World Journal of Applied Science, 2008, 3(1): 119-121.
- Donkor O.N., Henriksson A., Vasiljevic T., Shah N.P. Proteolytic activity of dairy lactic acid bacteria and probiotics as determinant of growth and in vitro angiotensin-converting enzyme inhibitory activity in fermented milk. Lait, 2007, 87(1): 21-38 (doi: 10.1051/lait:2006023).
- García-Cano I., Rocha-Mendoza D., Ortega-Anaya J. Lactic acid bacteria isolated from dairy products as potential producers of lipolytic, proteolytic and antibacterial proteins. Applied Microbiology and Biotechnology, 2019, 103: 5243-5257 (doi: 10.1007/s00253-019-09844-6).
- Parra L., Requena T., Casal V., Gymez R. Proteolytic activity of lactobacilli in a model goats' milk curd system. Letters in Applied Microbiology, 1996, 23(6): 375-378 (doi: 10.1111/j. 1472-765x.1996.tb01340.x).
- Bascomb S., Manafi M. Use of enzyme tests in characterization and identification of aerobic and facultatively anaerobic gram-positive cocci. Clinical Microbiology Reviews, 1998, 11(2): 318-340.
- Son S.-H., Jeon H.-I., Yang S.-J., Sim M.-H., Kim Y.-J., Lee N.-K., Paik H.-D. Probiotic lactic acid bacteria isolated from traditional Korean fermented foods based on p-glucosidase activity. Food Science and Biotechnology, 2018, 27(1): 123-129 (doi: 10.1007/s10068-017-0212-1).
- Colombo M., Castilho N.P.A., Todorov S.D., Nero L.A. Beneficial properties of lactic acid bacteria naturally present in dairy production. BMC Microbiology, 2018, 18: 219 (doi: 10.1186/s12866-018-1356-8).
- Kivanij M., Yilmaz M., Qakir E. Isolation and identification of lactic acid bacteria from boza, and their microbial activity against several reporter strains. Turkish Journal of Biology, 2011, 35: 313-324 (doi: 10.3906/biy-0906-67).
- Kim S., Huang E., Park S., Holzapfel W., Lim S.D. Physiological characteristics and anti-obesity effect of Lactobacillus plantarum K10. Korean Journal for Food Science of Animal Resources, 2018, 38(3): 554-569 (doi: 10.5851/kosfa.2018.38.3.554).
- Esteban-Torres M., Mancheco J.M., de las Rivas B., Mucoz R. Production and characterization of a tributyrin esterase from Lactobacillus plantarum suitable for cheese lipolysis. Journal of Dairy Science, 2014, 97(11): 6737-6744 (doi: 10.3168/jds.2014-8234).
- Esteban-Torres M., Reverón I., Santamaría L., Mancheco J.M., de Las Rivas B., Muñoz R. The Lp_3561 and Lp_3562 enzymes support a functional divergence process in the lipase/esterase toolkit from Lactobacillus plantarum. Frontiers in Microbiology, 2016, 19(7): 1118 (doi: 10.3389/fmicb.2016.01118).
- Ramakrishnan V., Goveas L.C., Narayan B., Halami P.M. Comparison of lipase production by Enterococcus faecium MTCC 5695 and Pediococcus acidilactici MTCC 11361 using fish waste as substrate: optimization of culture conditions by response surface methodology. International Scholarly Research Notices, 2013, 2013: 980562 (doi: 10.5402/2013/980562).
- El Soda M., Abd El Wahab H., Ezzat N., Desmazeaud M.J., Ismail A. The esterolytic and lipolytic activities of the Lactobacilli. II. Detection of the esterase system of Lactobacillus helvet-icus, Lactobacillus bulgaricus, Lactobacillus lactis and Lactobacillus acidophilus. Lait, 1986, 66: 431-443 (doi: 10.1051/lait:1986428).
- Kim H.M., Lee D.E., Park S.D., Kim Y.T., Kim Y.J., Jeong J.W., Jang S.S., Ahn Y.T., Sim J.H., Huh C.S., Chung D.K., Lee J.H. Oral administration of Lactobacillus plantarum HY7714 protects hairless mouse against ultraviolet B-induced photoaging. The Journal of Microbiology and Biotechnology, 2014, 24(11): 1583-1591 (doi: 10.4014/jmb.1406.06038).
- Hong Y.F., Lee H.Y., Jung B.J., Jang S., Chung D.K., Kim H. Lipoteichoic acid isolated from Lactobacillus plantarum down-regulates UV-induced MMP-1 expression and up-regulates type I procollagen through the inhibition of reactive oxygen species generation. Molecular Immunology, 2015, 67(2, Part B): 248-255 (doi: 10.1016/j.molimm.2015.05.019).
- Shirzad M., Hamedi J., Motevaseli E., Modarressi M.H. Anti-elastase and anti-collagenase potential of Lactobacilli exopolysaccharides on human fibroblast. Artificial Cells, Nanomedicine, and Biotechnology, 2018, 46: 1051-1061 (doi: 10.1080/21691401.2018.1443274)
- Шайхиев А.А. Аминокислотный состав молочнокислых бактерий. Вопросы питания, 1977, 2: 63-65.
- Nuryana I., Andriani A., Lisdiyanti P., Yopi. Analysis of organic acids produced by lactic acid bacteria. IOP Conference Series Earth and Environmental Science, 2019, 251(1): 012054 (doi: 10.1088/1755-1315/251/1/012054).
- Guedon E., Renault P., Ehrlich S.D., Delorme C. Transcriptional pattern of genes coding for the proteolytic system of Lactococcus lactis and evidence for coordinated regulation of key enzymes by peptide supply. Journal of Bacteriology, 2001, 183(12): 3614-3622 (doi: 10.1128/JB.183.12.3614-3622.2001).
- Gitton C., Meyrand M., Wang J. Proteomic signature of Lactococcus lactis NCDO763 cultivated in milk. Applied and Environmental Microbiology, 2005, 71(11): 7152-7163 (doi: 10.1128/AEM.71.11.7152-7163.2005).
- Williams A.G., Noble A.J., Tammam J., Lloyd D., Banks J.M. Factors affecting the activity of enzymes involved in peptide and amino acid catabolism in non-starter lactic acid bacteria isolated from Cheddar cheese. International Daily Journal, 2002, 12(10): 841-852 (doi: 10.1016/S0958-6946(02)00063-8).
- Liu M., Renckens B., Bayjanov J.R., Nauta A. The proteolytic system of lactic acid bacteria revisited: A genomic comparison. BMC Genomics, 2010, 11(1): 36 (doi: 10.1186/1471-2164-11-36).
- Gu Y., Majumder K., Wu J. QSAR-aided in silico approach in evaluation of food proteins as precursors of ACE inhibitory peptides. Food Research International, 2011, 44(8): 2465-2474) (doi: 10.1016/j.foodres.2011.01.051).


