Оценка генетических взаимосвязей пород крупного рогатого скота черно-пестрого корня с предковыми популяциями на основе полногеномного SNP-генотипирования современных и музейных образцов
Автор: Абдельманова А.С., Харзинова В.Р., Форнара М.С., Чинаров Р.Ю., Боронецкая О.И., Сермягин А.А., Трухачев В.И., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 4 т.59, 2024 года.
Бесплатный доступ
Холмогорская и ярославская породы крупного рогатого скота наиболее распространены среди российских локальных пород. Однако история их возникновения и развития до сих пор остается предметом дискуссий и требует уточнений. Масштабное использование голштинских быков в качестве улучшателей могло привести к существенному изменению аллелофонда локальных пород. Включение в анализ музейных образцов позволяет прояснить историю становления пород, а также оценить, сохранились ли в современных холмогорской и ярославской породах предковые компоненты. В настоящем сообщении впервые описаны результаты анализа генетического разнообразия и структуры современного черно-пестрого скота в сравнении с предковыми популяциями с использованием ДНК-чипа высокой плотности, дана оценка влияния зарубежных пород на формирование генофонда холмогорского и ярославского скота в конце XIX-начале XX века. Целью нашей работы был анализ генетических взаимосвязей между предковыми и современными популяциями крупного рогатого скота черно-пестрого корня, оценка доли предковых компонентов в современных породах, уточнение истории происхождения и развития холмогорской и ярославской пород крупного рогатого скота на основании данных генотипирования с использованием ДНК-чипа высокой плотности. Выделение ДНК из музейных образцов проводили с использованием набора COrDIS Extract DECALCINE (ООО «ГОРДИЗ», Россия) с некоторыми модификациями протокола выделения ДНК, рекомендуемого производителем (увеличено время лизиса и объем реактивов на один образец). С помощью системы iScan («Illumina, Inc.», США) было проведено полногеномное генотипирование 68 музейных образцов с использованием ДНК-чипа высокой плотности (Bovine GGP HD BeadChip, «Illumina, Inc.», США), содержащего около 150 тыс. SNPs (single nucleotide polymorphisms). Полученные SNP-генотипы использовали для формирования объединенного набора данных, включающего SNP-генотипы 31 музейного (холмогорская порода, H_KHLM, n = 17; ярославская порода, H_YRSL, n = 12; великоросский скот, H_GRUS, n = 1; остфризский скот, H_OFRZ, n = 1) и 132 современных образцов (ярославская, YRSL, n = 53; холмогорская, KHLM, n = 26; голштинская, HLST, n = 54). Для оценки генетического разнообразия в каждой породе рассчитывали наблюдаемую (HO) и несмещенную ожидаемую (UHE) гетерозиготность, коэффициент инбридинга (UFIS) и показатель аллельного разнообразия, скорректированный на размер выборки (AR) с использованием R-пакета diversity. Генетические различия между породами оценивали в PLINK v1.90 методом анализа главных компонент (PCA) с визуализацией с помощью R-пакета ggplot2. Попарные дистанции для идентичных по состоянию (identical-by-state, IBS) фрагментов использовали для построения дендрограммы «сеть соседей» в программе SplitsTree 4.14.5. Попарные значения F ST были рассчитаны в R-пакете diveRsity и использовались для построения дендрограмм «сеть соседей» в программе SplitsTree 4.14.5. Кластерную структуру популяций исследовали в программе ADMIXTURE v1.23. Визуализация результатов анализа была выполнена с помощью R пакета BITE. Для оценки наличия и степени интрогрессии между породами проводили расчет показателей D-статистики и F4-статистики с использованием R пакета admixr. Для оценки степени дивергенции популяций и обмена генами между породами применяли программное обеспечение TreeMix 1.12. Аутгруппой при построении TreeMix дерева максимального правдоподобия (maximum likelihood, ML) послужила якутская порода. Результаты проведенных исследований указывают на более существенные изменения в генофонде ярославской породы за XX век по сравнению с холмогорской как за счет селекционного процесса, так и вследствие использования ограниченного числа производителей в связи со значительным снижением поголовья. Установлено, что аллельное разнообразие (AR) в предковых популяциях холмогорского и ярославского скота было достоверно (p ST = 0,040; D = 0,257) по сравнению с аналогичными показателями для H_YRSL и YRSL (F ST = 0,099; D = 0,353). Результаты анализа интрогрессии с помощью D- и F-статистики и анализа TreeMix подтвердили генетические связи между H_GRUS и H_YRSL. Показано присутствие в H_KHLM и H_YRSL некоторой доли геномных компонентов, характерных для остфризского скота, что подтверждает использование импортированного племенного материала в работе с российским локальным скотом на рубеже XIX-XX веков. В то же время в современных популяциях холмогорской и ярославской пород практически отсутствует интрогрессия голштинской породы, но сохранились геномные компоненты предковых популяций.
Крупный рогатый скот, музейные образцы, ярославская порода, холмогорская порода, snp-генотипирование, днк-чип
Короткий адрес: https://sciup.org/142243757
IDR: 142243757 | УДК: 636.2:57.082.133:577.2 | DOI: 10.15389/agrobiology.2024.4.605rus
Текст научной статьи Оценка генетических взаимосвязей пород крупного рогатого скота черно-пестрого корня с предковыми популяциями на основе полногеномного SNP-генотипирования современных и музейных образцов
Промышленная революция XVIII-XIX века стимулировала процессы урбанизации и роста населения (1), в результате чего вырос спрос на продукты питания, который не мог удовлетворяться за счет бессистемного разведения малопродуктивных животных. В этот период началось формирование основных отечественных пород молочного скота — холмогорской и ярославской (2). В конце XIX века было организовано несколько экспедиций с целью систематизации и описания скота, разводимого в центральных и северных областях России (3). Однако точное происхождение холмогорской и ярославской пород остается предметом дискуссий. Так, предком ярославского скота считается северный великоросский скот. Он был хорошо приспособлен к холодным зимам и выживанию в условиях недостатка кормов. Предком холмогорской породы, вероятно, был скот, распространенный в верховьях Северной Двины (4).
Некоторые авторы указывают, что на формирование русских пород черно-пестрого корня (холмогорской, ярославской, черно-пестрой, исто-бенской, тагильской) мог оказать влияние голландский крупный рогатый скот (5). Другие исследователи полагают, что голландский скот внес незначительный вклад в генофонд вышеупомянутых пород (4, 6). В пользу последней гипотезы говорят данные о том, что хотя завоз иностранного скота с 1752 по 1819 год и имел место, крестьяне, получившие завезенных животных для разведения, стремились скорее избавиться от них из-за плохих воспроизводительных качеств. Кроме того, есть сведения, что продукция животноводства и живой скот вывозились с низовьев Северной Двины еще во времена Ивана Грозного (7). Исходя из записей таможенных книг, в начале XVIII века Поморье экспортировало сотни подвод с различными товарами, в числе которых упоминаются крупный рогатый скот, кожи и говяжье сало. По данным подворной переписи Архангельской губернии за 1785 год, в переписных книгах Холмогорской округи есть сведения, что крестьяне занимались откормом и продажей скота, а также о том, что в этой местности не было зарегистрировано потомков от гибридизации с иностранными породами (8). Изложенные факты позволяют предполагать малозначительный вклад зарубежных пород на ранних этапах формирования холмогорского скота. Тем не менее нельзя отрицать тот факт, что и холмогорский, и ярославский северный скот был достаточно разнотипным по экстерьерным показателям (упоминаются животные пестрой, красной, черной масти с различными отметинами) (3, 7, 9).
Работа по консолидации пород стала более эффективной в начале XX века с учреждением племенных книг. Однако с конца 1970-х годов в нашей стране проводились масштабные селекционные мероприятия по улучшению стад молочного скота посредством скрещивания с голштинской породой, что могло привести к существенному изменению аллелофонда локальных пород (10). В сложившейся ситуации включение в анализ музейных образцов позволяет прояснить историю создания и развития пород, а также оценить, сохранились ли в современных холмогорской и ярославской породах предковые компоненты. Сравнительные исследования современных 606
и предковых популяций черно-пестрого скота с использованием микроса-теллитных маркеров показали сохранение в геноме современных животных компонентов, свойственных музейным образцам (11). Однако микросателлиты обладают меньшей разрешающей способностью по сравнению с другими инструментами анализа. Согласно результатам молекулярно-генетических исследований с использованием разных типов ДНК-маркеров, современные российские локальные породы генетически отличаются от зарубежных пород крупного рогатого скота; а холмогорская и ярославская выделяются среди других российских пород (12, 13). С использованием ДНК-чипов высокой плотности было показано, что в холмогорской и ярославской породах обнаружена минимальная интрогрессия голштинских генетических компонентов (14).
В настоящей работе впервые описаны результаты анализа генетического разнообразия и структуры современного черно-пестрого скота в сравнении с предковыми популяциями с использованием ДНК-чипа высокой плотности, дана оценка влияния импортных пород на формирование генофонда холмогорского и ярославского скота в конце XIX—начале XX века.
Нашей целью был анализ генетических взаимосвязей между предковыми и современными популяциями крупного рогатого скота черно-пестрого корня, оценка доли предковых компонентов в современных породах, уточнение истории происхождения и развития холмогорской и ярославской пород крупного рогатого скота на основании данных генотипирования с использованием ДНК-чипа высокой плотности.
Методика. Материалом для исследования предковых популяций служили музейные экспонаты черепов крупного рогатого скота ( Bos taurus taurus ), датированные концом XIX—первой половиной XX века, из краниологической коллекции Музея животноводства им. Е.Ф. Лискуна (РГАУ—МСХА им. К.А. Тимирязева, г. Москва). В качестве групп сравнения использовали образцы современных пород, биологический материал которых депонирован в генетическом банке ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста.
Выделение и оценку качества ДНК музейных образцов проводили в лаборатории, оборудованной для работы с древней ДНК (15), с использованием набора COrDIS Extract DECALCINE (ООО «ГОРДИЗ», Россия) с некоторыми модификациями протокола выделения ДНК, рекомендуемого производителем (увеличено время лизиса и объем реактивов на один образец).
С помощью системы iScan («Illumina, Inc.», США) было проведено полногеномное генотипирование 68 музейных образцов черно-пестрого скота с использованием ДНК-чипа высокой плотности (Bovine GGP HD BeadChip, «Illumina, Inc.», США), содержащего около 150 тыс. SNPs. В связи с постмортальными изменениями качество ДНК для этих образцов было ниже, чем для современных. По этой причине часть музейных образцов, показавших низкие значения call-rate (доля генотипированных SNPs из всех, представленных на чипе), GenCall и GenTrain, характеризующих соответственно точность определения генетическим анализатором каждого аллеля в SNP и гомо- или гетерозиготность по каждому аллелю, были исключены из дальнейшего анализа.
Полученные SNP-генотипы использовали для формирования объединенного набора данных, включающего SNP-генотипы 31 музейного (холмогорская порода, H_KHLM, n = 17; ярославская порода, H_YRSL, n = 12; великоросский скот, H_GRUS, n = 1; остфризский скот, H_OFRZ, n = 1) и 132 современных образцов (ярославская порода, YRSL, n = 53; холмогорская порода, KHLM, n = 26; голштинская порода, HLST, n = 54).
С использованием программы PLINK (16) из общего набора данных последовательно были удалены образцы, имевшие менее 30 % успешно геноти-пированных SNPs (--mind 0,7), а также локусы, генотипированные менее чем у 90 % животных (--geno 0,1) или показавшие частоту минорного аллеля менее 5 % (--maf 0,05). Для некоторых типов анализа (оценка генетического разнообразия и структуры популяций) была проведена дополнительная фильтрация SNPs по параметру неравновесия по сцеплению (linkage disequilibrium, LD). Были удалены SNPs, для которых квадрат коэффициента корреляции (r2) между двумя соседними SNPs внутри скользящего окна из 50 SNP с шагом перекрытия в 5 SNP превышал 0,5 (--indep-pairwise 50 5 0.5). Кроме того, исключали SNPs, локализованные на половых хромосомах, а также с неизвестной локализацией в геноме (сборка генома крупного рогатого скота ARS_UMD 3.1.1, . В итоговый набор данных после всех фильтров вошло 67046 SNPs.
Для оценки генетического разнообразия в каждой породе были рассчитаны наблюдаемая (H O ) и несмещенная ожидаемая ( U H E ) гетерозиготность (17), коэффициент инбридинга ( U F IS ) (18), основанный на несмещенной ожидаемой гетерозиготности, и показатель аллельного разнообразия, скорректированный на размер выборки (A R ) (19) с использованием R-пакета diveRsity (20).
Генетические различия между изученными породами оценивали в PLINK v1.90 методом анализа главных компонент (PCA) с визуализацией с помощью R-пакета ggplot2 (21). Попарные дистанции для идентичных по состоянию (identical-by-state, IBS) фрагментов были использованы для построения дендрограммы «сеть соседей» в программе SplitsTree 4.14.5 (22). Попарные значения F ST (18) были рассчитаны в R-пакете diveRsity (20) и использовались для построения дендрограмм «сеть соседей» в программе SplitsTree 4.14.5 (22). Кластерную структуру популяций исследовали в программе ADMIXTURE v1.23 (23). Оптимальное число кластеров К было определено на основании наименьшей ошибки при процедуре перекрестной проверки, рассчитанной для значений К от 1 до 9. Визуализация результатов анализа была выполнена с использованием R пакета BIT» (24).
Для оценки наличия и степени интрогрессии между исследованными породами проводили расчет показателей D-статистики (25) и F4-статистики (26) с использованием R пакета admixr (27).
Для оценки D-статистики в группе популяций W, X и Y, соответствующих филогении (((W, X), Y) О), где О — аутгруппа, было рассчитано отношение предковых (аллель А ) и производных (аллель В ) аллелей, общих для W и Y ( BABA ) или X и Y ( ABBA ). Значительный избыток BABA или ABBA указывал на наличие адмиксии между популяциями соответственно W и Y или X и Y. В качестве аутгруппы использовали современные образцы якутской породы как группы, генетически удаленной от черно-пестрого скота. Мы провели несколько итераций расчетов, изменяя породу, принимаемую за Y. Все остальные породы последовательно принимались за W и X. На достоверное наличие адмиксии между парами пород указывало значение Z > |3|.
Для оценки доли предков в адмиксированных популяциях рассчитывали отношения F4 (26). Были протестированы варианты, когда в качестве интрогрессированной породы (Х), источника интрогрессии (B) и сестринских для них (С для Х и А для В) пород принимались разные сочетания современных и предковых популяций. Значения показателя α , отражающего долю предковых компонентов породы В в породе Х, считались достоверными при Z > |3|.
Для оценки степени дивергенции популяций и обмена генами между изученными породами применили программное обеспечение TreeMix 1.12 (28). В качестве аутгруппы при построении TreeMix дерева максимального правдоподобия (maximum likelihood, ML) использовали якутскую породу.
Результаты. Из 68 генотипированных музейных образцов только 31 прошел контроль качества и был пригоден для дальнейшего анализа (табл. 1). Возможной причиной «выпадения» значительного числа образцов может быть так называемая post mortem 5´-концевая деградация ДНК, описанная для археологических образцов возрастом 4000 лет и старше, которая обусловлена гидролитическим дезаминированием цитозина в урацил (29, 30). Принимая во внимание, что черепа в процессе подготовки к депонированию подвергались действию высоких температур и агрессивных растворов, процесс деградации ДНК мог происходить существенно быстрее. То есть невозможность включения образца в анализ при относительно высокой доле успешно генотипированных SNPs в нем связана с ошибочным определением системой iScan («Illumina, Inc.», США) генотипа в некоторых SNPs (цитозин, дезаминированный до урацила, определен как аденин) и избытком гомозигот в большом числе локусов.
-
1. Результативность полногеномного генотипирования ДНК, выделенной из музейных образцов крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня, с использованием ДНК-чипа высокой плотности Bovine GGP HD BeadChip
-
2. Параметры генетического разнообразия предковых и современных популяций крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня на основании данных полногеномного SNP-генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip
-
4. Результаты анализа показателей D-статистики для предковых и современных популяций крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня на основании данных полногеномного SNP-генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip
Итерация
W
X 1
Y
O
Z-score
BABA
ABBA
I
H_YRSL
YRSL
H_GRUS
YAKUT
8,424
1055
960
H_YRSL
KHLM
H_GRUS
YAKUT
8,161
1057
968
H_YRSL
HLST
H_GRUS
YAKUT
7,756
1093
993
H_KHLM
KHLM
H_GRUS
YAKUT
6,792
989
935
H_KHLM
YRSL
H_GRUS
YAKUT
5,668
1027
966
H_KHLM
HLST
H_GRUS
YAKUT
5,581
1054
988
H_YRSL
H_KHLM
H_GRUS
YAKUT
3,862
977
943
H_OFRZ
HLST
H_GRUS
YAKUT
3,692
1169
1095
H_OFRZ
YRSL
H_GRUS
YAKUT
3,494
1151
1081
H_OFRZ
KHLM
H_GRUS
YAKUT
3,203
1142
1079
II
H_YRSL
YRSL
H_OFRZ
YAKUT
34,277
1717
1276
H_YRSL
KHLM
H_OFRZ
YAKUT
28,952
1699
1309
H_YRSL
HLST
H_OFRZ
YAKUT
23,326
1729
1377
H_KHLM
KHLM
H_OFRZ
YAKUT
19,245
1503
1311
H_KHLM
YRSL
H_OFRZ
YAKUT
18,941
1579
1336
H_YRSL
H_KHLM
H_OFRZ
YAKUT
18,935
1534
1335
H_KHLM
HLST
H_OFRZ
YAKUT
11,638
1577
1424
H_YRSL
H_GRUS
H_OFRZ
YAKUT
10,907
1199
989
HLST
YRSL
H_OFRZ
YAKUT
6,608
1540
1451
H_GRUS
YRSL
H_OFRZ
YAKUT
4,461
1151
1062
H_KHLM
H_GRUS
H_OFRZ
YAKUT
4,056
1129
1052
KHLM
YRSL
H_OFRZ
YAKUT
4,053
1472
1421
HLST
KHLM
H_OFRZ
YAKUT
3,191
1500
1462
Продолжение таблицы 4
III
H_OFRZ
YRSL
H_YRSL
YAKUT
24,702
1717
1319
H_OFRZ
HLST
H_YRSL
YAKUT
24,031
1729
1331
H_OFRZ
KHLM
H_YRSL
YAKUT
24,025
1699
1314
H_KHLM
KHLM
H_YRSL
YAKUT
21,988
1353
1195
H_KHLM
YRSL
H_YRSL
YAKUT
18,248
1408
1237
H_KHLM
HLST
H_YRSL
YAKUT
16,779
1438
1267
H_OFRZ
H_KHLM
H_YRSL
YAKUT
15,026
1534
1307
H_OFRZ
H_GRUS
H_YRSL
YAKUT
11,001
1199
1015
H_GRUS
HLST
H_YRSL
YAKUT
5,969
1093
1004
H_GRUS
YRSL
H_YRSL
YAKUT
5,806
1055
972
H_GRUS
KHLM
H_YRSL
YAKUT
5,593
1057
982
IV
H_YRSL
YRSL
H_KHLM
YAKUT
17,331
1408
1241
KHLM
YRSL
H_KHLM
YAKUT
14,941
1376
1242
H_YRSL
HLST
H_KHLM
YAKUT
12,111
1438
1301
H_OFRZ
YRSL
H_KHLM
YAKUT
12,015
1579
1383
KHLM
HLST
H_KHLM
YAKUT
11,614
1382
1277
H_OFRZ
HLST
H_KHLM
YAKUT
10,482
1577
1412
H_OFRZ
H_GRUS
H_KHLM
YAKUT
5,396
1129
1043
H_YRSL
H_GRUS
H_KHLM
YAKUT
4,937
1006
943
KHLM
H_GRUS
H_KHLM
YAKUT
4,182
1045
989
H_OFRZ
KHLM
H_KHLM
YAKUT
3,944
1503
1442
H_YRSL
KHLM
H_KHLM
YAKUT
3,256
1353
1321
H_GRUS
YRSL
H_KHLM
YAKUT
3,145
1027
982
HLST
YRSL
H_KHLM
YAKUT
3,067
1354
1324
V
KHLM
YRSL
HLST
YAKUT
5,99
1372
1312
KHLM
H_YRSL
HLST
YAKUT
5,367
1388
1333
H_KHLM
H_YRSL
HLST
YAKUT
4,33
1301
1267
H_KHLM
YRSL
HLST
YAKUT
4,187
1354
1314
KHLM
H_GRUS
HLST
YAKUT
3,996
1071
1013
H_OFRZ
H_YRSL
HLST
YAKUT
3,381
1377
1331
H_OFRZ
YRSL
HLST
YAKUT
3,096
1540
1488
Примечани е. Предковые популяции:
H_KHLM —
холмогорская порода, H_YRSL — ярославская по-
рода, H_
GRUS — великоросский скот, H
OFRZ — остфризский скот; современные популяции: HLST —
|
Популяция |
Всего генотипировано |
Call-rate |
Число гол. после QC |
|
|
H_ |
KHLM |
28 |
0,682-0,969 |
17 |
|
H_ |
YRSL |
32 |
0,711-0,972 |
12 |
|
H_ |
GRUS |
7 |
0,746-0,977 |
1 |
|
H_ |
OFRZ |
1 |
0,718 |
1 |
Пр им еч ан и е. H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — вели-коросский скот, H_OFRZ — остфризский скот; call rate — доля генотипированных SNP от общего числа SNP до оценки контроля качества генотипирования, QC — quality control, образцы, прошедшие контроль качества и отобранные для дальнейшего анализа.
Необходимо отметить, что с целью сохранения как можно большего числа образцов предковых популяций для дальнейшего анализа были использованы менее жесткие фильтры контроля качества по сравнению с теми, что применяются для наборов данных современных образцов.
|
Популяция |
] n |
H o ( M ±SEM) |
u H e ( M ±SEM) 1 A r ( M ±SEM)т |
U F IS (CI) |
|
|
H_KHLM |
17 |
0,360±0,001* |
0,378±0,001* |
1,959±0,001* |
0,041 (0,039; 0,043) |
|
H_YRSL |
12 |
0,380±0,001* |
0,395±0,001* |
1,981±0,001* |
0,036 (0,033; 0,039) |
|
HLST |
54 |
0,373±0,001 |
0,374±0,001 |
1,947±0,001 |
0,003 (0,002; 0,004) |
|
KHLM |
26 |
0,378±0,001 |
0,370±0,001 |
1,936±0,001 |
- 0,021 ( - 0,023; - 0,019) |
|
YRSL |
53 |
0,360±0,001 |
0,357±0,001 |
1,917±0,001 |
- 0,009 |
( - 0,010; - 0,008)
Пр им еч ан и е. Предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода (параметры генетического разнообразия для великоросского и остфризского скота не рассчитывали, поскольку эти группы были представлены одним образцом); современные популяции: KHLM — холмогорская порода, YRSL — ярославская порода, HLST — голштинская порода.
* Различия с современными популяциями статистически значимы при p < 0,001.
Аллельное разнообразие (AR) в H_KHLM и H_YRSL было достоверно выше (p < 0,001) по сравнению с современными популяциями аналогичных пород (табл. 2). Значения показателя наблюдаемой (HО) гетерозиготности в KHLM оказалось достоверно (p < 0,001) выше, а в YRSL — ниже, чем в предковых популяциях. Это может быть связано с использованием голштинского скота для улучшения современной популяции холмогорской породы, а также со снижением численности популяции YRSL. В обеих предковых популяциях отмечался достоверный дефицит гетерозигот. Вероятно, это объясняется тем, что подбор родительских пар осуществлялся внутри одного помещичьего хозяйства или населенного пункта, а обмен племенным материалом был нерегулярным, что постепенно приводило к росту инбридинга.
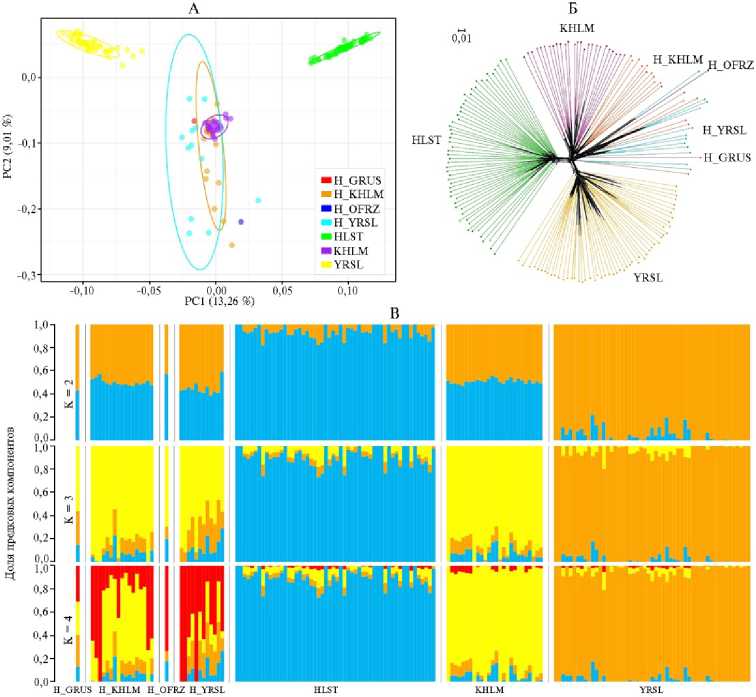
Рис. 1. Межпородные взаимосвязи и генетическая структура предковых и современных популяций крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня на основании данных полногеномного SNP-генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip: А — результаты анализа главных компонент, Б — дендрограмма Neighbor-Net, построенная на основании генетических дистанций IBS (dentical-by-state), В — результаты анализа структуры популяций (каждый образец представлен отдельным столбцом на гистограмме); предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — великоросский скот, H_OFRZ — остфризский скот; современные популяции: HLST — голштинская, KHLM — холмогорская, YRSL — ярославская порода.
Современные породы были гораздо более консолидированы, чем предковые (рис. 1). Большинство образцов H_KHLM оказались наиболее близки к современной холмогорской породе, о чем свидетельствовало формирование перекрывающегося массива при анализе PCA и расположение ветвей на индивидуальном дереве Neighbor-Net (см. рис. 1, А, Б). Современная ярославская порода достаточно сильно отличалась от предковой популяции, о чем свидетельствовало формирование обособленного породоспецифичного кластера (см. рис. 1, А, Б). Формирование общей ветви на инди- видуальном дереве Neighbor-Net (см. рис. 1, Б) между H_YRSL, H_GRUS и H_OFRZ указывало на их большую генетическую близость по отношению друг к другу по сравнению с холмогорским скотом. Некоторые образцы H_KHLM демонстрировали генетическую близость к предковым популяциям ярославского, остфризского и великоросского скота, формируя с ними единую ветвь, что может свидетельствовать о меньшей специализации пород в начале XX века по сравнению с современными.
Анализ структуры популяций указывает на участие четырех предковых популяций в формировании аллелофонда исследуемых групп скота (см. рис. 1, В). Три современные породы формировали четко дифференцированные кластеры. Предковые популяции несли значительную долю общих компонентов, позволивших объединить их в четвертый кластер. При этом в современных породах были компоненты, общие с предковыми. В наибольшей степени это проявлялось в холмогорской породе. У великоросского скота были обнаружены компоненты, характерные для всех четырех кластеров. Популяция H_YRSL представляла собой неоднородную группу, несущую компоненты холмогорского, остфризского и великоросского скота. В современной ярославской породе под давлением отбора закрепилась и сохранилась лишь некоторая часть предковых генетических компонентов. Образцы H_KHLM в большинстве случаев несли значительную долю компонентов, характерных для современных животных холмогорской породы. В образцах H_YRSL были выявлены компоненты, свойственные современным популяциям YRSL и KHLM, а также компоненты специфического кластера, общего для музейных образцов, в том числе остфризского и велико-росского скота.
Расчет генетических дистанций выявил наибольшую близость H_KHLM с современной породой и H_YRSL как по F ST , так и по J. Reynolds (табл. 3). Особи H_GRUS и H_OFRZ показали наименьшее генетическое расхождение с H_KHLM и H_YRSL, демонстрируя в то же время максимальную генетическую удаленность от других групп.
3. Генетические дистанции между предковыми и современными популяциями крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня на основании данных полногеномного SNP-генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip
|
Популяция \ H_GRUS |
\ H_KHLM |
H_OFRZ \ |
H_YRSL \ |
HLST |
\ KHLM |
\ YRSL |
|
|
H_GRUS |
0,517 |
0,561 |
0,516 |
0,538 |
0,525 |
0,534 |
|
|
H_KHLM |
0,084 |
0,407 |
0,291 |
0,333 |
0,257 |
0,335 |
|
|
H_OFRZ |
0,000 |
0,119 |
0,396 |
0,441 |
0,431 |
0,447 |
|
|
H_YRSL |
0,058 |
0,039 |
0,059 |
0,364 |
0,342 |
0,353 |
|
|
HLST |
0,138 |
0,091 |
0,189 |
0,105 |
0,321 |
0,350 |
|
|
KHLM |
0,121 |
0,040 |
0,191 |
0,084 |
0,090 |
0,326 |
|
|
YRSL |
0,138 |
0,094 |
0,216 |
0,099 |
0,114 |
0,094 |
|
Пр им еч ан и е. Предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — великоросский скот, H_OFRZ — остфризский скот; современные популяции: HLST — голштинская, KHLM — холмогорская, YRSL — ярославская порода. Над диагональю указаны генетические дистанции, рассчитанные по методике, предложенной J. Reynolds; под диагональю указаны генетические дистанции, рассчитанные по F ST .
На дендрограмме, построенной на основании генетических дистанций FST (рис. 2), предковые популяции великоросского, остфризского и ярославского скота сформировали общий кластер, удаленный от современных популяций. Популяция KHLM имела существенно большее генетическое сходство с H_KHLM (FST = 0,040; D = 0,257) по сравнению со степенью генетического сходства между H_YRSL и YRSL (FST = 0,099; D = 0,353). H_KHLM и KHLM сформировали общую ветвь, в то время как H_YRSL и YRSL локализовались на различных ветвях. Это позволяет пред- положить, что в современной ярославской породе действуют иные факторы отбора, чем в предковых популяциях.
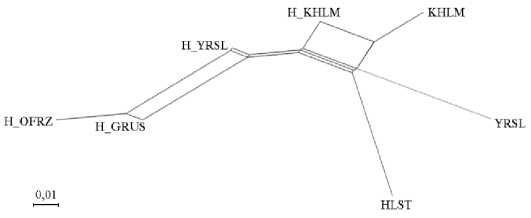
Рис. 2. Дендрограмма Neighbor-Net, построенная на основании генетических дистанций F ST и демонстрирующая взаимосвязи предковых и современных популяций крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня: предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — великоросский скот, H_OFRZ — остфризский скот; современные популяции: HLST — голштинская, KHLM — холмогорская, YRSL — ярославская порода.
Между H_KHLM и KHLM были обнаружены существенно меньшие генетические дистанции F ST по сравнению с H_YRSL и YRSL. Великорос-ский скот демонстрировал наибольшую генетическую близость к H_YRSL и YRSL, что свидетельствует об использовании северного великоросского скота на начальных этапах формирования породы. Локализация современных популяций на отдельных ветвях, выявленная при построении дендрограммы на основании дистанций F ST , свидетельствует о генетическом расхождении пород в результате селекционной работы, при этом видно, что холмогорская порода менее всех отошла от предковой популяции.
Результаты анализа показателей D-статистики для исследуемых пород, в котором за источник интрогрессии последовательно принимались предковые популяции скота, а также голштинская порода, представлены в таблице 4.
голштинская, KHLM — холмогорская, YRSL — ярославская порода; W и X — исследуемые популяции,
Y — источник интрогрессии, О — аутгруппа, Z-score — показатель достоверности наличия адмиксии между парами пород; BABA — число аллелей, общих для популяций W и Y, ABBA — число аллелей, общих для популяций X и Y.
В первой итерации в качестве источника интрогрессии (популяция Y) мы определили великоросский скот на основании предположения о его вкладе в формирование холмогорской и ярославской пород (см. табл. 4). Был обнаружен избыток общих аллелей для H_GRUS и других предковых популяций по сравнению с современными, что указывает на его отдаленность от современных пород. Однако при сравнении между предковыми популяциями достоверный избыток общих аллелей обнаружен только для H_YRSL при сравнении с H_KHLM. Это указывает на то, что между H_GRUS и H_YRSL существовала более тесная связь, чем с H_KHLM.
Во второй итерации в качестве Y был выбран остфризский скот с целью уточнения его возможного вклада в развитие изучаемых пород. Наибольшая доля генетических компонентов H_OFRZ наблюдалась в популяциях H_YRSL и H_KHLM. Великоросский скот показал наименьшее число общих с остфризским скотом SNPs среди предковых популяций. KHLM и YRSL несли меньше общих SNPs с остфризским скотом, чем H_KHLM и H_YRSL, что свидетельствует об элиминации аллелей, характерных для остфризского скота, в процессе селекционной работы. Среди современных популяций к остфризскому скоту наиболее близка голштинская порода, что не противоречит истории происхождения и развития породы.
В третьей и четвертой итерациях за Y принимали H_YRSL и H_KHLM как предковые группы для современных одноименных пород. Выявлен поток генов между H_YRSL и популяциями H_OFRZ, H_KHLM и H_GRUS, что свидетельствует о продолжавшемся процессе формирования ярославской породы в конце XIX—начале XX века. Тестирование в качестве Y популяции H_KHLM показало ее близость к H_YRSL и H_YRSL, чем под- тверждается использование остфризских производителей на поголовье холмогорской породы, а быков холмогорской породы для улучшения ярославского скота. Кроме того, были выявлены генетические компоненты, общие с современной популяцией холмогорской породы.
В пятой итерации мы определили как Y голштинскую породу вследствие ее интенсивного использования при улучшении изучаемых пород. Общие генетические компоненты были выявлены как для H_KHLM, так и для KHLM, а также для H_OFRZ. Интрогрессии голштинских генетических компонентов в ярославский скот мы не обнаружили.
Оценку доли предковых компонентов в популяциях с выявленной адмиксией осуществляли на основании расчета соотношения F4 (табл. 5). Были протестированы все возможные варианты, когда за источник интро-грессии/интрогрессированную породу последовательно принималась каждая из исследуемых групп. Комбинации пород, не показавшие достоверных значений показателя α , в таблицу включены не были.
5. Соотношение F4 при попарном сравнении предковых и современных популяций крупного рогатого скота ( Bos taurus taurus ) черно-пестрого корня на основании данных полногеномного SNP-генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip
|
A |
B |
X |
C |
O |
α |
±SE |
Z-score |
|
|
H_ |
OFRZ |
H_YRSL |
KHLM |
YRSL |
YAKUT |
0,1160 |
0,027258 |
4,256 |
|
H_ |
OFRZ |
H_KHLM |
KHLM |
YRSL |
YAKUT |
0,2121 |
0,044976 |
4,715 |
|
H_ |
OFRZ |
H_GRUS |
KHLM |
YRSL |
YAKUT |
0,4280 |
0,129216 |
3,312 |
|
H_ |
GRUS |
H_OFRZ |
H_KHLM |
KHLM |
YAKUT |
0,7857 |
0,242678 |
3,238 |
Пр и м еч ани е. X — интрогрессированная популяция, C — сестринская популяция для популяции X, B — источник интрогрессии, A — сестринская популяция для популяции B, O — аутгруппа, α — доля предковых компонентов B в X, SE — стандартная ошибка показателя α , Z-score — число стандартных ошибок α , отклоняющихся от нуля; предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — великоросский скот, H_OFRZ — остфризский скот; современные популяции: HLST — голштинская, KHLM — холмогорская, YRSL — ярославская порода.
Расчет соотношения F4 (см. табл. 5) показал, что KHLM несет 21,2 % предковых компонентов H_KHLM, 11,6 % компонентов H_YRSL и 42,8 % предковых компонентов H_GRUS. При исследовании в качестве ин-трогрессированной группы H_KHLM в ней было выявлено 78,6 % предковых компонентов, источником которых послужил остфризский скот, что, вероятно, связано с использованием импортного скота при развитии породы в XIX веке.
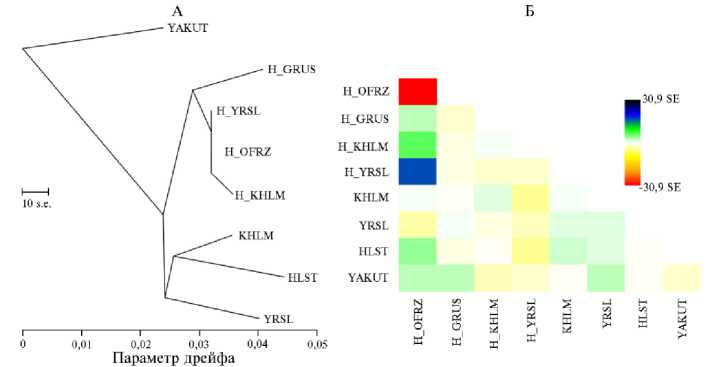
Рис. 3. Оценка взаимоотношений между исследуемыми популяциями на основании анализа
TreeMix: A — дерево максимального правдоподобия для предковых и современных популяций крупного рогатого скота (Bos taurus taurus) черно-пестрого корня на основании данных SNP- генотипирования с использованием ДНК-чипа Bovine GGP HD BeadChip. Шкала показывает 10-кратную среднюю стандартную ошибку (SE) записей в ковариационной матрице выборки (размер шкалы 10 s.e. соответствует значению 10-кратной стандартной ошибки, standard error, SE), Б — матрица остатков, построенная на основе анализа TreeMix и выраженная как число отклонений SE (положительные значения SE между парами популяций указывают на то, что популяции более тесно связаны друг с другом, чем в смоделированном дереве, отрицательные значения SE показывают, что наблюдаемая ковариация завышена). Предковые популяции: H_KHLM — холмогорская порода, H_YRSL — ярославская порода, H_GRUS — великорос-ский скот, H_OFRZ — остфризский скот; современные популяции: HLST — голштинская, KHLM — холмогорская, YRSL — ярославская, YAKUT — якутская порода.
Степень дивергенции исследуемых популяций и обмена генами между ними оценивали посредством анализа TreeMix на основании данных о частотах аллелей по всему геному. На основании результатов расчета TreeMix было построено дерево максимального правдоподобия, отражающее взаимоотношения исследуемых популяций (рис. 3, А). Из тридцати итераций на основании анализа регрессионных остатков модели (см. рис. 3, Б) была выбрана конфигурация дендрограммы, наилучшим образом отражающая оцененные взаимоотношения пород.
На дендрограмме были сформированы две крупные ветви: одна из них объединила все предковые популяции, причем H_GRUS образовал ветвь, отдельную от H_YRSL, H_OFRZ и H_GRUS; вторая крупная ветвь, объединившая все современные популяции, в свою очередь разделилась на более мелкие градации, выделив YRSL в отдельную ветвь.
Результаты D-, F-статистики и анализа TreeMix подтвердили генетические связи между H_GRUS и H_YRSL. Кроме того, было выявлено наличие общих предковых компонентов у ярославского скота с H_KHLM, что подтверждает историю формирования породы. Подтверждено использование остфризского скота на поголовье холмогорского и ярославского скота в конце XIX—начале ХХ века.
С момента начала исследований палеоДНК и до недавнего времени большинство работ с вовлечением древних образцов проводилось на основании анализа полиморфизмов митохондриальной ДНК (31-33) или микро-сателлитных маркеров (34, 35). Тенденции, выявленные при помощи этих инструментов анализа генома, неоднозначны. Так, M. Gargani с соавт. (35) сообщают о меньшей по сравнению с современными образцами наблюдаемой гетерозиготности в средневековых образцах крупного рогатого скота из Центральной Италии. Средневековые образцы крупного рогатого скота из Ирландии, описанные C.J. Edwards с соавт. (34), также показали несколько меньшую гетерозиготность по сравнению с современными породами различного происхождения. В предыдущих исследованиях локальных российских пород было показано, что предковые популяции холмогорского и ярославского скота превосходили современные одноименные породы по показателям генетического разнообразия (11). Статистически значимый избыток гетерозигот при оценке по микросателлитным маркерам был обнаружен только для предковой популяции холмогорской породы, в то время как в настоящей работе анализ по SNP-маркерам выявил недостаток гетерозигот в обеих предковых популяциях.
Некоторое расхождение оценки коэффициента инбридинга для разных типов маркеров (SNP-маркеры в настоящем исследовании и микроса-теллитные маркеры в упомянутых выше работах) могло быть следствием нескольких причин: многообразия микросателлитных аллелей из-за недостаточной по сравнению с современными образцами консолидированности холмогорской породы в конце XIX—начале XX веков, формирования участков гомозиготности в результате давления отбора на локусы, связанные с продуктивными и адаптационными качествами, а также постепенного нарастания инбридинга по причине подбора родительских пар внутри одного хозяйства или общины. Кроме того, определенную роль в расхождении результатов оценки по разным типам маркеров могут играть неодинаковая разрешающая способность используемых инструментов и разное селекционное воздействие (селекционно нейтральные микросателлитные локусы против находящихся под давлением отбора SNPs, локализованных вблизи или внутри генов), связанное с молекулярно-генетической структурой маркеров разного типа (36).
Таким образом, результаты полногеномного SNP-генотипирования с помощью ДНК-чипа высокой плотности указывают на более существенные изменения в генофонде ярославской породы за XX век по сравнению с холмогорской как за счет селекционной работы, так и вследствие использования ограниченного числа производителей в связи со значительным снижением поголовья. Показано присутствие в исторических образцах холмогорской и ярославской пород некоторой доли геномных компонентов, характерных для остфризского скота, что подтверждает использование импортного племенного материала в работе с российскими породами на рубеже XIX-XX веков. В то же время современные популяции холмогорской и ярославской пород обладают собственными уникальными породоспецифичными компонентами, в них практически отсутствует интрогрессия голштинской породы, однако сохранились геномные компоненты предковых популяций. Выявлен более заметный вклад великоросского скота в генофонд предкового ярославского скота по сравнению с холмогорским, что подтверждает гипотезу об общих предках ярославской породы и северного великоросского скота. Получить более полную информацию о том, использовались ли зарубежные породы на начальных этапах формирования российских локальных пород в XVII-XVIII веках или же гибридизация с иностранными породами пришлась на вторую половину XIX века, когда основные типы локального скота были уже относительно оформлены, вероятно, поможет исследование более древних образцов с использованием полногеномного секвенирования.
Список литературы Оценка генетических взаимосвязей пород крупного рогатого скота черно-пестрого корня с предковыми популяциями на основе полногеномного SNP-генотипирования современных и музейных образцов
- Промышленная революция. Режим доступа: https://old.bigenc.ru/world_history/text/3180201. Дата обращения: 11.04.2024.
- Эрнст Л.К., Дмитриев Н.Г., Паронян И.А. Генетические ресурсы сельскохозяйственных животных в России и сопредельных странах. СПб, 1994.
- Миддендорф А.Ф. Исследование современного состояния скотоводства в России. Выпуск I. Рогатый скот. М., 1884: 226-260 Режим доступа: https://elibrary.tambovlib.ru/?ebook=4383. Дата обращения: 11.04.2024.
- Диомидов А.М., Жиркович Е.Ф. Разведение и породы крупного рогатого скота. М., 1934.
- Сабанеев Л.П. Очерки Зауралья и степное хозяйство на башкирских землях. М., 1873 Режим доступа: https://elibrary.orenlib.ru/index.php?dn=down&to=avtoropen&id=3546. Дата обращения: 11.04.2024.
- Felius M. Cattle breeds — an encyclopedia. Misset, Doetinchem, Netherlands, 1995.
- Лискун Е.Ф. Отечественные породы крупного рогатого скота. М., 1949.
- Колесников П.А. Северная Русь. Выпуск 2. Архивные источники истории Европейского Севера России XVIII в. Вологда, 1973. Режим доступа: https://www.booksite.ru/fulltext/754332/text.pdf. Дата обращения: 12.04.2024.
- Отчет о Первой Всероссийской выставке рогатого скота 1869 года, составленный с соизволения Его Императорского Высочества Государя Великого Князя Николая Николаевича Старшего, комиссией по устройству выставки. СПб, 1869. Режим доступа: https://kp.rus-neb.ru/item/reader/otchet-o-pervoy-vserossiyskoy-vystavke-rogatogo-skota-1869-goda. Дата обращения: 12.04.2024.
- Сермягин А.А., Нарышкина Е.Н., Недашковский И.С., Ермилов А.Н., Богданова Т.В. Оценка эффекта голштинизации в популяции черно-пестрого скота Подмосковья. АгроЗооТехника, 2018, 1(3): 1 (doi: 10.15838/alt.2018.1.3.1).
- Abdelmanova A.S., Kharzinova V.R., Volkova V.V., Mishina A.I., Dotsev A.V., Sermyagin A.A., Boronetskaya O.I., Petrikeeva L.V., Chinarov R.Y., Brem G., Zinovieva N.A. Genetic diversity of historical and modern populations of russian cattle breeds revealed by microsatellite analysis. Genes, 2020, 11(8): 940 (doi: 10.3390/genes11080940).
- Волкова В.В., Кошкина О.А., Хожоков А.А. Изучение генетического разнообразия ярославской и холмогорской породы с использованием разных типов генетических маркеров. Труды Кубанского государственного аграрного университета, 2022, 98: 163-171 (doi: 10.21515/1999-1703-98-163-171).
- Столповский Ю.А., Бекетов С.В., Солоднева Е.В., Абсаликов B.М., Абдельманова А.С., Гладырь Е.А., Зиновьева Н.А. Генетическая структура аборигенного тагильского скота по STR- и SNP-маркерам. Сельскохозяйственная биология [Agricultural Biology], 2021, 56(6): 1123-1133 (doi: 10.15389/agrobiology.2021.6.1123rus).
- Абдельманова А.С., Сермягин А.А., Доцев А.В., Родионов А.Н., Столповский Ю.А., Зиновьева Н.А. Полногеномные исследования структуры популяций российских локальных пород черно-пестрого корня. Генетика, 2022, 58(7): 786-797 (doi: 10.31857/S0016675822070025).
- Абдельманова А.С., Волкова В.В., Харзинова В.Р., Форнара М.С., Чинаров Р.Ю., Боронецкая О.И., Трухачев В.И., Brem G., Зиновьева Н.А. Определение консенсусных генотипов по микросателлитам у музейных краниологических образцов крупного рогатого скота (Вos taurus). Сельскохозяйственная биология[Agricultural Biology], 2023, 58(6): 1035-1045 (doi: 10.15389/agrobiology.2023.6.1035rus).
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M.A., Bender D., Maller J., Sklar P., de Bakker P.I., Daly M.J., Sham P.C. PLINK: a tool set for whole-genome association and population-based linkage analyses. The American Journal of Human Genetics, 2007, 81(3): 559-575 (doi: 10.1086/519795).
- Nei M. Estimation of average heterozygosity and genetic distance from small number of individuals. Genetics, 1978, 89(3): 583-590 (doi: 10.1093/genetics/89.3.583).
- Weir B.S., Cockerham C.C. Estimating F-Statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 (doi: 10.1111/j.1558-5646.1984.tb05657.x).
- Kalinowski S.T. Counting alleles with rarefaction: private alleles and hierarchical sampling designs. Conservation Genetics, 2004, 5: 539-543 (doi: 10.1023/B:COGE.0000041021.91777.1a).
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodöhl P.A. diveRsity: An R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4(8): 782-788 (doi: 10.1111/2041-210X.12067).
- Wickham H. ggplot2: elegant graphics for data analysis. Springer, New York, 2009.
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 (doi: 10.1093/molbev/msj030).
- Alexander D.H., Novembre J., Lange K. Fast model-based estimation of ancestry in unrelated individuals. Genome Res., 2009, 19: 1655-1664 (doi: 10.1101/gr.094052.109).
- Milanesi M., Capomaccio S., Vajana E., Bomba L., Garcia J.F., Ajmone-Marsan P., Colli L. BITE: an R package for biodiversity analyses. bioRxiv, 2017 (doi: 10.1101/181610).
- Green R.E., Krause J., Briggs A.W., Maricic T., Stenzel U., Kircher M., Patterson N., Li H., Zhai W., Fritz M.H., Hansen N.F., Durand E.Y., Malaspinas A.S., Jensen J.D., Marques-Bonet T., Alkan C., Prüfer K., Meyer M., Burbano H.A., Good J.M., Schultz R., Aximu-Petri A., Butthof A., Höber B., Höffner B., Siegemund M., Weihmann A., Nusbaum C., Lander ES., Russ C., Novod N., Affourtit J., Egholm M., Verna C., Rudan P., Brajkovic D., Kucan Ž., Gušic I., Doronichev V.B., Golovanova L.V., Lalueza-Fox C., de la Rasilla M., Fortea J., Rosas A., Schmitz R.W., Johnson P.L.F., Eichler E.E., Falush D., Birney E., Mullikin J.C., Slatkin M., Nielsen R., Kelso J., Lachmann M., Reich D., Pääbo S. A draft sequence of the Neandertal genome. Science, 2010, 328(5979): 710-722 (doi: 10.1126/science.1188021).
- Patterson N., Moorjani P., Luo Y. Mallick S., Rohland N., Zhan Y., Genschoreck T., Webster T., Reich D. Ancient admixture in human history. Genetics, 2012, 192(3): 1065-1093 (doi: 10.1534/genetics.112.145037).
- Petr M., Vernot B., Kelso J. admixr — R package for reproducible analyses using ADMIXTOOLS. Bioinformatics, 2019, 35(17): 3194-3195 (doi: 10.1093/bioinformatics/btz030).
- Pickrell J.K., Pritchard J.K. Inference of population splits and mixtures from genome-wide allele frequency data. PLoS Genetics, 2012, 8: e1002967 (doi: 10.1371/journal.pgen.1002967).
- Dabney J., Meyer M., Pääbo S. Ancient DNA damage. Cold Spring Harbor Perspectives in Biology, 2013, 5(7): a012567 (doi: 10.1101/cshperspect.a012567).
- Gorden E. M., Sturk-Andreaggi K., Marshall C. Repair of DNA damage caused by cytosine deamination in mitochondrial DNA of forensic case samples. Forensic Science International: Genetics, 2018, 34: 257-264 (doi: 10.1016/j.fsigen.2018.02.015).
- Bailey J.F., Richards M.B., Macaulay V.A., Colson I.B., James I.T., Bradley D.G., Hedges R.E., Sykes B.C. Ancient DNA suggests a recent expansion of European cattle from a diverse wild progenitor species. Proceedings of the Royal Society of London. Series B: Biological Sciences, 1996, 263(1376): 1467-1473 (doi: 10.1098/rspb.1996.0214).
- Beja-Pereira A., Caramelli D., Lalueza-Fox C., Vernesi C., Ferrand N., Casoli A., Goyache F., Royo LJ., Conti S., Lari M., Martini A., Ouragh L., Magid A., Atash A., Zsolnai A., Boscato P., Triantaphylidis C., Ploumi K., Sineo L., Mallegni F., Taberlet P., Erhardt G., Sampietro L., Bertranpetit J., Barbujani G., Luikart G., Bertorelle G. The origin of European cattle: evidence from modern and ancient DNA. Proceedings of the National Academy of Sciences, 2006, 103(21): 8113-8118 (doi: 10.1073/pnas.0509210103).
- Delsol N., Stucky B.J., Oswald J.A., Cobb C.R., Emery K.F., Guralnick R. Ancient DNA confirms diverse origins of early post-Columbian cattle in the Americas. Sci. Rep., 2023, 13(1): 12444 (doi: 10.1038/s41598-023-39518-3).
- Edwards C.J., Connellan J., Wallace P.F., Park S.D.E., McCormick F.M., Olsaker I., Eythórsdóttir E., MacHugh D.E., Bailey J.F., Bradley D.G. Feasibility and utility of microsatellite markers in archaeological cattle remains from a Viking Age settlement in Dublin. Animal Genetics, 2003, 34(6): 410-416 (doi: 10.1046/j.0268-9146.2003.01043.x).
- Gargani M., Pariset L., Lenstra J.A., De Minicis E., European Cattle Genetic Diversity Consortium, Valentini A. Microsatellite genotyping of medieval cattle from central Italy suggests an old origin of Chianina and Romagnola cattle. Front. Genet., 2015, 6: 68 (doi: 10.3389/fgene.2015.00068).
- Глазко В.И., Косовский Г.Ю., Глазко Т.Т., Федорова Л.М. ДНК маркеры и «микросателлитный код» (обзор). Сельскохозяйственная биология[Agricultural Biology], 2023, 58(2): 223-248 (doi: 10.15389/agrobiology.2023.2.223rus).


