Оценка материнской изменчивости российских локальных пород овец на основе анализа полиморфизма гена цитохрома B
Автор: Кошкина О.А., Денискова Т.Е., Доцев А.В., Kunz E., Upadhyay M., Krebs S., Соловьева А.Д., Medugorac I., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Структура генома и геномные технологии
Статья в выпуске: 6 т.56, 2021 года.
Бесплатный доступ
Исследование полиморфизма митохондриальной ДНК (мтДНК) - это один из эффективных современных подходов к оценке генетического разнообразия сельскохозяйственных животных. У овец ( Ovis aries ) секвенирование мтДНК служит наиболее эффективным подходом для определения гаплогрупп мтДНК. Несмотря на то, что за рубежом он широко применяется, системного и всестороннего исследования российских пород овец с его помощью до сих пор не проводили. В настоящей работе впервые установлена принадлежность овец 25 российских пород к гаплогруппам и показаны гаплотипические связи между грубошерстными, тонкорунными и полутонкорунными породами на основе анализа полиморфизма последовательностей митохондриального гена CytB . Дана характеристика материнской изменчивости локальных пород овец в сравнении с трансграничными. Нашей целью было изучение генетического разнообразия и определение гаплотипической изменчивости и гаплогрупповой принадлежности российских локальных пород овец на основе последовательностей гена СytB . Исследования 25 российских пород овец проводили в 2020-2021 годах. Образцы ткани (ушной выщип) были взяты из биоколлекции «Банк генетического материала домашних и диких видов животных и птицы» (зарегистрирован Минобрнауки РФ № 498808), созданной и поддерживаемой в ФГБНУ ФИЦ животноводства - ВИЖ им. академика Л.К. Эрнста. Итоговая выборка для исследования включала девять тонкорунных пород: забайкальскую ( n = 3), дагестанскую горную ( n = 4), грозненскую ( n = 5), кулундинскую ( n = 5), манычского мериноса ( n = 5), сальскую ( n =5), советского мериноса ( n = 3), ставропольскую ( n = 5) и волгоградскую ( n = 5); пять полутонкорунных пород: горноалтайскую ( n = 5), куйбышевскую ( n = 1), северокавказскую ( n = 5), русскую длинношерстную ( n = 3) и цигайскую ( n = 2); одиннадцать грубошерстных пород: романовскую ( n = 3), андийскую черную ( n = 5), буубей ( n = 5), каракульскую ( n = 3), карачаевскую ( n = 5), кучугуровскую ( n = 3), лезгинскую ( n = 5), тушинскую ( n = 5), тувинскую короткожирнохвостую ( n = 4), эдильбаевскую ( n = 5) и калмыцкую ( n = 5). Полные последовательности гена CytB изучаемых пород овец определяли с использованием технологии секвенирования следующего поколения (next generation sequencing, NGS). Для этого были амплифицированы три перекрывающихся фрагмента мтДНК (область перекрытия более 290 п.н.) длиной 6500, 5700 и 6700 п.н. Полученные продукты полимеразной цепной реакции (ПЦР) использовали для подготовки библиотек, которые затем секвенировали методом парных концевых прочтений по 300 п.н. на приборе MiSeq («Illumina, Inc.», США). Последовательность гена СytB была восстановлена из полной последовательности мтДНК после ее выравнивания, выполненного с использованием MUSCLE алгоритма в программном обеспечении MEGA 7.0.26. Все изучаемые породы характеризовались высоким гаплотипическим (HD = 0,400-1,000) и нуклеотидным разнообразием (p = 0,00058-0,00760). В общей сложности идентифицировали 82 гаплотипа, при этом тувинская порода овец была представлена только одним. Анализ результатов AMOVA показал, что генетическое разнообразие в основном определяло внутрипородные различия (90,55 %). Были выявлены четыре гаплогруппы овец, включая A, B, C и D, что объясняется широким ареалом исследуемых животных. Наиболее распространенными среди локальных российских пород овец оказались гаплогруппы В ( n = 64) и А ( n = 34), характерные для овец европейского и азиатского происхождения. Всего 7 животных было отнесено к гаплогруппе С, а гаплогруппа D оказалась представлена одним животным. Полученные результаты внесут важный вклад в более глубокое понимание процессов миграции и расселения домашних овец на территории Евразии.
Домашние овцы, митохондриальная днк, ген цитохрома b, гаплогруппы, гаплотипы
Короткий адрес: https://sciup.org/142231904
IDR: 142231904 | УДК: 636.32/.38:575.162:577.2 | DOI: 10.15389/agrobiology.2021.6.1134rus
Текст научной статьи Оценка материнской изменчивости российских локальных пород овец на основе анализа полиморфизма гена цитохрома B
-
* При выполнении исследований было использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста. Работа проведена при финансовой поддержке Российского научного фонда (РНФ ¹ 21-66-00007). Пробы от овец были получены в рамках выполнения задания Министерства науки и высшего образования РФ (тема ¹ 0445-2019-0024).
овчина, смушки) (1). С момента одомашнивания (между 11000 и 10500 годами до н. э.) овцы распространились на всех континентах (2). В результате были получены разнообразные местные породы с уникальным набором признаков, обусловленных адаптацией и искусственным отбором (3).
Генетическое разнообразие (вариации аллелей и генотипов, присутствующих в популяции) отражает размер, историю, экологию и приспособляемость популяции (4) и играет важную роль в обеспечении формирования признаков, ответственных за улучшение, выживание и адаптацию биологического вида (5). Изменение климата, новые заболевания, дефицит земельных и водных ресурсов, меняющийся спрос рынка делают еще более важным сохранение и устойчивое использование генетических ресурсов животноводства (6). Изучение генетической изменчивости мирового генофонда современных аборигенных пород овец позволяет комплексно оценивать генетическое разнообразие и показатели отбора, углублять знания об их происхождении и расселении, а также о влиянии человеческой деятельности на этих животных с момента одомашнивания (7-9).
Однонуклеотидные полиморфизмы (single nucleotide polymorphisms, SNPs), хоть и широко используется при исследовании генома сельскохозяйственных животных (10, 11), представляют всего лишь один из типов общих геномных вариаций. Эффективным подходом к оценке генетического разнообразия, не потерявшим своей актуальности, остается исследование полиморфизма митохондриальной ДНК (мтДНК) (12, 13). мтДНК демонстрирует высокую степень полиморфизма и характеризуется отсутствием рекомбинации, что позволяет изучать генетические связи между породами и отслеживать как древние, так и относительно недавние эволюционные события.
Ранее филогенетические работы часто фокусировались на митохондриальных генах, кодирующих 12S и 16S рРНК, но их использование в широком таксономическом анализе сдерживается преобладанием вставок и де-леций (инделов), которые значительно усложняют выравнивание секвени-рованных нуклеотидных последовательностей (14). В связи с этим в качестве более подходящих мишеней в митохондриальном геноме рассматривают 13 белок-кодирующих генов, в которых редко встречаются инделы из-за сдвига рамки считывания.
Ген цитохрома b ( CytB ) обладает рядом преимуществ перед другими. Во-первых, он имеет более широкий диапазон филогенетического сигнала, чем остальные митохондриальные гены. Во-вторых, нуклеотиды в 3-й позиции кодонов гена CytB демонстрируют высокую частоту замен оснований, которая примерно в 3 раза превышает скорость для 12S или 16S рДНК, что приводит к ускорению молекулярной эволюции (15). В-третьих, этот ген достаточно быстро эволюционирует, что позволяет различать не только близкородственные виды, но и филогенетические группы внутри одного вида (16, 17).
Наиболее эффективным подходом для определения гаплогрупп мтДНК у овец служит секвенирование мтДНК. За рубежом этот подход широко применяется (18-20), однако системного и всестороннего исследования российских пород овец с его помощью до сих пор не проводили.
В 1996 году N.J. Wood с соавт. (19) идентифицировали две гапло-группы у домашних овец из Новой Зеландии, а в 1998 году S. Hiendleder с соавт. (20) после сравнения распределения гаплотипов в нескольких породах, отобранных в Германии, России и Казахстане, охарактеризовали выявленные гаплогруппы как азиатский (гаплогруппа А) и европейский (гаплогруппа В) типы. В 2005 году J. Guo с соавт. (18) и S. Pedrosa с соавт.
-
(21) на основании результатов исследований местных пород из Китая и Турции расширили состав гаплогрупп до трех общепризнанных филогенетических ветвей (гаплогруппа С). Последовательности гаплогруппы C были обнаружены с низкой частотой у овец, обитающих в Португалии (22), что позволяет предположить существование потока генов из Плодородного полумесяца на Пиренейский полуостров. Было также показано, что гапло-группа С содержит большее генетическое разнообразие, чем А или В (21), но в отличие от гаплогруппы В, она не соответствует ни одному из диких животных рода Ovis . В 2006 году M. Tapio с соавт. (23) обнаружили у одного карачаевского животного, отобранного на Северном Кавказе, последовательность контрольного региона, которая группировалась отдельно от трех определенных кластеров митохондриальной ДНК домашней овцы. Это исследование послужило доказательством наличия четвертой материнской линии, которая получила название D. В 2007 году на основе анализа полиморфизма фрагмента контрольной области и CytB мтДНК овец J.R. Meadows с соавт. (24) выделили пятую гаплогруппу Е.
Генетический анализ показал, что гаплогруппы А и В встречаются у домашних овец из всех географических регионов (средняя комбинированная частота 89 %). Гаплогруппа А в основном выявляется в азиатских популяциях (19, 25), однако гаплогруппа В имеет высокую частоту встречаемости в европейских и азиатских популяциях. Гаплогруппа С встречается реже (средняя частота 18 %): было выявлено лишь небольшое количество особей в Азии (в пределах Плодородного полумесяца) и Европе (в пределах Кавказа и Пиренейского полуострова) (23, 25, 26). Гаплогруппы D и E были идентифицированы совсем недавно и из пяти линий наименее представлены; овец с этими гаплогруппами до сих пор обнаруживали только на Кавказе и в Турции (23, 24).
Благодаря использованию мтДНК для определения генетического разнообразия овец было получено представление об истории одомашнивания овец и их глобальной миграции под влиянием человека (27). Так, в 2007 году индийские ученые V.C. Pardeshi с соавт. (28) впервые охарактеризовали разнообразие мтДНК трех пород индийских овец. Все породы принадлежали к материнской линии А. Индийская сеть не имела четко определенного центрального гаплотипа, было показано отсутствие обмена гаплотипами между индийскими популяциями и продемонстрирована устойчивая породная структура. Структура гаплотипов индийских овец свидетельствует о том, что история этих пород характеризовалась очень низкой частотой скрещивания между популяциями и полной репродуктивной изоляцией. Индийское овцеводство действительно основано на поддержании культурных и традиционных барьеров, которые препятствуют генетическому обмену между породами (28).
В 2013 году E. Zhao с соавт. (29) исследовали изменчивость мтДНК у местных овец, выращиваемых в семи регионах Китая. Филогенетический анализ последовательностей D-петли мтДНК 16 китайских коренных пород овец подтвердил наличие трех материнских гаплогрупп (А, В и С) с высоким генетическим разнообразием.
F.-H. Lv с соавт. (27) выделили два этапа миграции в истории восточно-евразийских овец. Авторы пришли к выводу, что регион Монгольского плато был вторичным центром расселения, действуя как «транспортный узел» в Восточной Евразии. Овцы из ближневосточного центра одомашнивания мигрировали через Кавказ и Центральную Азию и прибыли в Северный и Юго-Западный Китай (гаплогруппы А, В и С) и на Индийский субконтинент (гаплогруппы В и С) через этот регион (27).
Предполагаемое время расхождения между пятью основными гапло-группами произошло до одомашнивания, о чем свидетельствуют археологические данные (30). Например, время расхождения между двумя наиболее распространенными линиями A и B по оценкам, основанным на последовательностях гена цитохрома b ( CytB ), составляло 1,6-1,7 млн лет (20). Исходя из анализа последовательностей контрольной области CytB , S. Pedrosa с соавт. (21) предположили, что время расхождения линии C и линий A и B составляет соответственно ∼ 0,42-0,76 млн лет и ∼ 0,45-0,75 млн лет. Однако недавнее исследование (25) c использованием 12 белок-кодирующих генов продемонстрировало более позднее расхождение между линиями: 0,590±0,17 млн лет — между A и B, 0,26±0,09 млн лет — между C и E. В 2020 году J. Liu с соавт. (31) провели полногеномное исследование мтДНК тибетских овец и получили схожие результаты. Было доказано существование двух материнских линий (гаплогруппы А и В) с большим генетическим разнообразием у 15 популяций тибетских овец в Китае. Предками материнских линий могут быть муфлоны и аргали (31).
Исследования мтДНК овец за рубежом проводятся в широких масштабах, тогда как российские породы остаются малоизученными. Овцеводство всегда было важной отраслью животноводства в Российской Федерации, поскольку в первую очередь обеспечивало население шерстью, на которую был огромный спрос в связи с суровыми климатическими условиям. В 1990-е годы овцеводство России полностью обеспечивало внутренние потребности страны (32), однако к 2007 году наблюдается резкое сокращение численности овец (на 65 %) и текстильной отрасли (на 85-90 %) (33). К этому привело множество причин: невостребованность рынком тонкой и кроссбредной шерсти, смена форм собственности, диспаритет цен на промышленную и сельскохозяйственную продукцию, товарная интервенция (поступление дешевой импортной продукции из шерсти, хлопка, кожи на российский рынок), неподготовленность и незащищенность отечественного товаропроизводителя со стороны государства (34). В стране сократилось число лучших племенных заводов по разведению овец (35). Все это привело к экономическому упадку в отечественном овцеводстве.
В 35 регионах РФ разводят 43 породы овец, в том числе 15 тонкорунных (34,9 %), 12 — полутонкорунных (27,9 %), 2 — полугрубошерстных (4,7 %), 14 — грубошерстных (32,5 %) (36). Несмотря на проблемы, отечественное овцеводство начинает восстанавливаться. В современных условиях перспективным для повышения экономической эффективности овцеводческой отрасли считается максимальное использование потенциала мясной продуктивности разводимых пород. Это обусловлено существенной разницей в экономической значимости шерсти (5 % от общего дохода) и баранины (95 %). Основное внимание в настоящее время уделяется повышению мясной продуктивности овец. Из-за переориентации рынка доля шерстных пород значительно снизилась с 90,0 % в 1990 году до 55,2 % в 2020 году, а мясных, наоборот, увеличилась с 10,0 до 44,8 % (37, 38).
Подобные изменения влекут за собой серьезные последствия. Некоторые шерстные породы уже находятся на грани исчезновения. Большинство тонкорунных и полутонкорунных пород было создано на основе использования аборигенных овцематок в качестве материнской формы, которых улучшали баранами-производителями высокопродуктивных иностранных пород (39). Локальные грубошерстные породы создавались на основе генетических ресурсов аборигенных овец, история происхождения которых до сих пор до конца не выяснена.
В настоящей работе проведен анализ полиморфизма полной последовательности гена CytB у российских пород овец различного происхождения. Впервые установлена принадлежность овец из 25 российских пород к гаплогруппам и показаны гаплотипические связи между грубошерстными, тонкорунными и полутонкорунными породами на основе анализа полиморфизма последовательностей митохондриального гена CytB . Дана характеристика материнской изменчивости локальных пород овец в сравнении с трансграничными.
Нашей целью было изучение генетического разнообразия и определение гаплотипической изменчивости и гаплогрупповой принадлежности российских локальных пород овец на основе последовательностей гена CytB .
Методика . Исследования проводили в 2020-2021 годах. Образцы ткани 25 российских пород овец (ушной выщип) были взяты из биоколлекции «Банк генетического материала домашних и диких видов животных и птицы» (зарегистрирован Минобрнауки РФ ¹ 498808), созданной и поддерживаемой в ФГБНУ ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста.
Итоговая выборка включала девять тонкорунных пород: забайкальскую (BAKL, n = 3), дагестанскую горную (DGMT, n = 4), грозненскую (GRZY, n = 5), кулундинскую (KLND, n = 5), манычского мериноса (MNCM, n = 5), сальскую (SLSK, n =5), советского мериноса (SVTM, n = 3), ставропольскую (STVP, n = 5) и волгоградскую (VLGD, n = 5); пять полутонкорунных пород: горноалтайскую (ALTM, n = 5), куйбышевскую (KBSV, n = 1), северокавказскую (NCCS, n = 5), русскую длинношерстную (RSLH, n = 3) и цигайскую (TSIG, n = 2); одиннадцать грубошерстных пород: романовскую (RMNV, n = 3), андийскую черную (ANDB, n = 5), буубей (BUBI, n = 5), каракульскую (KRKL, n = 3), карачаевскую (KRCV, n = 5), кучугуровскую (KHGR, n = 3), лезгинскую (LZGN, n = 5), тушинскую (TSHN, n = 5), тувинскую короткожирнохвостую (TUVA, n = 4), эдильба-евскую (EDLB, n = 5) и калмыцкую (KLMY, n = 5).
ДНК выделяли с помощью набора ДНК-Экстран-2 (ООО «Синтол», Россия) в соответствии с рекомендациями производителя. Контроль качества полученных препаратов ДНК проводили в два этапа. На первом измеряли концентрацию (от 15 до 50 нг/мкл) на флуориметре Qubit 4.0 («Invit-rogen/Life Technologies», США), на втором — соотношение поглощения OD 260 /OD 280 (от 1,8 и выше) на спектрофотометре NanоDrop ™ 8000 («Ther-mo-Fisher Scientific, Inc.», США).
Полные последовательности гена CytB изучаемых пород овец определяли с использованием технологии секвенирования следующего поколения (next generation sequencing, NGS). Для этого были амплифицированы три перекрывающихся фрагмента мтДНК (область перекрытия более 290 п.н.) длиной 6500, 5700 и 6700 п.н. с использованием следующих пар праймеров: F1 5´-GTCCTCGCCCTAATCCTCTC-3´, R1 3´-AGGGTGCCGATATCTTT-GTG-5´; F2 5´-ACCCAAAACTCTTCGTGCTC-3´, R2 3´-GGAAGTCAGA-ATGCGATGGT-5´; F3 5´-ACACCAAACCCACGCTTATC-3´, R3 3´-GGGT-GTTGATAGTGGGGCTA-5´. Реакции проводили в конечном объеме 25 мкл: 10 мкл реакционного буфера (2,5½ HF Reaction buffer), 10,25 мкл H 2 O, 2,5 мкл dNTPs, 1 мкл смеси праймеров, 0,25 мкл SmartTaq HF-FuZZ ДНК полимеразы («Диалат Лтд.», Россия), 1 мкл ДНК. После начальной денатурации (2 мин при 94 ° С) проводили амплификацию в следующем температурно-временном режиме: 30 с при 94 ° С (1 цикл); 30 с при 61 ° С, 6,5 мин при 70 ° С (10 циклов); 30 с при 94 ° С, 30 с при 60 ° С, 3,5 мин при
70 ° С (25 циклов); заключительный этап — 10 мин при 72 ° С (термоциклер Applied Biosystems SimpliAmp («Thermo-Fisher Scientific, Inc.», США).
Полученные продукты полимеразной цепной реакции (ПЦР) очищали с помощью набора для очистки ДНК из агарозного геля и реакционных смесей Cleanup Standard (ЗАО «Евроген», Россия) и использовали для подготовки библиотек, которые затем секвенировали методом парных концевых прочтений по 300 п.н. на приборе MiSeq («Illumina, Inc.», США). Последовательность гена CytB была восстановлена из полной последовательности мтДНК после ее выравнивания, выполненного с использованием MUSCLE алгоритма (40) в программном обеспечении MEGA 7.0.26 (41).
Для построения медианной сети (42) использовали программное обеспечение PopART 1.7 (43). Наилучшие модели эволюции определяли в программе PartitionFinder 2 (44) с использованием скорректированного критерия информации Акайке (AICc) (45). Оптимальными оказались эволюционные модели HKY и HKY+I. Анализ AMOVA проводили в программе Arlequin 3.5.2.2 (46). Построение байесовского филогенетического дерева выполнили в программе MrBayes 3.2.7 (47) с последующей визуализацией в FigTree 1.4.3 (48). В качестве аутгруппы использовали последовательность гена CytB снежного барана ( Ovis nivicola ) (Gene Bank accession number NC_039431.1) (49).
Поиск по методу Монте-Карло с цепями Маркова был проведен с применением четырех цепей с использованием 10000000 шагов, деревья отбирались каждые 500 поколений (первые 25 % деревьев были отброшены как «горящие»). В программе DnaSP 6.12.01 (50) рассчитывали параметры генетического разнообразия: число полиморфных сайтов (S), среднее число нуклеотидных различий (K), число гаплотипов (H), гаплотипическое разнообразие (Hd), нуклеотидное разнообразие ( п ), ошибки средних арифметических (±SEM).
Результаты. Всего у изученных 106 домашних овец мы идентифицировали 82 гаплотипа. Группа тувинской короткожирнохвостой породы была представлена только одним гаплотипом.
1. Индексы генетического разнообразия в популяциях 25 российских локальных пород домашних овец ( Ovis aries ), рассчитанные на основе анализа нуклеотидной последовательности митохондриального гена CytB (ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста, Московская обл., 2020-2021 годы)
|
Популяция |
1 n 1 |
S |
K |
H |
Hd±SEM |
n ±SEM |
|
ALTM |
5 |
8 |
4,000 |
4 |
0,900±0,161 |
0,00351±0,00069 |
|
ANDB |
5 |
5 |
2,600 |
4 |
0,900±0,161 |
0,00228±0,00049 |
|
BAKL |
3 |
13 |
8,667 |
3 |
1,000±0,272 |
0,00760±0,00308 |
|
BUBI |
5 |
13 |
5,800 |
3 |
0,800±0,164 |
0,00509±0,00208 |
|
DGMT |
4 |
14 |
7,333 |
3 |
0,833±0,222 |
0,00643±0,00240 |
|
EDLB |
5 |
6 |
2,800 |
4 |
0,900±0,161 |
0,00246±0,00064 |
|
GRZY |
5 |
4 |
1,600 |
4 |
0,900±0,161 |
0,00140±0,00042 |
|
KBSV |
3 |
2 |
1,333 |
2 |
0,667±0,314 |
0,00117±0,00055 |
|
KHGR |
3 |
4 |
2,667 |
2 |
0,667±0,314 |
0,00234±0,00110 |
|
KLMY |
5 |
8 |
3,600 |
5 |
1,000±0,126 |
0,00316±0,00065 |
|
KLND |
5 |
5 |
2,000 |
4 |
0,900±0,161 |
0,00175±0,00051 |
|
KRCV |
5 |
13 |
5,600 |
4 |
0,900±0,161 |
0,00491±0,00220 |
|
KRKL |
3 |
6 |
4,000 |
3 |
1,000±0,272 |
0,00351±0,00141 |
|
LZGN |
5 |
15 |
8,000 |
5 |
1,000±0,126 |
0,00702±0,00160 |
|
MNCM |
5 |
7 |
2,800 |
5 |
1,000±0,126 |
0,00246±0,00051 |
|
NCCS |
5 |
3 |
1,200 |
2 |
0,400±0,400 |
0,00105±0,00062 |
|
RMNV |
3 |
2 |
1,333 |
2 |
0,667±0,314 |
0,00117±0,00055 |
|
RSLH |
3 |
4 |
2,667 |
3 |
1,000±0,272 |
0,00234±0,00068 |
|
SLSK |
5 |
6 |
2,400 |
4 |
0,900±0,161 |
0,00211±0,00065 |
|
STVP |
5 |
18 |
7,800 |
5 |
1,000±0,126 |
0,00684±0,00212 |
|
SVTM |
3 |
1 |
0,667 |
2 |
0,667±0,314 |
0,00058±0,00028 |
|
TSHN |
5 |
2 |
0,800 |
2 |
0,400±0,237 |
0,00070±0,00042 |
TSIG
TUVA
VLGD
2 11,000
4 00,000
5 187,600
2 1,000±0,500
1 0,000±0,000
5 1,000±0,126
Продолжение таблицы 1
0,00088±0,00044
0,00000±0,00000
0,00667±0,00183
Примечание. n — число образцов, S — число полиморфных сайтов, K — среднее число нуклеотидных различий, H — количество гаплотипов, Hd — гаплотипическое разнообразие, π — нуклеотидное разнообразие. ALTM — горноалтайская, ANDB — андийская, BAKL — забайкальская, BUBI — буубей, DGMT — дагестанская горная, EDLB — эдильбаевская, GRZY — грозненская, KBSV — куйбышевская, KHGR — кучугуровская, KLMY — калмыцкая, KLND — кулундинская, KRCV — карачаевская, KRKL — каракульская, LZGN — лезгинская, MNCM — манычский меринос, NCCS — северокавказская, RMNV — романовская, RSLH — русская длинношерстная, SLSK — сальская, STVP — ставропольская, SVTM — советский меринос, TSHN — тушинская, TSIG — цигайская, TUVA — тувинская, VLGD — волгоградская.
Наибольшее гаплотическое разнообразие (Hd = 1,000) наблюдалось у забайкальской, калмыцкой, каракульской, лезгинской, русской длинношерстной, ставропольской, цигайской и волгоградской пород и манычского мериноса (табл. 1). В северокавказской мясошерстной и тушинской породах обнаружили наименьшее разнообразие гаплотипов (Hd = 0,400). Самые низкие значения нуклеотидного разноообразия и наименьшее среднее число нуклеотидных различий зафиксировали в породе советский меринос ( π = 0,00058, K = 0,667). Забайкальская порода характеризовалась наивысшими значениями этих показателей ( π = 0,00760, K = 8,667).
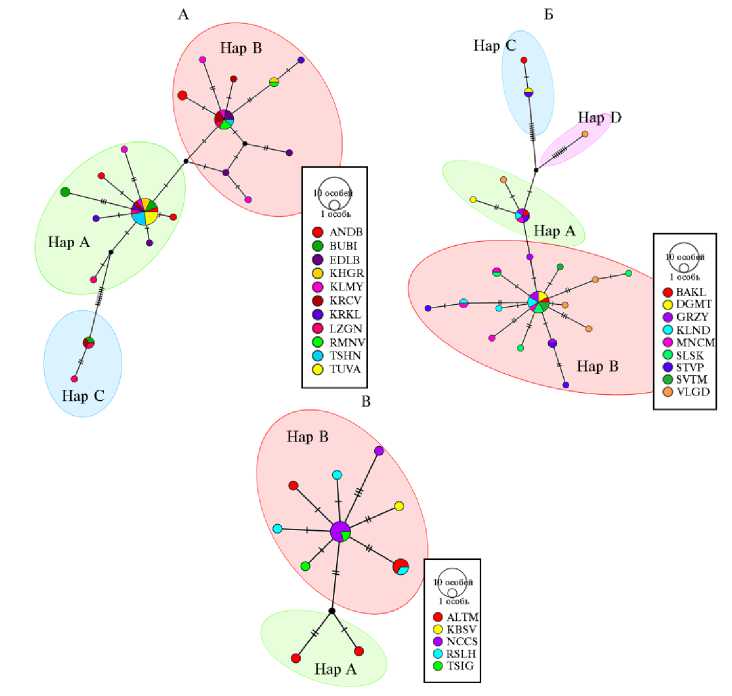
Рис. 1. Медианная сеть, характеризующая связи гаплотипов, идентифицированных у представителей 25 российских локальных пород домашних овец ( Ovis aries ) на основе анализа нуклеотидной последовательности митохондриального гена CytB (ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста, Московская обл., 2020-2021 годы) .
А — медианная сеть, построенная для овец грубошерстных пород: ANDB — андийская ( n = 5), BUBI — буубей ( n = 5), EDLB — эдильбаевская ( n = 5), KHGR — кучугуровская ( n = 3), KLMY — калмыцкая ( n = 5), KRCV — карачаевская ( n = 5), KRKL — каракульская
( n = 3), LZGN — лезгинская ( n = 5), RMNV — романовская ( n = 3), TSHN — тушинская ( n = 5), TUVA — тувинская короткожирнохвостая ( n = 4).
Б — медианная сеть, построенная для тонкорунных пород овец: BAKL — забайкальская ( n = 3), DGMT — дагестанская горная ( n = 4), GRZY — грозненская ( n = 5), KLND — кулундин-ская ( n = 5), MNCM — манычский меринос ( n = 5), SLSK — сальская ( n = 5), STVP — ставропольская ( n = 5), SVTM — советский меринос ( n = 3), VLGD — волгоградская (5).
В — медианная сеть, построенная для овец полутонкорунных пород: ALTM — горноалтайская ( n = 5), KBSV — куйбышевская ( n = 3), NCCS — северокавказская ( n = 5), RSLH — русская длинношерстная ( n = 3), TSIG — цигайская ( n = 2).
Hap A — гаплогруппа А, Hap B — гаплогруппа В, Hap C — гаплогруппа С, Hap D — гаплогруппа D. Диаметр круга соответствует числу особей, принадлежащих к соответствующему гаплотипу. Число поперечных линий указывает на число нуклеотидных замен. Черные круги в точках ветвления сети обозначают гипотетические гаплотипы.
Грубошерстные породы овец (рис. 1, А) сформировали три кластера, соответствующие гаплогруппам А, В и С. Породы характеризовались высоким генетическим разнообразием. Исключение составила тувинская короткожирнохвостая порода, все исследуемые особи которой принадлежали к одной гаплогруппе А. Животные остальных пород относились к различным гаплогруппам, что, вероятно, может косвенно указывать на смешанное происхождение изучаемых популяций. Тонкорунные породы овец (см. рис. 1, Б) также демонстрировали высокое гаплотипическое разнообразие. Как и в случае с грубошерстными овцами, животные одних и тех же пород, за исключением сальской, кластеризовались в разные гаплогруппы. В отличие от тувинской короткожирнохвостой, овцы сальской породы характеризовались более высоким нуклеотидным разнообразием и формировали различные гаплотипы внутри одной гаплогруппы В, которая оказалась наиболее многочисленной среди тонкорунных пород. Овцы, отнесенные к гаплогруппе С, отличались от животных, принадлежащих к гаплогруппе А, девятью нуклеотидными заменами. Среди тонкорунных овец одно животное волгоградской породы, отличающееся восьмью нуклеотидными заменами от гаплогрупп А и С, образовывало отдельный кластер — гаплогруппу D. Большинство овец полутонкорунных пород (см. рис. 1, В) относились к гаплогруппе В. Два животных горноалтайской породы кластеризовались отдельно в гаплогруппу А.
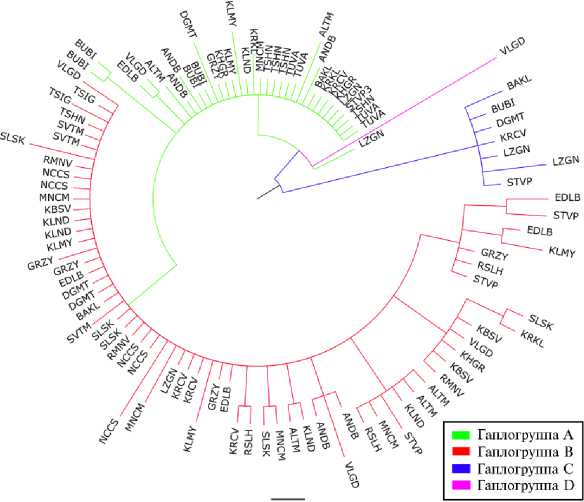
З.ОЕ-4
Рис. 2. Байесовское филогенетическое дерево, отражающее генетические связи представителей
25 российских локальных пород домашних овец ( Ovis aries ) на основе анализа нуклеотидной последовательности митохондриального гена CytB : ALTM — горноалтайская ( n = 5), ANDB — андийская ( n = 5), BAKL — забайкальская ( n = 3), BUBI — буубей ( n = 5), DGMT — дагестанская горная ( n = 4), EDLB — эдильбаевская ( n = 5), GRZY — грозненская ( n = 5), KBSV — куйбышевская ( n = 3), KHGR — кучугуровская ( n = 3), KLMY — калмыцкая ( n = 5), KLND — кулундинская ( n = 5), KRCV — карачаевская ( n = 5), KRKL — каракульская ( n = 3), LZGN — лезгинская ( n = 5), MNCM — манычский меринос ( n = 5), NCCS — северокавказская ( n = 5), RMNV — романовская ( n = 3), RSLH — русская длинношерстная ( n = 3), SLSK — сальская ( n = 5), STVP — ставропольская ( n = 5), SVTM — советский меринос ( n = 3), TSHN — тушинская ( n = 5), TSIG — цигайская ( n = 2), TUVA — тувинская ( n = 4), VLGD — волгоградская ( n = 5) (ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста, Московская обл., 2020-2021 годы).
Аналогичные выводы можно сделать на основании анализа байесов-сого филогенетического дерева (рис. 2). Наибольшее число животных были отнесены к гаплогруппе В; от гаплогруппы А отделялись два кластера, соответствующие гаплогруппам С и D.
Результаты анализа AMOVA, проведенного для трех групп овец (грубошерстные, тонкорунные и полутонкорунные породы), подтвердили наличие генетической дифференциации внутри пород, которые соответствовали 90,55 % изменчивости (табл. 2). Межпородные различия составили лишь 3,77 %. Между группами было обнаружено 5,68 % генетической вариации.
2. Результаты AMOVA (analysis of molecular variance) в популяциях 25 российских локальных пород домашних овец ( Ovis aries ), рассчитанные на основе анализа нуклеотидной последовательности митохондриального гена CytB ( n = 106, ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста, Московская обл., 20202021 годы)
|
Источник вариаций |
Степени свободы, df |
Сумма квадратов, SS |
Компоненты дисперсии, VS |
Процент вариации, V% |
|
Межгрупповые различия |
2 |
12,052 |
0,11615 |
5,68 |
|
Межпородные различия внутри группы |
22 |
47,906 |
0,77060 |
3,77 |
|
Внутрипородные различия |
81 |
150,033 |
1,85226 |
90,55 |
|
Общее |
105 |
209,991 |
2,04547 |
Домашняя овца — это традиционный и значимый вид сельскохозяйственных животных в России, который обеспечивает продуктами питания население и сырьем легкую промышленность (1). Однако генетические ресурсы российских популяций овец охарактеризованы в большей мере по ядерным молекулярно-генетическим маркерам, таким как однонуклеотидные полиморфизмы (SNPs) (51) и микросателлиты (52).
В выполненном нами исследовании полиморфизма CytB мтДНК получены новые знания о материнской изменчивости и генетическом разнообразии российских пород овец.
Разнообразие гаплотипов в российских популяциях овец (Hd = 0,4001,000) было сопоставимо со значениями, полученными в других исследованиях на тибетских (Hd = 0,464-1,000) (31) и марокканских овцах (Hd = 0,9630,996) (53). Нуклеотидное разнообразие ( π = 0,0000-0,00760) оказалось несколько ниже, чем у мексиканских ( п = 0,00041-0,90000) (54) и марокканских овец ( п = 0,01330-0,02260) (53), и близко к значениям, полученным у тибетских овец ( п = 0,00100-0,00600) (31). Следовательно, генетическое и нуклеотидное разнообразие российских овец не сильно отличалось от значений, представленных ранее, что может косвенно свидетельствовать об адекватности нашего подхода к расчетам показателей.
Согласно результатам анализа AMOVA, генетическое разнообразие в основном определяло внутрипородные различия (90,55 %). Мы получили результаты, схожие с данными J.A. Oliveira с соавт. (55), которые сообщали, что 91,54 % генетической вариации было обусловлено внутрипородными различиями у бразильских овец, выращенных в штате Мату-Гросу-ду-Сул
(Mato Grosso do Sul).
На основе нуклеотидной последовательности мтДНК были определены четыре гаплогруппы овец, включая A, B, C и D (18-21, 23). Наиболее распространенными среди локальных российских пород овец оказались гаплогруппы В и А, характерные для овец европейского и азиатского происхождения. Такой результат был ожидаем и согласуется с данными, полученными ранее N.J. Wood с соавт. (19), S. Hiendleder с соавт. (20) и J.R. Meadows с соавт. (25). В 1996 году N.J. Wood с соавт. (19) идентифицировали две гаплогруппы (А и В) у домашних овец из Новой Зеландии, причем в азиатских популяциях преобладала гаплогруппа А. В 1998 году S. Hiendleder с соавт. (20) охарактеризовали выявленные гаплогруппы как азиатский (гаплогруппа А) и европейский (гаплогруппа В) тип происхождения. Гаплогруппа В преобладала среди европейских пород, находясь в то же время в меньшинстве в Восточной Азии. В 2011 году J.R. Meadows с соавт. (25) получили схожие результаты: наиболее распространенными оказались гаплогруппы A и B ( ∼ 89 %). У гаплогруппы А наблюдалась высокая частота встречаемости ( ∼ 77 %) на Индийском субконтиненте, в то время как в Европе ее частота составляла менее 10 %. Линия B, напротив, встречалась в основном в Европе, с самой высокой частотой (> 90 %) — в Юго-Западной Европе.
В нашем исследовании среди местных российских пород овец также была обнаружена гаплогруппа С. Как и в более ранней работе (22, 23, 26), гаплогруппа С встречалась реже, и было выявлено лишь небольшое число особей в Азии и Европе. Кроме того, одно животное из Волгоградской области кластеризовалось с гаплогруппой D. В 2006 году M. Tapio с соавт. (23) обнаружили гаплогруппу D у одного животного карачаевской породы на Северном Кавказе, что свидетельствует о присутствии этого материнского типа на территории России.
Таким образом, проведенный нами анализ полиморфизма гена CytB мтДНК домашней овцы показал большое генетическое разнообразие российских пород овец. Были обнаружены четыре гаплогруппы (А, В, С и D), что можно объяснить широким ареалом исследуемых животных. Кроме того, разнообразие представленных гаплогрупп (в том числе присутствие азиатского и европейского филогенетического корня) косвенно указывает на миграцию домашних овец на территории Евразии, включая Российскую Федерацию, в двух направлениях.
Список литературы Оценка материнской изменчивости российских локальных пород овец на основе анализа полиморфизма гена цитохрома B
- Chessa B., Pereira F., Arnaud F., Amorim A., Goyache F., Mainland I., Kao R.R., Pember-ton J.M., Beraldi D., Stear M.J., Alberti A., Pittau M., Iannuzzi L., Banabazi M.H., Kazwala R.R., Zhang Y.P., Arranz J.J., Ali B.A., Wang Z., Uzun M., Dione M.M., Olsaker I., Holm L.E., Saarma U., Ahmad S., Marzanov N., Eythorsdottir E., Holland M.J., Ajmone-Mar-san P., Bruford M.W., Kantanen J., Spencer T.E., Palmarini M. Revealing the history of sheep domestication using retrovirus integrations. Science, 2009, 324(5926): 532-536 (doi: 10.1126/sci-ence.1170587).
- Zeder M.A. Domestication and early agriculture in the Mediterranean Basin: origins, diffusion, and impact. Proceedings of the National Academy of Sciences, 2008, 105(33): 11597-11604 (doi: 10.1073/pnas.0801317105).
- Diamond J. Evolution, consequences and future of plant and animal domestication. Nature, 2002, 418(6898): 700-707 (doi: 10.1038/nature01019).
- Sheriff O., Alemayehu K. Genetic diversity studies using microsatellite markers and their contri-bution in supporting sustainable sheep breeding programs: a review. Cogent Food & Agriculture, 2018, 4(1): 1459062 (doi: 10.1080/23311932.2018.1459062).
- Zhong Y., Tang Z., Huang L., Wang D., Lu Z. Genetic diversity of Procambarus clarkii popula-tions based on mitochondrial DNA and microsatellite markers in different areas of Guangxi, China. Mitochondrial DNA. Part A, 2020, 31(2): 48-56 (doi: 10.1080/24701394.2020.1721484).
- FAO. The state of food and agriculture: climate change, agriculture, and food security. Food and Agriculture Organization of the United Nations, Rome, 2016.
- Wei C., Wang H., Liu G., Wu M., Cao J., Liu Z., Liu R., Zhao F., Zhang L., Lu J., Liu C., Du L. Genome-wide analysis reveals population structure and selection in Chinese indigenous sheep breeds. BMC Genomics, 2015, 16(1): 194 (doi: 10.1186/s12864-015-1384-9).
- Wang H., Zhang L., Cao J., Wu M., Ma X., Liu Z., Liu R., Zhao F., Wei C., Du L. Genome-wide specific selection in three domestic sheep breeds. PLoS ONE, 2015, 10(6): e0128688. (doi: 10.1371/journal.pone.0128688).
- Fariello M.-I., Servin B., Tosser-Klopp G., Rupp R., Moreno C., International Sheep Genomics Consortium, San Cristobal M., Boitard S. Selection signatures in worldwide sheep popula-tions. PLoS ONE, 2014, 9(8): e103813 (doi: 10.1371/journal.pone.0103813).
- Денискова Т.Е., Доцев А.В., Форнара М.С., Сермягин А.А., Reyer H., Wimmers К., Brem G., Зиновьева Н.А. Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира. Сельскохозяйственная биология, 2020, 55(2): 285-294 (doi: 10.15389/agrobiology.2020.2.285rus).
- Костюнина О.В., Абдельманова А.С., Мартынова Е.У., Зиновьева Н.А. Поиск геномных областей, несущих летальные рецессивные варианты у свиней породы дюрок. Сельскохо-зяйственная биология, 2020, 55(2): 275-284 (doi: 10.15389/agrobiology.2020.2.275rus).
- Сулимова Г.Е., Столповский Ю.А., Рузина М.Н., Захаров-Гезехус И.А. Мониторинг генофондов популяций животных в связи с задачами селекции и изучения филогении. В сб.: Биоразнообразие и динамика генофондов. М., 2008: 211-214.
- Qiao G., Zhang H., Zhu S., Yuan C., Zhao H., Han M., Yue Y., Yang B. The complete mito-chondrial genome sequence and phylogenetic analysis of Alpine Merino sheep (Ovis aries). Mito-chondrial DNA. Part B, Resources, 2020, 5(1): 990-991 (doi: 10.1080/23802359.2020.1720536).
- Doyle J.J., Gaut B.S. Evolution of genes and taxa: a primer. Plant Molecular Biology, 2000, 42(1): 1-23.
- Knowlton N., Weigt L. New dates and new rates for divergence across the Isthmus of Panama. Proceedings of the Royal Society of London. Series B: Biological Sciences, 1998, 265: 2257-2263 (doi: 10.1098/rspb.1998.0568).
- Cox A.J., Hebert P.D. Colonization, extinction, and phylogeographic patterning in a freshwater crustacean. Molecular Ecology, 2001, 10(2): 371-386 (doi: 10.1046/j.1365-294x.2001.01188.x).
- Wares J.P., Cunningham C.W. Phylogeography and historical ecology of the North Atlantic in-tertidal. Evolution, 2001, 55(12): 2455-2469 (doi: 10.1111/j.0014-3820.2001.tb00760.x).
- Guo J., Du L.-X., Ma Y.-H., Guan W.-J., Li H.-B., Zhao Q.-J., Li X., Rao S.-Q. A novel maternal lineage revealed in sheep (Ovis aries). Animal Genetics, 2005, 36(4): 331-336 (doi: 10.1111/j.1365-2052.2005.01310.x).
- Wood N.J., Phua S.H. Variation in the control region sequence of the sheep mitochondrial genome. Animal Genetics, 1996, 27(1): 25-33 (doi: 10.1111/j.1365-2052.1996.tb01173.x).
- Hiendleder S., Mainz K., Plante Y., Lewalski H. Analysis of mitochondrial DNA indicates that domestic sheep are derived from two different ancestral maternal sources: no evidence for contri-butions from urial and argali sheep. The Journal of Heredity, 1998, 89(2): 113-120 (doi: 10.1093/jhered/89.2.113).
- Pedrosa S., Uzun M., Arranz J.J., Gutiérrez-Gil B., San Primitivo F., Bayón Y. Evidence of three maternal lineages in Near Eastern sheep supporting multiple domestication events. Proceedings of the Royal Society B: Biological Sciences, 2005, 272(1577): 2211-2217 (doi: 10.1098/rspb.2005.3204).
- Pereira F., Davis S.J., Pereira L., McEvoy B., Bradley D.G., Amorim A. Genetic signatures of a Mediterranean influence in Iberian Peninsula sheep husbandry. Molecular Biology and Evolution, 2006, 23(7): 1420-1426 (doi: 10.1093/molbev/msl007).
- Tapio M., Marzanov N., Ozerov M., Cinkulov M., Gonzarenko G., Kiselyova T., Murawski M., Viinalass H., Kantanen J. Sheep mitochondrial DNA variation in European, Caucasian, and Central Asian areas. Molecular Biology and Evolution, 2006, 23(9): 1776-1783 (doi: 10.1093/mol-bev/msl043).
- Meadows J.R., Cemal I., Karaca O., Gootwine E., Kijas J.W. Five ovine mitochondrial lineages identified from sheep breeds of the near East. Genetics, 2007, 175(3): 1371-1379 (doi: 10.1534/ge-netics.106.068353).
- Meadows J.R., Hiendleder S., Kijas J.W. Haplogroup relationships between domestic and wild sheep resolved using a mitogenome panel. Heredity, 2011, 106(4): 700-706 (doi: 10.1038/hdy.2010.122).
- Meadows J.R., Li K., Kantanen J., Tapio M., Sipos W., Pardeshi V., Gupta V., Calvo J.H., Whan V., Norris B., Kijas J.W. Mitochondrial sequence reveals high levels of gene flow between breeds of domestic sheep from Asia and Europe. The Journal of Heredity, 2005, 96(5): 494-501 (doi: 10.1093/jhered/esi100).
- Lv F.-H., Peng W.-F., Yang J., Zhao Y.-X., Li W.-R., Liu M.-J., Ma Y.-H., Zhao Q.-J., Yang G.-L., Wang F., Li J.-Q., Liu Y.-G., Shen Z.-Q., Zhao S.-G., Hehua E., Gorkhali N.A., Farhad Vahidi S.M., Muladno M., Naqvi A.N., Tabell J., Iso-Touru T., Bruford M.-W., Kan-tanen J., Han J.-L., Li M.-H. Mitogenomic meta-analysis identifies two phases of migration in the history of eastern Eurasian sheep. Molecular Biology and Evolution, 2015, 32(10): 2515-2533 (doi: 10.1093/molbev/msv139).
- Pardeshi V.C., Kadoo N.Y., Sainani M.N., Meadows J.R., Kijas J.W., Gupta V.S. Mitochondrial haplotypes reveal a strong genetic structure for three Indian sheep breeds. Animal Genetics, 2007, 38(5): 460-466 (doi: 10.1111/j.1365-2052.2007.01636.x).
- Zhao E., Yu Q., Zhang N., Kong D., Zhao Y. Mitochondrial DNA diversity and the origin of Chinese indigenous sheep. Tropical Animal Health and Production, 2013, 45(8): 1715-1722 (doi: 10.1007/s11250-013-0420-5).
- Zeder M.A. Domestication and early agriculture in the Mediterranean Basin: Origins, diffusion, and impact. Proceedings of the National Academy of Sciences, 2008, 105(33): 11597-11604 (doi: 10.1073/pnas.0801317105).
- Liu J., Lu Z., Yuan C., Wang F., Yang B. Phylogeography and phylogenetic evolution in Tibetan sheep based on MT-CYB sequences. Animals, 2020, 10(7): 117 (doi: 10.3390/ani10071177).
- Ульянов А.Н., Куликова А.Я. Состояние и резервы породного генофонда овцеводства Рос-сии. Овцы, козы, шерстяное дело, 2012, 1: 4-11.
- Абонеев В.В. Стратегия развития овцеводства в Российской Федерации. Достижения науки и техники в АПК, 2008, 10: 37-39.
- Ерохин А.И., Карасев Е.А., Ерохин С.А. Состояние, динамка и тенденции в развитии овцеводства в мире и в России. Овцы, козы, шерстяное дело, 2019, 3: 3-6.
- Абонеев В.В., Колосов Ю.А. О проблемах сохранения племенных ресурсов овцеводства России. Овцы, козы, шерстяное дело, 2020, 1: 43-46.
- Селионова М.И Сохранение и рациональное использование генетических ресурсов овец и коз. Животноводство и кормопроизводство, 2019, 102(4): 272-276.
- Lescheva M., Ivolga A. Current state and perspectives of sheep breeding development in Russian modern economic condition. Economics of Agriculture, 2015, 2(62): 467-480.
- Дунин И.М., Сафина Г.Ф., Чернов В.В., Григорян Л.Н., Хататаев С.А., Хмелевская Г.Н., Степанова Н.Г., Павлов М.Б. Состояние овцеводства России и его племенных ресурсов. В сб.: Ежегодник по племенной работе в овцеводстве и козоводстве в хозяйствах российской федерации (2019 год). М., 2020: 3-14.
- Эрнст Л.К., Дмитриев Н.Г., Паронян И.А. Генетические ресурсы сельскохозяйственных животных в России и сопредельных странах. СПб, 1994.
- Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nu-cleic Acids Research, 2004, 32(5): 1792-1797 (doi: 10.1093/nar/gkh340).
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874 (doi: 10.1093/mol-bev/msw054).
- Bandelt H.J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37-48 (doi: 10.1093/oxfordjournals.mol-bev.a026036).
- Leigh J.W., Bryant D. Popart: Full-feature software for haplotype network construction. Methods in Ecology and Evolution, 2015, 6(9): 1110-1116 (doi: 10.1111/2041-210X.12410).
- Lanfear R., Frandsen P.B., Wright A.M., Senfeld T., Calcott B. PartitionFinder 2: new methods for selecting partitioned models of evolution for molecular and morphological phylogenetic anal-yses. Molecular Biology and Evolution, 2017, 34(3): 772-773 (doi: 10.1093/molbev/msw260).
- Akaike H. A new look at the statistical model identification. IEEE Trans Auto Control, 1974, 19(6): 716-723 (doi: 10.1109/TAC.1974.1100705).
- Excoffier L., Lischer H.E. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10(3): 564-567 (doi: 10.1111/j.1755-0998.2010.02847.x).
- Ronquist F., Teslenko M., van der Mark P., Ayres D.L., Darling A., Höhna S., Larget B., Liu L., Suchard M.A., Huelsenbeck J.P. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 2012, 61(3): 539-542 (doi: 10.1093/sysbio/sys029).
- Molecular evolution, phylogenetics and epidemiology. Режим доступа: http://tree.bio.ed.ac.uk/soft-ware/figtree. Дата обращения: 30.07.2021.
- Dotsev A.V., Kunz E., Shakhin A.V., Petrov S.N., Kostyunina O.V., Okhlopkov I.M., Denis-kova T.E., Barbato M., Bagirov V.A., Medvedev D.G., Krebs S., Brem G., Medugorac I., Zi-novieva N.A. The first complete mitochondrial genomes of snow sheep (Ovis nivicola) and thin-horn sheep (Ovis dalli) and their phylogenetic implications for the genus Ovis. Mitochondrial DNA Part B, 2019, 4(1): 1332-1333 (doi: 10.1080/23802359.2018.1535849).
- Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C., Guirao-Rico S., Librado P., Ramos-On-sins S.E., Sánchez-Gracia A. DnaSP 6: DNA sequence polymorphism analysis of large data sets Molecular Biology and Evolution, 2017, 34: 3299-3302 (doi: 10.1093/molbev/msx248).
- Deniskova T.E., Abelmanova A.S., Dotsev A.V., Sambu-Khoo Ch.S., Reyer H., Selionova M.I., Fornara M.S., Wimmers K., Brem G., Zinovieva N.A. PSX-18 High-density genomic description of Russian native sheep breed of the Republic of Tyva. Journal of Animal Science, 2020, 98(4): 453-454 (doi: 10.1093/jas/skaa278.789).
- Денискова Т.Е., Селионова М.И., Гладырь Е.А., Доцев А.В., Бобрышова Г.Т., Костю-нина О.В., Брем Г., Зиновьева Н.А. Изменчивость микросателлитов в породах овец, разводимых в России. Сельскохозяйственная биология, 2016, 51(6): 801-810 (doi: 10.15389/agrobiology.2016.6.801rus).
- Kandoussi A., Boujenane I., Auger C., Serranito B., Germot A., Piro M., Maftah A., Badaoui B., Petit D. The origin of sheep settlement in Western Mediterranean. Scientific Reports, 2020, 10(1): 10225 (doi: 10.1038/s41598-020-67246-5).
- Alonso R.A., Ulloa-Arvizu R., Gayosso-Vázquez A. Mitochondrial DNA sequence analysis of the Mexican Creole sheep (Ovis aries) reveals a narrow Iberian maternal origin. Mitochondrial DNA. Part A, 2017, 28(6): 793-800 (doi: 10.1080/24701394.2016.1192613).
- Oliveira J.A., Egito A.A.D., Crispim B.D.A., Vargas Junior F.M., Seno L.O., Barufatti A. Im-portance of naturalized breeds as a base for the formation of exotic sheep (Ovis aries) breeds in tropical altitude regions. Genetics and Molecular Biology, 2020, 43(2): e20190054 (doi: 10.1590/1678-4685-GMB-2019-0054).


