Оценка меры информационного полиморфизма генетического разнообразия
Автор: Чесноков Ю.В., Артемьева А.М.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Биоинформатика и математическая статистика
Статья в выпуске: 5 т.50, 2015 года.
Бесплатный доступ
Одна из основных целей генетики растений и животных - это идентификация и картирование генов. При определении генетического сцепления обычно стараются установить, какие маркерные локусы (маркеры) имеют аллели, косегрегирующие с аллелями желаемого локуса. Пригодность маркера для указанных целей зависит от числа аллелей, которые имеет этот маркер, и их соответствующих относительных частот. Количественно степень полиморфизма обычно измеряется двумя различными величинами, или показателями (мерами), - гетерозиготностью (heterozygosity, Н), для которой объективный алгоритм оценки и формула изменчивости хорошо известны (M. Nei с соавт., 1974; M. Nei с соавт., 1979), и величиной информационного полиморфизма (рolymorphism information content, PIC) (D. Botstein с соавт., 1980). Исходя из этого в работе на основе данных литературы описаны статистические подходы, применяемые для анализа информационного полиморфизма. Рассмотрены меры информационного полиморфизма, гетерозиготности и некоторых сопутствующих величин, определяемых при оценке генетического разнообразия как на межвидовом, так и на внутривидовом популяционном уровне. Мера, или величина, информационного полиморфизма (PIC) определяется способностью маркера устанавливать полиморфизм популяции в зависимости от числа обнаруживаемых аллелей и распределения их частот (D. Botstein c соавт., 1980). Таким образом, PIC выявляет дискриминационную способность маркера, фактически зависит от числа известных (устанавливаемых) аллелей и распределения их частот и тем самым эквивалентна генному разнообразию. Для доминантных маркеров максимальное значение PIC составляет 0,5. Следует отметить, что в случае маркеров с равным распределением частот внутри популяции величина PIC выше. Маркеры с множественными аллелями имеют еще большие значения этого показателя, однако при этом величина PIC также зависит от распределения частот аллелей. С помощью 21 пары SSR (simple sequence repeats) и 12 пар S-SAP (sequence specific amplified polymorphism) праймеров у 96 образцов Brassica rapa L. из стержневой коллекции ВИР мы обнаружили 135 SSR и 123 S-SAP полиморфных маркера. Среднее значение PIC для обоих типов маркеров - 0,316, тогда как для микросателлитных маркеров - 0,257, для S-SAP маркеров - 0,379, то есть в среднем на 50 % выше. Ожидаемую (H E) гетерозиготность обычно определяют, когда описывают генетическое разнообразие, поскольку она менее чувствительна к размеру выборки, чем наблюдаемая гетерозиготность (Н О). Если H O и H E схожи (достоверно не различаются), скрещивание в популяции происходит практически случайно. При H OE, популяция инбредная. Если H O > H E, то в популяции система случайного скрещивания преобладает над инбридингом. Эффективное мультиплексное отношение (effective multiplex ratio, EMR) определяют как произведение общего числа полиморфных локусов (на праймер) и доли полиморфных локусов от их общего числа (W. Powell с соавт., 1996; J. Nagaraju с соавт., 2001). Маркерный индекс (marker index, MI) - статистическая величина, используемая для оценки суммарной пригодности маркерной системы (чем выше значение MI для методики, тем она лучше) (W. Powell с соавт., 1996; J. Nagaraju с соавт., 2001). Чтобы отразить способность сочетания «праймер-применяемая методика» устанавливать различия между большим числом генотипов, используют показатель разрешающей способности (resolving power, Rp) (J.E. Gilbert с соавт., 1999; A. Prevost с соавт., 1999). Представлена информация о некоторых продуктах программного обеспечения, которое может быть использовано для расчета величины информационного полиморфизма и гетерозиготности. Приведены формулы для установления эффективного мультиплексного отношения, маркерного индекса и показателя разрешающей способности комбинации «праймер-применяемая методика».
Гетерозиготность, величина информационного полиморфизма, эффективное мультиплексное отношение, маркерный индекс, показатель разрешающей способности, программное обеспечение
Короткий адрес: https://sciup.org/142133619
IDR: 142133619 | УДК: 575.17:575.118.5:575.162:57.087.1 | DOI: 10.15389/agrobiology.2015.5.571rus
Текст научной статьи Оценка меры информационного полиморфизма генетического разнообразия
Одна из основных целей генетики растений и животных — идентификация и картирование генов, определяющих проявление интересующих исследователя признаков. Существует большое число маркерных локусов, визуализируемых с помощью различных маркерных систем (кратко
Работа выполнена при частичной поддержке РФФИ (грант ¹ 13-04-00128-а).
их называют маркерами), чьи позиции и порядок, в котором они располагаются на хромосоме, хорошо известны. При определении генетического сцепления обычно стараются установить, какие маркерные локусы (маркеры) имеют аллели, косегрегирующие с аллелями желаемого локуса. Пригодность маркера для указанных целей зависит от числа аллелей, которые имеет этот маркер, и их соответствующих относительных частот. Качественно маркер характеризуется как полиморфный, если он представлен, как минимум, двумя аллелями и его наиболее часто встречающийся аллель в популяции имеет частоту не менее 99 %. Количественно степень полиморфизма обычно измеряется двумя различными величинами, или показателями (мерами). Одна известна как гетерозиготность (heterozygosity, Н) и ее объективный алгоритм оценивания и формула изменчивости хорошо известны (1, 2). Другой единицей измерения служит величина информационного полиморфизма (рolymorphism information content, PIC) (3).
Молекулярные маркеры стали эффективным инструментом и средством, с помощью которого оценивают и характеризуют как внутри-, так и межвидовое генетическое разнообразие. Маркерные системы различают по мере (то есть величине) их информативности, что, в свою очередь, зависит от степени их полиморфизма. Концепцию полиморфизма используют для определения генетической изменчивости в популяции, что в последние десятилетия стало предметом интенсивного изучения в различных научных дисциплинах — генетике, экологии, ботанике, зоологии и некоторых других. Примеры этому многочисленны и очевидны (4-10). Однако при планировании применения молекулярных маркеров для какого-либо исследования или для практического использования в селекционных программах неизбежно возникают вопросы, на которые исследователям зачастую приходится искать ответ. Насколько трудно будет найти пригодные для планируемой работы полиморфные локусы? Как много маркеров необходимо будет задействовать? Насколько полиморфным должен быть каждый подобранный маркер? На все эти вопросы можно найти ответ, оценив меру информативности маркеров. Двумя основными параметрами, определяемыми для этого, являются гетерозиготность (Н) и величина информационного полиморфизма (PIC). В дополнение к ним существуют некоторые сопутствующие показатели, с помощью которых можно также установить эффективность выбранной системы «праймер—маркер» и(или) избранного методического подхода.
В настоящей работе кратко обобщены описанные в научной литературе статистические подходы, используемые для установления меры информационного полиморфизма, гетерозиготности и некоторых сопутствующих величин, определяемых при оценке генетического разнообразия как на межвидовом, так и на внутривидовом популяционном уровнях.
Гетерозиготность (Н). Гетерозиготность локуса, которая определяется как вероятность того, что в популяции особь гетерозиготна по этому локусу (11), может быть рассчитана по формуле:
l
H = 1 - ∑ P i 2 , [1] i = 1
где P i — частота i -го аллеля среди общего числа l аллелей. Иными словами, геретозиготность может быть рассмотрена как средняя порция локусов с двумя различными аллелями в одном локусе у одной особи. Обычно это распространяется на всю популяцию или какую-то ее часть и подразделяется на наблюдаемую и ожидаемую гетерозиготность. Ожидаемая (expected) гетерозиготность (HE), или генное разнообразие по M. Nei (1), — это ожидаемая вероятность того, что особь будет гетерозиготна по соответст-572
вующему локусу в мультилокусных системах (для всех анализируемых локусов). Другими словами, это установленная фракция всех особей, которые были бы гетерозиготны по любому случайно выбранному локусу. Ее часто высчитывают, основываясь на установлении квадратного корня из частоты нуль-аллеля (рецессивного), следующим образом (подобно уравнению [1]): H E = 1 – ∑ i n p i 2 , где p i — частота i -го аллеля, n i — общее число аллелей во всех локусах. Наблюдаемая (observed) гетерозиготность (HO) — это часть генов, которые в популяции гетерозиготны. Она рассчитывается для каждого локуса как общее число гетерозигот, поделенное на размер выборки. Значения для HE и HO варьируют от 0 (нет гетерозиготности) до практически 1 (большое число аллелей с равной частотой встречаемости). Ожидаемую гетерозиготность обычно определяют, когда описывают генетическое разнообразие, поскольку она менее чувствительна к размеру выборки, чем наблюдаемая гетерозиготность. Если HO и HE схожи (достоверно не различаются), то скрещивание в популяции происходит практически случайно. При HO< HE, популяция инбредная. Если HO > HE, то в популяции система случайного скрещивания преобладает над инбридингом.
Мера информационного полиморфизма (PIC). Мера, или величина, информационного полиморфизма (рolymorphism information content — PIC) определяется способностью маркера устанавливать полиморфизм в популяции в зависимости от числа обнаруживаемых аллелей и распределения их частот (3). Таким образом, PIC выявляет дискриминационную способность маркера, фактически зависит от числа известных (устанавливаемых) аллелей и распределения их частот и тем самым эквивалентна генному разнообразию. В самой простой форме величина PIC может быть рассчитана подобно гетерозиготности (см. уравнение [1]):
n
PIC j = 1 - ∑ P i 2 , [2] i = 1
где i — i -й аллель j -го маркера, n — число аллелей j -го маркера, Р — частота аллелей. Примеры расчетов значения PIC для биаллельного и мультиаллель-ного маркеров представлены в таблице 1. В то же время для кодоминантных маркеров уравнение [2] может быть представлено следующим образом (12):
PIC = 1 - ( k ∑ i = 1 P i 2 ) - k - 1 ∑ i = 1k ∑ j = 1 2 P i 2 P j 2 , [3] где k — число аллелей, P i и P j — частота соответственно i -го и j -го аллеля в популяции. Для доминантных маркеров величину PIC рассчитывают согласно описанию (13):
PIC = 1 - [f2 + (1 - f)2], [4] где f — частота маркера в наборе данных. Для доминантных маркеров максимальное значение PIC составляет 0,5. Следует отметить, что в случае маркеров с равным распределением частот внутри популяции величина PIC выше. Маркеры же с множественными аллелями имеют еще большие значения этого показателя, однако при этом величина значения PIC также зависит от распределения частот аллелей (табл. 1).
1. Примеры расчета PIC для биаллельного и мультиаллельного маркеров
|
Частота аллелей |
1 Формула расчета по уравнению [2] [ |
Значение PIC |
|
Р 1 = 0,5; Р 2 = 0,5 |
Биаллельный маркер 1 - (0,52 + 0,52) |
0,50 |
|
Р 1 = 0,4; Р 2 = 0,6 |
1 - (0,42 + 0,62) |
0,48 |
|
Р 1 = 0,3; Р 2 = 0,7 |
1 - (0,32 + 0,72) |
0,42 |
|
Р 1 = 0,2; Р 2 = 0,8 |
1 - (0,22 + 0,82) |
0,32 |
|
Р 1 = 0,1; Р 2 = 0,9 |
1 - (0,12 + 0,92) |
0,18 |
Продолжение таблицы 1
|
Мультиаллельный маркер |
||||
|
Р 1 = 0,33; Р 2 = 0,33; Р 3 = 0,33 |
1 - (0,332 + 0,332 + 0,332) |
0,67 |
||
|
Р 1 = 0,4 |
; Р 2 = 0,3 |
; Р 3 = 0,3 |
1 - (0,42 + 0,32 + 0,32) |
0,66 |
|
Р 1 = 0,4 |
; Р 2 = 0,4 |
; Р 3 = 0,2 |
1 - (0,42 + 0,42 + 0,22) |
0,64 |
|
Р 1 = 0,5 |
; Р 2 = 0,3 |
; Р 3 = 0,2 |
1 - (0,52 + 0,32 + 0,22) |
0,62 |
|
Р 1 = 0,5 |
; Р 2 = 0,4 |
; Р 3 = 0,1 |
1 - (0,52 + 0,42 + 0,12) |
0,58 |
|
Р 1 = 0,6 |
; Р 2 = 0,2 |
; Р 3 = 0,2 |
1 - (0,62 + 0,22 + 0,22) |
0,56 |
|
Р 1 = 0,6 |
; Р 2 = 0,3 |
; Р 3 = 0,1 |
1 - (0,62 + 0,32 + 0,12) |
0,54 |
|
Р 1 = 0,7 |
; Р 2 = 0,2 |
; Р 3 = 0,1 |
1 - (0,72 + 0,22 + 0,12) |
0,46 |
|
Р 1 = 0,8 |
; Р 2 = 0,1 |
; Р 3 = 0,1 |
1 - (0,82 + 0,12 + 0,12) |
0,35 |
|
Прим |
еч ани е. PIC — polymorphism information content (информационный полиморфизм) |
. |
||
2. Выявленные аллели молекулярных маркеров SSR и S-SAP и их PIC при оценке стержневой коллекции Brassica rapa L., сохраняемой в ВИР (Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова)
|
Число аллелей |
Доля аллелей, % |
Значение PIC |
|
39 |
SSR маркеры 28,9 |
0-0,1 |
|
20 |
14,8 |
0,1-0,2 |
|
16 |
11,9 |
0,2-0,3 |
|
20 |
14,8 |
0,3-0,4 |
|
40 |
29,6 |
0,4-0,5 |
|
Всего 135 |
100 |
|
|
4 |
S-SAP м ар к е р ы 3,2 |
0-0,1 |
|
9 |
7,3 |
0,1-0,2 |
|
18 |
14,6 |
0,2-0,3 |
|
18 |
14,6 |
0,3-0,4 |
|
74 |
60,0 |
0,4-0,5 |
|
Всего 123 |
100 |
В нашей работе при оценке стержневой коллекции Brassica rapa L. (96 образцов), хранящейся в ВИР, с помощью 21 пары SSR (simple sequence repeats) и 12 пар S-SAP (sequence specific amplified polymorphism) праймеров найдено 135 SSR и 123 S-SAP полиморфных маркера, которые использовались для построения филогенетического древа (14). Для каждого маркера нами были подсчитаны значения PIC (табл. 2). Их распределение по частоте встречаемости
П р им еч ани е. SSR — simple sequence repeats, S-SAP — sequence для каждого типа маркеров specific amplified polymorphism. PIC — рolymorphism information content (информационный полиморфизм). представлены на диаграм мах (рис.). Среднее значение PIC для обоих типов маркеров 0,316, тогда как для микросателлитных маркеров — 0,257, для S-SAP маркеров — 0,379, то есть в среднем на 50 % выше. Таким образом, в наших исследованиях по оценке генетического полиморфизма в коллекции B. rapa с использованием двух типов маркеров наибольшую информативность отмечали у S-SAP маркеров.
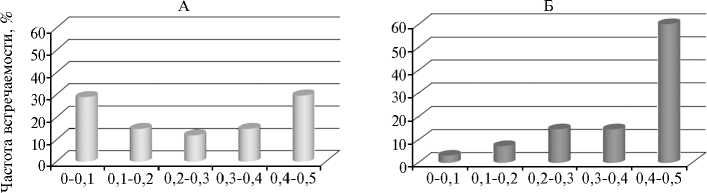
Значение PIC
Распределение значений PIC (рolymorphism information content) для молекулярных маркеров SSR (simple sequence repeats, А) и S-SAP (sequence specific amplified polymorphism, Б) при оценке стержневой коллекции Brassica rapa L., сохраняемой в ВИР (Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова).
В то же время при определении генетического разнообразия у риса (Oryza sativa L.) среднее значение PIC было в 2 раза выше в случае использования SSR маркеров (0,66), чем в варианте, когда применяли RFLP (restriction fragment length polymorphism) маркеры (0,36) (15). При исследовании молекулярного генетического разнообразия у сладкой кукурузы (Zea mays L.) с помощью RAPD (random amplified polymorphic DNA) и SSR маркеров получены результаты, указывающие на более высокую симилярность между изученными популяциями, чем внутри них (16). При этом авторы отмечают, что RAPD маркеры имели более низкие средние значения PIC (0,17), чем SSR маркеры (0,57). Абсолютные значения PIC для RAPD и SSR маркеров в описанном случае не подлежали сравнению из-за максимальной величины PIC 0,5 и 1,0 соответственно для RAPD и SSR локусов. RAPD и SSR маркеры также использовались для оценки генетической изменчивости и взаимоотношений у тунисских местных сортов ячменя (Hordeum vulgare L.) (17). Несмотря на то, что был выявлен высокий уровень полиморфизма как для RAPD, так и для SSR маркеров, а средние значения PIC составили 0,477 и 0,533 соответственно для RAPD и SSR маркеров, авторы заключают, что SSR маркеры все же лучше подходят для оценки генетического разнообразия ячменя, чем RAPD маркеры, поскольку SSR маркеры обладали более высоким полиморфизмом (90,7 %) по сравнению с RAPD маркерами (74,0 %).
И, наконец, еще один пример установления значения PIC, на который хотелось бы обратить внимание. Исследователи из Аргентины и США провели анализ генетического разнообразия аргентинских сортов мягкой пшеницы ( Triticum aestivum L.), созданных в период с 1932 по 1995 год (18). Используя SSR и AFLP (amplified fragment length polymorphism) маркеры, они установили, что нет существенных различий по генетическому разнообразию между группой сортов, полученных до 1960 года, и группами, выпущенными в каждое из трех последующих десятилетий. Среднее значение разнообразия, установленное с помощью SSR маркеров, было практически идентичным для всех четырех временн ы́ х периодов. Генетическое разнообразие, выявленное посредством AFLP маркеров, подтвердило отсутствие уменьшения генетического разнообразия во времени. Однако между сортами мягкой пшеницы, созданными в 1970-х (PIC = 0,28) и 1980-х (PIC = 0,34) годах были найдены достоверные различия (Р = 0,01). В целом результаты, полученные по PIC, указывают на то, что аргентинские сорта мягкой пшеницы поддерживались практически на одном и том же уровне генетического разнообразия на протяжении более 60 лет, а их различия обусловлены преимущественно реализуемыми селекционными программами, но никак не степенью генетического разнообразия полученных сортов. Таким образом, мера информационного полиморфизма служит важным компонентом при составлении планов селекционных программ и одним из ключевых информационно-статистических показателей при их выполнении.
Программное обеспечение для расчета Н и PIC. Для правильного составления плана генетических исследований и оценки полученных результатов зачастую приходится проводить расчеты величин гетерозиготности (Н) и информационного полиморфизма (PIC) для описания информативности маркеров, но до последнего времени не было простых и общедоступных калькуляторов для таких расчетов. Для упрощения работ по маркерным исследованиям группа венгерских ученых в 2012 году предложила разработанную ими интерактивную Интернет-программу PICcalc (19). Программа позволяет вычислять значения Н и PIC по аллельным частотам при введении показателей в ручном режиме или с использованием специального файла, содержащего бинарную матрицу данных. Дополнительные опции дают возможность рассчитывать значения для определенного числа локусов, используя для этого простой текстовый файл, что гарантирует установление Н и PIC для праймера или наборов праймеров, использованных для анализа различных генетических маркерных систем, имеющих дело с бинар- ными данными. Для мультилокусных маркеров, например AFLP, ISSR (inter simple sequence repeats) или RAPD, теоретически предполагается, что фрагменты равной длины амплифицируются на соответствующих локусах хромосом и что они представляют одиночный доминантный локус с двумя возможными аллелями (наличие/отсутствие ампликона). Максимальное значение Н и PIC для доминантных маркеров в этом случае будет равно 0,5, поскольку для такого типа маркеров допускается только два аллеля на локус и обе величины подвержены влиянию числа и частоты аллелей (13, 20, 21). С учетом этой особенности доминантных маркеров в программе специально предусмотрена возможность расчета Н и PIC для них (19).
Ранее группа американских ученых также предложила компьютерную программу, которая позволяла проводить расчет показателей Н и PIC (22). Основное отличие от упомянутого выше Интернет-ресурса заключается в том, что авторы вывели единообразно распределяемое минимальное отклонение несмещенной оценки PIC в соответствии с его точным значением дисперсии. Для того чтобы установить это, они получили формулу для расчета любого многочлена в наборе переменных, распределенных мультиномиально.
Сопутствующие величины. Эффективное мультиплексное отношение (effective multiplex ratio, EMR) определяют как произведение общего числа полиморфных локусов (на праймер) и доли полиморфных локусов от их общего числа (23, 24):
EMR = n p (n p /n), [5] где np — число полиморфных локусов, n — общее число локусов. Чем выше значения EMR, тем эффективней система «праймер—маркер».
Маркерный индекс (marker index, MI) — статистическая величина, используемая для оценки суммарной пригодности маркерной системы. Маркерный индекс есть произведение величины информационного полиморфизма (или ожидаемой гетерозиготности, НЕ) и эффективного мультиплексного отношения (23, 24):
MI = PIC ½ EMR, [6]
Чем выше значение MI для методики, тем она лучше. В то же время чтобы отразить способность сочетания «праймер—применяемая методика» устанавливать различия между большим числом генотипов, используют показатель разрешающей способности (resolving power, Rp) (25, 26):
Rp = ∑ I b , [7] где Ib = 1 - (2 ½ 0,5 - p) — информативность ампликона, р — доля особей, у которых выявлен ампликон I.
Таким образом, существует несколько подходов, позволяющих оценивать меру информационного полиморфизма и сопутствующие ей величины. ДНК-маркеры в настоящее время признаны довольно удобным и качественным инструментом оценки генетического разнообразия на молекулярном уровне. Однако перед использованием той или иной маркерной системы необходимо оценить техническую оснащенность лаборатории, потребность в применении выбранной маркерной системы и ее соответствие решаемым задачам, профессиональную подготовку персонала, а также предстоящие эксплуатационные расходы и доступные средства вспомогательного обслуживания. Требуемое программное обеспечение должно быть выбрано на основании расчета его пригодности для решения стоящих перед исследователем задач, в том числе задач по популяционной генетике, если речь идет об оценке информационного полиморфизма. Морфологические параметры весьма важны для интерпретации полученных результа-576
тов. Установление статистически достоверных ассоциативных и корреляционных связей между морфологическими и молекулярно-генетическими показателями служит ключевым обстоятельством при принятии окончательных решений. И, конечно же, нельзя не учитывать биологические особенности изучаемых видов при оценке исследуемых генетических параметров, поскольку один и тот же параметр может формироваться у разных видов неодинаково не только в фило-, но и в онтогенезе. Последнее особенно важно для эффекта взаимодействия «генотип—среда».
Список литературы Оценка меры информационного полиморфизма генетического разнообразия
- Nei M., Roychoudhury A.K. Sampling variances of heterozygosity and genetic distance. Genetics, 1974, 76: 379-390.
- Nei M., Li W.H. Mathematical model for studying genetic variation in terms of restriction endonucleases. PNAS USA, 1979, 76: 5269-5273 ( ) DOI: 10.1073/pnas.76.10.5269
- Botstein D., White R.L., Skalnick M.H., Davies R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphism. Am. J. Hum. Genet., 1980, 32: 314-331.
- Артемьева А.М., Чесноков Ю.В., Клоке Э. Морфолого-географический и молекулярно-генетический анализ коллекции белокочанной капусты ВИР. Доклады РАСХН, 2008, 5: 14-18.
- Артемьева А.М., Чесноков Ю.В., Клоке Э. Генетическое разнообразие и внутривидовые филогенетические взаимоотношения культур вида Brassica rapa L. по результатам анализа микросателлитов. Информационный Вестник ВОГиС, 2008, 12(4): 608-619.
- Артемьева А.М., Клоке Э., Чесноков Ю.В. Анализ филогенетических связей вида Brassica oleracea L. (капуста огородная). Информационный вестник ВОГиС, 2009, 13(4): 759-771.
- Вишнякова М.А., Бурляева М.О., Алпатьева Н.В., Чесноков Ю.В. RAPD-анализ видового полиморфизма рода Чина (Lathyrus L.) сем. Fabaceae Lindl. Информационный вестник ВОГиС, 2008, 12(4): 595-607.
- Артемьева А.М., Чесноков Ю.В. Коллекция капусты ВИР: этапы формирования и изучения. Вавиловский журнал генетики и селекции, 2012, 16(4/2): 1047-1060.
- Иванов А.А., Буренин В.И., Чесноков Ю.В. Оценка филогенетических отношений видов рода Beta L. с помощью RAPD-маркеров. Доклады Россельхозакадемии, 2012, 3: 20-22.
- Чесноков Ю.В., Буренин В.И., Иванов А.А. RAPD-анализ коллекционных образцов дикой и культурной свеклы (Beta L.). Сельскохозяйственная биология, 2013, 3: 28-36 ( , 10.15389/agrobiology.2013.3.28eng) DOI: 10.15389/agrobiology.2013.3.28rus
- Liu B.H. Statistical genomics: linkage, mapping and QTL analysis. CRC Press, Boca Raton, 1998.
- Anderson J.A., Churchill G.A., Autrique J.E., Tanksley S.D., Sorrells M.E. Optimizing parental selection for genetic linkage maps. Genome, 1993, 36(1): 181-186 ( ) DOI: 10.1139/g93-024
- De Riek J., Calsyn E., Everaert I., Van Bockstaele E., De Loose M. AFLP-based alternatives for the assessment of distinctness, uniformity and stability of sugar beet varieties. Theor. Appl. Genet., 2001, 103: 1254-1265 ( ) DOI: 10.1007/s001220100710
- Артемьева А.М., Будан Х., Клоке Э., Чесноков Ю.В. Использование мобильных генетических элементов САСТА для уточнения филогенетических взаимоотношений внутри вида Brassica rapa L. Вавиловский журнал генетики и селекции, 2011, 15(2): 398-411.
- Xu Y., Beachell H., McCouch S.R. A marker-based approach to broadening the genetic base of rice (Oryza sativa L.) in the US. Crop Sci., 2004, 44: 1947-1959 ( ) DOI: 10.2135/cropsci2004.1947
- Bered F., Freitas Terra T., Spellmeier M., Neto J.F.B Genetic variation among and within sweet corn populations detected by RAPD and SSR markers. Crop Breed. Appl. Biotechnol., 2005, 5: 418-425.
- Karim K., Rawda A., Hatem C.-M. Genetic diversity in barley genetic diversity in local Tunisian barley based on RAPD and SSR analysis. Biological Diversity and Conservation, 2009, 2/1: 27-35 ( ) DOI: 10.1590/S0100-204X2015000200008
- Manifesto M.M., Schlatter A.R., Hopp H.E., Suarez E.Y., Dubcovsky J. Quantitative evaluation of genetic diversity in wheat germplasm using molecular markers. Crop Sci., 2001, 41: 682-690 ( ) DOI: 10.2135/cropsci2001.413682x
- Nagy S., Poczai P., Cernak I., Gorji A.M., Hegedus G, Taller J. PICcalc: An online program to calculate polymorphic information content for molecular genetic studies. Biochem. Genet., 2012, 50: 670-672 ( ) DOI: 10.1007/s10528-012-9509-1
- Bolaric S., Barth S., Melchinger A.E., Posselt U.K. Genetic diversity in European perennial ryegrass cultivars investigated with RAPD markers. Plant Breed., 2005, 124: 161-166 ( ) DOI: 10.1111/j.1439-0523.2004.01032.x
- Henry R.J. Practical applications of plant molecular biology. Chapman and Hall, London, 1997.
- Shete S., Tiwari H., Elston R.C. On estimating the heterozygosity and polymorphism information content value. Theor. Popul. Biol., 2000, 57: 265-271 ( ) DOI: 10.1006/tpbi.2000.1452
- Powell W., Morgante M., Andre C., Hanafey M., Vogel J., Tingey S., Rafalski A. The utility of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis. Mol. Breed., 1996, 2: 225-238 ( ) DOI: 10.1007/BF00564200
- Nagaraju J., Damodar Reddy K., Nagaraja G.M., Sethuraman B.N. Comparison of multilocus RFLPs and PCR-based marker systems for genetic analysis of the silkworm, Bombyx mori. Heredity, 2001, 86: 588-597 ( ) DOI: 10.1046/j.1365-2540.2001.00861.x
- Gilbert J.E., Lewis R.V., Wilkinson M.J., Caligari P.D.S. Developing an appropriate strategy to assess genetic variability in plant germplasm collections. Theor. Appl. Genet., 1999, 98: 1125-1131 ( ) DOI: 10.1007/s001220051176
- Prevost A., Wilkinson M.J. A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars. Theor. Appl. Genet., 1999, 98: 107-112 ( ) DOI: 10.1007/s001220051046


