Оценка внутрисортового полиморфизма генов устойчивости к грибным болезням томата у сортов селекции Мичуринского ГАУ
Автор: Шамшин Иван Николаевич, Ильичев Алексей Сергеевич, Грошева Екатерина Владимировна
Журнал: Овощи России @vegetables
Рубрика: Селекция, семеноводство и биотехнология растений
Статья в выпуске: 6 (68), 2022 года.
Бесплатный доступ
Цель. Исследования направлены на оценку внутрисортового аллельного разнообразия генов устойчивости к фузариозу и кладоспориозу томата сортов селекции Мичуринского ГАУ с использованием молекулярных маркеров. Методология. Биологическими объектами исследования являются сорта томата селекции Мичуринского ГАУ. Всего проанализировано 10 сортов. Оценка внутрисортового полиморфизма определялась на выборке из 10 растений каждого сорта. Экстрагирование ДНК было проведено с использованием набора для экстрагирования ДНК Quick-DNA Plant/Seed Miniprep Kit (Zymo Research, USA) согласно протоколу производителя. Для идентификации гена устойчивости к кладоспоризу Cf-19 использовали ДНК-маркер Р7. Наличие гена устойчивости к фузариозному увяданию I-2 определяли с помощью маркера I-2/5. Визуализацию результатов амплификации осуществляли с помощью электрофореза в 2% агарозном геле. Результаты. Было проанализировано по 10 растений 10 сортов томата селекции Мичуринского ГАУ с использованием молекулярных маркеров генов устойчивости к кладоспориозу и фузариозу. Проведена оценка внутрисортового полиморфизма. У большинства исследуемых сортов анализируемые гены идентифицированы в гетерозиготном состоянии. По гену I-2 выявлены три сорта (Виват, Каротинка,Красавец), содержащих в генотипе только аллели восприимчивости к возбудителю заболевания. Отмечены отдельные образцы гомозиготные по аллелю устойчивости к фузариозу. Анализ гена Cf-19 устойчивости к кладоспориозу показал, что большинство исследуемых сортов являются гетерозиготными формами. У части сортов все десять растений были гетерозиготны. К ним относятся Красавец, Виват, Орлик, Буй-Тур. Остальные образцы имели различный аллельный состав. Аллель устойчивости к возбудителю был идентифицирован у растений сорта Непрядва и Черныш. Доминантный аллель в гомозиготном состоянии выявлен у сорта Япончик. При этом все анализируемые растения этого сорта были однообразны. Заключение. Установлено, что значительная часть сортов томата селекции Мичуринского ГАУ является полиморфной по генам устойчивости к кладоспориозу и фузариозу. При этом значительная часть сортов может быть использована в селекции при проведении предварительного молекулярного анализа. По гену устойчивости к фузариозу выделены следующие сорта источники аллеля устойчивости: Сокол, Непрядва, Япончик, Орлик, Золотничок, Черныш. У них ген I-2 представлен в гетерозиготном состоянии. Внутрисортовой полиморфизм отмечен и для гена Cf-19. Большинство образцов имеют два аллеля. Исключение составляет сорт Япончик, который является доминантной гомозиготой по данному гену.
Томат, маркер-опосредованная селекция, днк-маркер
Короткий адрес: https://sciup.org/140296495
IDR: 140296495 | УДК: 635.64:631.526.32:631.524.86 | DOI: 10.18619/2072-9146-2022-6-35-39
Текст научной статьи Оценка внутрисортового полиморфизма генов устойчивости к грибным болезням томата у сортов селекции Мичуринского ГАУ
Современный процесс селекции базируется на знаниях фундаментальных аспектов наследования ключевых признаков растений, поэтому широкое распространение получили методы маркер-опосредованной селекции. ДНК-маркеры позволяют ускорить анализ генетических коллекций и отобрать источники ценных генов. Это дает возможность вести отбор не только по внешнему проявлению признака, но и опираясь на знания о закономерностях его наследования. Маркерная селекция может быть успешно применена для создания устойчивых форм томата.
Для создания новых генотипов чаще используют исходные формы с комплексом значимых признаков. При этом отдельные гены, в том числе гены устойчивости, могут находиться в рецессивном гомозиготном состоянии или гетерозиготе. Поэтому одной из задач отбора исходных форм является анализ генетической однородности сорта. Это позволяет избежать предварительной оценки растений одного сорта перед гибридизацией.
К числу наиболее распространенных болезней томата, снижающих урожайность плодов, относятся кладоспо-риоз. Это грибное заболевание, возбудителем которого является Cladosporium fulvum Cooke. Он поражает как плоды, так и само растение, вызывая его гибель. Создание устойчивых сортов и гибридов – одно из основных направлений селекционной работы.
Исследователи отмечают, что у дикорастущих видов идентифицировано более 20 генов семейства Cf , дающие устойчивость к данному заболеванию. В культурные сорта были интродуцированы гены Cf-1 – Cf-9 [1].
Работа по маркированию генов устойчивости к C. fulvum начата с появлением первых ДНК-маркеров. Исследования были сосредоточены на картировании отдельных хромосом томата и нанесения на них маркеров генов Cf . Так, на коротком плече хромосомы 1 идентифицированы два RFLP-маркера TG236 и GP46, расположенные в 5 сМ от генов Cf-4 и Cf-9 [2]. На хромосоме 6 картированы гены Cf-2 и Cf-5 . Для них идентифицированы RFLP-маркеры CT119 и GP79 [3]. Ряд маркеров AFLP идентифицирован для гена Cf-9 [4].
Однако эти маркеры являются мультилокусными. Использование их в анализе является трудоемким и требует значительных затрат времени. Наибольшую ценность для маркер-опосредованной селекции имеют маркеры уникальных последовательностей ДНК (доминантные и кодоминантные маркеры)способные выявлять аллельное состояние генов. Большинство таких маркеров разработаны для генов способных придавать устойчивость к нескольким расам возбудителя заболевания. К таким генам относится Cf-10. Ning и коллеги идентифицировали микросателлитный маркер LEtaa001, расположенный на расстоянии 9,7 сМ от гена и дающий возможность различать гомо и гетерозиготные растения [5]. SCAR-маркеры выявлены и для гена Cf-9. Маркеры 8F/10R и 8F/12R были апробированы на 42 линиях томата и 59 гибридах F1 и показали свою актуальность для маркерного отбора [6]. Наибольший интерес представляет ген Cf-19. Установлено, что он является доминантным и индуцирует сверхчувствительность у растений томатов, инокулированных 1–4 физиологическими расами C. fulvum. Для идентификации гена Cf-19 и скрининга генетических коллекций томата был создан молекулярный маркер Р7. Этот маркер был протестирован на растениях F2 и различных линиях F3 томата,его достоверность в идентификации генотипа достаточна для маркер-опосредованной селекции [7].
Еще одним грибной болезнью томата, наносящим значительный ущерб, является фузариоз. Возбудитель фузариоза почвенный гриб Fusarium oxysporum f. sp. lycopersici. Гриб инфицирует растения через корни посредством прямого внедрения или через раны.После этого происходит заселение ксилемной ткани растения и вызывает его гибель [8].
В культурные сорта томата интродуцированы три гена устойчивости к болезни - I, I-2, I-3 . Гены I и I-2 локализованы на 11 хромосоме и получены от S. pimpinellifolium. Они обеспечивают устойчивость к расам 1 и 2соответ-ственно. Ген I-3 , идентифицированный на 7 хромосоме, впервые выявлен у вида S. pennellii. Он создает резистентность к расам 1-3 возбудителя [9].
Для идентификации генов устойчивости разрабатываются ДНК-маркеры, способствующие ускорению создания иммунных генотипов.
Наиболее актуальна разработка кодоминантных маркеров, в частности маркера гена I-2 , обеспечивающего невосприимчивость к большому количеству рас возбудителя. Такие работы были проведены и идентифицированы несколько CAPS-маркеров на хромосоме 11 тома-та.Однако часть из них была на значительном расстоянии от идентифицируемого гена [10]. Для отбора устойчивых форм был разработан маркер TAO1,с помощью которого было проведено генотипирование 100 контрастных форм [11]. Он был так же использован при анализе линий, сортов и гибридов томата турецкой селекции [12, 13]. Еще одним маркером, рекомендованным для селекции, является I-2/5 [ 14]. Его успешно применяют для анализа исходного материала в Республике Беларусь [15, 16], России [17], [18]. Он позволяет проводить оценку аллельного состояния гена I-2 и отбирать источники устойчивости для селекции.
Большинство работ с использованием маркеров генов устойчивости к кладоспориозу и фузариозу томата сосредоточены на анализе генетических коллекций сортов и гибридов. Работ по оценке внутрисортового полиморфизма отдельных сортов недостаточно. Это и послужило основой для выбора темы нашего исследования.
В Мичуринском ГАУ работа по селекции томата ведется на протяжении нескольких десятилетий. Создано более 10 сортов для открытого грунта. Однако исследований, полученных сортов, с использованием молекулярных маркеров практически не проводилось. Для определения возможности использования в качестве исходных форм в селекции на устойчивость к грибным болезням, необходима оценка аллельного разнообразия по селектируемым генам. Это облегчает поиск генетических источников ценных признаков. Ранее нами проведена работа по идентификации генов устойчивости к грибным болезням среди наших сортов [18]. Однако аллельное разнообразие внутри одного генотипа не было оценено.Для работы были отобраны наиболее распространенные в Центрально-черноземном регионе сорта селекции университета.Два сорта из них уже используются в селекционном процессе как доноры ряда ценных признаков [18]. Поэтому целью нашей работы является анализ внутрисортового полиморфизма сортов томата селекции Мичуринского ГАУ по генам устойчивости к кладоспориозу и фузариозу с использованием молекулярных маркеров.
Материалы и методы
Работа выполнена на базе учебно-исследовательского тепличного комплекса и лаборатории молекулярно-генетического анализа плодовых растений Мичуринского ГАУ. Растения томата культивировались в торфо-почвенной смеси в емкостях объемом 7 литров в условиях поликар-бонатного укрытия. Были проанализированы по десять следующие фрагменты: фрагмент размером 633 пары нуклеотидов (п.н.) свидетельствующий о наличии аллеля устойчивости, фрагмент размером 693 п.н., выявляет восприимчивость к возбудителю болезни. Нередко данный фрагмент амплифицируется в паре с фрагментом 7 6 0 п.н., также свидетельствующий о неустойчивости генотипа.Наличие всех трех фрагментов (760, 693 и 633 п.н.) говорит о гетерозиготном состоянии анализируемого гена [14, 15,19].
В ходе анализа сортов селекции Мичуринского ГАУ установлено, что большинство образцов являются
Таблице 1. Нуклеотидная последовательность праймерных пар используемых в работе Table 1. Nucleotide sequence of primer pairs used in the work
Экстрагирование ДНК было проведено с использованием набора для экстрагирования ДНК Quick-DNA Plant/Seed Miniprep Kit (Zymo Research, USA) согласно протоколу производителя.
Для оценки полиморфизма использованы молекулярные маркеры гена устойчивости к фузариозу (I-2) [14] и кладоспоризу [7] томата. Последовательность праймерных пар представлена в таблице 1.
Реакционная смесь для ПЦР со всеми праймерами объемом 15 мкл содержала: 20 нг ДНК, 1,5 мМ dNTP, 2,5 мМ MgSO 4 , 10 пМ каждого праймера, 1 ед. Taq-полимеразы и 10х стандартного ПЦР-буфера. Реакцию для обоих пар праймеров проводили в приборе SimpliAmp (Life Technology) по программе:
-
- для маркера P7 - 94 о С - 4 мин, 35 циклов 94 о С - 30 с, 60 о С - 30 с, 72 о С - 1 мин и финальная элонгация в течение 7 мин при 72 о С;
-
- для маркера I-2/5 - 5 мин 94°С, 35 циклов 30 с 94 о С, 30 с 55 о С, 1 мин 72 о С и финальная элонгация в течение 7 мин при 72°С.
Результаты амплификации разделялись путем электрофореза в 2% агарозном геле. После электрофореза гель анализировали в ультрафиолетовом свете с использованием трансиллюминатора.
Результаты исследований и обсуждения
Для определения внутрисортового полиморфизма по генам устойчивости к грибным болезням были взяты 10 растений каждого сорта. Для идентификации гена I-2 устойчивости к фузариозу был использован маркер I-2/5. Этот маркер был апробирован нами в предыдущих работах и показал хорошо воспроизводимые результаты. Его широко используют для анализа томатных линий и гибридного потомства [15, 17]. В результате работы были получены четкие, воспроизводимые в ряде повторности результаты.
Согласно авторам оригинальной статьи [14] при амплификации с маркером I-2/5 могут быть выявлены гетерозиготами.Наиболее часто встречается комбинация фрагментов 693 и 633 п.н. как, например, у сорта Орлик где все 10 образцов имеют оба аллеля (рис.1).
У отдельных сортов, таких как Каротинка, Черныш и Виват наряду с аллелем устойчивости (693 п.н.) амплифи-цируется фрагмент размером 760 п.н. При этом все анализируемые растения сорта Виват имеют такой аллельный состав. У сорта Черныш выявлен один образец, имеющий все три фрагмента.
Идентифицированы сорта, у которых амплифицируется только один фрагмент. Среди них можно выделить сорт Буй-Тур. Из 10 анализируемых растений этого сорта у всех выявлен только фрагмент размером 693 п.н., соответствующий рецессивному аллелю. Это свидетельствует о восприимчивости его к фузариозу. Этот фрагмент идентифицирован также у одного образца сорта Черныш и двух образцов сорта Непрядва. Аллель, соответствующий устойчивому генотипу в гомозиготной форме, отмечен у одного растения сорта Непрядва. В табл. 2 показано распределение аллелей у растений томата изучаемых сортов.
Анализ полученных данных показывает, что у сортов селекции Мичуринского ГАУ присутствует внутрисортовой полиморфизм по гену I-2 устойчивости к фузариозу.
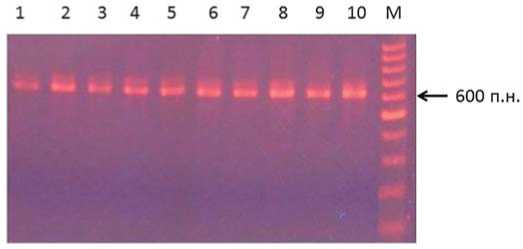
Рис. 1. Результаты анализа внутрисортового полиморфизма сорта Орлик по гену I-2 1 – 10 – растения томата сорта Орлик,М – маркер молекулярного размера 100 п.н.
Fig. 1. Results of analysis of the intrasortalpolymorphism of the Orlyk variety by gene I-2
1 - 10 - plants of Orlyk tomato, M - 100 bp molecularsize marker
Таблица 2. Распределение аллелей гена I-2 у растений томата селекции Мичуринского ГАУ Table 2. Distribution of alleles of the I-2 gene in tomato plants of Michurinsky GAU selection
|
№ п/п |
Название сорта Variety name |
Количество растений с фрагментом (шт.) Number of plants with a fragment |
|||
|
633 п.н. 633 bp. |
693 п.н. 693 bp. |
633/693 п.н. 633/693 bp. |
693/760 п.н. 693/760 bp. |
||
|
1 |
Сокол |
2 |
8 |
||
|
2 |
Каротинка |
1 |
9 |
||
|
3 |
Непрядва |
1 |
2 |
7 |
|
|
4 |
Япончик |
1 |
9 |
||
|
5 |
Красавец |
2 |
8 |
||
|
6 |
Виват |
10 |
|||
|
7 |
Орлик |
10 |
|||
|
8 |
Золотничок |
5 |
5 |
||
|
9 |
Буй-Тур |
10 |
|||
|
10 |
Черныш |
1 |
5 |
4 |
|
Большинство растений гетерозиготны и имеют оба аллеля. Отмечены образцы, которые содержат только аллели восприимчивости (Виват, Каротинка, Красавец). Однако, большинство сортов (Сокол, Непрядва, Япончик, Орлик, Золотничок, Черныш) являются гетерозиготами с аллелем устойчивости. Использовать такие образцы в качестве исходных форм можно, но предварительно проанализировав каждое растение из большой выборки перед гибридизацией.
В проводимой нами селекционной работе мы анализировали отдельные растения перед гибридизацией для получения устойчивого сорта [18]. Так нами была использована комбинация скрещивания Красавец × Сибирскийтигр. Был проведен поиск растений исходных сортов с аллелем устойчивости (фрагмент размером 633 п.н.) непосредственно перед опылением. Идентифицированы гетерозиготные образцы, которые были использованы в качестве родительских форм. Полученные гибриды имели ген устойчивости.
Анализ гена Cf-19 устойчивости к кладоспориозу показал следующие результаты. При амплификации с праймером Р7 синтезируется два фрагмента размером 250 п.н. и 300 п.н. Устойчивые генотипы томата имеют фрагмент 300 п.н., неустойчивые – 250 п.н. [7]. Для большинства исследуемых сортов отмечены гетерозиготные формы, т.е. имеющие оба аллеля в своем генотипе. У части сортов все десять растений были гетерозиготны. К ним относятся Красавец, Виват,
Орлик, Буй-Тур. Остальные образцы имели различный аллельныйсостав. Распределениеаллелейу растенийтома-та селекции Мичуринского ГАУ отмечены в таблице 3.
Анализ результатов показывает, что среди растений отдельных сортов были идентифицированы гомозиготные формы. Так у сортов Сокол, Каротинка и Золотничок из 10 растений два были гомозиготны по рецессивному аллелю, а остальные гетерозиготны. Фрагмент250 п.н. был идентифицирован у растений сорта Непрядва и Черныш. Доминантный аллель в гомозиготном состоянии выявлен у
Таблица 3. Распределение аллелей гена Cf-19 у растений томата селекции Мичуринского ГАУ Table 3 Distribution of alleles of the Cf-19 gene in tomato plants of Michurinsky GAU selection
|
№ п/п |
Название сорта Variety name |
Количество растений с фрагментом (шт.) Number of plants with a fragment |
||
|
250 п.н. 250 bp. |
300 п.н. 300 bp. |
250/300 п.н. 250/300 bp. |
||
|
1 |
Сокол |
2 |
8 |
|
|
2 |
Каротинка |
2 |
8 |
|
|
3 |
Непрядва |
1 |
9 |
|
|
4 |
Япончик |
10 |
||
|
5 |
Красавец |
10 |
||
|
6 |
Виват |
10 |
||
|
7 |
Орлик |
10 |
||
|
8 |
Золотничок |
2 |
8 |
|
|
9 |
Буй-Тур |
10 |
||
|
10 |
Черныш |
1 |
9 |
|
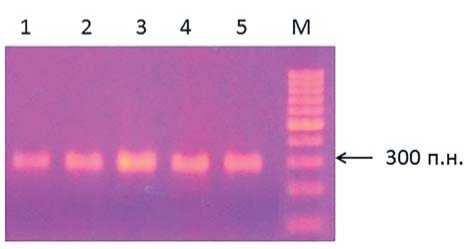
Рис. 2. Результаты анализа внутрисортового полиморфизма сорта Япончик по гену Cf-19 1 – 5 – растения томата сорта Япончик, М – маркер молекулярного размера 100 п.н.
Fig. 2. Results of analysis of intrasortum polymorphism of the variety Japanese by gene Cf-19
1 - 5 - plants of tomato varieties Japonchik, M -markerof molecularsize 100 bp сорта Япончик (рис 2). При этом все анализируемые растения этого сорта были однообразны.
Результаты анализа внутрисортового полиморфизма по гену Cf-19 устойчивости к кладоспориозу показывает, что практически все сорта селекции Мичуринского ГАУ являются полиморфными. Исключение составляет сорт Япончик. Из 10 исследованных растений этого сорта все были гомозиготны по доминантному аллелю. Большинство растений гетерозиготны. У пяти выявлены гомозиготные рецессивные генотипы. Для использования их в качестве исходных форм для селекции необходим предварительный анализ непосредственно перед гибридизацией. Это даст возможность оценить аллельный состав и выбрать растение с нужным аллелем. Сорт Япончик представляет интерес как возможный источник гена устойчивости. Использование его в качестве родительской формы позволит получать устойчивые гибриды. При этом предварительный анализ растений не нужен.
Заключение
Таким образом, установлено, что часть анализируемых сортов селекции Мичуринского ГАУ полиморфна по гену устойчивости к кладоспориозу Cf-19. Это сорта Сокол, Каротинка, Непрядва, Золотничок, Черныш. Они имеют оба аллеля в своем генотипе. Сорт Япончик является доминантной гомозиготой по данному гену.
По гену устойчивости к фузариозу выделены следующие сорта источники аллеля устойчивости: Сокол, Непрядва, Япончик, Орлик, Золотничок, Черныш.У них ген I-2 представлен в гетерозиготном состоянии. Сорт Виват гомозиготен по рецессивному аллелю.
Из представленных результатов видно, что для отбора исходных форм томата необходим анализ аллельного состояния селектируемых генов. При этом важна оценка разнообразия внутри сорта. Для получения устойчивых гибридов целесообразно проведение предварительного молекулярно-генетического анализа.
Об авторах:
Aboutthe Authors:
Ivan N. Shamshin – Cand. Sci. (Biology), the Head of the Laboratory of the Molecular and Genetic Analysis of Fruit Plants, ,
Aleksey S. Ilyichev – Post-Graduate Student of the Department of Horticulture, Biotechnology and Crop Breeding,
Ekaterina V. Grosheva – Researcher of the Laboratory of the Molecular and Genetic Analysis of Fruit Plants, , Researcher ID ААА7122-2020.
Список литературы Оценка внутрисортового полиморфизма генов устойчивости к грибным болезням томата у сортов селекции Мичуринского ГАУ
- Polyxenova V.D. Induced resistance of plants to pathogens and abiotic stress factors: on the example of tomato. Bulletin of the Belarusian State University. 2009;(1):48-60. (in Russ.).
- Van der Beek J. G., Verkerk R., Zabel P., Lindhout P. Mapping strategy for resistance genes in tomato based on RFLPs between cultivars: Cf9 (resistance to Cladosporium fulvum) on chromosome 1. Theoretical and Applied Genetics. 1992;84(1):106-112. https://doi.org/10.1007/BF00223988.
- Grushetskaya Z.E., Lemesh V.A., Khotyleva L.V., Poliksenova V.D. Mapping of the Cf-6 tomato leaf mould resistance locus using SSR markers. Russian Journal of Genetics. 2007;43(11):1266-1270. https://doi.org/10.1134/S1022795407110099. (in Russ.).
- Thomas C.M., Vos P., Zabeau M., Jones D. A., Norcott K. A., Chadwick B. P., Jones J. D. Identification of amplified restriction fragment polymorphism (AFLP) markers tightly linked to the tomato Cf-9 gene for resistance to Cladosporium fulvum. The Plant Journal. 1995;8(5):785-794. https://doi.org/10.1046/j.1365-313X.1995.08050785.x.
- Ning L., Jing-bin J., Jing-fu L., Xiang-yang X. Development of molecular marker linked to Cf-10 gene using SSR and AFLP method in tomato. Journal of Northeast Agricultural University (English Edition). 2012;19(4):30-36. https://doi.org/10.1016/S1006-8104(13)60047-1.
- Truong H.T.H., Choi H., Cho M.C., Lee H.E., Kim J.H. Use of Cf-9 genebased markers in marker-assisted selection to screen tomato cultivars with resistance to Cladosporium fulvum. Horticulture, Environment, and Biotechnology. 2011;52(2):204-210. https://doi.org/10.1007/s13580-011-0164-y.
- Zhao T., Jiang J., Liu G., He S., Zhang H., Chen X., Xu X. Mapping and candidate gene screening of tomato Cladosporium fulvum-resistant gene Cf-19, based on high-throughput sequencing technology. BMC plant biology. 2016;16(1):1-10. https://doi.org/10.1186/s12870-016-0737-0.
- Singh V.K., Singh H.B., Upadhyay R.S. Role of fusaric acid in the development of ‘Fusarium wilt’symptoms in tomato: Physiological, biochemical and proteomic perspectives. Plant physiology and biochemistry. 2017;(118):320-332. https://doi.org/10.1016/j.plaphy.2017.06.028.
- Takken F., Rep M. The arms race between tomato and Fusarium oxysporum. Molecular plant pathology. 2010;11(2):309-314. https://doi.org/10.1111/j.1364-3703.2009.00605.x.
- El-Kharbotly A., Leonards-Schippers C., Huigen D. J., Jacobsen E., Pereira A., Stiekema W. J., Gebhardt C. Segregation analysis and RFLP mapping of the R1 and R3 alleles conferring race-specific resistance to Phytophthora infestans in progeny of dihaploid potato parents. Molecular and General Genetics MGG. 1994;242(6);749-754. https://doi.org/10.1007/BF00283432.
- Staniaszek M., Kozik E.U., Marczewski W. A CAPS marker TAO1902 diagnostic for the I-2 gene conferring resistance to Fusarium oxysporum f. sp. lycopersici race 2 in tomato. Plant Breeding. 2007;126(3):331-333. https://doi.org/10.1111/j.1439-0523.2007.01355.x.
- Tanyolaç B., Akkale C. Screening of resistance genes to fusarium root rot and fusarium wilt diseases in F3 family lines of tomato (Lycopersicon esculentum) using RAPD and CAPs markers. African Journal of Biotechnology. 2010;9 (19):2727-2730.
- Simsek D., Pinar H., Mutlu N. Development of Fusarium oxysporum f. Sp. Lycopersici (FOL) and Fusarium oxysporum f. sp. Radicis lycopersici (FORL) resistant tomato lines with the aid of marker assisted selection. Current Trends in Natural Sciences. 2018;7(13):281-285.
- Yu S.C., Zou Y.M. A co-dominant molecular marker of Fusarium wilt resistance gene I-2 derived from gene sequence in tomato. Yi Chuan= Hereditas. 2008;30(7):926-932. https://doi.org/10.3724/sp.j.1005.2008.00926.
- Adzhieva V.F., Grushetskaya Z.E., Malyshev S.V., Nekrashevich N.A., Babak O.G., Kilchevsky A.V. Creation of a complex of DNA markers for tomato genes that determine the content of carotenoids and resistance to diseases and pests. II International. научн. - практ. conf. "Modern trends in the selection and seed production of vegetable crops. Traditions and perspectives." Moscow, Russia. August 2-4, 2010. P. 47. (in Russ.)
- Grushetskaya Z.E., Lemesh V.A., Poliksenova V.D. Possible sources of new genes for resistance to the pathogen of tomato cladosporiosis in species Solarium L. Vegetable growing. 2010;(18):112.
- Eroshevskaya A.S., Egorova A.A., Milyukova N.A., Pyrsikov A.S. Molecular-genetic analysis of tomato hybrids F1 on resistance to fuzariosis. Potatoes and vegetables. 2021;(5):37-40. (in Russ.)
- Shamshin I.N., Grosheva E.V., Maslova M.V., Samoilova R.M. Creation of new tomato forms with fungal disease resistance genes based on marker selection. Vegetable crops of Russia. 2021;(6):16-21. (In Russ.) https://doi.org/10.18619/2072-9146-2021-6-16-21
- Babak O.G., Nekrashevich N.A., Pugacheva I.G., Dobrodkin M.M., Kilchevsky A.V. Study of allelic polymorphism for resistance to fusariosis in cherry tomatoes. Biotechnological techniques in preserving biodiversity and plant breeding. Collection of articles of the International Scientific Conference. Minsk, August 18-20, 2014. 2014. Р.38. (in Russ.)


